枣转录组序列的微卫星特征分析
2015-12-20魏琦琦贾宝光李承想
魏琦琦,林 青,贾宝光,吴 炼,李承想,张 琳
(中南林业科技大学 a. 经济林培育与保护省部共建教育部重点实验室;b. 林学院,湖南长沙 410004)
枣转录组序列的微卫星特征分析
魏琦琦a,b,林 青a,b,贾宝光a,b,吴 炼b,李承想a,b,张 琳a,b
(中南林业科技大学 a. 经济林培育与保护省部共建教育部重点实验室;b. 林学院,湖南长沙 410004)
运用Illumina测序平台的RNA-seq技术对中秋酥脆枣的花、果实和枣吊进行了转录组测序,并对测序获得的Unigene进行微卫星特征分析。经序列组装和拼接,共获得34 587个Unigene,运用Misa软件分析发现12 624个微卫星。在所得转录组序列的单碱基至五碱基微卫星中,以单碱基微卫星最多(6 314个,50.02%),并以A/T(6 217个,98.46%)为主要重复单元;二碱基微卫星(3 335个,26.42%)次之,其中以AG/CT(2 532个,75.92%)类型最多;再次是三碱基微卫星(2 871个,22.74%);最后是四碱基和五碱基微卫星,二者仅占所有微卫星信息的0.82%。单碱基微卫星所占比例最多,为枣最优势微卫星,而且其重复单元次数的变化明显高于其他重复类型,表明单碱基在整个枣转录组中变异最为活跃。
枣;转录组测序;RNA测序;微卫星特征
枣Ziziphus jujubaMill.是我国重要的经济林树种之一,迄今已有7 000多年的栽培利用历史,具有重要的经济和生态价值[1]。枣果实营养价值高,亦可药用,枣产业在我国经济林果产业中发挥着重要作用。我国枣资源丰富,品种繁多,近年来国内外学者利用SSR、RAPD、AFLP等分子标记技术对枣的品种分类、鉴别以及遗传多样性方面开展了研究[2-4]。
SSR即为微卫星标记,是均匀分布于真核生物基因组中的简单重复序列[5],由1~6个核苷酸的串联重复片段构成,由于重复单位的重复次数在个体间呈高度变异性并且数量丰富,因此微卫星标记的应用非常广泛。微卫星标记可鉴别杂合体和纯合子,对隐性性状的选择十分有利。与其他分子标记相比较,SSR具有以下优点:(1)共显性,能够提供比显性标记更多的信息;(2)位点丰富且随机均匀地分布在整个基因组中;(3)以PCR为基础,技术简便,易于操作,重复性及稳定性好[6]。SSR具有影响转录、基因调节、蛋白质功能以及基因组构建等功能[7-8]。它具有比其它分子标记更多的可检测等位基因,被公认为目前遗传学研究中最令人信赖的分子标记之一。目前,在枣中开发了一些SSR标记,如马秋月等[9]利用454高通量测序技术对枣(‘金丝小枣’)基因组进行了部分测序,在基因组水平上分析了枣微卫星的特点。
为进一步开发微卫星标记资源,本研究利用Illumina测序平台的Hiseq 2000高通量测序技术对枣花、枣果实和枣吊进行了转录组测序,并对获得的Unigene序列进行微卫星特征分析,以期为我国枣品种鉴别以及遗传研究提供标记和序列资源。
1 材料与方法
1.1 植物材料
采集中秋酥脆枣的花、果实和枣吊作为实验材料,迅速于液氮中冷冻后,在-80 ℃中保存。
1.2 文库构建及转录组测序
采用Ambion试剂盒结合CTAB法提取各枣材料的总RNA,RNA等量混合后,构建cDNA文库,而后用Illumina平台的Hiseq 2000 RNASeq技术对枣的转录组进行测序,再对其测序数据进行处理与分析,采用Trinity软件对clean reads进行组装,通过序列之间的overlap信息组装得到contig,再根据序列的paired-end信息和contig之间的相似性对contig进行聚类,然后在局部进行组装得到转录本,最后从局部中挑选最长的转录本作为unigene。对于多样品的组装,由于后续的表达丰度、差异基因分析等内容均建立在同一套参考基因的基础上,对样品间得到的基因序列作进一步的聚类,整合得到对应这个物种的unigene数据库。
1.3 转录组序列中微卫星的数据统计
采用MISA软件对组装获得的Unigene序列进行微卫星特征分析,查找重复基元包括:单碱基≥10次,二碱基≥6次,三碱基≥5次,四碱基≥5次,五碱基≥5次,六碱基≥5次的重复序列。在统计各重复序列时,将基序的所有可能的+1移码及其互补序列都视为同一个序列类型[10-11]。
2 结果与分析
2.1 转录组测序组装结果及统计
转录组测序共获得37 107 202条Clean Reads,对Clean Reads进行组装拼接获得2 098 231条contig序列。Contig序列长度主要分布在0~2 000 bp之间,其中以0~300 bp序列数量居多,约占总contig序列数量的98.63%,大于2 000 bp的序列数量约占总contig序列数量的0.2%。转录本(Transcript)长度主要分布在200~2 000 bp之间,在1 000~2 000 bp序列数量最多,约占总转录本数量的27.98%。(见表1)。
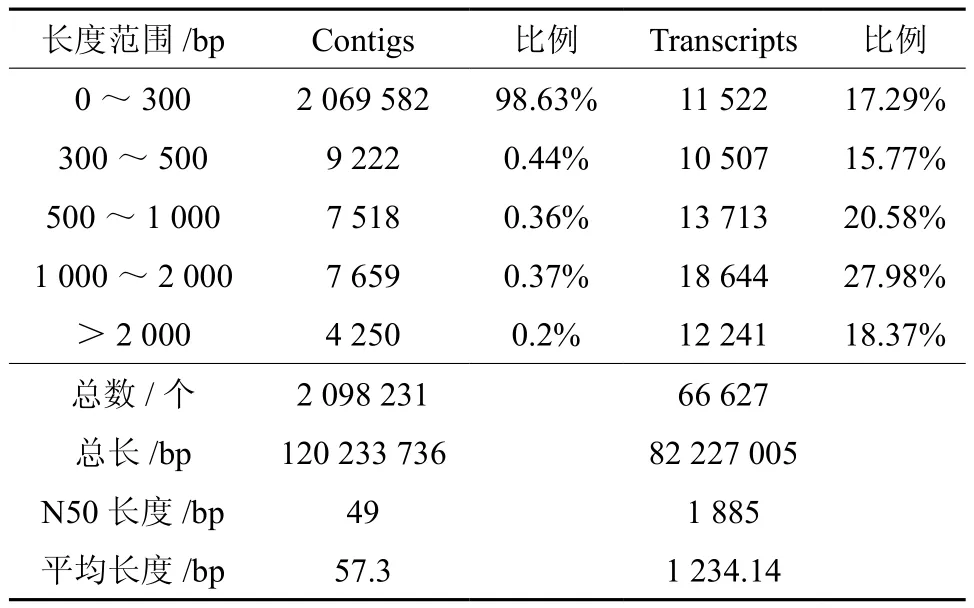
表1 中秋酥脆枣转录组组装测序结果Table 1 Assembly sequencing results of transcriptome of Z.jujuba cv. ‘Zhongqiusucui’
组装拼接获得34 587条unigene,序列长度主要分布在200~3 000 bp范围内,平均长度为953.25 bp,200~2 000 bp序列数量最多,占全部unigene序列数量的87.47%,2 000~3 000 bp的unigene序列有2 866条,占全部unigene序列数量的8.29%,大于3 000 bp的unigene序列有1 471条,占4.25%(见表2)。
2.2 微卫星类型及数目统计
使用Misa软件,共发现12 624条微卫星重复序列,对枣转录组中单碱基至六碱基重复完整型微卫星和复合型微卫星进行分析发现,以完整型微卫星为主,共有10 453条,占总微卫星的82.8%,而复合型微卫星只有2 171条,占17.2%。在完整型微卫星中,单碱基完整型微卫星(5 076个,40.21%)最多,其次是二碱基完整型微卫星(2 788个,22.08%)和三碱基完整型微卫星(2 501个,19.81%),而四碱基和五碱基完整型微卫星最少(88个,0.7%)。本研究中未发现六碱基重复的微卫星(见图1)。
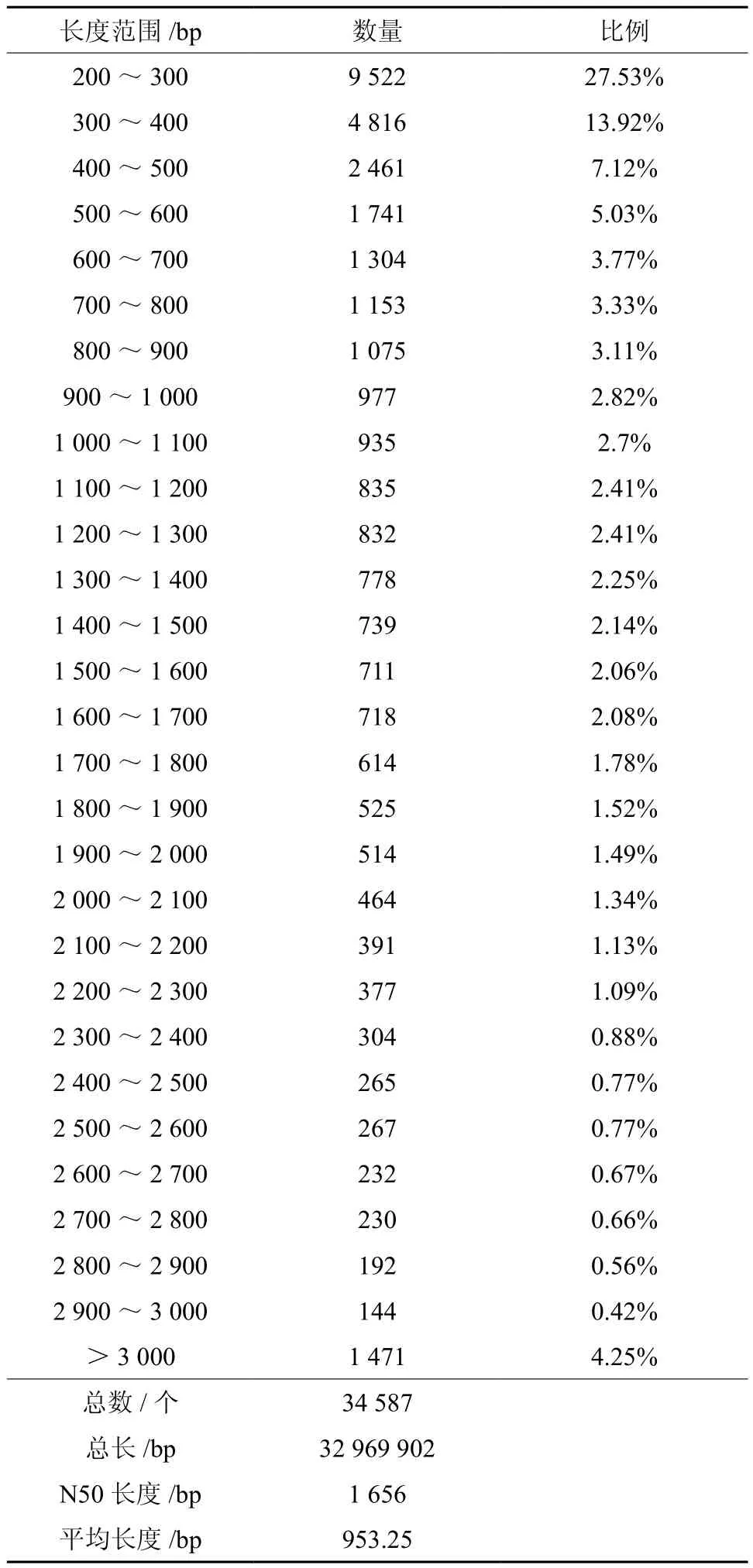
表2 中秋酥脆枣的Unigene的长度统计Table 2 Numbers and proportions of Unigene of Z.jujuba cv. ‘Zhongqiusucui’ in different lengths
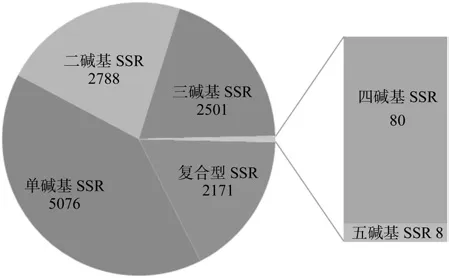
图1 SSR类型及数目Fig.1 Types and numbers of microsatellite in SSR database
2.3 不同重复基序微卫星的频率
在枣转录组微卫星数据库中,共有12 624条微卫星序列。单碱基微卫星最多(6 314个,50.02%),并以A/T类型为主;其次是以AG/CT类型最多的二碱基微卫星(3 335个,26.42%);再次是三碱基微卫星(2 871个,22.74%);四碱基和五碱基微卫星最少,共有104个,只占所有微卫星信息的0.82%(见表3)。

表3 不同类型微卫星的频率Table 3 Frequency of classified repeat SSRs
在对枣不同类型重复单元微卫星中各重复单元数量的变化情况的统计得出:在2种单碱基重复微卫星中,以A/T为最主要的重复单元,共有6 217个,占98.46%,而C/G只占1.54%(见图2)。

图2 单碱基微卫星的频率Fig.2 Frequency of mononucleotide repeat SSRs
二碱基重复类型有4种(AC/GT、AG/CT、AT/AT和CG/CG),其中AG/CT重复的数量最多,共有2 532个,占总数的75.92%;其次是AT/AT(531个,15.92%);再次是AC/GT(271个,8.12%);而CG/CG只有一个(见图3,封三)。
三碱基重复类型有10种,AAG/CTT重复的数量最多,共有1 070个,占37.27%;其次是ATC/ATG(375个,13.06%)和ACC/GGT(373个,12.99%);再次是AAC/GTT(260个,9.05%)、AAT/ATT(224个,7.8%)和AGG/CCT(220个,7.66%),其他重复碱基则相对较少(见图4,封三)。
在18种四碱基重复类型中,以AAAT/ATTT重复数量为最多,共31个(33.33%),其次为AAAG/CTTT(21个,22.58%)(见图5,封三)。
五碱基重复类型有9种,AAAAT/ATTTT重复数量最多,有3个,占27.27%(见图6,封三)。
由分析结果知,枣的微卫星数量随着重复次数增加而呈递减趋势,其中以单碱基微卫星最为明显,二碱基微卫星次之,再次是三碱基微卫星,四碱基微卫星和五碱基微卫星重复次数最少。
3 结论与讨论
(1)枣不同重复序列结构微卫星类型
高通量RNA-seq测序具有高精度、不需要参考基因组信息、低成本、应用范围广等优势,尤其可为非模式植物转录组学的研究提供捷径。本研究利用Illumina高通量测序技术对枣转录组进行测序,共获得约2 098 231条contig序列和34 587个unigene序列。而后再对unigene数据库得到的unigene序列进行SSR标记开发的分析。
按照微卫星重复序列结构的不同,将其分为完整型微卫星、不完整型微卫星以及复合型微卫星。完整型微卫星一般是由1种串联重复序列以不间断的重复方式构成的单一重复类型的微卫星;不完整型微卫星是指2个或2个以上的同种重复序列被3个或3个以下的非重复碱基分隔开;复合型微卫星指2个或2个以上的串联核心序列被3个或者3个以上连续的非重复碱基所间隔,但这种连续性的核心序列重复数不得少于5[12]。
根据其重复序列结构的重复类型不同将微卫星重复序列进行分类,并对由此获得的12 624条微卫星重复序列中单碱基至六碱基重复完整型和复合型进行分析得出:大部分为完整型微卫星,共有10 453条,占总微卫星的82.79%;而复合型微卫星只有2 171条,占总微卫星的17.2%。又针对完整型微卫星进行分析得出:以单碱基重复完整型微卫星为最多,其次是二碱基重复完整型微卫星,再次是三碱基重复完整型微卫星,四碱基和五碱基重复完整型微卫星最少,未发现六碱基。在枣的基因组序列微卫星特征研究中,六碱基重复微卫星出现的频率(40.1%)明显高于其他类型,之后依次为复合碱基(18.0%)、单碱基(17.1%)、四碱基(8.1%)、二碱基(7.5%)、三碱基(7.0%)、五碱基(2.2%)[9:83]。可见,枣转录组比基因组低级基元频率高,而高级基元比基因组的低。
(2)枣优势重复碱基类型分析
本研究中,单碱基微卫星出现的频率(50.02%)明显高于其他类型,其次是二碱基微卫星(26.42%),再次是三碱基微卫星(22.74%),四碱基微卫星(0.74%)和五碱基微卫星(0.08%)最少。从基元重复次数来看,枣微卫星数量随着重复次数增加而呈递减趋势,碱基重复次数越少,微卫星数量下降速率越快。其中以单碱基微卫星最为明显,二碱基微卫星次之,再次是三碱基微卫星,四碱基微卫星和五碱基微卫星重复次数最少。单碱基微卫星集中在10~23次重复,二碱基微卫星是6~12次重复,三碱基微卫星是5~8次重复,四碱基微卫星是5~6次重复,五碱基微卫星仅集中于5次重复。通常认为SSR位点的变异频率与基元重复数存在一定的正相关,即重复次数越多,SSR产生变异的可能性越大[13]。本研究中单碱基重复微卫星为枣最优势微卫星,所占比例最多,而且单碱基微卫星重复单元次数的变化明显高于其他重复类型,其次是二碱基微卫星,这在一定程度上说明单碱基在整个枣转录组中变异最为活跃。
(3)枣优势重复单元碱基组成分析
在2种单碱基重复微卫星中,以A/T为最主要的重复单元;4种二碱基重复类型是AG/CT重复的数量最多,其次为AT/AT;10种三碱基重复类型中AAG/CTT重复的数量最多,其次为ATC/ATG和ACC/GGT,再次为AAC/GTT、AAT/ATT和AGG/CCT,其他重复碱基则相对较少;在18种四碱基重复类型中, AAAT/ATTT的重复数量最多,其次是AAAG/CTTT;五碱基重复类型有9种,AAAAT/ATTTT重复的数量最多。由此发现枣不同重复碱基类型优势重复单元的共同特点是富含A和T碱基。不同植物中优势重复单元不同:杜仲转录组微卫星中,出现频率高的2个重复类型是AG/TC和CT/GA,其次是AC/TG和AGA/TCT[14];南方红豆杉转录组中,出现频率高的重复类型是AAG/CTT、AGG/CCT、AGC/CTG、ATC/ATG、AG/CT、AT/AT[15]。同一物种不同组织器官优势重复单元不同:在茶树转录组微卫星中占优势的前3种重复类型是CT/AG 、TC/GA和AT/TA,其次是A/T[16];在茶树花转录组中单碱基至六碱基重复单元中出现频率最高的类型分别是A/T、AG/CT、AAG/CTT、AAAG/CTTT、AAAAT/ATTTT 和 AAAAAC/GTTTTT[17]。
在微卫星重复序列中,如CA、GA、GT等重复可以通过影响DNA的结构而影响DNA重组,因此微卫星中的重复单元碱基组成在很大程度上会影响生物的生命活动[18]。造成不同植物基因组中不同重复碱基类型及重复单元偏好性的原因除了与不同物种间的真实微卫星信息差异有关外,可能还与不同微卫星查找工具中的参数设置有一定关系[19]。
(4)微卫星技术在枣研究中的应用前景
微卫星序列广泛分布于真核生物基因组的编码区和非编码区,通过对枣转录组中微卫星序列特征的分析,对开发大量高效的微卫星分子标记提供了重要的信息资源,另外对于在转录组序列中发掘的微卫星序列,它是具有基因功能的序列,有助于开发出枣的重要基因关联的微卫星标记。本研究中所得到的枣转录组微卫星对以后该物种的进化、遗传多样性、枣品种的鉴别及分子标记辅助育种等方面的研究奠定基础。
[1] 原勤勤, 文亚峰, 刘 儒, 等. 枣优良品种亲缘关系的ISSR分析[J]. 经济林研究, 2012, 30(1): 56-61.
[2] 王永康, 田建保, 王永勤, 等. 枣树品种品系的AFLP分析[J]. 果树学报, 2007, 24(2): 146-150.
[3] 智福君, 贾彦丽, 梁海永, 等. 利用RAPD技术进行枣树的品种鉴定[J]. 华北农学报, 2009, 24(增刊): 110-114.
[4] 麻丽颖, 孔德仓, 刘华波, 等. 36份枣品种SSR指纹图谱的构建[J]. 园艺学报, 2012, 39(4): 647-654.
[5] Mrazek J, Guo X, Shah A. Simple sequence repeats inprokaryotic genomes [J]. PNAS, 2007, 10(4): 8472-8477.
[6] 许靖诗, 乌云塔娜, 叶生晶, 等. 梨种质资源SSR引物的筛选与评价[J]. 中南林业科技大学学报, 2012, 32(7): 80-85.
[7] Kashi Y, King D G. Simple sequence repeat as advantageous mutators in evolution [J]. Trents in Gentic, 2006, 22(5): 253-259.
[8] Lawson M J, Zhang L. Patterns of SSR distribution in the Arabidopsis thaliana and rice genomes [J]. Genome Biology,2006, 7(2): R14.
[9] 马秋月, 戴晓港, 陈赢男, 等. 枣基因组的微卫星特征[J]. 林业科学, 2013, 49(12): 81-87.
[10] Jurka J, Pethiyagoda C. Simple repetitive DNA sequence from primates: complication and analysis [J]. Joumal of Molecular Evolution, 1995, 40(2): 120-126
[11] Cardle L, Ramsay L, Miboume D,et al.Computional and experimental characterization of physically clustered simple sequence repeats in plants [J]. Genetics, 2000, 156(2): 847-854
[12] Weber J L. Informativeness of human (dC-dA) n’ (dG-dT) n Polymorphisms [J]. Genomics, 1990, 7(4): 524-530.
[13] Schlötterer C. Evolutionary dynamics of microsatellite DNA [J].Chromosoma, 2000, 109(8): 365-371.
[14] 黄海燕, 杜红岩, 乌云塔娜, 等. 基于杜仲转录序列的SSR分子标记的开发[J]. 林业科学, 2013, 49(5): 176-181.
[15] 李炎林, 杨星星, 张家银, 等. 南方红豆杉转录组SSR挖掘及分子标记的研究[J]. 园艺学报, 2014, 41(4): 735-745.
[16] 杨 华, 陈 琪, 韦朝领, 等. 茶树转录组中SSR位点的信息分析[J]. 安徽农业大学学报, 2011, 38(6): 882-886.
[17] 王丽鸳, 韦 康, 张成才, 等. 茶树花转录组微卫星分布特征[J], 作物学报, 2014, 40(1): 80-85.
[18] Biet E, Sun J S, Dutreix M. Conserved sequence preference in DNA binding among recombination proteins: an effect of ssDNA secondary structure [J]. Nucleic Acids Research, 1999, 27(2):596-600.
[19] 阎毛毛, 戴晓港, 李淑娴, 等. 松树、杨树及桉树表达基因序列微卫星比对分析[J]. 基因组学与应用生物学, 2011, 30(1):103-109.
Microsatellites characteristics of transcriptomic sequences fromZiziphus jujubacv. ‘Zhongqiusucui’
WEI Qi-qia,b, LIN Qinga,b, JIA Bao-guanga,b, WU Lianb, LI Cheng-xianga,b, ZHANG Lina,b
(a. Key Lab. of Cultivation and Protection for Non-wood Forest Tree Co-constructed by China Education Ministry and Hunan;b. College of Forestry, Central South University of Forestry and Technology, Changsha 410004, Hunan, China)
Transcriptome sequencing was conducted on fruits, fl owers, and bearing shoots of eliteZiziphus jujubacv. ‘Zhongqiusucui’by using Illumina-based RNA-seq technology. The microsatellites characteristics were then analyzed from the obtained unigenes. The assembled unigenes were totally 34 584, from which 12 624 microsatellites repeats were detected with Misa software. In the 1~5 bases repeat microsatellites, the mononucleotide repeat microsatellites (MNRs) were the maximum in quantitative terms (6 314, 50.02%), of which the A/T (6 217, 98.46%) was the main repeating MNRs; the next was the the dinucleotide repeat microsatellites(3 335, 26.42%), in which AG/CT was the most common DNRs(2 532, 75.92%); the third was trinucleotide repeat microsatellites(2 871,occupying 22.74%); the last was tetranucleotide and pentanucleotide repeat microsatellites( the least, accounting for 0.82% of the total microsatellites). It is concluded that the MNRs was the preponderance among Z. jujuba, and changes in the number of the MNRs repeat motifs was higher than the others, which indicates that the mononucleotide is the most active motif in the variation from theZ. jujuba’s transcriptome.
Ziziphus jujuba; transcriptome sequencing; RNA sequencing; microsatellite characteristics
S727.3;S665.1
A
1673-923X(2015)06-0093-05
10.14067/j.cnki.1673-923x.2015.06.017
2014-09-24
国家“十二五”农村领域科技计划课题(2013BAD14B03)
魏琦琦,硕士研究生 通讯作者:张 琳,副教授,博士;E-mail: triwoodtim918@126.com
魏琦琦,林 青,贾宝光,等. 枣转录组序列的微卫星特征分析[J].中南林业科技大学学报,2015,35(6):93-97.
[本文编校:吴 彬]
