双翅目摇蚊科昆虫分子系统学研究进展
2019-07-17余海军陈佳林黄鑫张钰莹田召志王茜
余海军,陈佳林,黄鑫,张钰莹,田召志,王茜
双翅目摇蚊科昆虫分子系统学研究进展
余海军,陈佳林,黄鑫,张钰莹,田召志,王茜通信作者
(天津农学院 水产科学学院,天津 300384)
摇蚊科昆虫属于完全变态昆虫,其整个生活史要经过卵、幼虫、蛹、成虫4个阶段,每个生活阶段都有其单独的特征,故基于形态对摇蚊科昆虫分类研究存在一定的挑战性。随着分子技术的提出,运用DNA测序技术对物种进行相关研究已经成为一种趋势,其中分子系统学就被广泛用于动、植物各类群的研究。本文整理总结了分子系统学在摇蚊科昆虫系统发育中的研究进展。
摇蚊科;分子系统学;基因片段
摇蚊科昆虫(Chironomidae)隶属双翅目(Diptera),长角亚目(Nematocera),常被人称为“不咬人的蚊子”,是淡水生态系统中最多样、最丰富的无脊椎动物[1],广泛分布于全球各大生物地理区,现已记述的种类超过5 000种[2]。中国已记录的摇蚊种类在1 000种左右[3],隶属于摇蚊亚科(Chironominae)、直突摇蚊亚科(Orthocladiinae)、长足摇蚊亚科(Tanypodinae)、寡角摇蚊亚科(Diamesinae)、寡脉摇蚊亚科(Podonominae)、原寡角摇蚊亚科(Prodiamesinae)6个亚科。摇蚊是一类完全变态昆虫,一生中经过卵、幼虫、蛹、成虫4个阶段,栖息地范围很广,对环境有特殊的适应性,从而能很好地检测水生生态系统和水质变化[4-5]。因此,长期以来摇蚊科一直是国际上倍受重视的昆虫类群之一。
双翅目昆虫种类繁多,不少物种之间的形态特征十分接近,基于传统形态学进行物种鉴定存在一定困难[6],且对一些特征相似种,其雌雄差异和虫态变化等情况,如:翅斑的位置、固定室数目、上附器刚毛数量和位置都存在变异,从而导致错误鉴定时有出现。伴随着分子系统学的快速发展,可利用分子系统学技术,以达到对双翅目中科、亚科、族、属和种水平系统发生关系的研究。与此同时,分子系统学技术还能帮助探索物种的起源,有学者通过对摇蚊科中所有亚科的取样证实了除原寡角摇蚊亚科(Prodiamesinae)以外其余摇蚊亚科均是单系[7],而通过摇蚊亚科的多样化速率可以说明其起源于二叠纪,在三叠纪中晚期到白垩纪早期之间,有其亚科群的起源[8]。在许多昆虫类群的系统发育研究中,分子系统学特征和数值分析方法已被广泛应用;至今为止,摇蚊科昆虫DNA序列也有部分报道,但分子系统发育研究仍很少。本文在现有文献的基础上,对摇蚊科昆虫分子系统学研究现状作一综述。
1 分子系统学研究技术
目前用于昆虫分子系统学研究的主要技术有核酸序列分析(DNA sequence analysis)、限制性片段长度多态性分析(Restriction frangment length polymorphic,RFLP)、扩增片段长度多态性分析(Amplified fragment length polymorphism,AFLP)、随机扩增DNA多态性分析(Random amplified polymorphic DNA,RAPD)、单链构象多态性分析(Single-strand conformation polymorphism,SSCP)、双链构象多态性分析(Double-strand conformation polymorphism,DSCP)等。
核酸序列分析方法是通过研究不同类群中个体同源序列的核苷酸排列顺序,构建分子系统发育树,并推断类群间的系统演化关系,是目前分子进化和系统发育研究的热点[9-10]。目前,国际上存储核酸序列的3大数据库为美国GenBank(www.ncbi.nlm.nih.gov)、欧洲EMBL(www.ebi.ac. uk/embl)和日本DDBJ(www.ddbj. nig.ac.jp)。在摇蚊分子系统学研究中大部分采用了核酸序列分析方法,Ekrem等通过3种线粒体基因(COI、COII、16S)和两种核基因(CAD和EF-1α)对摇蚊科双翅目昆虫的系统学关系进行了分析[11]。Krosch等通过核糖体基因28S、核蛋白质编码基因CAD和线粒体基因COI对长足摇蚊亚科昆虫的系统发育关系进行分析[12]。这些工作对于摇蚊科内高级阶元的分子系统学分析有重要的指导意义。
伴随着PCR技术的兴起,PCR-RFLP、AFLP和RAPD分析手段得到广泛应用。RFLP和AFLP通常用于种类鉴定或种内种群间的遗传进化分析,但在科级以上高级阶元间的系统发育研究应用较少[13]。应用RFLP方法进行昆虫分子系统学研究的主要双翅目昆虫类群是果蝇科,在摇蚊科中应用未见报道[14]。RAPD技术主要用于分类学和系统学的研究,其中在小型昆虫中应用较多,RAPD技术可通过特异性扩增图谱来确定未知标本的分类地位。Vanessa等通过RAPD法评估了摇蚊属()的遗传多样性[15]。但RAPD在系统发育分析中的应用还有一定的争论,王银东用16个随机引物对半褶皱摇蚊()、红裸须摇蚊()和刺铗长足()3 种摇蚊幼虫类群进行RAPD 扩增,结果表明刺铗长足摇蚊与红裸须摇蚊的亲缘关系较近[16]。
SSCP和DSCP是一种DNA检测技术,两种方法原理相同:突变引起DNA分子双螺旋构象的改变,从而导致他们在聚丙酰胺凝胶中电泳速度发生改变[17]。由于潜在序列的差异不能估计,SSCP和DSCP不能用于系统发育分析。但是SSCP和DSCP确实是一种快速有效的分析方法,主要用于昆虫种群遗传和进化生物学的研究。Atkinson等在白蚁种群研究中,采用DSCP技术对mtDNA控制区的DNA片段进行了分析,同时还用双翅目果蝇及膜翅目蚂蚁进行了验证[18]。
2 分子系统学研究的基因片段
2.1 线粒体DNA(mtDNA)
mtDNA拥有许多适合于进化和系统学研究的特性:(1)母性遗传;(2)结构简单;(3)序列趋异的速率相对较快;(4)与细胞核基因组相比小得多,因此成为研究进化的重要材料[19]。昆虫mtDNA大小为15.4~16.3 kb左右[20],含有12~13 个蛋白质编码基因,编码22 个tRNA的基因,编码2 个核糖体RNA亚单位的基因(12S rDNA和16S rDNA)及包括复制起点的非编码区(D-loop区)[21]。
在mtDNA中,存在高突变区域和高度保守区域,因此可选用不同区域的基因片段对不同的分类阶元进行研究,对昆虫系统发育分析,研究范围从种内、种群间至高级阶元之间均有报道。12S rDNA和16S rDNA基因大小适中,由于进化速率较慢,且可以使用通用引物,所以这两个基因是昆虫研究中使用较多线粒体基因,较适用于研究属间、不同种间及分化时间较早的种间系统关系[22-23]。Cranston 等[24]基于线粒体16S rDNA基因的IV和V域,以蠓科(Ceratopogonidae)和蚋科(Simuliidae)种类作为外群,对澳大利亚地区摇蚊科昆虫作了系统发育分析。细胞色素C氧化酶亚基I()是分子量最大、功能结构域最为保守的基因[25-26],适合于研究属、种及种下阶元的系统发育关系,而对于较高的分类阶元如科、亚科及族间的系统发育关系不能很好的解决。Makarchenko等通过选用基因来鉴定、和原寡角摇蚊亚科(Prodiamesinae)的部分物种[27]。姜丽等通过测定23种摇蚊亚科基因的部分序列,研究摇蚊亚科物种的分类和系统进化关系[28]。相关文献报道,基因适合研究动物类群属、种及种以下阶元的系统发育关系,但不能很好地解决较高的阶元如族间、亚科及科的系统发育关系。因为基因具有变异多、易被通用引物扩增、序列本身又很少存在插入和缺失等特点[29],其在物种界定和识别方面具有良好的应用性,所以将其作为DNA条形码的分子标记,用于物种鉴定。Lin等[30]通过使用基因作为DNA条形码的分子标记对长跗摇蚊属121个形态种进行系统发育分析,结果表明DNA条形码明确区分了先前通过形态学研究分类的94.6%长跗摇蚊属种类。Song等[31]通过对多足摇蚊属()27 个形态种的114 个样本,选取基因作为DNA条形码的分子标记,研究DNA条形码在多足摇蚊属中的应用,研究结果支持DNA条形码作为一种有效的方法来鉴别同类物种。Silva等[32]利用基因作为条形码的分子标记对棒脉摇蚊属()14 个形态种的86 个样本进行物种鉴定,结果显示物种与形态学描述的11 个物种一致。Amora等[33]选用基因作为DNA条形码的分子标记对形态相似的日本、韩国和巴西摇蚊属类群进行物种鉴定。细胞色素b(Cyt b)基因在线粒体基因中进化速度适中,被广泛应用于昆虫系统学的研究;Cyt b位于线粒体内膜磷脂双层中,是线粒体13个蛋白质编码基因中结构和功能被研究得最为清楚的基因之一[34]。Cyt b 基因已被证明在不同门类不同分类阶元中均含有亲缘信号,其基因片段含有缓慢和快速的进化密码子位点、保守区及多个可变区或结构域,所以其可被用于研究种内到种间,甚至科间的物种关系。Carew等[35]使用两个线粒体基因(COI和Cyt b)和1个核编码蛋白基因()来区分来自澳大利亚的前突摇蚊属()类群。结果显示,在基于线粒体基因的NJ树中,前突摇蚊属样品至少有5 个类群,且支持率大于99%,基于核基因的NJ树表明有6 个类群。
2.2 rDNA
rDNA是编码核糖体RNA的基因,是一类中度重复序列,以串联多拷贝形式存在于染色体DNA中,且每个重复单位由非转录间隔区(NTS)、转录间隔区(ITS1和ITS2)和3 种RNA(18S、5.8S和28S rRNA)基因编码区组成。所以rDNA是昆虫系统进化研究中一个非常有用且普遍的分子标记[36]。
28S rDNA是rDNA中最长的片段(4~5 kb),属于高保守区,但在其中夹有一些多变异的序列,称为D区,此序列被广泛应用于系统发育分析。Moulton[37]通过对蚋科类群的28S、EF-1α、DDC、PEPCK、12S部分基因片段分析,结果表明以上部分基因片段可以对大部分蚋类群的系统学关系进行合理解释。Allegrucci等[38]运用28S rDNA D1和28S rDNA D3-D5区片段对南极地区摇蚊科部分类群进行了系统学分析后提出,这两个基因片段可以帮助解决摇蚊科高级阶元的系统学关系。王茜等[39]利用28S rRNA部分基因序列对直突摇蚊亚科代表属级阶元进行了分子系统学研究,测定12个代表性属及长足摇蚊亚科中两个外群属的28S rRNA D1部分基因序列,分子研究结果与基于形态学的系统发育研究结果相一致。18S rDNA全长约2 kb,大小适中,既能提供足够的系统发育信息,两端序列又非常保守,利于设计通用引物进行序列扩增,但由于18S rDNA的不同区域进化速率不同,因此在不同分类层次上都有应用。Lin等[8]通过18S、AATS1、CAD、PGD和TPI基因对长跗摇蚊属进行系统发育分析,结果能明显划分长跗摇蚊属各物种之间的关系。转录间隔区ITS包括 ITS1和ITS2,全长不超过1 Kb,进化速率比较快,是区分种或种下阶元的理想分子标记[40]。Asari等[41]利用核糖体内转录间隔区ITS2对水摇蚊属()中的3 个日本种进行区分,这与形态学研究的结果一致。因为不同物种ITS1和ITS2序列在种间、种内和个体间变异不同,特别是当该序列中A或T有较多重复时,会干扰测序结果,且其序列长度变化较大会在序列比对时造成误差,所以在实际应用中此序列很少单独使用,而是联合其他基因及形态性状一起分析。Sharma[42]基于核基因ITS1和ITS2序列对摇蚊的7 个种做了详细分析,结合基因库中获得的15 个物种序列,研究表明这些物种具有很高的种间变异,可能存在隐种。
2.3 其他基因
目前一些编码核蛋白的基因被应用于昆虫系统发育分析中,其中编码EF-1α的基因使用最多,研究表明此序列在科(亚科)内的属和种团间非常有用;葡萄糖-6-磷酸脱氢酶基因(PGD)基因用于属或目级水平的系统发育研究,但不能用于近缘种的系统发育分析;Silva等[43]利用CAD的部分序列结合形态学特征对拉布长足摇蚊属()中22 个种的系统发育进行了验证,结果表明利用CAD基因序列与基于形态特征数据分析的结果一致。Lin等[44]也通过COI、CAD、PGD和AATS1(丙氨酰-tRNA合成酶)基因片段探索了长跗摇蚊属()的种内界限。综上所述,编码核蛋白的基因用于昆虫高级阶元的系统发育分析是很有效的[8]。
3 分子系统学研究类群
分子系统学的研究内容主要包括种群遗传变异及进化、种及种下阶元的分类鉴定、种上阶元的系统发育分析等[45]。其中系统发育分析是系统学研究的热点,通过分子系统发育研究对传统分类有疑问的类群或形态分类不能解决的系统发育问题进行分析和探讨,也可对传统的分类系统进行验证。目前已有许多昆虫类群从种级至目级阶元进行了分子系统发育的研究。闫春财等[46]通过比较常用的基因序列在摇蚊科昆虫系统发育研究中发现18S rDNA 和28S rDNA适合研究从界到科所有高级阶元的系统发育关系;CAD基因能解决从亚科到属之间各分类阶元的系统发育关系;ITS和EF-1α可以解决种或属间的系统发育关系;COI和COII适合研究属、种及种以下阶元的系统发育关系,但不能很好地解决较高阶元如族间、亚科及科间的系统发育关系;16S rDNA适用于种、属阶元水平的系统学研究,但不适用于种内;Cyt b适合研究种内到种间甚至科间的系统发育关系。在昆虫纲中直翅目、同翅目、半翅目、鞘翅目、鳞翅目在分子系统学方面取得了较多的研究进展。但双翅目及膜翅目的研究相对较少,且对双翅目昆虫的研究大多以果蝇为主,摇蚊科报道很少,近10年摇蚊科分子系统学研究中出现的类群与基因序列如表1所示。
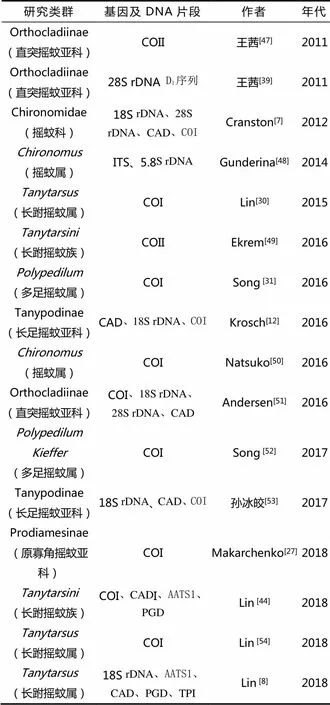
表1 摇蚊科分子系统学研究
4 数据联合分析
在数据的联合分析方面,核基因间、线粒体基因间、核基因和线粒体基因间以及分子数据和形态数据都进行了联合分析的尝试。16S rDNA、18S rDNA、COI和EF-1α是一组包含许多信息的分子标记,Caterino等[55]主张将这4个基因作为昆虫分子系统研究中的标准标记,以增加同源序列之间的可比性。Andersen等[51]使用COI、18S rDNA、28S rDNA、CADI和CADIV 5种基因对和进行系统发育分析。孙冰皎[53]选取CADI、CADIV、18S rRNA和COI基因片段对长足摇蚊亚科6 族25 属58 种进行系统发育关系的分析,研究中基于最大似然法和贝叶斯法原理对单基因序列和数据联合分析,分别构建ML和BI系统发育树,结果表明联合数据集两种方法所建系统发育树解析力优于单基因序列所建系统发育树。目前,一些学者认为用形态学方法和分子生物学方法研究系统发育,实际上是从不同的方面取得证据来阐明同一个问题,二者结合所得到的结果将更全面更具有说服力。Whiting等[56]在18S rDNA、28S rDNA和形态学的基础上,分析了包括全部全变态昆虫和20 种非变态昆虫的85 个分类单元的发育关系,绘出一幅相当完整的昆虫系统发育图。但同时,一些例子也显示形态学与分子数据在一些情况下得出的结论不一致,因此形态特征和分子特征在构建系统发育时,哪种特征更好一直是分子系统学研究中争论的话题。Moritz等[57]认为在解决某个问题时,DNA序列数据的长处在于具有明确的遗传基础,形态数据的优点在于可以从化石和保存的标本上得到,并可以从个体发育的角度进行解释。因此大量研究结果表明,将分子与形态结合起来比用单一方法对问题作出的描述和解释更好。
5 结语
随着分子系统学的不断发展,将其应用于摇蚊分类,能够有效地解决摇蚊科昆虫系统研究上遇到的一些问题,但分子系统学也有着自身的局限性。首先,虽然分子系统学对摇蚊类群具有良好的鉴别能力,但若单一依靠分子系统学对摇蚊科昆虫进行系统分类,完全脱离形态特征、生长特性等相关因素的物种鉴定,得到的结果也不能让人完全信服;其次是在选用基因标记时,不管是mtDNA基因片段还是rDNA基因片段,或其他基因片段,都还需进一步验证;且DNA序列所归属的聚类只能是分子单元或进化种,不是有生命的物种;此外,mtDNA基因在有些物种类群中可能会存在假基因(NUMT);最后,针对某些已经遗失的摇蚊物种模式标本,或是因年代过于久远而无法取证的化石样本,这些标本具有很重要的物种分类价值,如果采用分子手段提取样本DNA,会破环其样本且无法恢复,因此,把分子系统学应用于一些珍贵的模式标本是不合适的[58]。总体来说,只有将形态学系统分类与分子生物学技术结合起来,才能快速、准确、科学的鉴别摇蚊科昆虫系统发育。
[1] MiloševićD,SimićV,StojkovićM. Chironomid faunal composition represented by taxonomic distinctness index reveals environmental change in a lotic system over three decades[J]. Hydrobiologia,2012,683(1):69-82.
[2] Armitage P,Cranston P S,Pinder L C V,et al. The Chironomidae:the Biology and Ecology of Non-biting Midges[M]. London:Chapman & Hall,1995.
[3] Wang X. A revised checklist of Chironomids from China (Diptera). Late 20th Century Rarearch on Chimnomidae:an Anthology from the 13th International Symponium on Chironomidae[R]. Aachen: Shaker Verlag,2000.
[4] Nicacio G,Juen L. Chironomids as indicators in freshwater ecosystems:An assessment of the literature[J]. Insect Conservation and Diversity,2015,8(5):393-403.
[5] Sæther O A. Zoogeographical patterns in Chironomidae (Diptera)[J]. SIL Proceedings,2000,27(1):290-302.
[6] Chukwunonso O N,Abraham G C,Martín J A,et al. DNA barcoding for identification of sand fly species (Diptera:Psychodidae)from leishmaniasis-endemic areas of Peru[J]. Acta Tropica,2015,145:45-51.
[7] Cranston P S,Hardy N B,Morse G E. A dated molecular phylogeny for the Chironomidae(Diptera)[J]. Systematic Entomology,2012,37(1):172-188.
[8] Lin X L,Stur E,Ekrem T. Molecular phylogeny and temporal diversification of Tanytarsus van der Wulp (Diptera:Chironomidae)support generic synonymies,a new classification and centre of origin[J]. Systematic Entomology,2018,3:195.
[9] 聂瑞娥,杨星科. 鞘翅目高级阶元分子系统学:研究现状及存在的问题[J]. 昆虫学报,2013,56(9):1055- 1062.
[10] 李爱林,张健,张晓军. 鞘翅目昆虫分子系统学研究概况[J]. 吉林农业,2011(12):236-237.
[11] Ekrem T,Willassen E,Stur E . Phylogenetic utility of five genes for dipteran phylogeny:A test case in the Chironomidae leads to generic synonymies[J]. Molecular Phylogenetics and Evolution,2010,57(2):561-571.
[12] Krosch M N,Cranston P S,Bryant L M. Towards a dated molecular phylogeny of the Tanypodinae (Chironomidae,Diptera)[J]. Invertebrate Systematics,2017,31:302-316.
[13] 张媛,郭晓华,刘广纯,等. DNA 条形码在鞘翅目昆虫分子系统学研究中的应用[J]. 应用昆虫学报,2011,48(2):410-416.
[14] Lyttle T W,Haymer D S. The role of the transposable element hobo in the origin of endemic inversions in wild populations of Drosophila melanogaster[J]. Khwer Academic Publishers,1992,86(1-3):113-126.
[15] Vanessa C C,Guilherme R G,Thais S F,et al. Genetic diversity loss in(Diptera:Chironomidae) exposed to pyrimethanil fungicide:an analysis using RAPD technique[J]. Water Air Soil Pollut,2017,228(10):399.
[16] 王银东. 武汉市南湖大型底栖动物生态学和优势种群的遗传多样性[D]. 武汉:华中农业大学,2005.
[17] 黄建华,陈斌,周善义. 蚁科昆虫分子系统学研究进展[J]. 广西师范大学学报(自然科学版),2004,22(3):91-96.
[18] Atkinson L,Adams E S. Double-strand conformation polymorphism(DSCP)analysis of the mitochondrial control region generates highly variable markers for population studies in a social insect[J]. Insect Molecular Biology,1997,6(4):369-376.
[19] 张亚平,施立明. 动物线粒体DNA多态性的研究概况[J].动物学研究,1992,13(3):289-298.
[20] Clary D O,Wolstenholme D R. The mitochond rial DNA molecule of Drosophila yakuba:nucleotide sequence,gene organization and genetic code[J]. Molecular Phylogenetics and Evolution,1985,22:252-271.
[21] Fauron C M R,Wolstenholme D R. Intraspectific diversity of nucleotide sequences within the adenine thyminerich region of mitoch ond rial DNA molecules of Drosophila simulans[J]. Nucleic Acids Res,1900,8:5391-5410.
[22] 窦向梅,肖晖,黄大卫. DNA分类概述[J]. 生物学通报,2008,43(6):23-26.
[23] 徐庆刚,花保祯. 线粒体DNA在昆虫系统学研究中的应用[J]. 西北农林科技大学学报(自然科学版),2001,29(1):79-83.
[24] Cranston P S,Edward D,Cook L G. New status,species,distribution records and phylogeny for Australian mandibulate Chironomidae(Diptera)[J]. Australian Journal of Entomology,2002,41(4):357-366.
[25] Beard C B,Hamm D M,Collins F H. The mitochondrial genome of the mosquito Anopheles gambiae: DNA sequence,genome organization,and comparison with mitochondrial sequences of other insects[J]. Insect Mol Biol,1993,2(2):103-124.
[26] Gennis R B. Site-directed mutagenesis studies on subunit I of the aa3-type cytochrome coxidase of Rhodobacter sphaeroides:a brief review of progress to data[J]. Biochim Biophys Acta,1992,1101:184-187.
[27] Makarchenko E A, Velyaev O A,Yavorskaya N M. Morphologlcal redescription and DNA barcoding of monodiamesa kamora makarchenko ET yavorskaya (Diptera:chironomidae) from the amur river basin[J]. Far Eastern Entomologist,2018,350:1-8.
[28] 姜丽,闫娇,王婷婷,等. 基于COI基因的摇蚊亚科部分属的系统发育分析(双翅目,摇蚊科)[J]. 天津师范大学学报(自然科学版),2015,35(3):7-11.
[29] 王鑫,黄兵. DNA条形编码技术在动物分类中的研究进展[J]. 生物技术通报,2006(4):67-72.
[30] Lin X L,Stur E,Ekrem T. Exploring genetic divergence in a species-rich insect genus using 2790 DNA barcodes[J]. Plos One,2015,10(9):1-24.
[31] Song C,Wang Q,Wang X H. Exploring the utility of DNA barcoding in species delimitation of Polypedilum(Tripodura)non-biting midges(Diptera:Chironomidae)[J]. Zootaxa,2016,4079(5):534-550.
[32] Silva F L,Wiedenbrug S. Integrating DNA barcodes and morphology for species delimitation in thegroup(Diptera:Chironomidae:Orthocladiinae)[J]. Bulletin of Entomological Research,2014,104(1):65-78.
[33] Amora G,Hamada N,Fusari L M,et al. An asiatic chironomid in Brazil:morphology,DNA barcode and bionomics[J]. Zookeys,2015,514:129-144.
[34] Gray M W. Origin and evolution of mitochondrial DNA[J]. Annu Rev Cell Biol,1989,5:25-50.
[35] Carew M E,Marshall S E,hoffmann A A. A combination of molecular and morphological approaches resolves species in the taxonomically difficult genus Procladius Skuse(Diptera:Chironomidae)despite high intraspecific morphological variation[J]. Bull Entomol Res,2011,101(5):505-519.
[36] 张筠. 四角蛤蜊分子群体遗传学和系统发育[D]. 大连:辽宁师范大学,2008.
[37] Moulton J K. Can the current molecular arsenal adequately track rapid divergence events within Simuliidae(Diptera)Mol[J]. Phylogenet Evol,2003,27(1):45-57.
[38] Allegrucci G,Carchini G,Todisco V,et al. Amolecular phylogeny of antarctic chironomidae and itsimplications for biogeogra phical history[J]. Pol Bio,2006,29(4):320-326.
[39] 王茜,王新华. 直突摇蚊亚科部分属28S rDNA D1序列的分子系统发育关系(双翅目,摇蚊科)[J]. 动物分类学报,2011,36(1):93-98.
[40] 陈士林,姚辉,韩建萍,等. 中药材DNA 条形码分子鉴定指导原则[J]. 中国中药杂志,2013,38(2):141-147.
[41] Asari H,Kasuyas T K. Identilication of closely related Hydrobaenus species(Diptera:Chironomidae)using the second internal transcribed spacer(ITS2)region of ribosomal DNA[J]. Aquatic Insects,2004,26(3):207- 213.
[42] Sharma M. Molecular Identification of Chironomid Species Based on ITS1 and ITS2 Regions of rDNA[D]. Dayton:Wright State University,2007.
[43] Silva F L,Ekrem T R,Fonseca-gessner A A. Out of south America:phylogeny of non-biting midges in the genus Labrundinia suggests multiple dispersal events to central and north America[J]. Zoologica Scripta,2015,44(1):59-71.
[44] Lin X L,Stur E,Ekrem T. Exploring species boundaries with multiple genetic loci using empirical data from non-biting midges[J]. Zoologica Scripta,2018,47:325- 341.
[45] 袁锋,袁向群. 蝶类分子系统学研究进展[J]. 西北农业学报,2013,22(12):1-14.
[46] 闫春财,郭琴,赵广君,等. 常用基因序列在摇蚊科昆虫系统发育研究中的应用进展[J]. 天津师范大学学报(自然科学版),2016,36(6):54-61.
[47] 王茜,王新华. 直突摇蚊亚科部分属基于COII基因部分序列的系统发育关系(双翅目,摇蚊科)[J]. 四川动物,2011,30(5):711-716.
[48] Gunderina L I. Design of Molecular markers for identification of species of the genus(Diptera: Chironomidae)[J]. Entomological,2014,94(1):140-148.
[49] Ekrem T,Willassen E. Exploring Tanytarsini relationship (Diptera,Chironomidae) using mitochondrial COII gene sequences[J]. Insect Syst Evol,2016,35(3):264-276.
[50] Natsuko I K,Ryuhei U,Kako O,et al. DNA barcoding supports reclassification of Japanesespecies (Diptera: Chironomidae)[J]. Entomological Science,2016,19:337-350.
[51] Andersen T.,Baranov V,Hagenlund L K. Blind flight? a new troglobiotic orthoclad(Diptera,Chironomidae) from the lukina jama-trojama cave in croatia[J]. Plos One,2016,11(4):1-15.
[52] Song C,Lin X L,Wang X H,et al. DNA barcodes successfully delimit morphospecies in a superdiverse insect genus[J]. Zoological Scripta,2018,47(3):1-14.
[53] 孙冰皎. 长足摇蚊亚科高级阶元分子系统学研究[D].天津:南开大学,2017.
[54] Lin X L,Stur E,Ekrem T. DNA barcodes and morphology reveal unrecognized species in Chironomidae(Diptera)[J]. Insect Systematics & Evolution,2017,49(4):1-70.
[55] Caterino M S,Cho S,Sperling F A H,et al. The current state of insect molecular systematice:a thriving Tower of Babel[J]. Annu Rev Entomol,2000,45:1-54.
[56] Whiting M F,Carpenter J C,Wheeler Q D,et al. The Strepsiptera problem:phylogeny of the holometabolous insect orders inferred from 18S and 28S ribosomal DNA sequences and morphology[J]. Systematic Biology,1997,46(1):1-68.
[57] Moritz C,Dowling T E,Brown W M. Evolution of animal mitochondrial DNA:relevance for population biology and systematics[J]. Annual Review of Ecology and Systematics,1987,30(1):371-403.
[58] 宋南,刘杰,彩万志,等. DNA 条形码在昆虫分类中的应用[J]. 四川动物,2013,32(3):470-474.
Research progress on molecular phylogeny of the dipteran family Chironomidae
YU Hai-jun, CHEN Jia-lin, HUANG Xin, ZHANG Yu-ying, TIAN Zhao-zhi, WANG QianCorresponding Author
(College of Fisheries, Tianjin Agricultural University, Tianjin 300384, china)
The insect of Chironomidae belongs to complete metamorphosis, and its whole life cycle goes through four stages: egg, larva, pupa and adult. Each stage of life has its individual characteristics, which is challenging for the study of Chironomidae insect based on morphology. With the development of molecular technology, it has become a dendency to use DNA sequencing technology for species research, and molecular phylogeny is widely used in the study for animal and plant classification. This study summarized the research progress which are mentioned in molecular phylogeny relative to Chironomidae.
Chironomidae; molecular phylogeny; gene segment
S931.1
A
1008-5394(2019)02-0077-07
10.19640/j.cnki.jtau.2019.02.018
2019-01-04
国家自然科学基金(31672264,31672324,31272284)
余海军(1994-),男,硕士在读,主要从事分子系统学研究。E-mail:yhj18322040463@163.com。
王茜(1971-),女,副教授,博士,主要从事水生动物及昆虫分子系统学的研究。E-mail:wqgt1999@163.com。
责任编辑:张爱婷
