基于MiSeq技术的枯草期放牧蒙古羊瘤胃细菌多样性分析
2019-04-23范文斌李长青高仙灵张金然刘永斌
范文斌,李长青,高仙灵,张金然,刘永斌
(1.呼和浩特职业学院 生物化学工程学院,内蒙古 呼和浩特 010051;2.内蒙古自治区农牧业科学院,内蒙古 呼和浩特 010031)
蒙古羊是我国优良的绵羊品种,主要分布在我国北方蒙古高原常年进行放牧的草原地区。蒙古羊具有抗病能力强、耐寒冷气候、耐粗饲等特点,同时拥有较好的产肉和产皮毛性能。从古至今,牧民主要通过放牧的手段来饲养蒙古羊,它们是草原牧民赖以生存的生活保障和经济发展的主要物质基础。在饲养蒙古羊的北方草原地区,冬春季节草原的枯草期长达7~8个月,研究表明,枯草期草原的牧草产量下降,牧草的蛋白质含量下降,粗纤维的含量增加[1],在该时期仅仅通过放牧,蒙古羊摄取的营养已经不能满足其自身的需要[2],最终导致牧民的收益降低。因此,在枯草期对蒙古羊进行补饲,以补充冬春季节枯草期蒙古羊的营养摄入量,是提高蒙古羊养殖经济效益的可行方法[3-4]。
一般情况下,蒙古羊依靠采食天然牧草来维持生存和繁殖,但到了冬春季节的枯草期,牧草逐渐枯黄,品质显著下降。在这种环境胁迫作用下,蒙古羊对牧草的利用反而变得更加高效,主要是由于蒙古羊体内的微生物种群结构在自然环境的长期选择下,具有更合理的配置,从而对饲料完成了更加高效的消化、吸收与利用。瘤胃细菌数量增多和种群结构调整,均可以显著改善蒙古羊的瘤胃内环境,提高其对劣质牧草的消化率,从而提高蒙古羊的日增体质量[5]。在冬季枯草期,牧草中的纤维素含量较高,因此,发挥微生物的协同作用来增加绵羊对牧草的消化率,可能是目前提高蒙古羊对牧草利用率低的有效途径之一。因此,本研究通过对枯草期(1月中旬)放牧蒙古羊的瘤胃微生物菌群DNA进行高通量(MiSeq)测序,明确枯草期蒙古羊瘤胃微生物菌群的构成情况,以及枯草期牧草对蒙古羊瘤胃生理功能的影响和瘤胃消化的限制性因素,以期为研究蒙古羊瘤胃细菌的调控机制和探索科学补饲方法奠定理论基础。
1 材料和方法
1.1 试验动物
在枯草期(1月中旬),随机选取锡林郭勒盟苏尼特左旗草原上终年放牧(不补饲)的体况良好、体质量(50 kg)接近的6只蒙古羊(3~4岁)作为供试羊只。出牧前用真空瘤胃液收集装置通过口腔分别抽取6只供试蒙古羊瘤胃液各约50 mL,用孔径0.15 mm的4层灭菌纱布进行过滤,分装于5 mL离心管中,迅速置于-80 ℃冰箱中保存,用于瘤胃细菌菌群DNA的提取。
1.2 试验方法
1.2.1 基因组DNA的提取 采用十二烷基硫酸钠-十六烷基三甲基溴化铵(SDS-CTAB)方法提取细菌样本基因组DNA[2]。取2 μL的DNA样品,1%琼脂糖电泳检测。DNA溶液于-20 ℃冰箱保存。
1.2.2 16S rDNA扩增及MiSeq测序 细菌基因组文库构建及MiSeq测序由上海美吉生物公司完成。以总DNA为模板,对细菌16S rDNA的V3—V4区进行PCR扩增,建立DNA文库,所采用的细菌通用引物为338F(5′-ACTCCTACGGGAGGCAGCA-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)。PCR采用TransGen AP221-02:TransStartFastpfu DNA Polymerase,PCR仪为ABI GeneAmp®9700型。将检测合格的DNA片段经电泳后,使用AxyPrepDNA凝胶回收试剂盒(AXYGEN公司)回收,并进行Tris-HCl洗脱,然后经2%琼脂糖电泳检测。参照电泳初步检测的结果,将PCR产物用QuantiFluorTM-ST蓝色荧光定量系统(Promega公司)进行检测定量,然后按照每个样品的测序量要求,进行相应比例的混合,采用Illumina的TruSeq Nano DNA LT Library Prep Kit构建Illumina测序文库,合格的文库采用Illumina MiSeq进行2×300 bp的双端测序。
1.2.3 基因序列的生物信息学分析 原始测序数据经质量控制后,舍弃低质量序列(Read尾部碱基质量小于20,质控后的Read小于50 bp),获得的高质量序列使用UPARSE version 7.1软件(http://drive5.com/uparse/)进行OTU聚类,以16S rDNA序列97%的相似度作为OTU的划分标准。获得的OTU与RDP数据库(http://rdp.cme.msu.edu/)进行比对,采用RDP classifier贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析,并在各个分类水平(门、纲、目、科、属、种)统计每个样品的群落组成。用Mothur version v.1.30.1指数分析软件完成Alpha多样性分析,包括丰富度指数(Chao、Ace)和多样性指数(Shannon、Simpson)[6-8]。
2 结果与分析
2.1 蒙古羊瘤胃细菌OTU聚类和Alpha多样性分析
对枯草期放牧蒙古羊的瘤胃细菌进行OTU聚类和Alpha多样性计算,共获得103 162条高质量的16S rDNA序列,平均每个样品17 193.67条,平均长度为442.33 bp。序列经拼接优化后,根据97%的相似度水平,对所有序列进行OTU划分,共获得2 774个OTU,由表1可知,平均每个样品OTU为462.30,Chao指数为559.00,Ace指数为546.00,Shannon指数为3.11,Simpson指数为0.22。
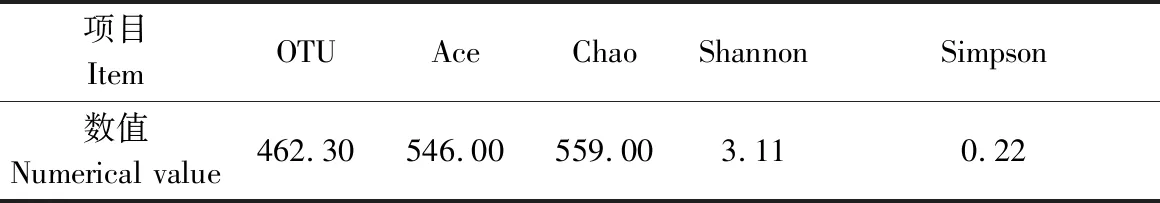
表1 枯草期放牧蒙古羊瘤胃细菌OTU聚类和Alpha多样性指数
2.2 蒙古羊瘤胃细菌菌群结构分析
对蒙古羊瘤胃细菌共13个门、24个纲、30个目、59个科、85个属进行鉴定,由表2可知,在门水平上,厚壁菌门(Firmicutes,58.59%)和拟杆菌门(Bacteroidetes,18.18%)是放牧绵羊瘤胃中占主导地位的细菌,而软壁菌门(Tenericutes,5.99%)、变形菌门(Proteobacteria,5.62%)相对含量也较高。瘤胃菌科(Ruminococcaceae,15.43%)、毛螺菌科(Lachnospiraceae,14.86%)和克里斯蒂娜科(Christensenellaceae,11.90%)在科的水平上为绝对的优势菌。在属水平上,除了未分类和RC9外,普雷沃氏菌属(Prevotella,2.89%)和Candidatussaccharimonas(2.89%)是瘤胃中占主导地位的细菌。这些微生物在枯草期蒙古羊瘤胃消化牧草的过程中均起着重要的作用。
3 结论与讨论
在北方牧区枯草期放牧蒙古羊瘤胃细菌中,厚壁菌门在门水平上,瘤胃菌科和毛螺菌科在科水平上为绝对的优势菌,表现出典型的纤维消化菌群特征,因此,在冬春季为这2种菌科的细菌提供营养,将有效改善蒙古羊瘤胃的发酵功能,提高其对牧草的消化率。
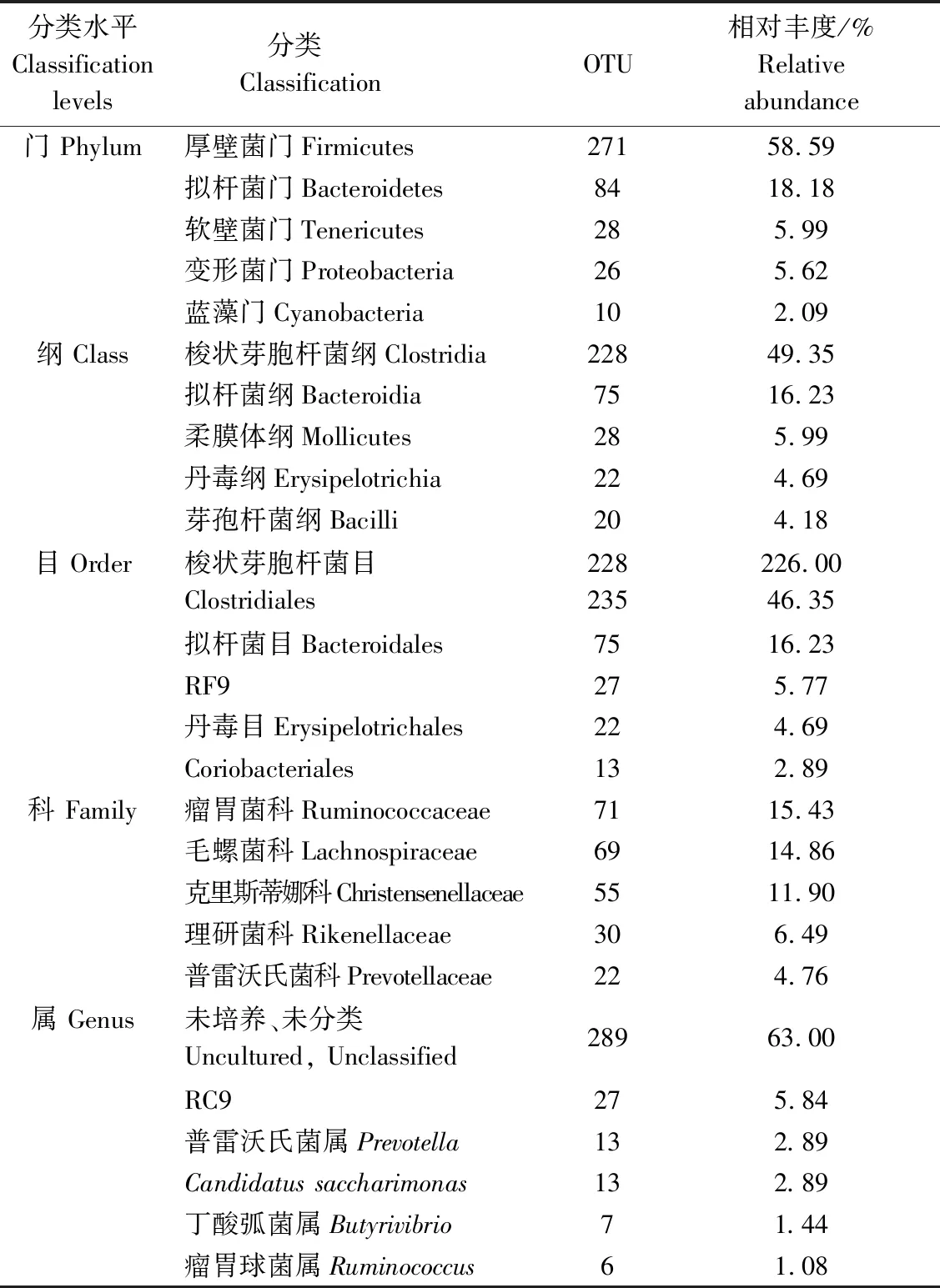
表2 不同分类水平相对丰度较高的瘤胃细菌Tab.2 Rumen bacteria with higher relative abundance at different classification levels
蒙古羊在长期的自然进化过程中形成了独特的瘤胃发酵系统,其内栖息着多种多样的微生物,与宿主之间形成了一种相互制约、相互依赖的复杂动态平衡,在对营养物质消化吸收中起着举足轻重的作用。特别是在冬季枯草期,牧草的纤维素含量较高,发挥蒙古羊瘤胃微生物的协同作用来增加牧草的消化率,是提高牧草利用率的有效手段,可以提高放牧家畜补饲的效果并降低养殖成本。因此,了解枯草期放牧蒙古羊瘤胃微生物的情况是提高蒙古羊放牧养殖效益的第一步。
本研究通过对蒙古羊瘤胃细菌种群DNA进行MiSeq测序,明确了蒙古羊在枯草季节瘤胃微生物菌群构成情况。研究结果表明,在门水平上,放牧蒙古羊瘤胃细菌中厚壁菌门(58.59%)和拟杆菌门(18.18%)为放牧羊瘤胃中的优势细菌种群,其中厚壁菌门为绝对的优势菌。这种菌群组成类似于其他品种羊的瘤胃微生物结构,孙国雷[9]的研究表明,济南动物园喜马拉雅塔尔羊瘤胃细菌相对丰度最高的为厚壁菌门(66.11%)、拟杆菌门(25.91%),欧洲盘羊瘤胃细菌相对丰度最高的为厚壁菌门(57.17%)、拟杆菌门(33.67%)、岩羊瘤胃细菌相对丰度最高的为厚壁菌门(57.22%)、拟杆菌门(33.48%),与本研究的结果相近,它们均表现出典型的纤维消化菌群特征,因此,这2个种群瘤胃细菌的变化将影响羊瘤胃的消化能力。但在陈芸等[10]研究中,川中黑山羊瘤胃细菌相对丰度最高的为拟杆菌门(40.87%)和厚壁菌门(27.19%),在曾燕[11]研究中,四川雅安市蒙古羊瘤胃相对丰度最高的为拟杆菌门(67.00%)和厚壁菌门(23.00%),本研究中,放牧蒙古羊瘤胃相对丰度最高的为厚壁菌门(58.59%)和拟杆菌门(18.18%),与前者存在较大的差异。这个结果可能与地区差异引起的饲喂饲料不同有关,此外,不同品种羊瘤胃内的优势细菌相对丰度也存在一定的差异。蒙古羊常年放牧于草原,大多数时间采食的是富含纤维的牧草,因此瘤胃内细菌以分解纤维的厚壁菌门为主,厚壁菌门中瘤胃球菌属、丁酸弧菌属和假丁酸弧菌属等纤维降解菌促进纤维物质的消化吸收[12]。而生产中舍饲羊一般以饲喂富含淀粉的精料居多,因此以分解淀粉、可溶性碳水化合物的拟杆菌门占优势。拟杆菌门能分解饲料中的碳水化合物和蛋白质等物质,并协助宿主利用饲料中其本身不能消化的多糖成分,促进饲料中营养物质的消化吸收,从而提高营养物质的利用率[13]。
在属水平上,本研究发现,放牧蒙古羊瘤胃细菌的60.00%以上为不能培养或者不能命名的种类,明确鉴定的菌属主要包括普雷沃氏菌属(2.89%)、Candidatussaccharimonas(2.89%)和丁酸弧菌属(1.44%)。BEKELE等[14]研究发现,绵羊瘤胃中大部分普雷沃氏菌(87.80%)的序列与已知普氏菌序列的相似度低于97.00%,表明不可培养的普氏菌数量远超过已知的普氏菌,其特定的生理功能也有待于进一步探索[15]。川中黑山羊瘤胃相对丰度最高的菌属为普雷沃氏菌属(25.53%),其次是琥珀酸弧菌属(7.56%)[10],四川雅安市蒙古羊瘤胃相对丰度最高的普雷沃氏菌属所占比例为53.00%[11],均与本研究有较大的差异。SINGH等[16]研究表明,水牛瘤胃中普雷沃氏菌在纤维饲料精粗比为0∶100的营养条件下数量最高,说明瘤胃普雷沃氏菌在反刍动物瘤胃中具有非常重要的作用。另外,品种差异也会导致瘤胃微生物的组成发生变化,有研究发现,不同品种反刍动物瘤胃内微生物多样性差别较大[17]。但瘤胃内微生物对反刍动物的影响是不容忽视的。
