河东乌麦低分子量谷蛋白基因的生物信息学分析
2019-02-21,,,
, ,,
(1.湖南农业大学 农学院,湖南 长沙 410128;2.桃源县第三中学,湖南 桃源 415701;3.湖南文理学院 生命与环境科学学院,湖南 常德 415000)
我国小麦在种植面积、总产量和消费量上,皆居世界首位,是我国第三大粮食作物。小麦的蛋白质占小麦籽粒重量的8%~20%,分为麦谷蛋白和醇溶蛋白,它们是决定面团弹性和延伸性的物质基础。面食的品质优劣主要依赖于小麦籽粒贮藏蛋白——面筋蛋白的结构及其相互作用。用水或者稀盐溶液冲洗面团,剩下的物质称为面筋。面筋中有80%由蛋白质组成,它们称为面筋蛋白,由醇溶蛋白和麦谷蛋白组成[1]。面筋蛋白之间通过很强的共价键和非共价键相互作用,赋予面团粘弹特性[2]。醇溶蛋白是单体蛋白,主要影响面团的粘性和延展性;而麦谷蛋白可以形成多聚体蛋白,主要影响面团的弹性,并最终影响小麦面粉的加工品质。因此对麦谷蛋白组成、结构和功能的研究一直是小麦品质改良领域的研究热点[3]。麦谷蛋白根据分子量大小可分为高分子量麦谷蛋白(HMW-GS)和低分子量麦谷蛋白(LMW-GS)。LMW-GS基因没有内含子,是一个单一的开放阅读框,其编码序列一般含有900~1200个碱基,其编码的成熟蛋白亚基的分子量大小约30~45kDa。重复区域一般以PPFSQQ为主要的重复单元,重复单元的个数直接影响着LMW-GS分子量的大小;C-I区比较保守,是半胱氣酸富集的区域,C-II区则是脯氨酸富集的区域,序列结构不保守,C-III区域含有一个半胱氨酸,其氨基酸序列也比较保守[4]。我国小麦蛋白质含量并不低,经过多年定向选择,也不乏优质亚基,但面筋质量仍然较差,说明贮藏蛋白组成不合理,组份比例及绝对含量可能是主要问题。谷蛋白总量及组份含量主要受品种遗传特性决定,而麦谷蛋白含量与面团特性和面包加工品质高度相关,可以通过培育谷蛋白高表达量的品种,提高品质性状的环境稳定性。本研究以河东乌麦为实验材料,对其低分子量麦谷蛋白编码基因进行生物信息学分析,为小麦低分子量谷蛋白基因研究增添新内容,为优化小麦加工品质提供参考。
1 材料与方法
1.1 供试材料
培养保存于湖南文理学院生命与环境科学学院种质资源库的河东乌麦的黄化幼苗提取其总基因组DNA。
1.2 研究方法
利用CTAB法提取供试材料总基因组DNA,1%琼脂糖凝胶检测DNA质量。根据NCBI数据库中已报道的低分子量谷蛋白基因序列的保守区,分别设计可扩增低分子量谷蛋白基因完整编码区的兼并性引物,对其进行PCR扩增,1%琼脂糖电泳检测扩增结果,用PCR产物琼脂糖凝胶回收试剂盒(天根生化科技有限公司)对目的片段进行回收纯化。
将回收纯化的PCR产物送专业公司进行测序,本研究序列测定由南京金斯瑞生物科技有限公司完成。测序结果比较分析采用NCBI网址中的Blast(http://www.ncbi.nlm.nib.gov/BLAST/)程序进行;DNA序列分析、系统演化、蛋白质序列分析等用DNAMAN8.0软件完成。
2 结果与分析
2.1 PCR扩增结果
对提取获得的河东乌麦总基因组DNA进行扩增,扩增产物用1%琼脂电泳进行检测,结果表明扩增出1条约920bp的亮带(图1)。
2.2 核酸序列信息
通过测序,获得一条长918bp,分子量为565.94kDa的核苷酸序列(图2)。
序列包括275个腺嘌呤核糖核苷酸(A),占整条DNA核苷酸链的30.0%;294个胞嘧啶核糖核苷酸(C),占32.0%;156个鸟嘌呤核糖核苷酸(G),占17.0%;193个胸腺嘧啶核糖核苷酸(T),占21.0%(表1)。
该DNA序列GC含量约为49.0% ,AT含量约为51.0%。其中GC在序列含量越高,DNA的双螺旋结构越稳定,在PCR扩增过程中所需要的退火温度就越高。
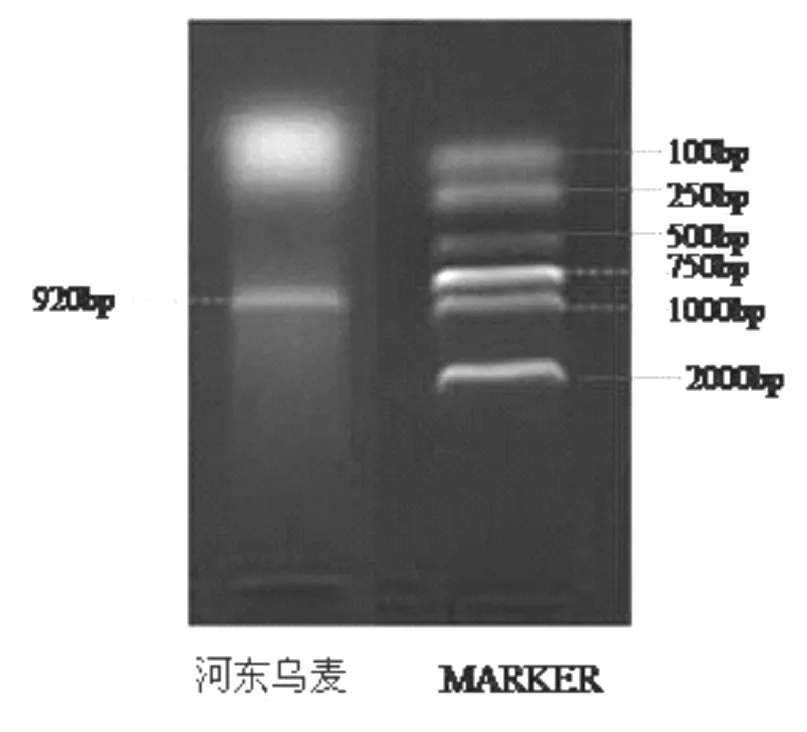
图1 PCR扩增产物琼脂糖凝胶电泳结果 表1 核苷酸序列的各种碱基的数目及百分数
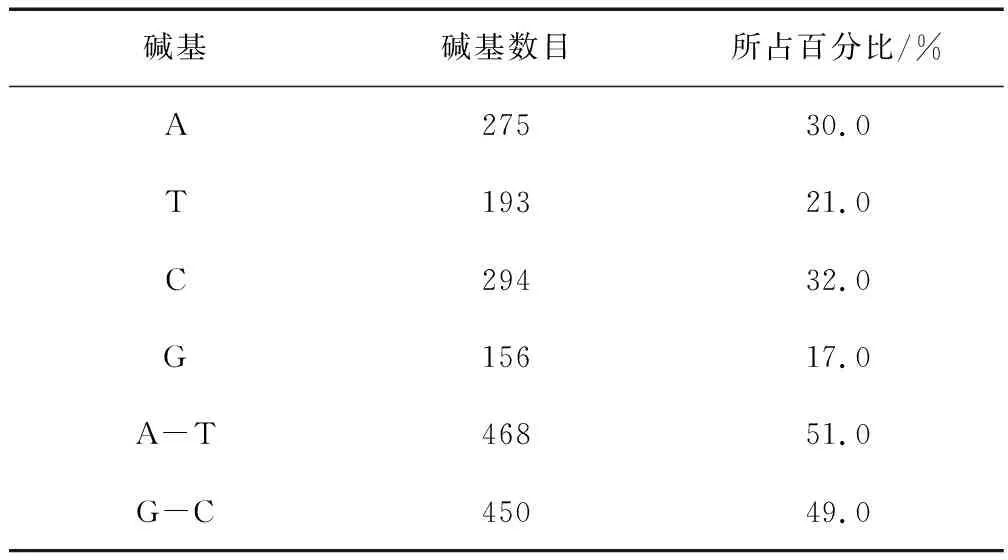
碱基碱基数目所占百分比/%A27530.0T19321.0C29432.0G15617.0A-T46851.0G-C45049.0

图2 河东乌麦低分子量谷蛋白基因编码的核酸序列
2.3 核酸限制性酶切图谱
在河东乌麦低分子量谷蛋白编码的基因核酸序列中通过DNAMAN用了117种常见酶进行筛选。共筛选出18个限制性酶切位点,其中包括了17种酶,ApaBI在序列中有两个酶切位点,分别在47bp和566bp处,其它16种酶Acc65I,AlwNI,Asp718I,BbvII,BglI,Bpu1102I,Bsc91I,Bsp1407I,Eco31I,EspI,KpnI,MstI,NcoI,SpeI,XcmI,XmnI在序列中各有一个位点(图3)。
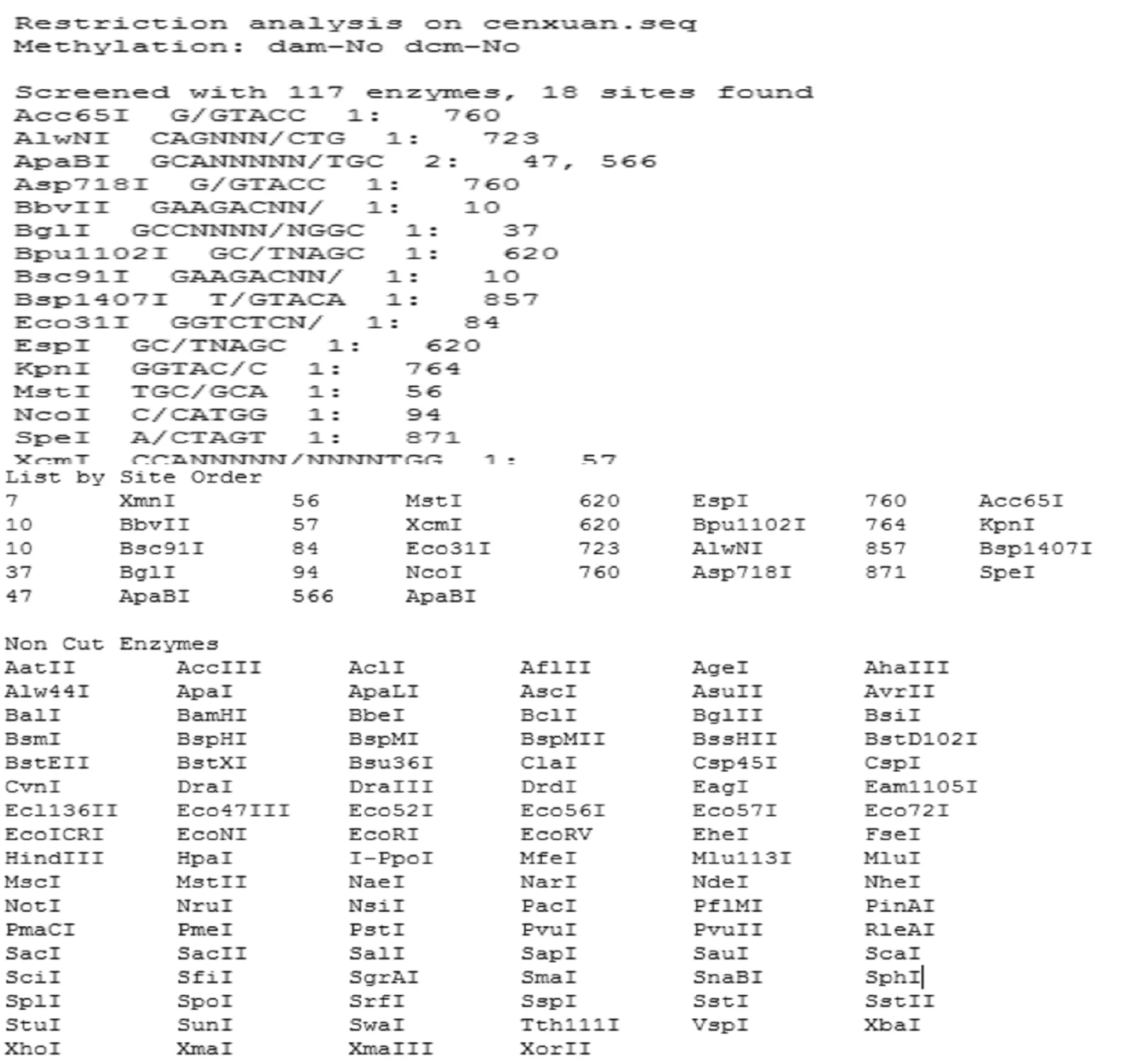
图3 河东乌麦低分子量谷蛋白编码基因DNA限制酶切图谱
2.4 核酸序列比对碱基同源性分析
利用NCBI对河东乌麦低分子量谷蛋白基因序列进行基因比对,发现有9条基因序列与河东乌麦基因的相似性达到99%。随机选取相似性较高的8条序列进行比对分析。结果发现与河东乌麦相似性最高的是普通小麦,两者同源性最高,其次是乌拉尔图小麦、Taeniatherum crinitum、节节麦(表2)。

表2 河东乌麦LMW-GS基因序列的同源性比对
用这8条核酸序列和研究的目的核算序列利用DNAMAN软件进行碱基同源性比对分析,构建系统发育树(图4)。
结果显示FJ549935和KM085255的该基因碱基排列顺序及种类极其相似,具有很高的同源性,最先聚为一类。说明这2个基因序列可能是由于环境等因素变化引起该种群的基因频率发生了变化,即趋异进化而形成的两个基因。它们拥有一个共同的祖先,因此可聚为一类。而河东乌麦的低分子量谷蛋白基因最先与FJ549935,KM085255,KX879103,FJ755302,KU522466聚成一类,最后才与JX828341聚成一类,说明该河东乌麦的基因与FJ549935,KM085255,KX879103,FJ755302,KU522466亲缘关系较近,与后者亲缘关系较远。
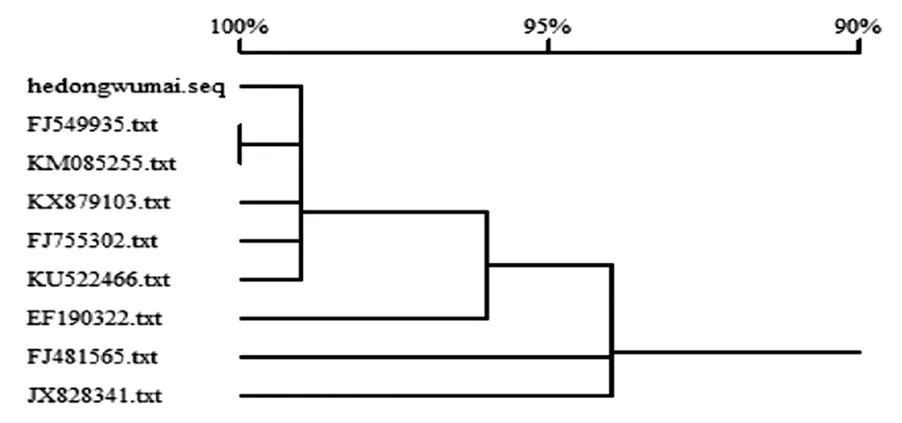
图4 系统发育树
利用DNAMAN软件对同源相似性程度较高的8个序列进行碱基同源性分析(图5)。碱基高度保守的区域:位点在388~403bp、415~429bp、447~470bp、491~506bp、508~524bp、620~644bp、665~719bp、775~790bp处碱基序列完全一致,所以这几种不同生物体的不同基因在这些区域内是高度保守的DNA序列,不易突变。碱基发生缺失的区域:EF190322在347~352bp处,KM085255在534~536bp处都发生了一个碱基的缺失,而在348~350bp处7条序列都出现了碱基的基因缺失。
目的序列在区域中一共发生了5处碱基突变。在490bp,A突变成C,在539bp,C突变成T,在556bp,G突变成C,在663bp,A突变成G,591bp处,A突变成G。

图5 基因核酸序列比对
2.5 蛋白质结构分析
蛋白质二级结构是蛋白质复杂空间构象的物质基础,是蛋白质可以行使各种各样生命活动功能的前提。利用DNAMAN分析软件对目的核酸序列编码的氨基酸序列进行分析。其中Gln谷氨酰胺的含量最高,为35.00%,其次Pro脯氨酸含量较高,为10.34%,Asp天冬氨酸含量最低,仅为0.33%(图6)。
对该序列所翻译的蛋白质二级结构进行了预测,结果发现,该蛋白质二级结构主要为无规则卷曲(coils),主要分布于第20~154个氨基酸片段内,有零星片层结构。其中氨基酸为306个,分子量大小为34588.0kDa(图7)。
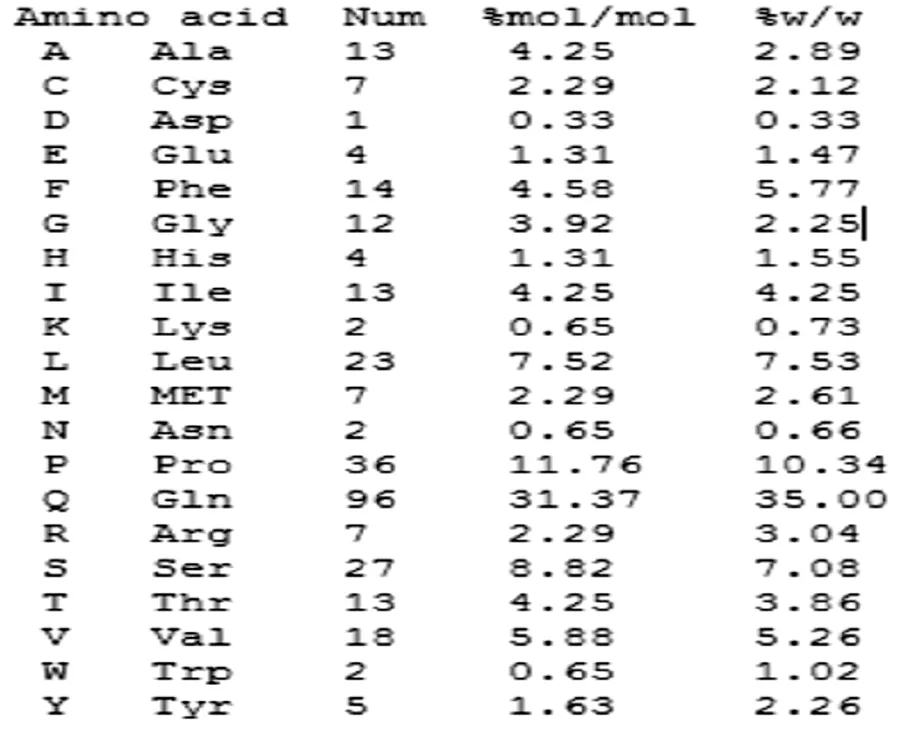
图6 河东乌麦低分子量谷蛋白编码基因氨基酸序列信息
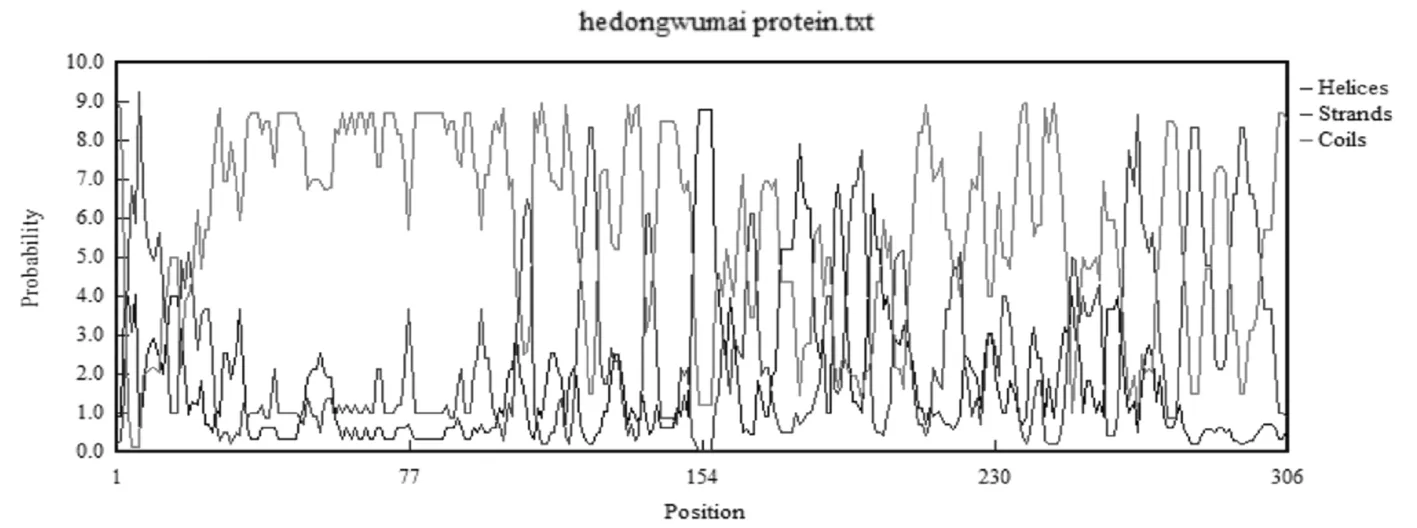
图7 蛋白质二级结构预测
2.6 蛋白质疏水性分析
利用DNAMAN分析软件对该序列编码的氨基酸序列进行了疏水性分析,结果显示,1~25bp,256~306bp处为疏水性,26~120bp,190~250bp处为亲水性(图8)。
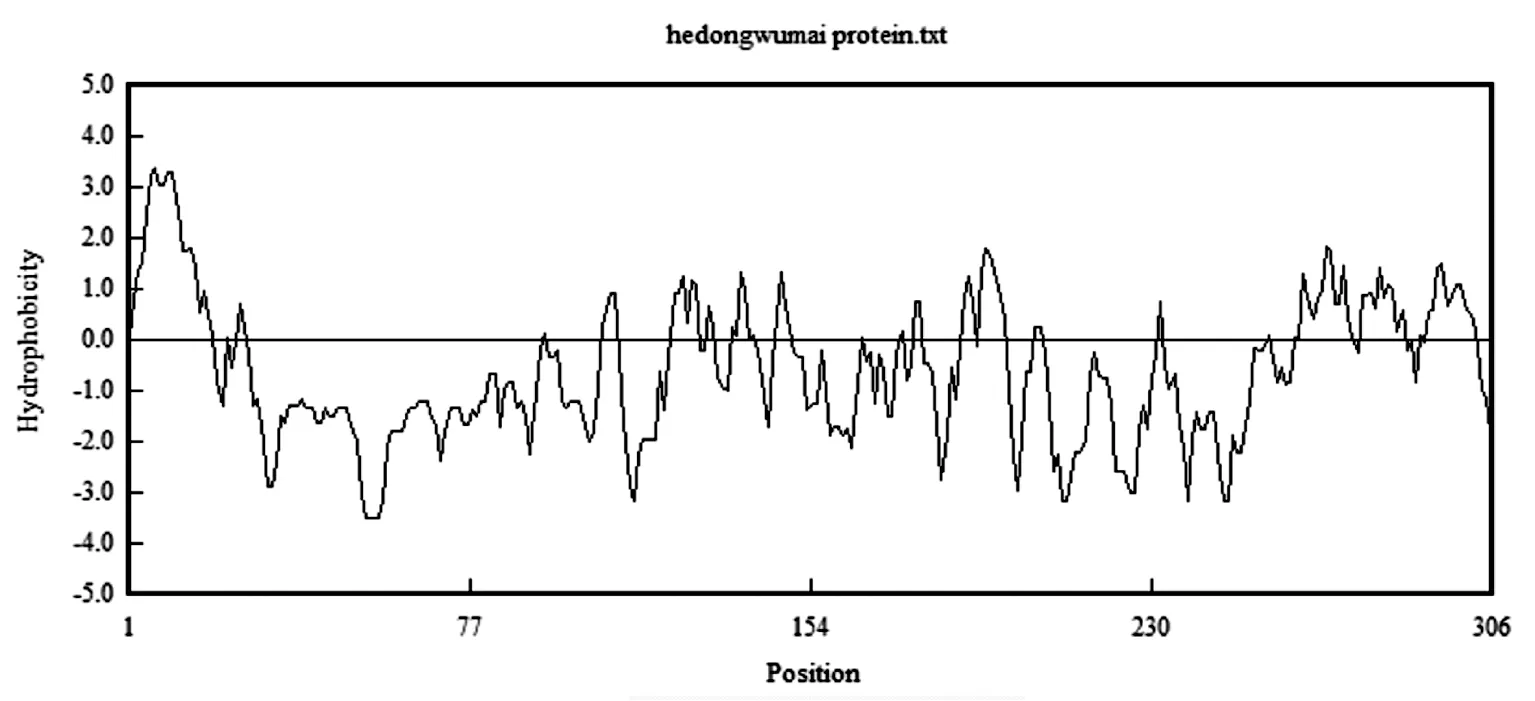
图8 蛋白质疏水性分析结果
2.7 蛋白质信号肽分析
利用DNAMAN分析软件对该序列编码的氨基酸序列进行了分析,结果得出河东乌麦蛋白质信号肽序列为MKTFLVFALLAVVATSAIA(图9)。

图9 蛋白质信号肽序列
3 结论与讨论
利用PCR扩增技术,从河东乌麦基因中分离得到一条长度为918bp的低分子量谷蛋白编码基因序列,其上共有18个酶切位点,GC含量为49.0%。
利用NCBI网站对该段序列BLAST同源比对结果表明,该基因与来自于普通小麦低分子量麦谷蛋白中得亚基GluA3-21和GluA3-1基因高度相似,相似值达到99%。系统演化分析表明,该基因与编号1、2、3、4、5的序列先聚到一类,表明这6个基因片段的碱基序列同源程度高,其亲缘关系较近。
利用DNAMAN分析软件对该核苷酸序列编码得氨基酸序列进行分析,结果得出河东乌麦蛋白质信号肽序列为MKTFLVFALLAVVATSAIA。进行人工翻译后,得到306个氨基酸,分子量大小为34588.0kDa。其中,含量最高的氨基酸为Gln谷氨酰胺,含量为35.00%,周琴等人[5]研究中表明,随着谷氨酰胺水平的提高,籽粒的淀粉含量呈现递减趋势,与淀粉含量变化趋势相反,籽粒蛋白质的含量随谷氨酰胺供应水平的的提高而迅速上升,而对于单个籽粒中的淀粉和蛋白质的积累量有促进作用,但是对蛋白质的促进作用大于淀粉的促进作用。二级结构分析表明,该蛋白质主要由卷曲(coil)结构组成,在第20-154号氨基酸区段分布最为密集,其次为片层结构。蛋白质的结构主要影响小麦的加工品质。
