棉花陆海杂种标记偏分离染色体上重要农艺性状的QTL定位
2018-12-12戴宝生郭欢乐尤春源张献龙林忠旭
戴宝生,郭欢乐,尤春源,张献龙,林忠旭 *
(1.华中农业大学植物科学与技术学院/作物遗传改良国家重点实验室,武汉430070;2.黄冈市农业科学院,湖北黄冈438000;3.石河子农业科技开发研究中心棉花研究所,新疆石河子832000)
棉属栽培种包括2个重要的异源四倍体种,分别是陆地棉(Upland cotton)和海岛棉(Sea island cotton)。陆地棉具有高产稳产、适应性广等特点,在世界范围内广泛种植,其产量占世界棉花总产量的90%以上,在棉花生产中具有十分重要的地位[1-3]。海岛棉具有纤维品质优良等优点,是高档纺织品和特种棉纺织品的重要原料,在国际市场上占有一定的份额,在中国、美国、埃及和乌兹别克斯坦等植棉国仍有一定的种植面积[3]。
近年来,棉花育种进展缓慢,产量水平提升不明显,早熟性难以满足生产需要,棉花育种进入平台期,传统的常规育种已经难以适应现代棉花育种的要求。分子标记技术的发展及覆盖全基因组的饱和遗传图谱的构建,使得研究人员能够在分子水平对产量和熟性等复杂的数量性状进行遗传解析,能够定位与棉花熟性、产量相关性状的数量性状位点(Quantitative trait locus,QTL)[4-8]。用海岛棉和陆地棉构建的群体遗传多态性好,利用海陆种间群体定位棉花重要农艺性状QTL一直是研究热点[9-11]。但是目前定位的QTL多是使用来自于2个亲本的单一群体,没有在多个群体中进行验证[9-11]。
本研究使用海岛棉3-79和陆地棉鄂棉22构建了4个BC1F2群体。在田间考查各个群体的产量性状和熟性相关性状,室内检测各株行的纤维品质性状。本实验室在构建棉花种间连锁图过程中,发现海陆种间群体中第2、16和18号3条染色体上偏分离标记数目较多(50%以上),并且大多数标记偏向杂合型,并且偏分离标记聚集形成偏分离区域[3]。利用上述4个BC1F2群体的偏分离严重的3条染色体的基因型数据进行产量、熟性和纤维品质相关性状的QTL定位,挖掘这2个材料基因组上的有利等位变异,以期检测到稳定的QTL位点,用于QTL精细定位、基因克隆和分子标记辅助选择。
1 材料与方法
1.1 试验材料和田间种植
利用海岛棉3-79和陆地棉品种鄂棉22(Emian 22)为亲本及其正反交F1构建了4个陆地棉基因组成分比例较大的BC1F1回交群体:Emian 22/ (Emian 22/3-79)、Emian 22/(3-79/Emian 22)、 (Emian 22/3-79)/Emian 22、 (3-79/Emian 22)/Emian22,分别简写为 E (E3)、E(3E)、(E3)E、(3E)E。2013 年收获 BC1F1单株种子,2014 年在长江大学试验地(湖北省荆州市荆州区)种植BC1F2家系和亲本材料,其中E (E3)118行、E(3E)94 行、(E3)E 102 行、(3E)E 103 行。株行材料和亲本按照单行小区种植,长5 m,宽0.8 m,种植10株,采用随机区组排列,每个家系种植2个重复。
1.2 农艺性状调查
调查记载各株行的出苗期、开花期、吐絮期,计算出苗至开花期(Days from seedling to flowering,SF)、吐絮期(Date of boll opening,DBO)、开花至吐絮期(Days from flowering to boll opening,FBO)等熟性相关性状。在棉花吐絮期,收获棉株中部吐絮良好的棉铃20个,室内测定铃重(Boll weight,BW)、衣分(Lint percent,LP)、籽指(Seed index,SI)、衣指(Lint index,LI)等产量相关性状。考察纤维长度 (Fiber length,FL)、断裂比强度(Fiber strength,FS)、马克隆值(Micronaire value,MIC)、纤维长度整齐度指数 (Fiber length uniform,FLU)、纤维成熟度(Fiber maturity,FM)、纺纱均匀性指数 (Spinning consistency index,SCI)及短纤维率 (Short fiber ratio,SFR)7个纤维品质性状。
1.3 数据分析
4个BC1F2群体的熟性相关性状、产量性状和纤维品质性状使用统计学软件R包library(pastecs)的 stat.desc函数[12]进行描述性统计分析,包括每个性状的最大值、最小值、平均值、标准差、变异系数、偏度系数、峰度系数和正态性检验结果等。亲本间的性状差异通过t测验检验。性状间的相关分析采用统计学软件R包library(psych)中的corr.test函数[13]进行。选取本实验室发表的棉花海陆种间图谱上第2、16和18号染色体上的共显性标记对全部群体进行了基因型分析。基于4个群体的表型数据,对各个群体的熟性相关性状、产量性状和纤维品质相关性状进行QTL检测。采用软件Windows QTL Cartographer 2.5[14]中的复合区间作图法(Composite interval mapping,CIM)进行QTL区间定位和效应估计,进行1 000次置换检验,对数优势比(Logarithm of the odd score,LOD)大于 2.5 时认为存在QTL。QTL命名按照Mc Couch等[15]的规则进行。
2 结果与分析
2.1 亲本及BC1F2群体的产量、纤维品质和熟性相关性状的表现
表1为4个群体及亲本的种植表现,对产量、纤维品质和熟性相关性状进行了统计分析。从表1可知,2个亲本的断裂比强度、纺纱均匀性指数、铃重、衣分和出苗期至开花期历时5个性状存在极显著差异,纤维长度和短纤维率2个性状存在显著差异,而其他7个性状差异不显著。正反交F1表型分析结果表明,正反交F1的产量相关性状和熟性相关性状差异不显著,这说明鄂棉22和3-79杂交F1产量性状和熟性相关性状受核基因组控制,与细胞质基因组无关;但断裂比强度存在显著差异,采用海岛棉3-79做母本的F1纤维断裂比强度较好。
由表2和表3可知,短纤维率在4个BC1F2群体中变异较大,变异系数为18.0%~23.0%;纤维成熟度在4个群体中变异较小,变异系数为1.0%~2.0%。与亲本相比,4个群体每个性状的最大值都超过了高值亲本,具有正向超亲优势,同时最低值都比低值亲本还低,表明这些性状同时具有正向和负向超亲优势,显示了超亲分离现象。

表2 4个BC1F2群体的纤维品质相关性状表型数据的统计描述Table 2 Descriptive statistics of fiber quality-related traits in the four BC1F2populations
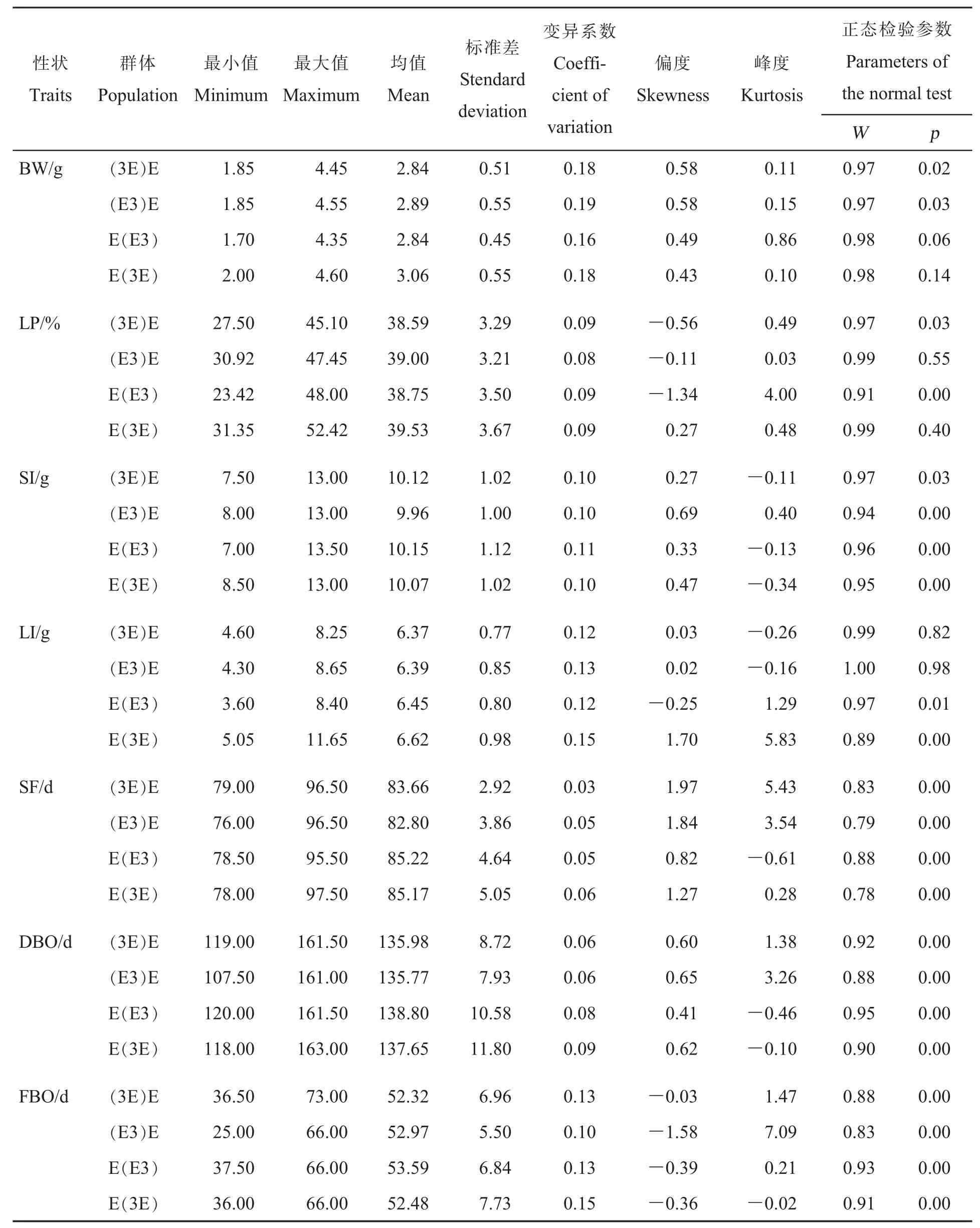
表3 4个BC1F2群体的产量和熟性相关性状表型数据的统计描述Table 3 Descriptive statistics of yield-and matarity traits in the four BC1F2populations
Shapiro-Wilk正态分布检验显示,就纤维品质相关性状而言,短纤维率在4个群体中均不符合正态分布,马克隆值在E(E3)群体不符合正态分布,其他6个纤维品质性状在4个群体中均符合正态分布。但是与棉花熟性相关的3个性状在4个群体中都不符合正态分布,说明熟性相关性状可能与环境有更强的互作效应。在产量性状中,铃重在4个群体中均符合正态分布;衣分在除E(E3)外的其他3个群体中均符合正态分布;籽指仅在(3E)E群体中符合正态分布;衣指在除E(3E)外的其他3个群体中均符合正态分布。峰度和偏度分析表明,除衣分在E(E3),衣指在E(E3)、E(3E),SF 在 E(3E)、(E3)E、(3E)E,FBO在(E3)E、(3E)E 外,其他性状在各群体中偏度和峰度的绝对值都小于1,说明这些性状近似于正态分布,表明这些性状是受多基因控制的数量性状。
2.2 棉花熟性、产量和纤维品质性状的相关分析
棉花熟性、产量与纤维品质的相关性分析结果(表4)表明:纤维长度与多数品质相关性状极显著相关,其中极显著正相关的有纤维断裂比强度、长度整齐度指数、纺纱均匀性指数,极显著负相关的有马克隆值、短纤维率。纤维长度与籽指呈显著正相关,与衣分呈极显著负相关。纤维断裂比强度与纺纱均匀性指数呈极显著正相关,与短纤维率呈极显著负相关。马克隆值与纤维成熟度呈极显著正相关,与纺纱均匀性指数呈极显著负相关。纤维长度整齐度指数与纺纱均匀性指数呈极显著正相关,与短纤维率呈极显著负相关。纺纱均匀性指数与短纤维率呈极显著负相关。短纤维率与籽指呈极显著负相关。衣分与衣指呈极显著正相关,与籽指呈显著负相关。出苗至开花期与吐絮期、开花至吐絮期呈极显著正相关。吐絮期与开花至吐絮期呈极显著正相关。
2.3 棉花熟性、产量和纤维品质性状的QTL定位
利用复合区间作图法对4个BC1F2分离群体的纤维品质、产量及熟性相关性状进行QTL定位分析,检测到 47个 QTLs(表 5、表 6和图 1),在 E(E3)、E(3E)、(E3)E 和(3E)E 中分别检测到12、7、15和 13个 QTLs, 解释 8.8%~30.9%的表型变异,LOD值为2.535~7.468。在这47个QTL中,27个与纤维品质性状相关,7个与产量性状相关,13个与熟性相关。
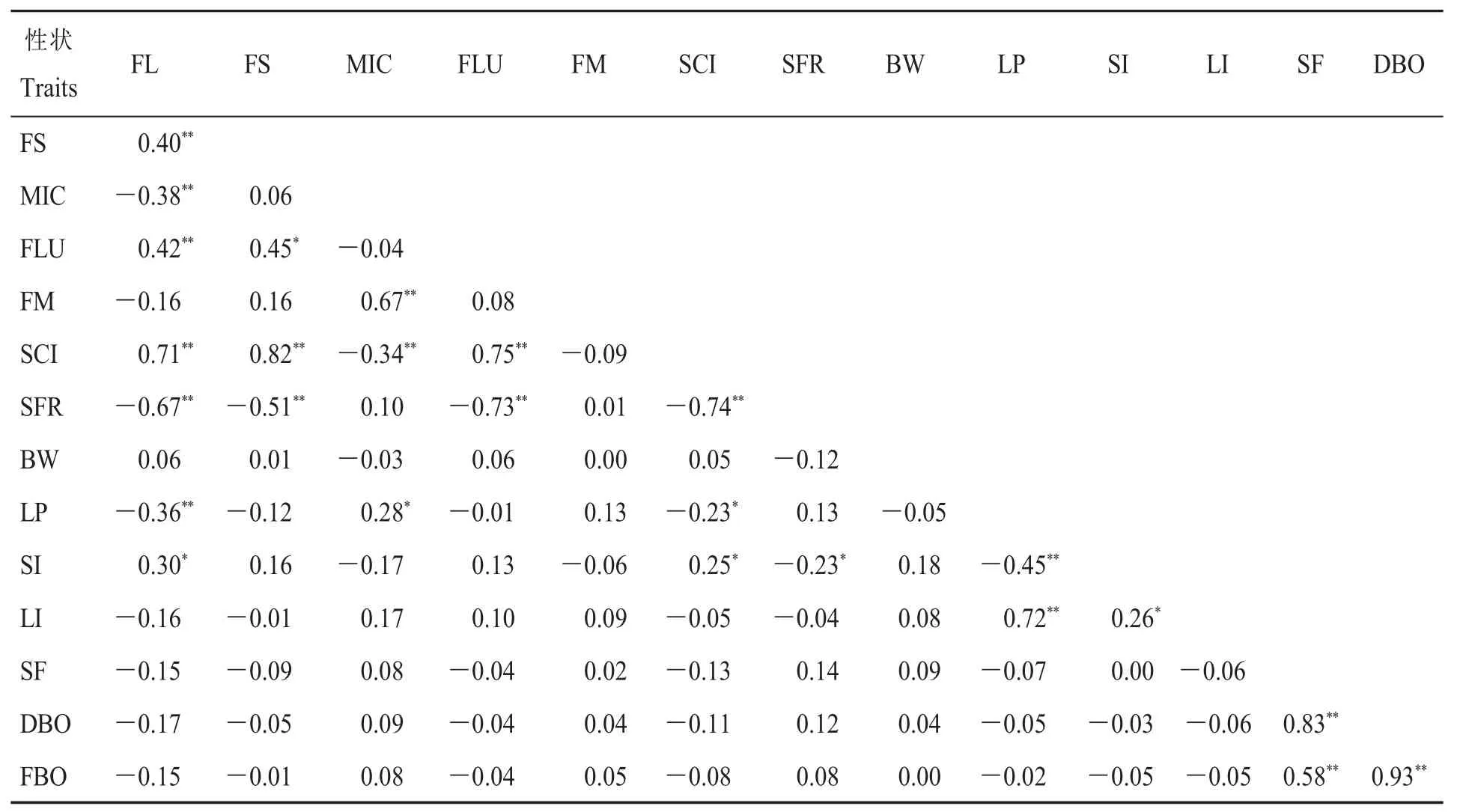
表4 4个群体性状间相关分析Table 4 Correlation analysis between traits in the four BC1F2populations
2.3.1纤维品质性状的QTL定位。利用4个BC1F2分离群体数据通过CIM法共定位到27个纤维品质相关性状QTLs(表5)。检测到5个与纤维长度相关的QTLs,1个位于2号染色体,1个位于16号染色体,3个位于18号染色体。单一QTL解释 10.8%~19.7%表型变异,LOD值为2.535~3.558。其中qFL-c2-1和qFL-c18-1的加性效应均为负值,分别为-2.208和-2.170,其他3个QTL的加性效应为正值。有4个与断裂比强度有关的QTLs,2个位于16号染色体,2个QTL位于18号染色体。单一QTL解释13.3%~30.9%表型变异,LOD值为3.322~7.468。qFS-c16-2和qFS-c18-2的加性效应为负值,而qFS-c16-1和qFS-c18-1的加性效应为正值。检测到2个与马克隆值有关的QTLs,分别位于2号和18号染色体。单一QTL解释14.2%~14.6%表型变异,LOD值为3.079~3.327。qMIC-c2和qMIC-c18的加性效应为负值,分别为-0.341和-0.397。有4个与纤维长度整齐度指数有关的QTLs,2、16号染色体各1个,18号染色体上2个。单一QTL解释10.7%~27.9%表型变异,LOD值为 2.985~4.350。其中qFLU-c16的加性效应为负值,其他3个QTLs的加性效应为正值。有3个与纤维成熟度有关的QTLs,均位于18号染色体。单一QTL解释14.0%~19.0%表型变异,LOD值为2.699~4.109。其中qFM-c18-1的加性效应为负值,其他2个QTLs的加性效应为正值。有4个与纺纱均匀性指数有关的QTLs,1个位于2号染色体,2个位于16号染色体,1个QTL位于18号染色体。单一QTL解释9.0%~24.4%表型变异,LOD值为 2.532~4.831。其中qSCI-c16-2、qSCI-c18-1的加性效应均为负值,其他2个QTL的加性效应为正值。检测到5个与短纤维率有关的QTLs,2个位于16号染色体,3个位于18号染色体。单一QTL解释 11.4%~18.1%表型变异,LOD值为2.705~3.779。其中qSFR-c18-1和qSFR-c18-3的加性效应均为负值,分别是-1.899和-1.946,其他3个QTLs的加性效应为正值。

表5 4个群体中纤维品质相关性状的QTL定位Table 5 QTL for fiber quality related traits in the four BC1F2populations
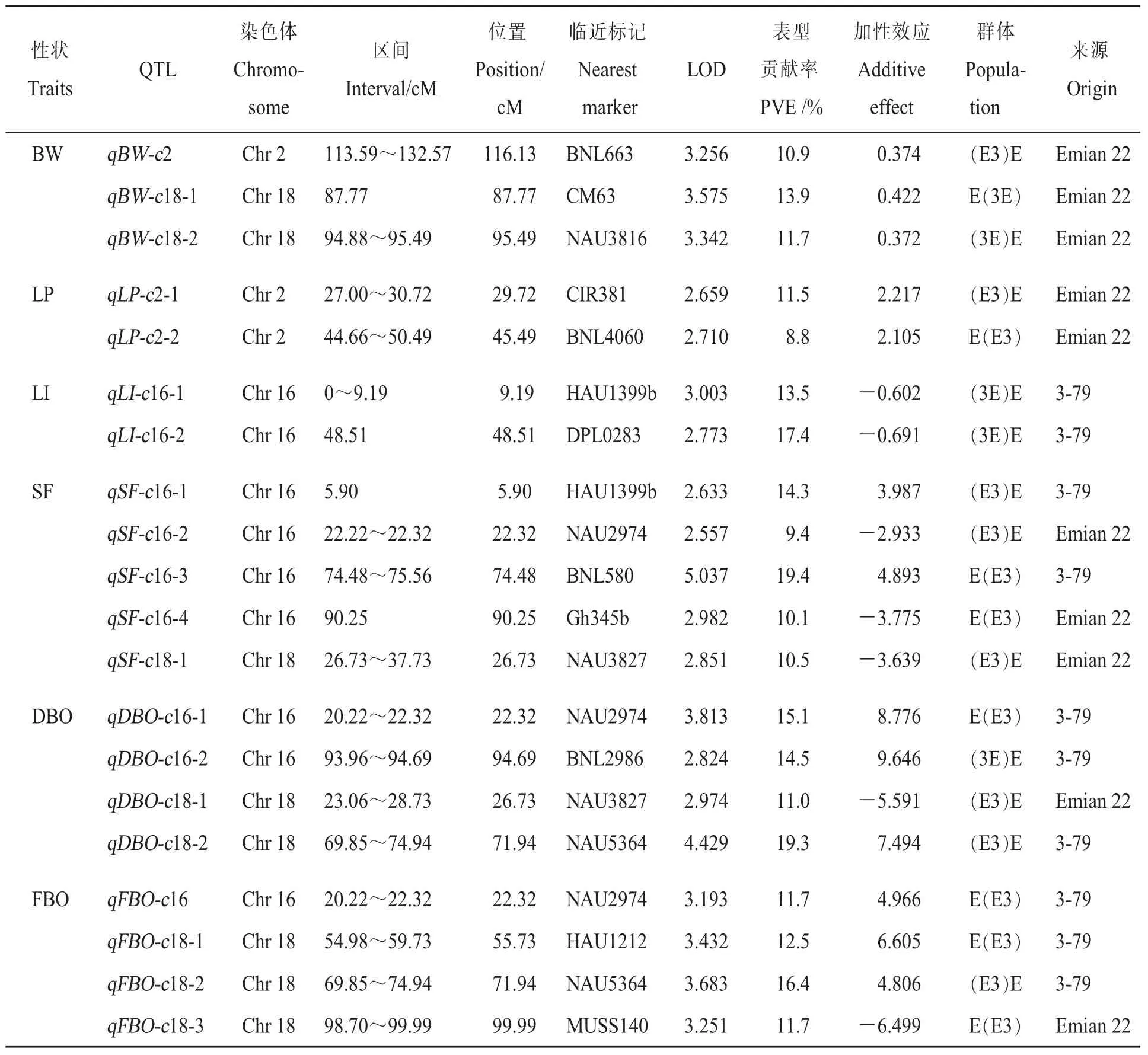
表6 4个群体产量及熟性相关性状QTL定位Table 6 QTL for yield and earliness traits in the four BC1F2populations

图1 4个群体棉花重要农艺性状在2号、16号和18号染色体上的QTL定位Fig.1 QTL mapping on chromosome 2(A),16(B)and 18(C)for cotton important traits in the four BC1F2populations
2.3.2产量性状的QTL定位。利用4个BC1F2分离群体产量性状数据,通过CIM法共定位7个与棉花产量相关性状QTLs(表6)。检测到3个与铃重相关的QTLs,1个位于2号染色体,2个位于18号染色体;单一QTL解释10.9%~13.9%表型变异,LOD 值为 3.256~3.575。qBW-c2、qBW-c18-1和qBW-c18-2的加性效应均为正值,分别是0.374、0.422和0.372。检测到2个与衣分相关的QTLs,均位于2号染色体。单一QTL解释8.8%~11.5%表型变异,LOD值为 2.659~2.710。qLP-c2-1和qLP-c2-2的加性效应均为正值,分别是2.217和2.105,有利等位基因和增效作用均来源于鄂棉22。2个与衣指有关的QTLs均位于16号染色体。单一QTL解释13.5%~17.4%表型变异 ,LOD 值 为 2.773~3.003。qLI-c16-1 和qLI-c16-2的加性效应均为负值,分别是-0.602和-0.691。
2.3.3熟性相关性状的QTL定位。利用4个BClF2分离群体熟性数据,通过CIM法共定位13个与棉花熟性相关性状QTLs(表6)。检测到5个与出苗至开花期有关的QTLs,4个位于16号染色体,1个位于18号染色体。单一QTL解释9.4%~19.4%表型变异,LOD值为2.557~5.037。qSF-c16-2、qSF-c16-4和qSF-c18-1的加性效应均为负值,分别为-2.933、-3.775、和-3.639,有利等位基因和增效作用均来源于鄂棉22;qSF-c16-1和qSF-c16-3的加性效应为正值,分别为3.987和4.893,有利等位基因和增效作用均来源于3-79。检测到4个与出苗至吐絮期有关的QTLs,16、18号染色体上各2个。单一QTL解释11.0%~19.3%表型变异,LOD值为 2.824~4.429。qDBO-c16-1、qDBO-c16-2 和qDBO-c18-2的加性效应均为正值,分别为8.776、9.646和7.494,增效作用均来源于3-79。而qDBO-c18-1的加性效应为负值,为-5.591,有利等位基因和增效作用均来源于鄂棉22。检测到4个与开花至吐絮期相关的QTLs,1个位于16号染色体,3个位于18号染色体。单一QTL解释11.7%~16.4%表型变异,LOD值为 3.193~3.683。qFBO-c16、qFBO-c18-1和qFBO-c18-2的加性效应均为正值,分别为 4.966、6.605和4.806,增效作用均来源于3-79。qFBO-c18-3的加性效应为负值(-6.499),有利等位基因和增效作用均来源于鄂棉22。
3 讨论
3.1 作图群体选择
随着分子标记技术的发展,定位重要性状的QTL在多种作物中进展快速。海岛棉和陆地棉都是世界范围重要的棉花栽培种,其中陆地棉贡献了90%以上的棉花产量,而海岛棉品质优良,在高档纺织品工业中有重要的应用。海岛棉和陆地棉遗传差异较大,分子标记多态性好,因此海陆种间群体广泛用于定位棉花重要性状相关QTL。
早在1998年Jiang等[16]利用陆地棉CAMD-E和海岛棉Sea Island Seaberry杂交产生的F2群体(含有271个单株),构建了1张含有261个限制性片段长度多态性 (Restriction fragment length polymorphism,RFLP)标记、27个连锁群的连锁图,这个连锁图全长为3 767 cM,标记间距为14.4 cM,还有16个标记没有被定位到连锁图上。对纤维长度、纤维强度等多个纤维品质性状进行QTL定位,共检测到14个相关的QTL。研究发现多数QTL定位在D基因组上,认为在棉花驯化和遗传改良过程中D基因组可能对纤维产量和品质的改良起到了巨大的作用。
随后棉花科研工作者利用不同的亲本构建各种群体进行棉花QTL定位,包括BCnFn、回交重组自交系(Backcross inbred lines,BIL)等[17-22]。Chee等[23-24]利用陆地棉Tamcot 2111和海岛棉Pima S6培育了高代回交群体,对24个BC3F1单株基因型分析结果表明,平均来说每个单株基因组中海岛棉等位位点约占总位点的7.3%,24个单株覆盖了海岛棉基因组的70%。由24个单株产生了24个BC3F2家系。利用这些家系进行了纤维品质性状的QTL定位,分别检测到22、8、6和2个与纤维长度、长度整齐度指数、短纤维率、断裂伸长率相关的QTLs。
本研究利用2个亲本鄂棉22和3-79构建了4个BC1群体。海岛棉3-79基因组已被测序[3],根据定位的QTL区间,很容易得到相应的基因组序列信息,因此利用3-79作为亲本能够加速相关QTL的定位和克隆。
根据交配方式可将这4个群体分为雄性分离群体和雌性分离群体,这种交配设计有助于检测母体效应和花粉或者胚囊产生的细胞质对QTL表达的影响。在本研究中仅qFLU-c18同时在 2个群体(3E)E和 E(3E)中被检测到,这个QTL临近的标记均为NAU3232,可能是3-79细胞质基因对分离配子产生了一定的影响所致。qFL-c18-2 和qFL-c18-3 分别在 (3E)E 和(E3)E中被检测到,这2个QTLs均分布于18号染色体且相距较近,可能是同1个位点,这2个QTLs仅在雌性分离群体中被检测到,说明它们在母体环境中得到了有效的表达。其他的纤维品质QTLs均仅在1个群体中被检测到。
3.2 群体间QTL比较分析
为剖析棉花产量、熟性和纤维品质等复杂数量性状的遗传基础,很多研究者进行了QTL定位研究。由于棉花熟性、产量和纤维品质等性状都是数量性状,受环境影响大,所以不同的作图亲本、群体类型和单株数量、标记类型和QTL定位方法等[25-33]会导致研究结果不一致,不同研究相互之间不能交叉验证。即使采用相同的亲本,也可能由于不同世代、不同群体类型导致QTL定位结果不一样,也可能存在QTL不能同时被检测到及QTL效应大小不一样的问题[34]。
为克服这些问题,比较图谱和元分析等综合分析方法被用来整合分析不同研究中检测到的QTL,为后续的QTLs图位克隆和分子辅助选择(Molecular marker-assisted selection,MAS) 提供有用的参考信息[34-37]。Said等[34,38-39]搜集了59篇文献中的1 075个陆地棉种内QTLs和30篇文献中1 059个海陆种间QTLs进行了综合的比较元QTL 分析(Comprehensive comparative meta QTL analysis)。表7分析了本研究定位的QTLs与Said等[38-39]综合分析QTL的染色体分布,结果表明在本研究中检测到的大部分QTLs在综合分析中也被检测到。如位于2号染色体的BW、FL、FLU、LP、MIC 等性状的 QTLs, 位于 16号的 FL、FS、FLU等性状的QTLs,位于18号染色体的BW、DBO、FL、FS、FLU、MIC 等性状相关的 QTLs。本研究也鉴定出了一些在综合图谱上没有报道的QTLs,如 16号染色体的qDBO-c16-1、qDBO-c16-2 和qLI-c16-1、qLI-c16-2,18号染色体的qSF-c18-1。这些新鉴定的QTLs可能为QTL比较分析提供新的参考信息。

表7 QTL比较结果Table 7 QTL comparison between our study and Meta QTL
4 结论
本研究在陆海种间群体中利用复合区间作图方法对4个BC1F2分离群体的产量、熟性相关及纤维相关性状进行QTL定位,共检测到47个QTLs,其中检测到27个纤维性状相关QTLs,单个QTL解释9.0%~30.9%的表型变异;检测到7个产量相关性状QTLs,单个QTL解释8.8%~17.4%的表型变异;检测到13个熟性相关QTLs,单个QTL解释9.4%~19.4%的表型变异。本研究鉴定出一些新的QTL,如16号染色体的qDBO-c16-1、qDBO-c16-2 和qLI-c16-1、qLI-c16-2,18号染色体的qSF-c18-1。
