基于重测序开发的InDel标记定位陆地棉矮化突变体
2018-12-12季高翔何守朴潘兆娥龚文芳贾银华王立如王朋朋耿晓丽杜雄明
季高翔,何守朴,潘兆娥,龚文芳,贾银华,王立如,王朋朋,耿晓丽,杜雄明
(中国农业科学院棉花研究所/棉花生物学国家重点实验室,河南安阳455000)
植物矮化是重要的农艺性状,矮化品种的选育可有效改善种植密度、提高抗倒伏能力、便于机械化管理,对于减少劳动强度、提高肥料利用率和提高产量具有重要意义。棉花具有无限生长的习性,培育矮化品种对于棉花尤为重要。目前棉花生产仍然需要人工打顶,从而控制棉花的株高,大面积植棉区如新疆主要利用喷施缩节胺来代替人工打顶,但是缩节胺的使用严重污染自然环境。因此,培育矮化品种不仅可以给生产带来很多的便利,而且可以减少对环境的污染。
分子标记是构建遗传图谱实现基因定位和分子标记辅助选择育种的强有力工具,20世纪70年代Grodzicker等[1]首先创立了基于酶切技术的RFLP(限制性片段长度多态性,Restriction fragment length polymorphism),到后来基于PCR(聚合酶链式反应,Polymerase chain reaction)技术的 SSR[2](简单重复序列,Simple sequence repeat)、AFLP[3](扩增长度多态性,Amplified fragment length polymorphism),再到后来的InDel(插入 /缺失,Insertion-deletion)、SNP[4](单核苷酸多态性,Single nucleotide polymorphism),分子标记技术已经发展到了第三代。随着测序技术的迅速发展,利用重测序的技术开发分子标记在水稻[5]、黄瓜[6]、棉花[7]等上均有应用。
本课题组在棉花种间杂交后代中发现1株自然矮化突变体AS98。张超等[8]研究表明极端矮化材料AS98的矮化性状是受1对不完全显性基因控制的质量性状,随后又利用AFLP和SSR标记对矮化基因进行定位分析,结果找到了与目标基因距离较近的2个分子标记,1个SSR标记(距离目标基因为4.9 cM)和1个AFLP标记(距离目标基因为9.7 cM)。随着棉花基因组测序的完成[9-10],基因组的信息为前人初定位的结果提供了验证依据,再加上分子标记技术的发展,也为实现基因定位提供了很大的帮助。本研究利用基因组重测序的数据开发覆盖全基因组的InDel标记,并利用新开发的InDel标记实现矮化基因的重新定位。
1 材料与方法
1.1 试验材料与群体构建
本试验所用亲本AS98和LHF10来自中国农业科学院棉花研究所的种质资源,其中AS98由陆地棉自然突变体多代自交纯化而来,性状已纯合稳定。LHF10为陆地棉正常高度材料。2016年4月在中国农业科学院棉花研究所实验基地(河南省安阳县)种植AS98半矮株和LHF10,利用AS98分离出来的半矮株作为父本与LHF10(母本)进行杂交,收获杂交种子。2017年4月在安阳种植AS98半矮株以及收获的杂交种子,分别作为2个群体,一个(群体1)用作定位,另一个(群体2)用作分离比的调查(表1)。
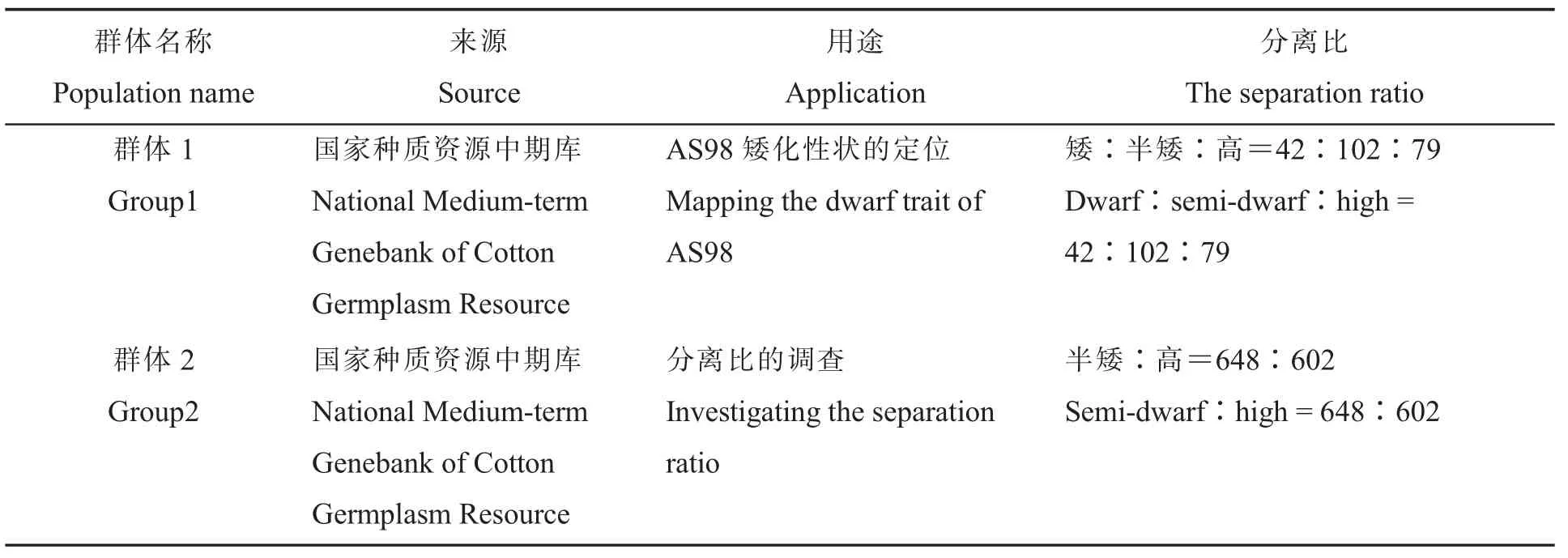
表1 2个分离群体信息Table 1 The information of the two segregation populations
1.2 试验方法
1.2.1性状调查与采样。2017年7月,田间植株已基本实现高矮分化,判断标准为:与亲本AS98性状相同的,即出土10~15 cm,无侧枝,且节间极度缩短的为极矮株;与LHF10性状相同的,即出土50 cm以上,节间正常,株型不紧凑为高株;介于2个亲本之间的,即出土15~50 cm,节间缩短,株型紧凑为半矮(图1)。其中群体1,亲本各选10株挂牌并对F2的每个单株挂牌编号,每个挂牌的植株各取2~3片大小适中、无病虫害的嫩叶作为提取DNA的样品材料,并记录每一株的高矮类型;群体2只调查植株性状。并用SAS软件对分离群体的高矮株数进行卡方检验。

图1 2个亲本及其杂交种的形态学特征Fig.1 The morphology of two parents and their hybrid
1.2.2基因组DNA的提取。采用宋国立等[11]改良的CTAB法提取DNA,并用1%(质量分数)琼脂糖对核酸质量进行检测。
1.2.3InDel标记的开发及多态性筛选。分别取极端矮化株AS98和正常高株LHF10各3株的DNA混样测序,AS98和LHF10平均测序深度分别为11.85×、13.32×,覆盖度均在97%以上,测序质量正常。基于测序结果,进行InDel标记的开发,采用GATK3.3软件的Unified Genotyper模块进行InDel的检测,使用Variant Filtration进行过滤,过滤参数为filterExpression"QD<4.0||FS>200.0"。随后对2个亲本中的InDel进行基因分型,去掉在2个亲本中的同一个位置上发生相同插入或者缺失的InDel位点作为差异InDel。根据亲本的InDel信息,提取InDel上下游各100 bp(base pairs)的序列,并根据上下游序列设计引物,引物的TM值在50~60℃,长度为18~22 bp,产物为80~288 bp。根据张超等[8]初定位的结果,找出与目标基因在一个连锁群上的所有SSR标记,分别用AS98和LHF10进行多态性检测,随后挑选该区段内新开发的所有InDel标记,同样用2个亲本对多态性进行筛选,筛选出多态性引物之后对F2群体的223单株进行基因分型,结合其表型,实现矮化基因的定位。
1.2.4PCR反应与凝胶电泳。SSR引物序列来自CottonDB(http://www.cottondb.org/)数据库,InDel引物序列由自己开发,均由上海生工生物工程股份有限公司合成。反应体系为10 μL,包括10×buffer(含 20 mol·L-1Mg2+)1.0 μL, dNTP(10 mol·L-1)0.5 μL,Taq DNA 聚合酶(2.5 U·μL-1)0.1 μL,50 mg·L-1的模板 DNA 1.5 μL, 前后引物(5 μmol·L-1)各 0.75 μL,双蒸水补至 10 μL。反应程序为:94℃预变性3 min;然后94℃变性30 s,55℃退火30 s,72℃延伸1 min,30个循环;最后72℃再延伸5 min;4℃保存。随后用8%(质量分数)的聚丙烯酰胺凝胶电泳,银染后拍照并记录带型。
1.2.5遗传连锁图谱的构建。对群体带型进行统计,与AS98带型相同的记为1,与LHF10相同的记为2,同时具有2个亲本带型的记为3,缺失记为“-”,随后利用Joinmap4.0构建遗传连锁图,其中对数优势比 (Logarithm of the old some,LOD) 设为3.0,选用Kosambi函数进行遗传距离(cM)的计算。
2 结果与分析
2.1 性状调查与分离比的卡方检验
根据方法中所述的高矮判断标准,统计得到群体1的极矮株、半矮株、高株,3种表型的分离比为42∶102∶79,群体2有高株和半矮株2种表型,它们的比例为648∶602,经卡方适合性检验,群体 1 不符合1∶2∶1(P<0.05)的分离比,但矮株和半矮株之和与高株呈2∶1(P>0.05)的分离比;群体2符合1∶1(P>0.05)的分离比。这说明矮秆性状为显性,高株为隐性,但后代的纯合显性棉株可能因长势过弱而致死,其机理还有待进一步研究。
2.2 InDel标记的筛选
对张超等[8]初定位与目标性状连锁的SSR标记在AS98(亲本矮株)以及LHF10(亲本高株)中进行多态性筛选,发现其中一个SSR引物NAU5204在亲本间表现多态性,利用CottonFGD(https://cottonfgd.org/)查阅该标记的信息,结果显示该标记在D12染色体上(起始位置:55 255 848 bp;结束位置:55 256 113 bp;覆盖度:100%; 同一性100%)。因此,本试验以NAU5204标记开始的物理位置(D12,55 255 848 bp)为坐标在新开发的InDel数据库中距离NAU5204前后各1 Mb(54~56 Mb之间)挑选114对InDel标记,经亲本筛选共有20对表现多态性,多态性频率为17.5%。
2.3 InDel标记分析与遗传连锁图谱的构建
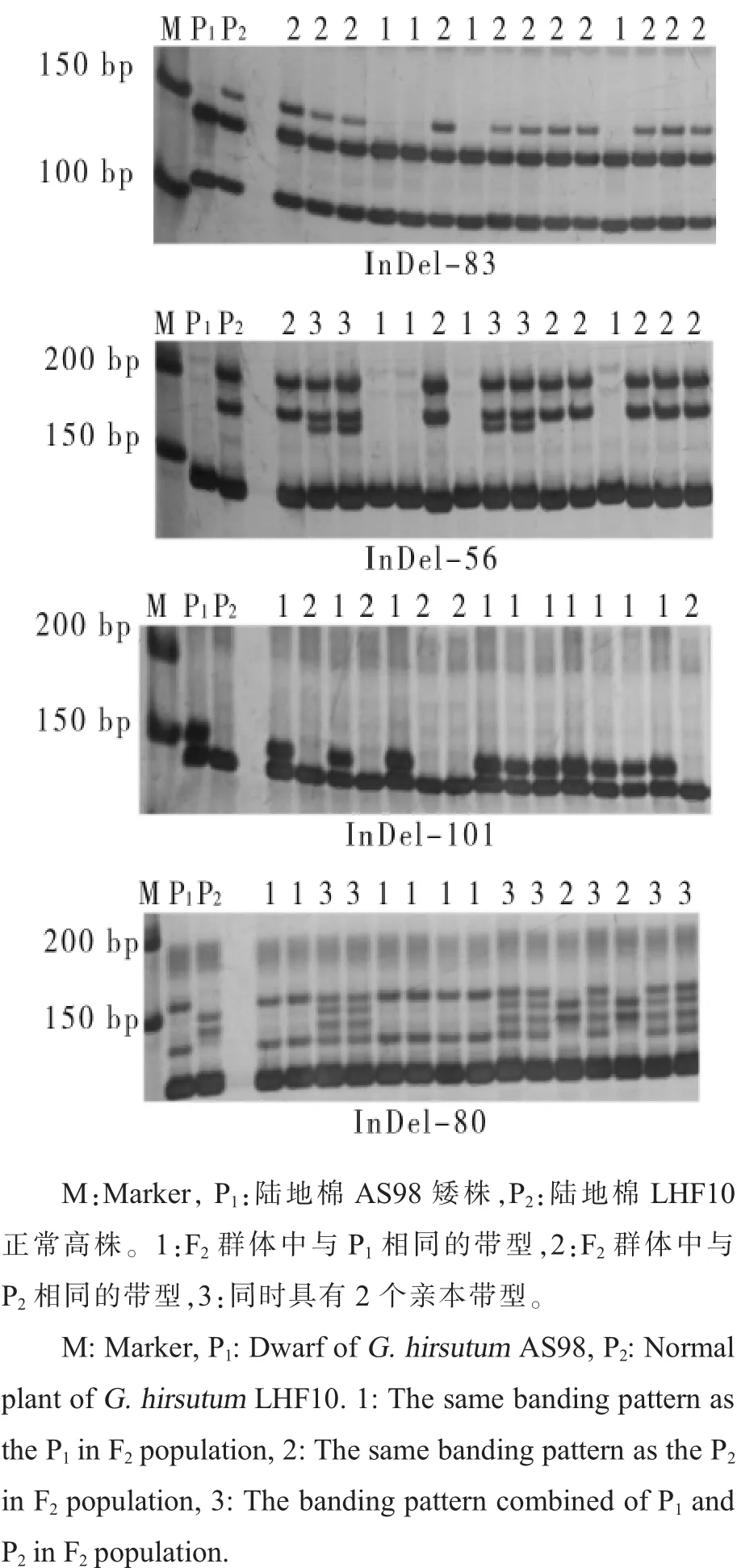
图2 F2群体InDel和SSR标记部分检测结果Fig.2 Partial detection results of InDel and SSR markers in F2
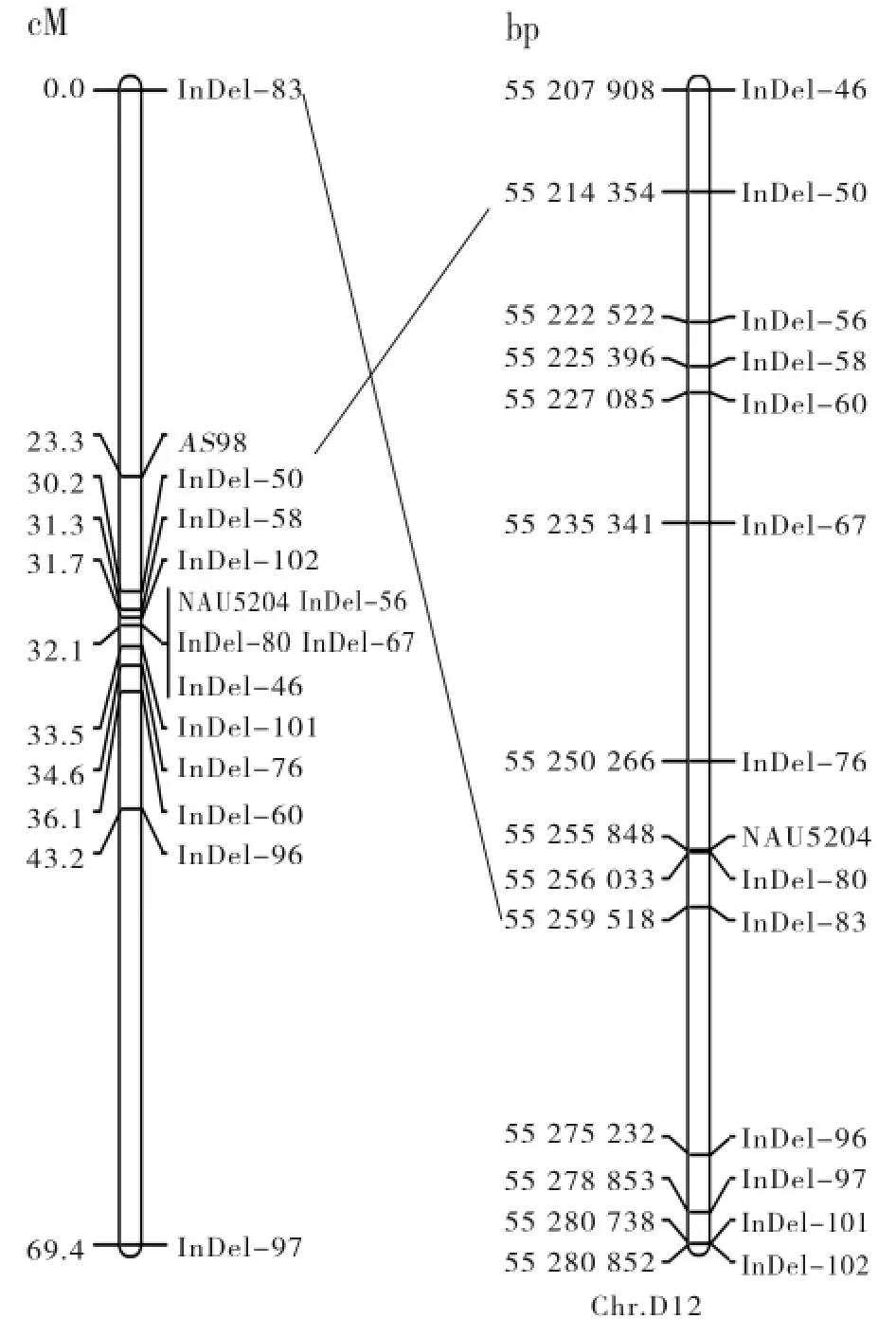
图3 AS98遗传连锁图以及物理图谱Fig.3 Linkage map and physical map ofAS98
用上述具有多态性的20对引物,加上NAU5204,总共21对引物,对 F2中223个单株进行检测(图2)。利用Joinmap4.0进行遗传连锁图谱构建,在LOD≥3的情况下,共有14个标记在一个连锁群上(图3)。与目标基因AS98连锁的标记群体分离情况(表2)显示,除了InDel-60、InDel-83、InDel-97、InDel-101偏分离比较严重外,其他标记在群体中均正常分离。连锁图谱显示矮化性状标记位于InDel-50和InDel-83之间,其中距离InDel-50的遗传距离为6.9 cM,距离InDel-83的遗传距离为23.3 cM。与目标性状连锁的InDel以及SSR标记相关信息见表3。
3 讨论
棉花的矮化突变体资源有限,自1918年Harland[12]在海岛棉中发现1个“矮皱突变体”(Crinkled dwarf)以来,只有10多个棉花矮化突变体被发现及鉴定。Hutchinson等[13]在陆地棉中也发现了“矮皱突变体”,McMichael等[14]在陆地棉Acala中发现1个 “矮皱与红叶”突变体。Quisenberry等[15]鉴定了1个具有数量性状遗传特点,且植株矮小,早熟,纤维短、细、强度差的突变体Lubbock dwarf。而Abzalov等[16]发现了纯合显性基因型致死的矮秆系L-69。戴日春等[17]在陆地棉品种协作二号中,发现1株丛生矮化突变体。何鉴星等[18]在陆地棉科遗2号(Gossypium hirsutumL.)×中棉(Gossypium arboreumL.)的种间杂交后代中分离出1株矮秆小叶突变体,育成了“矮早1号”。陈旭升等[19]在陆地棉C119-2分离群体中发现株高只有10.5 cm的突变体 “超矮1号”。刘爱玉等[20]报道“陆矮1号”的矮化特性主要为细胞核数量性状遗传,可能还存在部分细胞质效应。虽然棉花矮化突变体形态遗传研究已有初步进展,但对矮秆基因定位的分子研究少见报道。景超等[21]对1个陆地棉隐性超矮秆突变体基因进行了初步定位。Liu等[22]对陆地棉组合CRI12×J8891的重组自交系株高QTL分析表明,其株高受主基因和微效基因共同作用。Yang等[23]在棉花中鉴定出来1个油菜素内酯代谢途径上的基因PAG1,可以调节纤维发育。本研究对棉花矮化突变体的基因定位研究有助于进一步的精细定位及分子标记辅助选择育种。
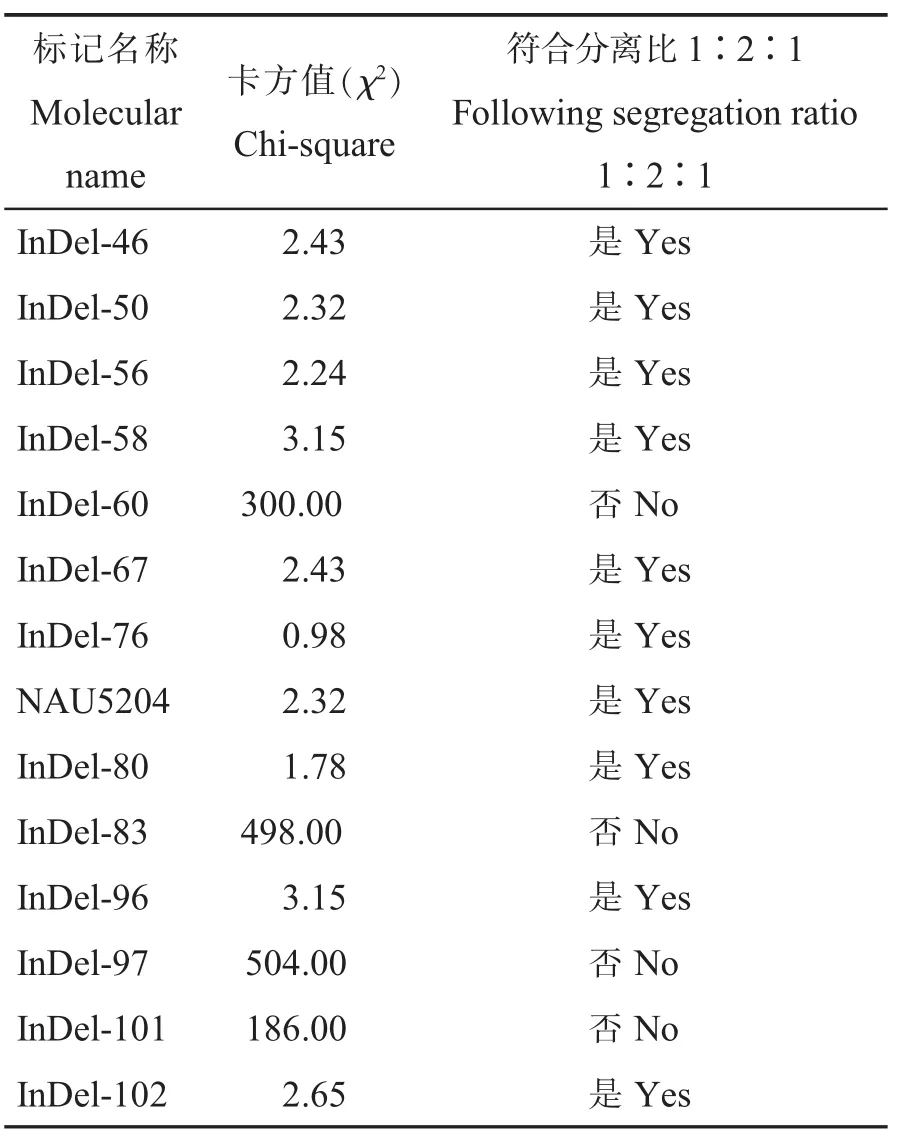
表2 14对标记在F2群体(223株)中的分离情况Table 2 The separation of 14 pairs of markers in the F2population(containing 223 individuals)
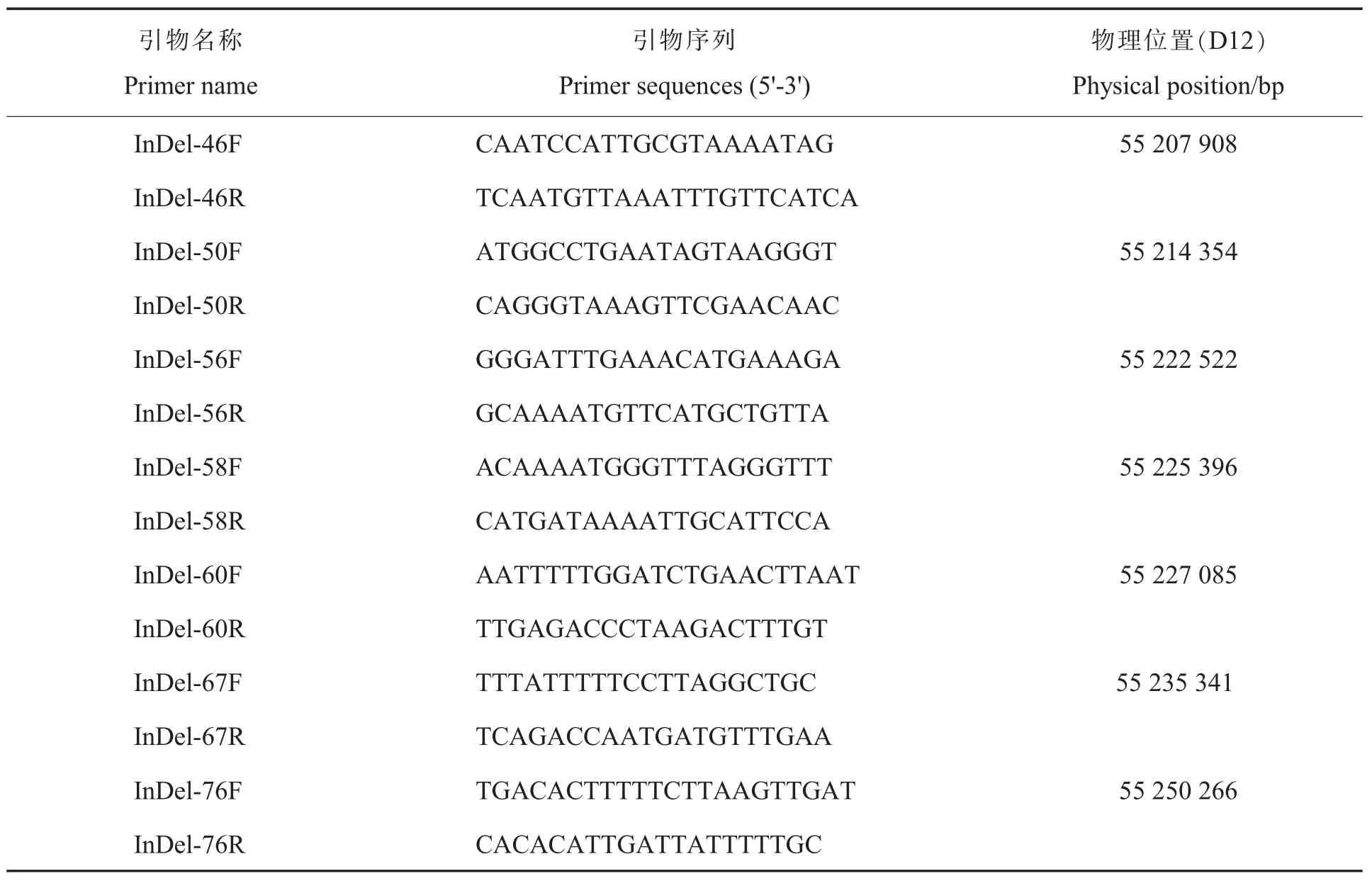
表3 与AS98连锁的InDel以及SSR引物序列Table 3 InDel and SSR primer sequences of linkage mapping withAS98

表3 (续)Table 3 (Continued)
InDel标记具有准确性高、多态性丰富、分布广泛等特点,而且利用InDel标记可基于PCR扩增技术对群体进行分型。相对于SNP复杂的分型系统,InDel标记就显得简单快捷,利用普通的聚丙烯酰胺凝胶电泳就可以实现。除此之外,InDel标记的扩增产物带型清晰,易于记录,而且对DNA的质量要求不高,样品量要求也比较小[24],而有些SSR标记的扩增结果会受到DNA质量的影响,从而影响基因分型结果[25]。InDel标记也有其局限性,对于未知基因组信息的物种来说,InDel标记的开发只能根据已知基因的序列,这样开发的InDel数量有限,且覆盖面窄。但是随着很多物种基因组计划的完成,结合基因组信息大量开发InDel标记得以实现。
近年来棉花基因组测序[9-10]已完成,基于基因组信息的测序技术也得到了迅猛发展,利用测序技术开发分子标记在黄瓜[6]和棉花[7]上都有实现。本试验利用重测序技术在基因组水平上检测极端矮化材料AS98和正常高株LHF10之间的序列差异,并在2个材料插入缺失位点开发设计InDel标记。为了进一步定位控制棉花矮化突变体AS98的基因,本试验在前人[8]的基础上利用新开发的部分标记对AS98中控制矮化性状的基因进行了重新定位。相较于前人的研究,重新定位的结果把该基因定位到具体的染色体上,即D12染色体上,位于InDel50和InDel83标记之间,这2个标记的物理间距仅有45 kb,但是遗传距离却有30.2 cM,而且遗传图谱与根据基因组信息[10]构建的物理图共线性不理想。我们猜想有可能是AS98极端矮化材料特殊,在该区段内有染色体结构上的变异,或者会有其他不明的原因,有待进一步研究。
在我们选择的区间内筛选的InDel标记,多态性频率仅有17.5%,这说明本试验开发的InDel标记存在一定的假阳性。究其原因可能是研究对象是多倍体的陆地棉,在检测插入和缺失突变的时候会受到亚组之间差异的影响[26]。总之,利用重测序开发覆盖全基因组的InDel标记,并利用这些标记实现基因的定位是可以实现的。本试验最终利用新开发的InDel标记对AS98矮化性状基因实现了重新定位,重新定位的结果可以为进一步的精细定位奠定基础。
