葡萄KEA家族基因的克隆、鉴定及表达分析
2018-12-11王壮伟王庆莲夏瑾王西成宋志忠吴伟民
王壮伟,王庆莲,夏瑾,王西成,宋志忠,2,吴伟民
葡萄KEA家族基因的克隆、鉴定及表达分析
王壮伟1,王庆莲1,夏瑾1,王西成1,宋志忠1,2,吴伟民1
(1江苏省农业科学院果树研究所/江苏省高效园艺作物遗传改良实验室,南京 210014;2鲁东大学农学院,山东烟台 264025)
【目的】从葡萄中克隆并鉴定KEA家族基因,在转录水平探索其组织特异性表达特征及对缺钾、脱落酸(ABA)、氯化钠(NaCl)与山梨糖醇(sorbitol)等胁迫的响应情况,明确主效基因。【方法】通过同源克隆法,在葡萄基因组中筛选并鉴定KEA家族基因;利用MEGA 7.0软件中的邻接法建立9种不同植物(葡萄、拟南芥、水稻、玉米、高粱、短柄草、白杨、梨和苹果)KEA家族成员的系统进化树;借助多种生物信息学软件分析葡萄KEA家族基因及其编码蛋白的详细特征;检索EST数据库,分析葡萄KEA家族基因的表达谱;利用实时荧光定量PCR分析KEA家族基因在葡萄不同组织部位的表达模式及对缺钾、ABA、NaCl与sorbitol等胁迫的响应情况,明确主效基因。【结果】在葡萄基因组中检索并克隆获得4个KEA家族基因,命名为,均含有典型的K/H交换结构域(K/H exchanger domain)和TrkA-N domain功能结构域,属于典型的植物K+/H+逆向转运体;9种不同植物的KEA家族蛋白在氨基酸水平具有33.10%的一致性,并可分为2个亚族(GroupⅠ和GroupⅡ),其中,葡萄VvKEA1和VvKEA2属于GroupⅠ,均含有7个Motif基序,而VvKEA3和VvKEA4属于GroupⅡ,只含有4个Motif基序;系统进化树表明葡萄VvKEA1、VvKEA2和VvKEA4成员分别与拟南芥AtKEA2、AtKEA3和AtKEA5紧密聚在一起,而葡萄VvKEA3则与梨PbrKEA5和苹果MdoKEA7紧密聚在一起,水稻、玉米、高粱和短柄草4种禾本科植物KEA家族成员更倾向于聚在一起,而木本植物白杨、苹果、梨KEA家族成员紧密聚在一起;葡萄KEA家族蛋白拥有相似的三级结构,主要定位于细胞质膜,均含有12—13个跨膜区,均为稳定蛋白,等电点均小于7.00,且只有VvKEA3具有信号肽;在葡萄启动子区域鉴定到至少15种的顺式作用元件,主要包括胁迫响应、营养和发育、激素响应、昼夜规律等不同生命活动相关的调控元件;EST表达谱结果表明葡萄KEA家族基因在多种组织或器官中均有表达,在果实中的表达水平最高,其次是叶、种子、根和雌蕊;实时荧光定量PCR分析表明在8年生‘白罗莎里奥’葡萄树不同组织中的整体表达水平最高,在幼果中的表达量最为突出,而其他3个的整体表达水平相对较低,且较为接近;幼苗中,在转录水平对NaCl处理没有响应,但对ABA处理最为敏感,的表达量在检测幼苗的地上部和根部均被显著诱导,在检测幼苗的地上部和根部及在地上部的表达量受缺钾处理诱导而显著增强,在检测幼苗的地上部和根部及在根部的表达量受sorbitol渗透处理诱导而显著上升。【结论】从葡萄中克隆并鉴定了4个KEA家族基因,主要在葡萄果实、叶片和种子中表达,其成员与拟南芥KEA成员在遗传距离上较近;在成年葡萄树中的整体表达水平最为丰富(幼果中最高),幼苗中受缺钾、ABA和sorbitol渗透胁迫的调控;VvKEA3是葡萄果实中重要的K+/H+逆向转运体。
葡萄;K+/H+逆向转运体;生物信息学;胁迫处理
0 引言
【研究意义】钾素营养与果树生长、花开放及果实发育密切相关,但其分子基础研究报道鲜少。钾离子(K+)是植物细胞中含量最为丰富的阳离子之一,维持细胞渗透调节平衡,控制细胞的基础膜电位水平,在气孔运动、光合作用、蒸腾作用、信号传导和胁迫抗逆等生命过程中均起着至关重要的作用[1-2]。由于K+控制着细胞的基础膜电位水平,然而,功能活跃细胞的真实膜电位水平往往偏离K+的扩散平衡电位。另一方面,离子或分子跨膜运输过程的激活,则依赖于这一基本驱动力并大多同时受细胞内质子(H+)浓度的调控。细胞质膜质子泵和液泡膜质子泵控制着细胞内质子H+浓度的电化学势,是造成细胞膜电位偏离的重要原因[3-6]。由此可见,K+和H+浓度的动态平衡对于细胞和植物的生命过程具有至关重要的作用。【前人研究进展】阳离子-质子逆向转运体CPAs(cation proton antiporters)广泛存在于植物、动物、真菌和细菌中,主要定位于细胞质膜、液泡膜和植物中的细胞器膜,可将细胞中的Na+、K+、Li+等阳离子排出,并引起细胞内H+的内流和积累[7-8],维持K+和H+浓度的动态平衡。植物中,CPAs可介导细胞中离子和质子的动态平衡,在植物的离子平衡、生长发育和信号转导中起重要作用[7-9]。植物CPAs分为2个亚族:CPA1和CPA2,其中,CPA1包括NHX(Na+/H+exchanger),CPA2包括KEA(K+efflux antiporter,K+/H+逆向转运体)和CHX(Cation/H+exchanger)[7-9]。特别地,植物CPAs中有关KEA亚族的研究最为稀少,主要体现在模式作物拟南芥中。2001年,Maser等[10]最早提出拟南芥中有6个AtKEA转运体基因,但其功能依然未知。最近研究表明,拟南芥AtKEA转运体定位于维管组织、保卫细胞和花萼等不同组织部位[11],在植物体内K+动态平衡和渗透调节中起主要作用[12-13];此外,有报道表明拟南芥AtKEA1-3转运体在叶绿体渗透调节、光合作用及pH调控等方面起关键作用[12-15]。然而,果树中有关K+/H+动态平衡问题的研究报道鲜少,仅见于蔷薇科梨基因组中KEA家族基因的鉴定及生物信息学分析[16],具体功能依然未知。【本研究切入点】近30年来,关于植物K+吸收转运以及质子泵的研究较为详尽,然而,对于同时转运K+和H+的K+/H+逆向转运体的研究鲜有报道。果树作物中,有关K+/H+动态平衡及KEA家族基因的功能依然未知。【拟解决的关键问题】本研究以‘白罗莎里奥’葡萄为材料,克隆并鉴定了4个葡萄KEA家族基因及其编码蛋白的特征,明确该家族基因在葡萄不同组织中的表达特征及对缺钾、ABA、NaCl和sorbitol等胁迫处理的响应,为研究果树K+/H+平衡与钾素动态营养机制提供了基因资源和理论基础。
1 材料与方法
1.1 试验时间、地点
试验于2017年4—12月在江苏省农业科学院/江苏省高效园艺作物遗传改良实验室完成。
1.2 试验材料与处理
供试材料为江苏省农业科学院溧水植物科学基地的8年生‘白罗莎里奥’葡萄(),树体健壮,南北行向,株行距为1 m×3 m,H型平棚架式,联栋大棚避雨设施栽培,常规田间管理。分别于特定日期采集8年生‘白罗莎里奥’的花蕾(4月30日,花序中部的花蕾)、花(5月3日,花序中部,盛开期)、新生叶片(5月24日,自顶端数起的第3片一年生新叶)、新生根(5月24日,近地表土层中侧根顶端的新生细根)、幼果(5月24日,幼果迅速膨大期)和熟果(9月7日,浆果成熟期)等组织材料,随后立即液氮冷冻并保存于-80℃冰箱中备用。胁迫试验供试材料为‘白罗莎里奥’试管幼苗,来源于江苏省农业科学院果树研究所浆果研究室。2016年8月,将生长情况一致的试管幼苗(长约12 cm)转接于GS液体培养基中,于人工气候箱(条件设置为25℃,200 μmol·m-2·s-1光照16 h;20℃,暗培养8 h)中预培养3 d,然后分别进行缺钾(用NaNO3和NaH2PO3分别代替GS配方中的KNO3和KH2PO4)、脱落酸(ABA,200 μmol·L-1)、氯化钠(NaCl,160 mmol·L-1)、山梨糖醇(sorbitol,320 mmol·L-1)等处理,每个处理6株幼苗。胁迫处理48 h后,分别采集不同处理后的幼苗地上部和根部材料,立即液氮冷冻并保存于-80℃冰箱中备用。
1.3 葡萄KEA基因克隆
以拟南芥TAIR数据库(http://www.arabidopsis. org/browse/genefamily/index.jsp)的6个KEA家族基因的氨基酸序列为参考序列,在Phytozome grape genome database (http://www.phytozome.net)中检索葡萄基因组中可能的KEA家族基因。检索结果在Pfam(http://pfam.xfam.org/search)在线服务器预测功能结构域。根据Phytozome获得的葡萄的CDS序列(coding sequence),分别设计引物,利用Prime STARTM HS DNA聚合酶(TaKaRa,大连)进行PCR扩增,测序验证后,提交NCBI GenBank获得登录号。
1.4 葡萄KEA家族基因的生物信息学分析
在Phytozome葡萄基因组数据库中获得KEA家族各基因的CDS编码区序列及基因组DNA序列,然后通过Gene Structure Display(http://gsds.cbi.pku.edu. cn/index.php)在线服务器进行基因结构分析;利用在线软件TMpredict(http://ch.embnet.org/software/ TMPRED_form.html)分析葡萄KEA家族蛋白的跨膜结构域;使用在线服务器MEME(v4.8.1)(http://meme. nbcr.net/meme/cgi-bin/meme.cgi)预测葡萄KEA家族蛋白的保守结构域;使用在线工具ProtParam(http:// expasy.org/tools/protparam.html)评估KEA蛋白成员的理论等电点、分子量、稳定性等理化性质;利用在线软件SignalP4.0(http://www.cbs.dtu.dk/services/ SignalP-4.0/)预测KEA家族基因的信号肽情况;利用PSORT在线服务器(http://psort.hgc.jp/form.html)预测葡萄KEA家族基因的亚细胞定位;利用Phyre2在线服务器(http://www.sbg.bio.ic.ac.uk/phyre2/html/page. cgi?id=index)分析葡萄KEA家族蛋白的三级结构;利用ClustalX 2.0软件对葡萄KEA转运体与拟南芥(6个)、水稻(6个)、玉米(8个)、高粱(7个)、短柄草(5个)、杨树(4个)、梨(12个)、苹果(7个)等[16-17]已知物种的同源KEA转运体进行氨基酸比对分析,用分子进化遗传分析软件MEGA 7.0中的邻接法(Neighbor-joining)构建系统进化树;利用NCBI的dbEST在线数据库(http://www.ncbi nlm. nih.gov/dbEST/),通过Blastn比对,检索葡萄KEA家族基因全长CDS序列和EST数据,选择匹配率大于95%、长度大于160 bp,并且E≤10-10的结果作为对应的EST序列,按照不同组织或器官来源进行分类,从而获得葡萄KEA家族基因的表达谱信息;为分析葡萄KEA家族基因的启动子区域,在Phytozome葡萄基因组数据库检索目的基因CDS区域起始密码子ATG的上游1.5 kb左右的片段为启动子区域,并通过PLACE(http://www.dna.affrc.go.jp/ PLACE/)在线软件分析启动子序列中含有的-顺式作用元件。
1.5 总RNA提取与实时荧光定量PCR分析
分别采集实生幼苗和8年生葡萄树体不同部位的组织材料,液氮冷冻后-80℃冰箱保存,通过MiniBEST Plant RNA Extraction Kit(TaKaRa,大连)提取样品的总RNA,并利用PrimeScriptTM RT reagent Kit 反转录试剂盒(TaKaRa,大连)合成第一链cDNA作为模板,用于实时荧光定量PCR。利用NCBI/Primer- BLAST在线服务器,设计葡萄的特异性表达引物(表1),以葡萄Ubiquitin(GenBank No. MH114011),通过ABI 7500实时荧光定量PCR仪检测在葡萄树不同组织部位的表达特征。荧光染料使用SYBR Green(TaKaRa,大连),反应体系参照商品说明书的描述,反应程序为95℃30 s;95℃5 s,60℃34 s(40个循环);72℃10 s。每个样品进行3次重复,不同样品在实时荧光定量PCR仪获得相应的Ct值,经内参基因均一化处理后,采用2-ΔΔCt法计算基因的相对表达量[18]。

表1 实时荧光定量PCR所用特异性引物
1.6 数据显著性分析
所有数据通过SPSS 13.0(SPSS Chicago,美国)软件进行显著性分析,即葡萄幼苗在处理条件与对照条件下2个独立样品间进行-检验(检验水平0.01<*<0.05, **<0.01)。
2 结果
2.1 葡萄KEA家族基因的克隆
以6个拟南芥KEA家族基因的氨基酸序列为参考序列,在Phytozome grape genome database(http:// www.phytozome.net)葡萄基因组数据库中检索到4个KEA家族基因,检索结果在Pfam在线服务器预测到K/H交换结构域(K/H exchanger domain,PF00999)和TrkA-N domain(PF02254)功能结构域,均属于典型的KEA家族蛋白。检索获得各基因的电子序列后,以CDS序列始密码子ATG和终止密码子TAG所在位置22 bp左右的序列作为上下游引物,以‘白罗莎里奥’葡萄幼苗叶片RNA反转录获得的cDNA为模板,PCR扩增各个候选基因的CDS序列,分别连接到pGEM-T载体,转化大肠杆菌感受态菌株DH5α,筛选阳性转化子送上海生工公司测序。经测序验证后,获得‘白罗莎里奥’葡萄的KEA家族各基因的CDS序列及相应翻译得到的氨基酸序列,分别命名为,提交NCBI GenBank获得登录号(表2)。
2.2 9种不同科属植物KEA家族成员的系统发育树
将葡萄(葡萄科)、拟南芥(十字花科)、水稻(禾本科)、玉米(禾本科)、高粱(禾本科)、短柄草(禾本科)、白杨(杨柳科)、梨(蔷薇科)和苹果(蔷薇科)9种不同科属的物种的KEA家族基因(表3),通过ClustalX 2.0进行氨基酸水平的多重序列比对。结果表明,供试物种的KEA家族基因之间具有较高的同源性,同源关系较近的两者之间的序列一致性均高于70%(数据未展示);9种植物56个KEA家族成员在氨基酸水平依然具有28.30%的一致性,在核苷酸水平具有33.10%的一致性(数据未展示);4个葡萄KEA家族成员在氨基酸水平具有42.20%的一致性(图1),在核苷酸水平具有57.03%的一致性(数据未展示)。

表2 葡萄KEA家族成员基本信息

图1 葡萄KEA氨基酸序列一致性分析
利用MEGA 7.0建立系统进化树,结果表明九种植物KEA家族基因在遗传进化关系上有差异,葡萄和拟南芥2种双子叶植物遗传距离上是较近的,葡萄VvKEA1、VvKEA2和VvKEA 4分别与拟南芥AtKEA2、AtKEA3和AtKEA 5紧密聚在一起,而葡萄VvKEA3则与梨PbrKEA5和苹果MdoKEA7紧密聚在一起(图2);水稻、玉米、高粱和短柄草4种禾本科植物KEA家族的相关成员,更倾向于聚在一起,而木本植物白杨、苹果、梨KEA家族更倾向于聚在一起,特别是同属蔷薇科的苹果和梨,遗传距离上更为相近(图2)。此外,9种植物KEA家族成员可分为2个亚族,GroupⅠ和Ⅱ,其中,葡萄VvKEA1和VvKEA2属于GroupⅠ,而VvKEA3和VvKEA4属于GroupⅡ(图2和表2)。
2.3 葡萄KEA基因定位、编码蛋白特征与Motif分析
葡萄KEA家族基因定位于不同染色体上(VvKEA2具体染色体未知),含有13—19个长度不一的内含子(图3和表2);其中,VvKEA1基因CDS编码区最长,其次是VvKEA3和VvKEA2,VvKEA4最短,其编码氨基酸数目和分子量与CDS长度成正比(表2);葡萄KEA家族蛋白的等电点均<7.00,表明其含有的酸性氨基酸较多;葡萄KEA家族蛋白均含有12—13个跨膜区,均为稳定蛋白,且只有VvKEA3具有信号肽,位于第1—26氨基酸区域(表2)。

表3 9种植物KEA家族蛋白信息
保守基序分析结果表明葡萄VvKEA3和VvKEA4蛋白均含有7个Motif基序,即Motif1—Motif7,而VvKEA1和VvKEA2均只含有4个Motif基序,即Motif3、Motif4、Motif6和Motif7,缺少Motif1、Motif2和Motif 5(图3和表2);其中,Motif2、Motif3和Motif5最长,均含有50个特征氨基酸序列,而Motif6最短,含有31个氨基酸序列(图4)。蛋白三级结构预测分析表明所有葡萄KEA家族蛋白拥有相似的三级结构(图5),暗示该家族基因可能拥有相近的功能。
2.4 葡萄KEA基因亚细胞定位预测及表达谱分析
亚细胞定位预测结果表明葡萄KEA家族基因定位趋势一致,均主要定位于细胞质膜,其次是内质网膜(除外)和液泡膜(除外)(表4)。EST表达谱分析结果表明,葡萄KEA家族基因可能在果实中的表达水平最高,其次是叶、种子、根和雌蕊,在花、果皮、花粉和木质部中亦有表达(图6)。
2.5 葡萄KEA基因启动子顺式作用元件预测
启动子区域-顺式作用元件预测结果表明,葡萄KEA家族基因启动子区域鉴定到至少15种的顺式作用元件,包括胁迫响应、营养和发育、激素响应、昼夜规律等不同生命活动相关的调控元件,且在不同中数量差异显著(表5)。其中,5种(光感应、胚乳特异表达、水杨酸响应、热胁迫响应、防御和胁迫响应)转录因子在全部4个的启动子中均能预测到,赤霉素响应作用元件在3个基因的启动子区域能检测到(除),厌氧感应、昼夜规律、茉莉酮酸甲酯、玉米蛋白代谢、乙烯响应和干旱响应相关的顺式作用元件均能在不同的2个的启动子区域鉴定到,此外,脱落酸响应()、低温感应()和真菌激发子()分别在一个的启动子区域出现(表5)。

红色圆点标注为葡萄KEA家族蛋白,红色线条表明亚族GroupⅠ和Ⅱ的分界线

图3 葡萄KEA家族成员Motif和基因结构分析

图4 不同Motif的特征序列及长度

图5 葡萄KEA家族蛋白三级结构预测
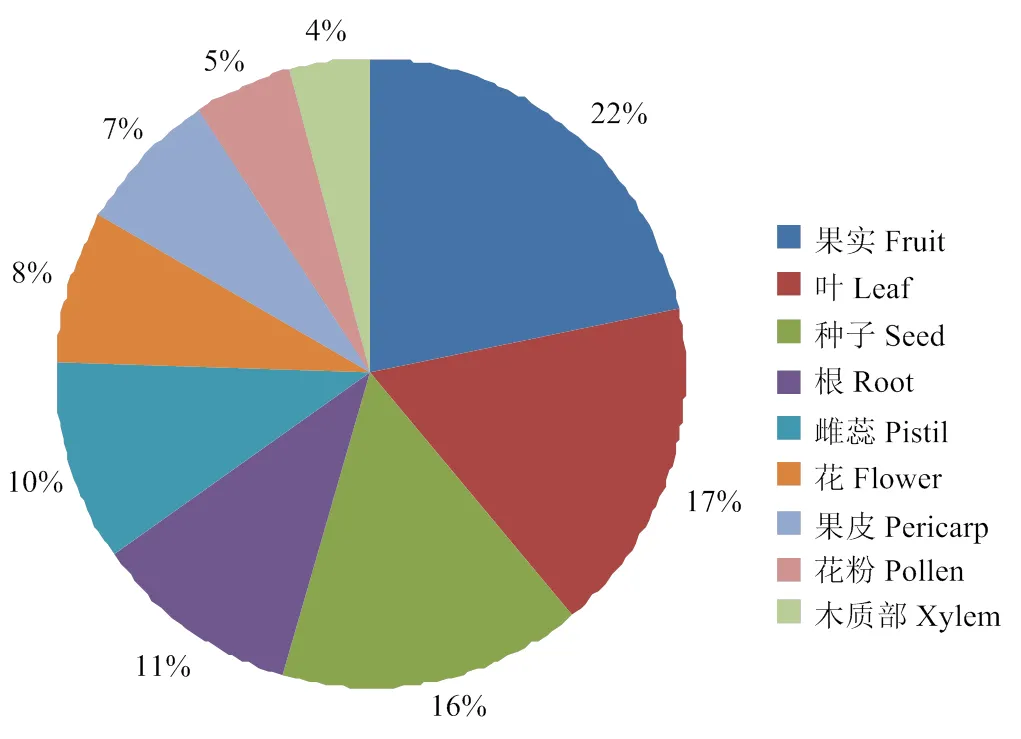
图6 葡萄KEA家族基因的EST表达谱分析

表4 葡萄KEA家族基因的亚细胞定位预测
2.6 葡萄KEA家族基因表达特征分析
通过实时荧光定量PCR分析,—在8年生‘白罗莎里奥’葡萄不同组织中的表达量有差异,在不同组织中的整体表达水平最高(图7),其他3个基因在葡萄不同组织中的表达水平较为接近,均显著低于;在不同组织中,在花器官中的表达量最高,和在幼果中的表达量最高,在果实和叶片中的表达量较高,其中,在幼果中的表达水平最高,约为其他3个基因的4—10倍(图7),暗示其可能在幼果发育过程中发挥作用。
2.7 幼苗中葡萄KEA家族基因对胁迫处理的响应
幼苗中,—在转录水平对不同胁迫处理的响应有差异:—对ABA处理最为敏感,其表达水平在检测的地上部和根部均被显著诱导;在地上部及在地上部和根部的表达水平对缺钾处理较为敏感,其表达水平均显著被诱导;在地上部和根部及在根部对sorbitol渗透处理较为敏感,其表达水平显著被诱导;此外,—在转录水平对NaCl处理没有响应,其表达水平没有显著差异(图8)。
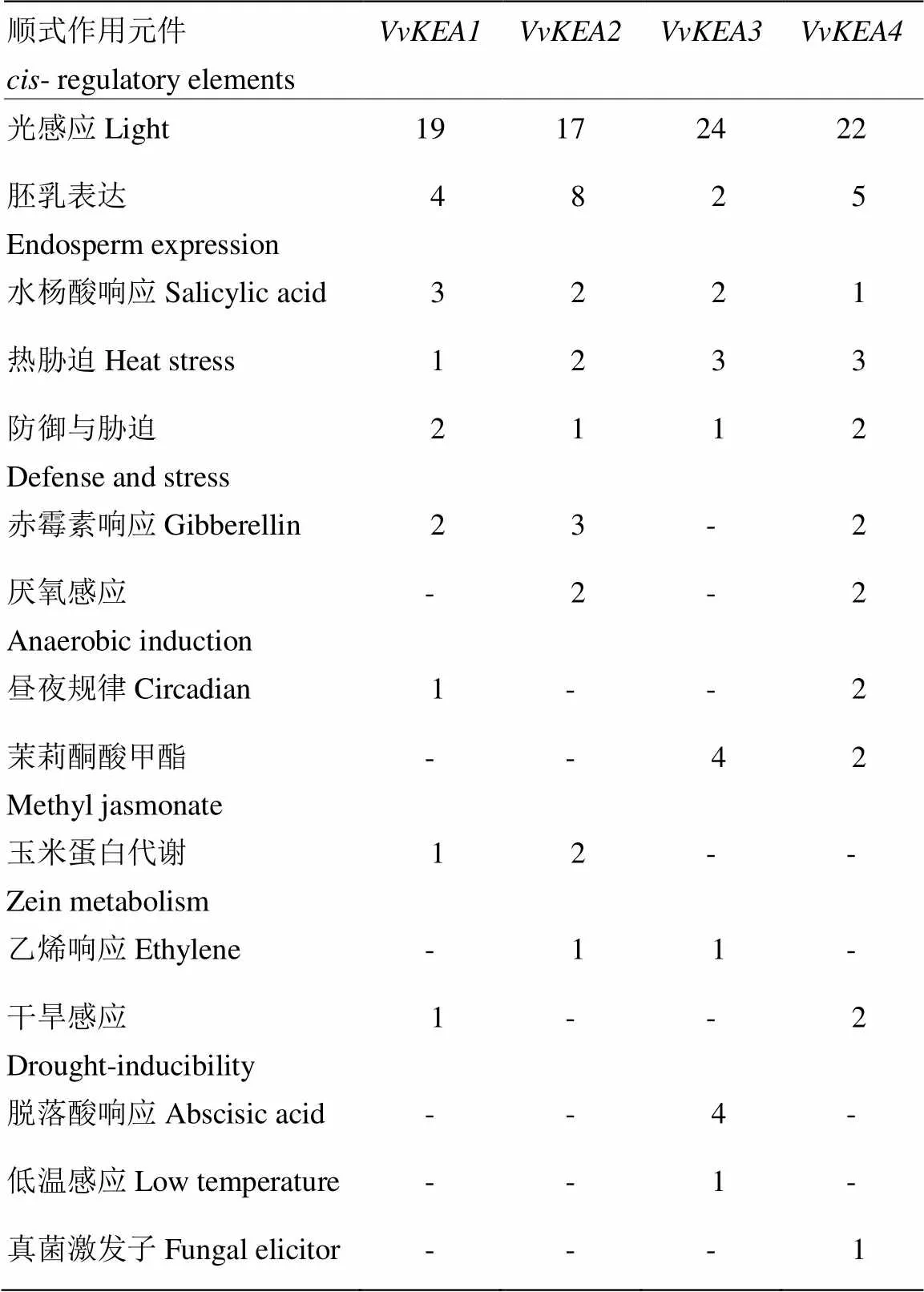
表5 葡萄KEA家族基因启动子顺式作用元件分析
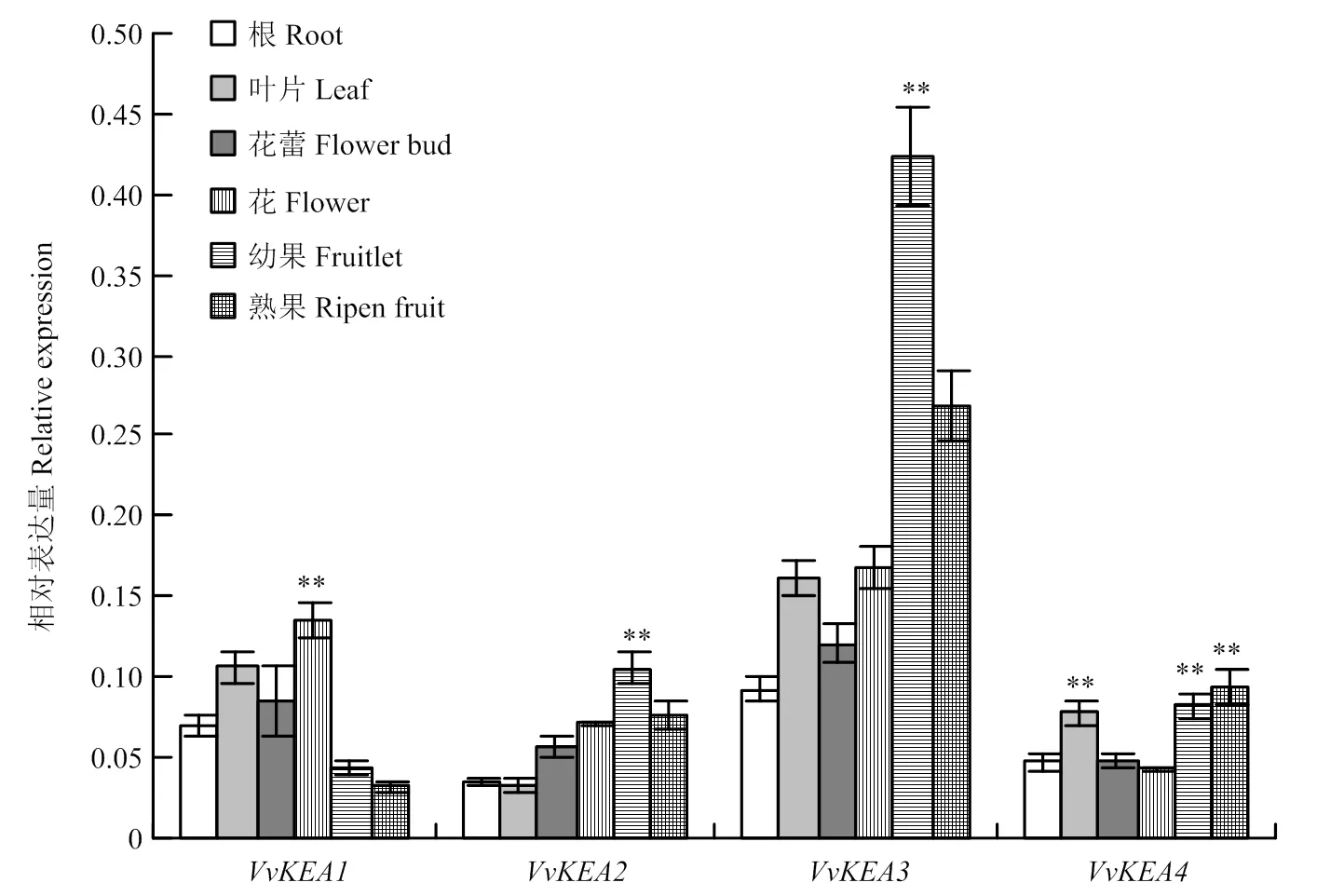
**表示在P<0.01水平差异极显著。下同
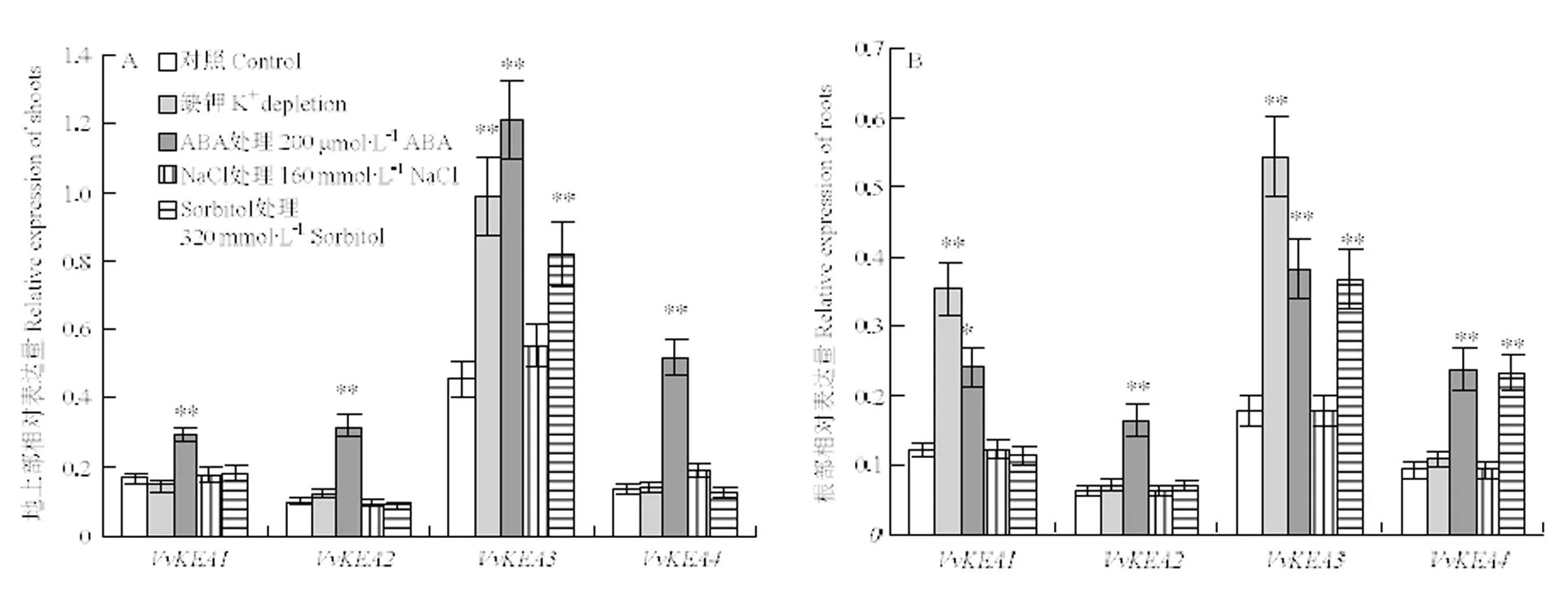
A:地上部相对表达量;B:根部相对表达量。*表示在P<0.05水平差异显著
3 讨论
果园中,适量的钾肥施用有助于果树发育、开花和果实品质形成[19-22],相关报道主要集中在生理生化层面,钾素营养分子基础的研究报道鲜少。植物中,KEA转运蛋白是一类维持体内K+动态平衡并参与叶绿体功能、光合作用和渗透调节作用的K+/H+逆向转运体,相关报道主要集中在模式作物拟南芥[8, 13-15]。果树中有关K+/H+动态平衡的研究依然是未知的。
近年来,植物基因组测序技术迅速发展,为植物科学研究提供了丰富的基因资源。不同物种之间,同一家族基因成员之间数目差异显著。本研究在葡萄中克隆并鉴定了4个转运体基因,与白杨(4)和草莓(5)中KEA成员数目接近,而远低于蔷薇科果树梨(12)和苹果(7)。此外,不同物种KEA家族成员在遗传进化关系上存在差异,2种双子叶植物(葡萄和拟南芥)KEA成员在遗传距离上是较近的,4种禾本科植物(水稻、玉米、高粱和短柄草)KEA成员更倾向于聚在一起,而3种木本植物(白杨、苹果和梨)KEA成员更倾向于聚在一起(图2),反映了不同物种在长期进化中依然是保守的,遗传距离也是较近的。在4个葡萄KEA转运体中,VvKEA1和VvKEA2同属于GroupⅠ,且具有6个相似的Motif,而VvKEA3和VvKEA4同属于GroupⅡ,具有7个相似的Motif(图2和图3),这些结果表明,进化关系上相近的(同一亚族)且具有类似蛋白Motif基序的两个转运体之间可能具有相似的功能。
前人研究表明拟南芥—主要定位于细胞质膜上,其中和定位于叶绿体膜上,定位于类囊体膜上[13,15],亚细胞定位预测结果表明葡萄—主要在细胞质膜上(表4),与Kunz等[15]报道一致,需要进一步的试验验证。Han等[11]报道指出拟南芥和主要在地上部表达,而其他4个成员基因在植物全身均有表达,与之情况一致的是,EST表达谱表明葡萄KEA家族基因在葡萄多种组织中均有表达,其中,在果实中的表达水平最高(图6);荧光定量PCR验证结果与表达谱分析是一致的,即在8年生‘白罗莎里奥’葡萄树不同组织中,3个基因(、和)的表达量均在果实中是最高的,特别是,其表达量在所有基因、不同检测组织中是最高的,暗示VvKEA3可能在果实发育中(特别是幼果时期)发挥重要作用的K+/H+逆向转运体。
植物KEA转运体在K+动态平衡、渗透调节、光合作用及pH调控中起主要作用[12-15]。荧光定量PCR结果表明葡萄幼苗中—在转录水平的表达量易受缺钾、ABA、NaCl或sorbitol等胁迫的诱导而上调,对不同胁迫处理的响应有差异(图8),其中作为葡萄KEA家族基因中整体表达量最为丰富的基因,对胁迫处理最为敏感,其表达水平极易受缺钾、ABA和sorbitol处理的影响而显著增强,与拟南芥[13]和大豆[23]中报道的现象类似;此外,Chen等[23]报道表明大豆KEA基因易受NaCl处理诱导表达,然而,本研究中—对NaCl处理没有响应,说明禾本科植物与木本科植物同源家族基因的功能或调控机制方面可能存在差异;尽管与对NaCl处理没有响应,但对等渗作用的sorbitol处理较为敏感,直接表明葡萄与易受渗透作用的胁迫诱导,而不受盐离子作用(Na+),这些发现与拟南芥中和的报道是一致的[13]。
此外,-顺式作用元件可通过与启动子区域的关键元件结合来控制启动子效率,进而调控靶标基因的表达[24-26]。本文结果发现,葡萄KEA家族基因启动子区域鉴定到多种顺式作用元件(表5),特别地,启动子区域含有脱落酸(ABA)响应的作用元件,而该基因的表达水平在葡萄幼苗全身组织中是受ABA处理诱导的;—启动子区域均含有光感应、胚乳特异表达、水杨酸响应、热胁迫响应及防御与胁迫相关的作用元件,这些发现与同为钾离子转运体的KT/HAK/KU家族转运体的报道是类似的[23-25],也为进一步研究葡萄KEA转运体的功能及其调控机理提供了理论支持。
本文为研究果树K+/H+平衡及钾素动态平衡机制提供了分子基础,并为高效园艺作物的遗传改良与育种奠定了理论依据。
4 结论
从葡萄中克隆并鉴定了4个KEA家族基因,该家族基因主要在葡萄果实、叶片和种子中表达;葡萄KEA成员与拟南芥KEA成员同源性较高;在成年葡萄树中的整体表达水平最为丰富(幼果中最高)。幼苗中在转录水平受缺钾、ABA和sorbitol渗透胁迫的调控;VvKEA3是葡萄果实中重要的K+/H+逆向转运体。
[1] VÉRY A A, SENTENAC H. Molecular mechanisms and regulation of K+transport in higher plants., 2003, 54: 575-603.
[2] GRABOV A. Plant KT/KUP/HAK potassium transporters: Single family - multiple functions., 2007, 99: 1035-1041.
[3] PALMGREN M G. Plant plasma membrane H+-ATPases: Powerhouses for nutrient uptake., 2001, 52: 817-845.
[4] FELLE H H. pH: Signal and messenger in plant cells., 2001, 3: 577-591.
[5] SZE H, SCHUMACHER K, Muller M L, PADMANABAN S, TAIZ L. A simple nomenclature for a complex proton pump: VHA genes encode the vacuolar H+-ATPase., 2002, 7:157-161.
[6] FELLE H H. pH regulation in anoxic plants., 2005, 96: 519-532.
[7] CHANROJ S, LU Y, PADMANABAN S, NANATANI K, UOZUMI N, RAO R, SZE H. Plant-specific cation/H+exchanger 17 and its homologs are endomembrane K+transporters with roles in protein sorting., 2011, 286(39): 33931-33941.
[8] CHANROJ S, WANG G, VENEMA K, ZHANG M W, DELWICHE C F, SZE H. Conserved and diversified gene families of monovalent cation/H+antiporters from algae to flowering plants., 2012, 3: 25.
[9] BASSIL E, BLUMWALD E. The ins and outs of intracellular ion homeostasis: NHX-type cation/H+transporters., 2014, 22: 1-6.
[10] MASER P, THOMINE S, SCHROEDER J I, WARD J M, HIRSCHI K, SZE H, TALKE IN, AMTMANN A, MAATHUIS F J, SANDERS D, HARPER J F, TCHIEU J, GRIBSKOV M, PERSANS M W, SALT DE, KIM S A, GUERINOT M L. Phylogenetic relationships within cation transporter families of., 2001, 126: 1646-1667.
[11] HAN L, LI J L, WANG L, SHI W M, SU Y H. Identification and localized expression of putative K+/H+antiporter genes in., 2015, 37(5): 1-14.
[12] ARANDA-SICILIA M N, CAGNAC O, CHANROJ S, SZE H, RODRÍGUEZ-ROSALES M P, VENEMA K.KEA2, a homolog of bacterial KefC, encodes a K+/H+antiporter with a chloroplast transit peptide., 2012, 1818(9): 2362-2371.
[13] ZHENG S, PAN T, FAN L G, QIU Q S. A novelgene family, homolog of bacterial K+/H+antiporters, plays potential roles in K+homeostasis and osmotic adjustment in., 2013, 8(11): e81463.
[14] ARMBRUSTER U, CARRILLO L R, VENEMA K, PAVLOVIC L, SCHMIDTMANN E, KORNFELD A, JAHNS P, BERRY J A, KRAMER D M, JONIKAS M C. Ion antiport accelerates photosynthetic acclimation in fluctuating light environments., 2014, 5: 5439.
[15] KUNZ H H, GIERTH M, HERDEAN A, SATOH-CRUZ M, KRAMER D M, SPETEA C, SCHROEDER J I. Plastidial transporters KEA1, -2, and -3 are essential for chloroplast osmoregulation, integrity, and pH regulation in., 2014, 111(20): 7480-7485.
[16] ZHOU H, QI K, LIU X, YIN H, WANG P, CHEN J, WU J, ZHANG S. Genome‑wide identification and comparative analysis of the cation proton antiporters family in pear and four otherspecies., 2016, 291: 1727-1742.
[17] 韩蕾, 宋志忠, 王莉, 苏彦华. 7种植物K+/H+逆向转运体蛋白的生物信息学分析. 基因组学与应用生物学, 2011, 30(3): 372-378.
HAN L, SONG Z Z, WANG L, SU Y H. Bioinformatics analysis of 7 plants’ K+/H+antiporters., 2011, 30(3): 372-378. (in Chinese)
[18] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCTmethod., 2001, 25(4): 402-408.
[19] DEMIRAL M A, KÖSEOGLU A T. Effect of potassium on yield, fruit quality, and chemical composition of green house-grown aalia melon., 2005, 28: 93-100.
[20] YURTSEVEN E, KESMEZ G D, ÜNLÜKARA A. The effects of water salinity and potassium levels on yield, fruit quality and water consumption of a native central anatolian tomato species ()., 2005, 78: 128-135.
[21] HARTZ T K, JOHNSTONE P R, FRANCIS D M, MIYAO E M. Processing tomato yield and fruit quality improved with potassium fertigation., 2005, 40: 1862-1867.
[22] 宋志忠, 郭绍雷, 马瑞娟, 俞明亮. KT/HAK/KUP家族基因在桃开花期的表达及对钾肥施放的响应分析. 中国农业科学, 2015, 48(6): 1177-1185.
SONG Z Z, GUO S L, MA R J, YU M L. Analysis of expression of KT/HAK/KUP family genes and their responses to potassium fertilizer application during peach flowering., 2015, 48(6): 1177-1185. (in Chinese)
[23] CHEN H T, CHEN X, WU B Y, YUAN X X, ZHANG H M, CUI X Y. Whole-genome identification and expression analysis of K+efflux antiporter (KEA) and Na+/H+antiporter (NHX) families under abiotic stress in soybean., 2015, 14(6): 1171-1183.
[24] GUPTA M, QIU X, WANG L, XIE W, ZHANG C J, XIONG L Z, LIAN X M, ZHANG Q F. KT/HAK/KUP potassium transporters gene family and their whole-life cycle expression profile in rice ()., 2008, 280: 437-452.
[25] SONG Z Z, MA R J, YU M L. Genome-wide analysis and identification of KT/HAK/KUP potassium transporter gene family in peach ()., 2015, 14(1): 774-787.
[26] ZHANG Z, ZHANG J, CHEN Y, LI R, WANG H, WEI J. Genome- wide analysis and identification of HAK potassium transporter gene family in maize (L.)., 2012, 39: 8465-8473.
Cloning, Characterization and Expression Analysis of K+/H+Antiporter Genes in Grape
WANG ZhuangWei1, WANG QingLian1, XIA Jin1, WANG XiCheng1, SONG ZhiZhong1,2, WU WeiMin1
(1Institute of Pomology, Jiangsu Academy of Agricultural Sciences/Jiangsu Key Laboratory of Horticultural Crop Genetic Improvement, Nanjing 210014;2School of Agriculture, Ludong University, Yantai 264025, Shandong)
【Objective】Isolation and characterization of KEA family genes from grape. Analysis of the tissue-specific expression patterns of KEA family genes and response to K+depletion, ABA, NaCl and sorbitol treatments. Screen the potential major KEA genes in grape. 【Method】By carring out homology-based cloning, putative KEA family genes were isolated and characterized from grape.A phylogenetic tree was constructed by multiple alignment of KEA family proteins from 9 known plants (grape,, rice, maize, sorghum, slender false brome, polar, pear, and apple) using the neighbor-joining method via MEGA7.0 software. Details of grape KEA family genes and encoded proteins were analyzed with the help of bioinformatical analysis softwares. By screening the EST database, electrical expression profiles of grape KEA genes were determined. Quantitative real-time PCR (qRT-PCR) was carried out to analyze the expression patterns of KEA family genes and response to K+depletion, ABA, NaCl, and sorbitol treatments, and obtained the major genes. 【Result】Four KEA family genes were isolated from grape, entitled by—, which were all containing the K/H exchanger and TrkA-N functional domains that belonging to the classic plant KEA family antiporters. The amino acid sequences of KEA proteins from 9 plants shared an overall identity of 33.10%. These KEA members were classified into 2 major groups (Groups Ⅰ and Ⅱ), and VvKEA1and VvKEA2 belong to Group Ⅰ that containing 7 Motifs, while VvKEA3 and VvKEA4 belong to GroupⅡ that just containing 4 Motifs. Phylogenetic tree analysis showed that VvKEA1, VvKEA 2 and VvKEA 4 of grape were closely clustered with AtKEA2, AtKEA 3 andAtKEA 5 of, respectively, and VvKEA3 was clustered with PbrKEA5 of pear and MdoKEA7 of apple. KEA members of 4 grass family plants (rice, maize, sorghum and slender false brome) were prone to clustered together, while three woody plants (polar, apple and pear) KEA members were prone to clustered together. Mainly localized in plasma membrane, all predicted VvKEA proteins possessed similar tertiary structures, contained 12 or 13 transmembrane domains (TMs), and the theoretical isoelectric point () were all less than 7.0. In particular, only VvKEA3 possessed the signalpeptide. Fifteen-acting regulatory elements, including the stress response, nutrition and development, hormone response and circadian rhythm regulations, et al., were identified in the promoter region ofgenes. Expression profile analysis showed thatfamily genes were expressed in different tissues or organs in grape, and the highest percentage was predicted in fruit, followed by leaf, seed, root and pistil. qRT-PCR analysis showed thatwas the most abundant expressed gene during different parts of 8-year-old ‘’ on the whole, especially in fruitlet, and the other 3 genes were less expressed with similar amount.In grape seedlings,—genes were more sensitive to ABA treatment, whose expression were all induced in both tested shoots and roots, but had no response to NaCl treatment. The expression ofVvKEA3 in both shoots and roots and VvKEA1 in shoots were up-regulated by K depletion treatment, and the expression of VvKEA3 in both shoots and roots and VvKEA4 in roots were increased by sorbitol treatment. 【Conclusion】Four predicted KEA family genes were cloned and characterized from grape, which were majorly expressed in fruit, leaf and seed. Notably,was the most abundant gene in 8-year old grape tree, especially in fruitlet, whose expression was prone to be regulated by K+depletion, ABA, and sorbitol osmotic stress. VvKEA3 may be a crucial
grape; K+/H+antiporter; bioinformaticsanalysis; stress treatment
10.3864/j.issn.0578-1752.2018.23.011
2018-05-14;
2018-06-21
江苏省农业科技自主创新项目(CX(17)3031)、国家自然科学基金(31501743)、现代农业产业技术体系建设专项资金(CARS-29-11)、江苏省政府留学奖学金(JS-2016-190)
王壮伟,Tel:025-83491977;E-mail:wzhuangzhuang@hotmail.com。
宋志忠,Tel:025-84390255;E-mail:szhzh2000@163.com
(责任编辑 李莉)
