团数的DNA折纸术计算模型
2018-12-03崔建中殷志祥
崔建中,殷志祥,杨 静
(1.安徽理工大学 电气与信息工程学院, 安徽 淮南 232001;2.淮南联合大学 计算机系, 安徽 淮南 232038;3.安徽理工大学 数学与大数据学院, 安徽 淮南 232001)
DNA折纸术[1]是近年来提出的一种新的DNA自组装方法,是目前DNA自组装领域研究的热点问题.与DNA模块自组装相比,DNA折纸术能构造出更复杂、精细的纳米结构,且DNA链的设计简单,组装效率较高.它的原理是利用较短的DNA订书钉链(Staple),按碱基配互补对原则,对一条较长的脚手架链(Scaffold)进行折叠,从而构造出理想的纳米结构.文献[2]利用DNA 折纸术原理成功地构造出纳米中国地图,证明了DNA折纸术具备构造非对称图案的能力.文献[3]将文献[1]的方法加以推广,并成功地构造出方螺帽等6种三维结构.文献[4]利用折纸术折叠成长方形的纳米结构来编码有向图的顶点,给出了通过该长方形结构的自组装寻找最短哈密顿路的方法.文献[5]采用DNA 纳米折纸结构编码无向图的顶点,借助纳米结构之间的粘性末端进行自组装,给出了图着色问题的一种解决方法.文献[6] 利用方形DNA折纸作为基本单元,以递归方式进行多次自组装,得到了微米级蒙娜丽莎的图案.文献[7]设计了三维砖形DNA基本折纸单元,可以组装成更大尺寸的三维结构.文献[8]利用V形DNA折纸作为基本单元,通过控制基本单元之间的几何形状和作用,可以构造更大的组装体.文献[9]提出了利用噬菌体制备DNA折纸所需的DNA链,为DNA折纸的应用和量产提供了保障.
最大团问题(Maximum Clique Problem)是图论中经典的组合优化问题,也是一类NP完全问题.最大团问题在计算机视觉、市场分析、编码理论中都有非常广泛的应用.文献[10] 使用双链DNA分子编码无向图的顶点,建立初始数据池,根据补图中相邻的顶点不可能同时出现在极大团中,在初始数据池中并行地删除非解.最后,初始数据池中长度最短的DNA分子编码的顶点即为所求的最大团.文献[11]给出了利用质粒求解最大团问题的算法.文献[12]将剪枝策略运用到DNA计算中,给出了基于粘贴模型的最大团问题的算法.文献[13]给出了基于环形DNA分子求解最大团问题的计算模型.文献[14]提出了最大团问题的Tile自组装模型.
图G中最大团的阶数即团数,记为ω(G),它与图的色数x(G)密切相关.易知,若图的团数为K,则该图至少是K-着色的.目前,DNA折纸术主要应用在纳米技术领域.将DNA折纸术应用于搜索简单无向图的最大团,进而求解图的团数.该模型利用订书钉链折叠脚手架链形成发夹结构,凝胶电泳检测发夹结构的变化来建立初始数据池、删除非解、读解.该模型简单、读解方便、可行性高.文中提出的计算模型一方面证明了DNA折纸术可以用来求解组合优化问题,另一方面也证明了订书钉链与脚手架链的杂交,结合链置换,凝胶电泳从算法的角度是完备的.它不仅拓宽了DNA折纸术的应用领域,也为解决组合优化问题提供了一种新的思路和方法.
1 团数问题
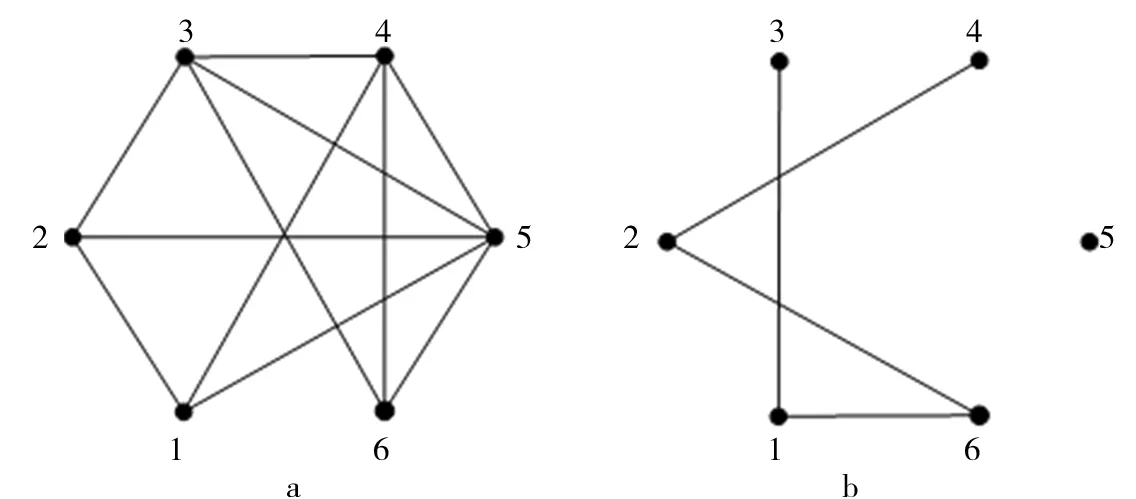
图1 简单无向图及其补图
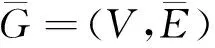
2 团数问题的DNA折纸术计算模型
2.1 脚手架链和订书钉链的设计
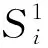
2.2 团的表示
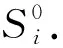
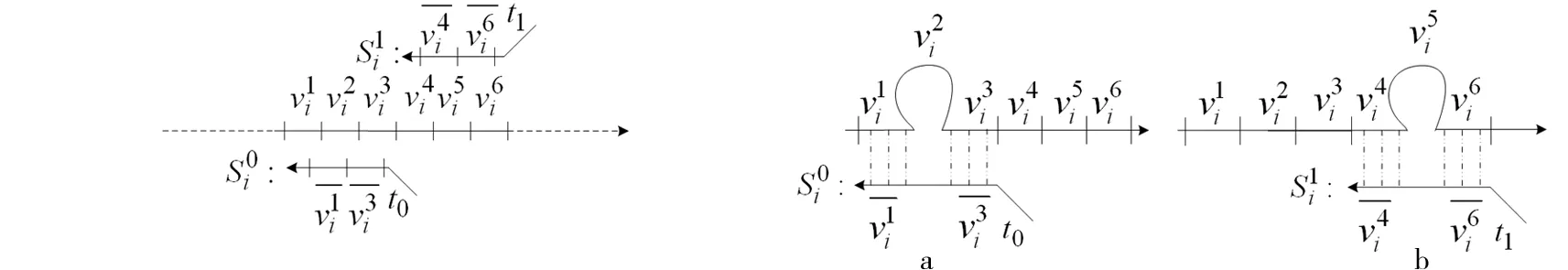
图2 脚手架链(中)与订书钉链上(下) 图3 团的表示
按照这种方法,最大团{3,4,5,6}的示意图如图4所示.
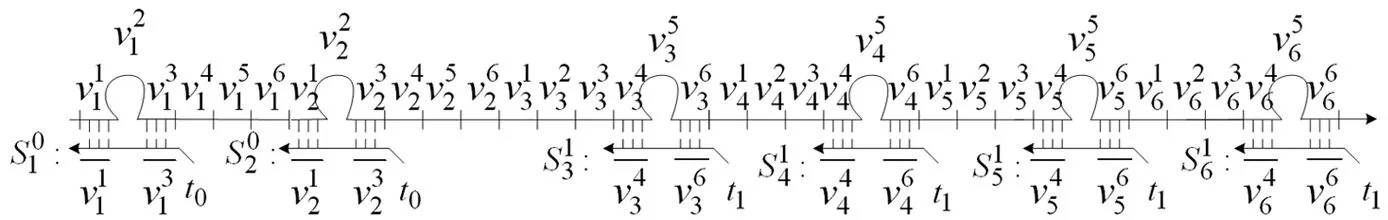
图4 最大团
2.3 算法
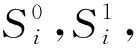
步骤1:
Fori=1 ton
Divide(T0→T1,T2)
Merge(T1,T2→T0)
Gel(T0)=l+ny→T0
Endfor
步骤2:
Fori=1 tom
Forj=i+1 tom
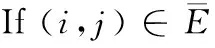
Divide(T0→T1,T2,T3)
Merge(T1,T2,T3→T0)
Endif
Gel(T0)=l+ny→T0
Endfor
Endfor
步骤3:
Gel(T0)=lmin→T0
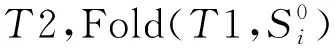
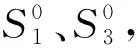
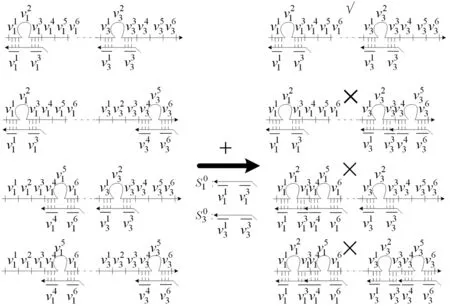
图5 再次折叠脚手架链选择编码正确的团
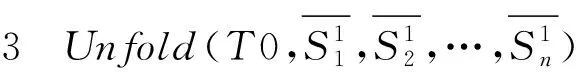
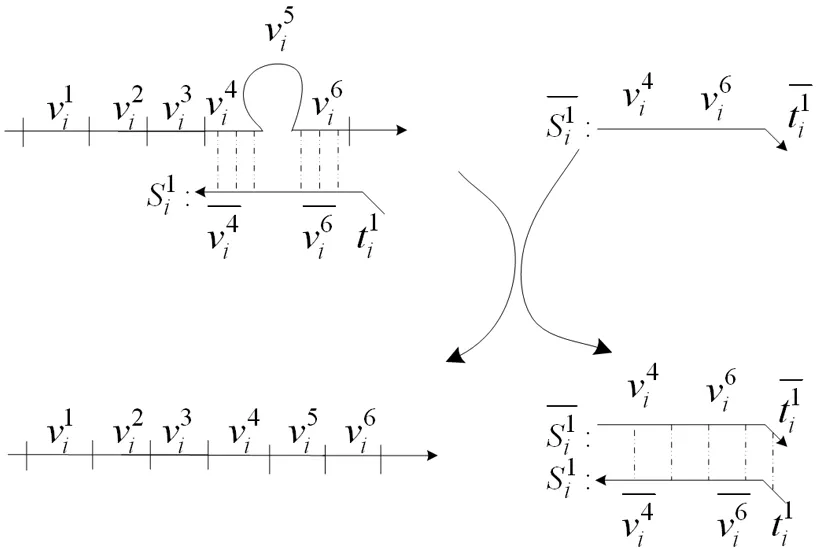
图6 链置换打开表示顶点i在团中的发夹结构
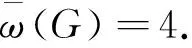
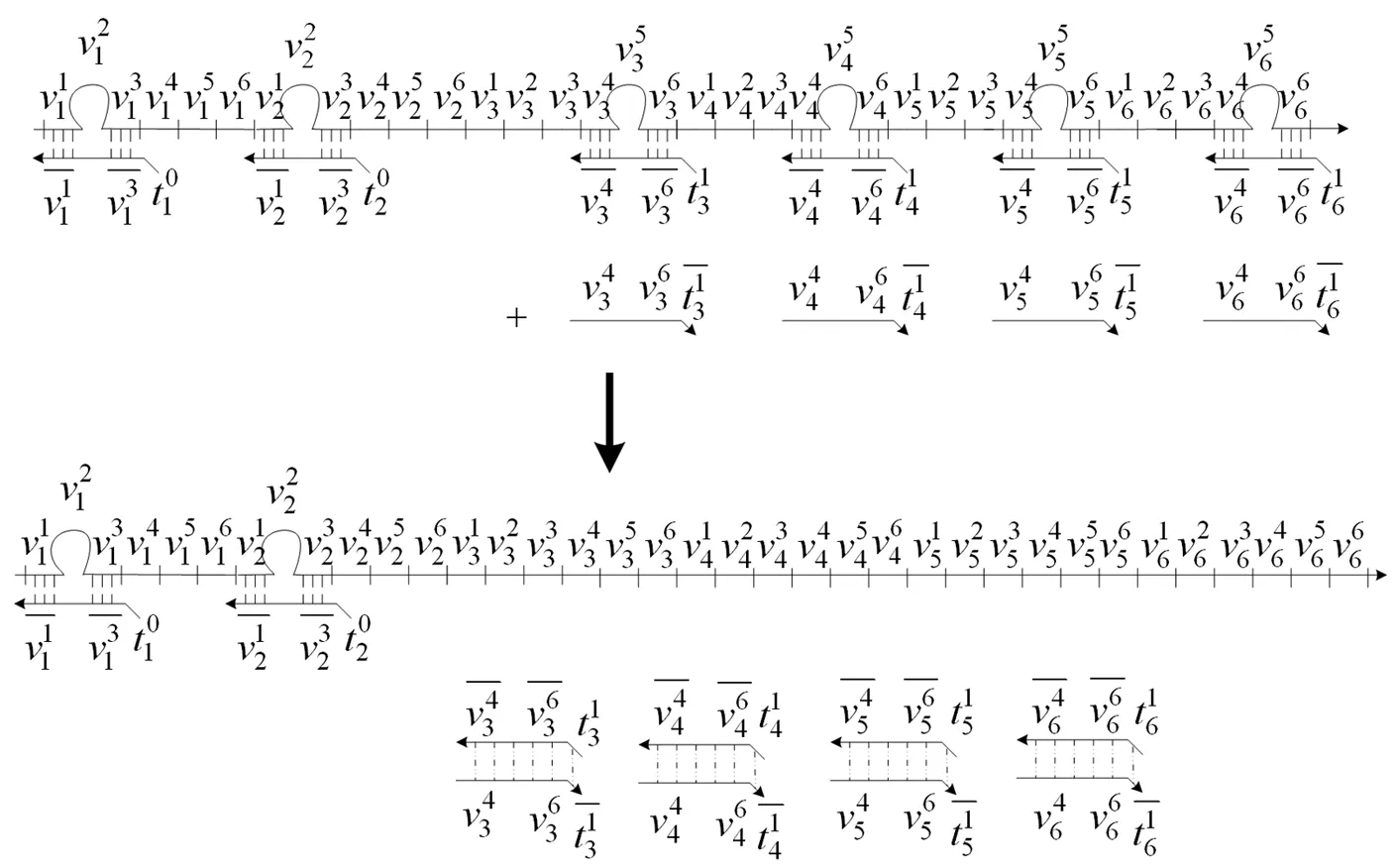
图7 打开表示顶点在团中的发夹结构的示意图
2.4 算法复杂度
对于简单无向G(V,E),其中|V|=n,|E|=m,文中提出的计算模型需要编码n种寡聚核苷酸片断表示给定图中的顶点,将这n种寡聚核苷酸片断连接构成脚手架链.当表示顶点的寡聚核苷酸片断编码完毕,相应的订书钉链及钉书钉链的补链的编码也随之确定.因此,文中提出的模型的编码复杂度为O(n).
在初始数据池中,为表示所有可能的团,模型需要n次折叠脚手架链,1次凝胶电泳,生成脚手架链上发夹结构的组合.然后,针对补图中的每条边,需要3次折叠脚手架链,1次凝胶电泳,删除非团.因此,共需3m次折叠脚手架链,m次凝胶电泳.最后为确定团数,需要1次打开脚手架链上的发夹结构,1次凝胶电泳.故模型的时间复杂度为O(n2).
与其它的DNA计算模型相比,文中提出的计算模型初始数据池中仅含有一种类型脚手架链,与问题的规模无关,脚手架链上发夹结构的组合为2n,它对应编码的顶点是否在团中的所有可能情况.脚手架链上发夹结构的变化可通过脚手架链的长度反映出来,凝胶电泳可以准确、可靠地判断发夹结构的变化,故模型的可靠性大大增加,可行性更高.
3 结论
应用DNA 折纸术求解团数问题,提出了团数的DNA折纸术计算模型.其核心思想是利用DNA折纸术,结合链置换,将团数映射为脚手架链上表示顶点在团中的发夹结构的数目,通过凝胶电泳检测脚手架链的长度,进而判断发夹结构的变化.DNA 折纸术的最大优点在于DNA链的编码简单,脚手架链和订书钉链杂交反应的相对浓度要求不高,实验简单,组装效率高.将DNA折纸术的核心思想应用于组合优化问题的求解,结合凝胶电泳判断发夹结构的变化,读解更简单,从而提高了DNA计算模型的可行性、可靠性.文中提出的模型目前仅能求出给定无向图的团数,而最终确定最大团中的顶点仍需要结合其他的读解方法,如分子信标,这也是下一步要进行的工作.文章从理论层面证明了订书钉链与脚手架链的杂交,结合链置换,凝胶电泳从算法的角度是完备的.它不仅扩宽了DNA折纸术的应用领域,也为解决组合优化问题提供了一种新的思路和方法.
