甘蓝型油菜芸薹素唑耐受因子(BnaBZR1/BnaBES1)全长CDS克隆与生物信息学分析
2018-11-29官春云
冯 韬 官春云
甘蓝型油菜芸薹素唑耐受因子(BnaBZR1/BnaBES1)全长CDS克隆与生物信息学分析
冯 韬 官春云*
湖南农业大学农学院 / 国家油料改良中心湖南分中心, 湖南长沙 410128
芸薹素唑耐受因子(brassinazole-resistant, BZR)是油菜素内酯信号转导过程中的关键转录因子, 目前已知BZR1与BZR2 (BES1)两种亚型。本文在甘蓝型油菜湘油15号cDNA中克隆到3个全长编码序列(coding sequence, CDS), 经比对鉴定分别为定位于A07染色体的1拷贝BZR1和定位于A06染色体的2拷贝BES1, 分别命名为、和, 序列长分别为996、993和996 bp, 各自编码331、330、331个氨基酸。这3个基因编码蛋白具有典型植物BZR/BES结构域, 亚细胞定位预测主要位于细胞核。多序列比对和进化分析表明,基因编码蛋白与甘蓝、白菜、拟南芥、亚麻芥等BZR/BES蛋白具有较高的同源性, 且同源物种间BZR1或BES1相似度高于同一物种或者近缘物种中BZR1与BES1间相似度, 表明BZR1与BES1分化是一个早期进化事件。湘油15号中这3个基因表达规律相似, 苗期和花期具有高水平的基因表达, 抽薹期和角果成熟期表达稍低; 且基因表达水平在地上部分的叶、茎、花和角果中相对于地下部分的根中较高。
甘蓝型油菜; 芸薹素唑耐受因子; 基因克隆; 基因表达; 生物信息学分析
油菜素内酯信号途径是植物中重要的激素途径, 其活性调控与植物生长发育以及抗逆能力息息相关。芸薹素唑耐受因子BZR是油菜素内酯信号与其他信号互作的核心元件, 在油菜素内酯信号转导中BZR1/BES1复合体与PIF4和DELLA等转录因子互作, 分别介导油菜素内酯信号途径与光信号[1]、赤霉素信号[2]、茉莉酸信号[3]等互作。在拟南芥中, AtBZR1与AtBES1形成异源复合体并相互磷酸化, 磷酸化后的异源二聚体与AtPIF4结合, 形成三元复合物介导下游基因表达调控, 从而完成油菜素内酯信号与光信号之间的传递, AtBZR1/AtBES1通过磷酸化修饰结合DELLA蛋白, 从而影响赤霉素的生物合成与赤霉素信号通路产生互作[4]。
拟南芥[5]、水稻[6]等植物中油菜素内酯信号途径的研究比较深入, 不仅完成了信号途径中主要基因的克隆, 而且对信号转导、传递有了较全面的认识。拟南芥和水稻中油菜素内酯信号转导过程基本一致, 油菜素内酯(BRs)结合膜表面受体BRI1激活其激酶活性, 并诱导BRI1结合BAK1形成二聚体后相互磷酸化, BSKs蛋白通过活化的BRI1磷酸化其230位丝氨酸而激活, 活化的BSKs蛋白激活BSU1活性, 进而BSU1阻断BIN2对BZR/BES蛋白的过磷酸化, 去磷酸化的BZR/BES蛋白在细胞核积累并与PIF4和DELLA等转录因子互作, 同时参与下游靶基因转录调控。
有研究表明, 油菜素内酯可以促进油菜对高盐环境耐受性, 调节油菜真菌抗性, 影响油菜脂代谢。油菜素内酯处理可明显改善油菜幼苗对渗透胁迫的抗性[7], 2,4-表油菜素内酯可增强油菜对丁香假单胞菌的抗性[8], 同时2,4-表油菜素内酯影响油菜脂代谢, 调控酯质信号的产生和转导[9]。过表达油菜素内酯合成基因可以促进油菜提高产量[10], 表明油菜素内酯与甘蓝型油菜生长发育息息相关, 油菜素内酯在油菜生产上有广阔的应用前景。然而甘蓝型油菜中油菜素内酯信号途径的相关研究却比较薄弱, 信号通路的构成元件大部分都还处于未知状态, 严重制约了油菜素内酯信号相关基因筛选在高含油、高抗性油菜新品种选育过程中的作用, 限制了油菜分子育种中相关基因工程的应用, 同时由于理论研究的空白限制了油菜素内酯在油菜生产上的应用。
本文通过甘蓝型油菜基因组数据库中预测的基因序列设计引物, 从甘蓝型油菜品种湘油15号中克隆获得3个基因CDS序列, 一个鉴定为BZR1型, 2个鉴定为BES型, 均位于A染色体组。
1 材料与方法
1.1 试验材料
甘蓝型油菜双低品系湘油15号由国家油料改良中心湖南分中心提供。
1.2 RNA提取与cDNA合成
将湘油15号在22℃、光周期为16 h/8 h条件下培养至2片真叶, 取幼苗50~100 mg。加液氮充分研磨后, 使用TRIzol RNA提取试剂盒(TransGen生物技术有限公司)按其所示方法提取总RNA, 以此RNA为模板参照Easy Script First-Strand cDNA Synthesis SuperMix试剂盒(TransGen生物技术有限公司)说明书合成第1链cDNA。
1.3 BnaBZR基因的克隆及序列分析
从BRAD数据库(http://brassicadb.org/brad/)甘蓝型油菜全转录组中提取以及的转录本序列。分析比对, 设计含起始密码子的5'端引物和含有终止密码子的3'端引物, 合成备用。设计的引物序列为BnaBZR1-F: 5'-ATGACGTCAGA TGGAGCTAC-3', BnaBZR1-R: 5'-TCAACCACGAG CTTTGCCG-3', BnaBES1-F: 5'-ATGACGTCTGACG GTGCG-3', BnaBES1-R: 5'-CTAACGACCTTTAGCG TTTCC-3'。以1.2所述cDNA为模板进行PCR扩增。PCR体系含50 ng μL–1模板1 μL、10 mmol L–1dNTPs 0.5 μL、2 μmol L–1BnaBZR1-F 0.75 μL、2 μmol L–1BnaBZR1-R 0.75 μL、10×反应缓冲液1 μL、HiFi高保真DNA聚合酶0.5 μL和ddH2O 15.5 μL。程序为预变性95℃ 5 min; 95℃ 50 s, 58℃ 45 s, 72℃ 3 min, 35个循环; 72℃后延伸10 min。PCR产物经1.5%琼脂糖凝胶电泳, 用凝胶成像系统(Bio-Rad)检测和记录结果。将PCR产物回收后与pMD18-T载体(TransGen生物技术有限公司)连接, 转化大肠杆菌感受态细胞Trans1-T1 (北京天根生化有限公司), 筛选阳性克隆进行基因测序, 通过BRAD数据库(http://brassicadb.org/brad/)对获得的克隆序列进行染色体步移分析, 随后使用DNAman对克隆获得的和基因全长CDS序列进行序列比对。通过NCBI核酸数据库(http://www.ncbi.nlm. nih.gov/nuccore/)下载甘蓝型油菜BZR相关基因转录本序列及植物BZR相关蛋白, 构建HMM文件, 使用HMMER软件检索甘蓝型油菜预测蛋白组, 并就BZR相关转录本在甘蓝型油菜基因组中的分布进行分析。
1.4 BnaBZR蛋白生物信息学分析
1.4.1 BnaBZR1_A07、BnaBES1_A06F和BnaBES1_ A06R氨基酸序列特征与二级结构预测 通过ANTHEPROT 6.2软件分析来源于甘蓝型油菜的BnaBZR1和BnaBES1基因所编码的蛋白质。统计编码蛋白的氨基酸残基组分、分析编码蛋白的等电点、信号肽序列和亲疏水特征并使用Gariner模型进行二级结构预测。使用PSORT (http://psort.hgc.jp/form. html)和Plant-mPLoc (http://www.csbio.sjtu.edu.cn/ cgi-bin/PlantmPLoc.cgi)进行亚细胞定位预测。
1.4.2 BnaBZR蛋白同源结构域分析 使用DNAman对BnaBZR蛋白进行同源比对分析; 同时使用NCBI (http://blast.ncbi.nlm.nih.gov/)分别对不同的BnaBZR蛋白序列进行保守结构域分析; 使用SMART (http://smart.embl-heidelberg.de/)、MOTIF (http://www. genome.jp/tools/motif/)及InterPro (http://www.ebi.ac. uk/interpro/)分析BnaBZR包含的特殊结构域。
1.4.3 BZR蛋白聚类分析 通过NCBI蛋白数据库(http://www.ncbi.nlm.nih.gov/protein)、String数据库(http://string.embl.de/)和PDB数据库(http://www. pdb.org/pdb/home/home.do)检索来源于不同物种的BZR相关蛋白序列, 并使用MEGA6.0对来源不同的BZR1相关蛋白进行聚类分析。
1.5 甘蓝型油菜中BnaBZR1_A07、BnaBES1_ A06F和BnaBES1_A06R基因时空表达分析
以1.2方法提取甘蓝型油菜湘油15号不同时期根、茎、叶、花和角果皮总RNA, 使用宝生物工程(大连)有限公司PrimeScript RTreagent Kit with gDNA Eraser (Perfect Real-time)试剂盒进行反转录, 获得cDNA第1链作为qRT-PCR模板。分析1.3中克隆获得的片段, 根据序列特征设计分型引物, 以作为内参基因[11], 用于和时空表达分析, 每样品进行3次重复。QRT-PCR在ABI 7500荧光定量PCR系统上运行。PCR体系包含1 μL cDNA、10 μL 2×FastStart Universal SYBR GreenMaster with ROX、10 μmol L–1正向引物和反向引物各0.5 μL、8 μL ddH2O。PCR程序为95℃ 10 min; 95℃ 15 s, 60℃ 15 s, 72℃ 32 s, 35个循环。反应完成后进行95℃ 20 s, 60℃ 20 s, 95℃ 20 s, 59℃ 20 s绘制熔解曲线检测扩增产物的正确性和引物二聚体。
2 结果与分析
2.1 BnaBZR基因全长CDS序列的克隆
以湘油15 cDNA为模板, 克隆到3个CDS序列, 长度分别为996、993和996 bp (图1), 分别定位于A07、A06和A06染色体上, 分别命名为、和。通过BRAD数据库比对, 确认3个CDS序列的完整性, 以3条CDS序列为模板分别对BRAD数据库进行检索, 发现1条同源序列位于C06染色体, 相似度82.5%, 发现1条和共有同源序列位于C08染色体, 相似度分别为85.2%和84.9%; 同时对、和进行多序列比对发现,与和相似度分别为82.28%和82.26%,和相似度98.79%。以上结果表明, 克隆获得的与和分属2个不同的家族亚类群, 并在C亚基因组上存在对应的同源基因; 甘蓝型油菜基因组具异源多倍性, 其A、C两个亚基因组之间高度同源, 在A染色体组上的基因大多存在C染色体组同源基因, C染色体组基因亦然。
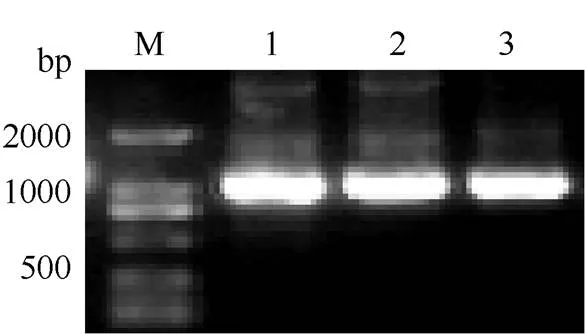
图1 BnaBZR1_A07、BnaBES1_A06F和BnaBES1_A06R 基因克隆
M: 2K DNA marker; 1:编码序列; 2:编码序列; 3:编码序列。
M: 2K DNA marker; 1:CDS; 2:CDS; 3:CDS.
从NCBI共获取甘蓝型油菜BZR/BES及BZR/BES类似序列转录本13个, 7个位于C染色体组, 6个位于A染色体组, 主要集中于A06、A07、A08、C06、C08和C09染色体; HMMER检索甘蓝型油菜预测蛋白组数据库共发现31条具有BZR/BES家族特征结构域的序列, 其中17个位于A染色体组, 14个位于C染色体组; 表明具有特征的基因在甘蓝型油菜A/C基因组间分布较均匀, 具有特征的家族基因在甘蓝和白菜2个亚组间可能具有相似的进化规律。
2.2 BnaBZR生物信息学分析
和基因各自编码长度331、330和331个氨基酸残基的蛋白, 分别命名为BnaBZR1_A07、BnaBES1_ A06F和BnaBES1_A06R (表1)。

表1 BnaBZR1、BnaBES1_F和BnaBES1_R蛋白信息汇总表
BnaBZR蛋白主要表现疏水性, 未发现信号肽序列, 暗示其不存在信号肽介导的跨膜转运。PSORT与Plant–mPLoc亚细胞定位预测显示, BnaBZR1_A07与BnaBES1_A06F和BnaBES1_A06 R 均主要定位于细胞核, 可信度0.789, 这与BZR蛋白作为转录因子的功能一致。
二级结构预测结果如图2, BnaBZR1_A07有27% α螺旋、14% β折叠、29% β转角和32%无规卷曲; BnaBES1_A06F分别有24%、16%、29%和31%; BnaBES1_A06R分别有23%、16%、29%和32%。

图2 BnaBZR1_A07、BnaBES1_A06F和BnaBES1_A06R蛋白二级结构
通过ANTHEPROT分析了BnaBZR1_A07、BnaBES1_A06F和BnaBES1_A06R蛋白具有的序列特异性识别位点。由表2可知, BnaBES1_A06F和BnaBES1_A06R具有仅含微弱差别的特异性识别序列, BnaBES1_A06R相对于BnaBES1_A06F仅175– 177位置识别序列存在一个氨基酸残基的差别, 其他位置的特异性识别序列完全一致, 表明BnaBES1_ A06F和BnaBES1_A06R蛋白相似度极高。BnaBZR1_A07蛋白缺少PS00016特异性序列, 同时PS00006特异性序列数量也更少。3条蛋白序列中特异性识别序列主要为糖基化修饰、磷酸化修饰、酰化修饰和细胞连接序列, 表明BnaBZR蛋白可能受多种激酶介导的糖基化、磷酸化、酰基化修饰调节, 并可能介导细胞间连接, 这与拟南芥BZR蛋白通过磷酸化水平改变实现核质穿梭与互作因子结合, 调控下游靶基因表达的功能相吻合。

表2 BnaBZR1_A07、BnaBES1_A06F和BnaBES1_A06R蛋白特殊识别位点序列总表
(续表2)
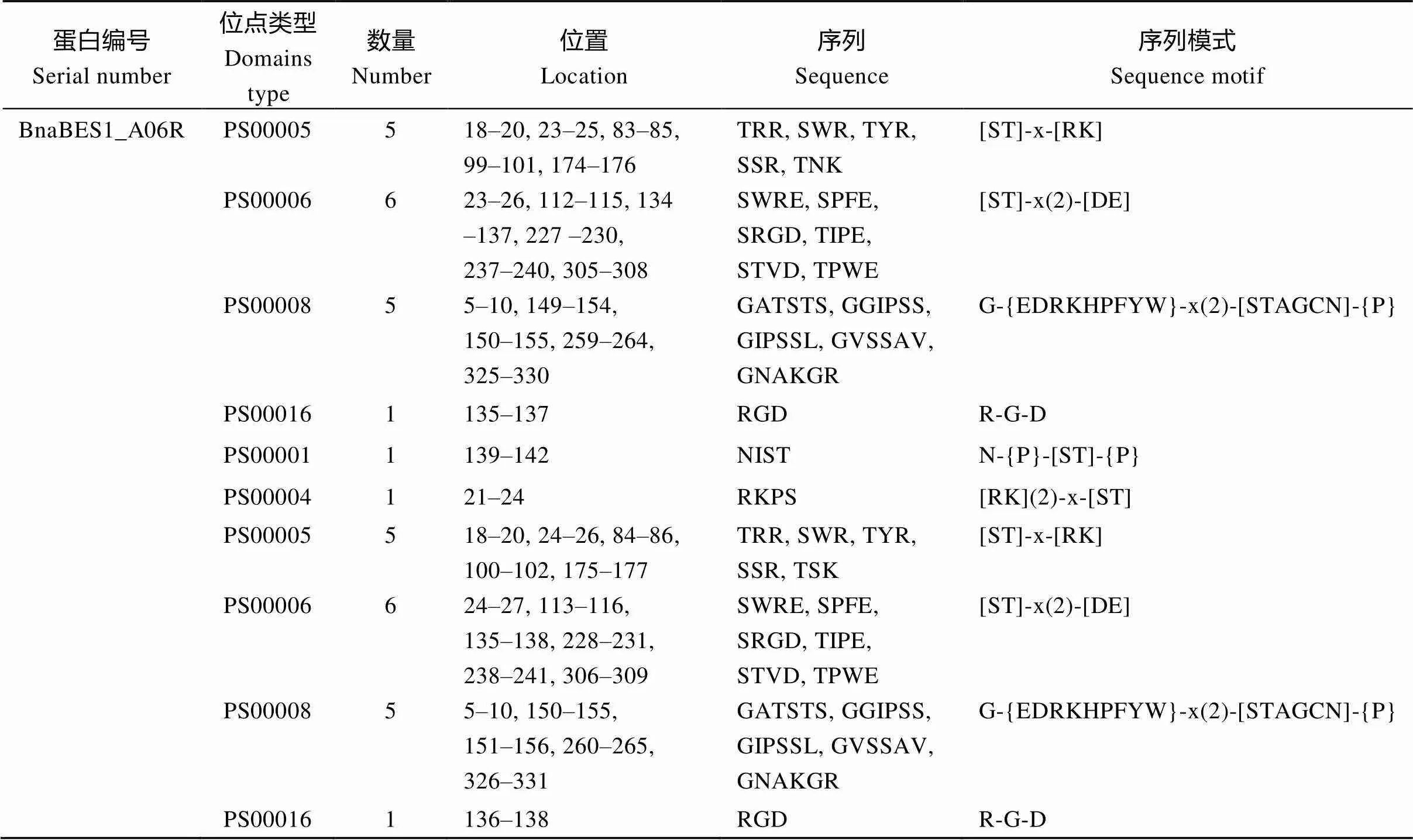
蛋白编号Serial number位点类型Domains type数量Number位置Location序列Sequence序列模式Sequence motif BnaBES1_A06RPS00005518–20, 23–25, 83–85, 99–101, 174–176TRR, SWR, TYR, SSR, TNK[ST]-x-[RK] PS00006623–26, 112–115, 134 –137, 227 –230, 237–240, 305–308SWRE, SPFE, SRGD, TIPE, STVD, TPWE[ST]-x(2)-[DE] PS0000855–10, 149–154, 150–155, 259–264, 325–330GATSTS, GGIPSS, GIPSSL, GVSSAV, GNAKGRG-{EDRKHPFYW}-x(2)-[STAGCN]-{P} PS000161135–137RGDR-G-D PS000011139–142NISTN-{P}-[ST]-{P} PS00004121–24RKPS[RK](2)-x-[ST] PS00005518–20, 24–26, 84–86, 100–102, 175–177TRR, SWR, TYR, SSR, TSK[ST]-x-[RK] PS00006624–27, 113–116, 135–138, 228–231, 238–241, 306–309SWRE, SPFE, SRGD, TIPE, STVD, TPWE[ST]-x(2)-[DE] PS0000855–10, 150–155, 151–156, 260–265, 326–331GATSTS, GGIPSS, GIPSSL, GVSSAV, GNAKGRG-{EDRKHPFYW}-x(2)-[STAGCN]-{P} PS000161136–138RGDR-G-D
PS00001: 糖基化位点; PS00004: cAMP/GMP 依赖激酶磷酸化位点; PS00005: 蛋白激酶C磷酸化位点; PS00006: 酪蛋白激酶磷酸化位点; PS00008: 豆蔻酰化位点; PS00016: 细胞粘连序列。
PS00001: N-glycosylation site; PS00004: cAMP- and cGMP-dependent protein kinase phosphorylation site; PS00005: Protein kinase C phosphorylation site; PS00006: Casein kinase II phosphorylation site; PS00008: N-myristoylation site; PS00016: Cell attachment sequence.
同源比对BnaBZR1_A07、BnaBES1_A06F和BnaBES1_A06R, 同时使用SMART和MOTIF分析结构域发现, 3条BnaBZR蛋白具有基本相同的结构域, N端表现明显的植物BZR/BES结构特征序列, C端结构差异相对明显; 全长蛋白序列与pfam05687、PLN02905、cd00609等结构域相似度高, 3条蛋白序列中均存在多个激酶特征域, 可能参与磷酸化、酰基化, 蛋白本身又具丰富的磷酸化位点, 这与BZR/BES蛋白可以通过结合并相互磷酸化进行构象调控介导核质穿梭的功能一致(图3)。
通过不同数据库, 共获得70种来源于不同植物的BZR相关蛋白序列, 使用MEGA6.0进行聚类分析, 绘制进化树(图4)。
BZR/BES相关蛋白聚类表现出了明显单、双子叶植物差异, 十字花科植物BZR/BES蛋白明显聚于同一亚群。对十字花科BZR/BES相关蛋白亚群分析可以发现, BZR1及其相似蛋白明显聚类, BZR2与其相似蛋白明显聚类, 蛋白类别之间的差异远大于物种差异, 同一亚类蛋白在不同的十字花科植物中相似度极高, 分枝距离极近, 然而, 甚至在同一物种中BZR1与BZR2亚群的分枝距离都较远, 相似度相对较低, 表明BZR1与BZR2的分化是一个早期进化事件。但物种亲缘关系极远的单双子叶植物中BZR/BES蛋白同样具有较高的相似度, 较近的亲缘关系, 表明BZR/BES蛋白家族在进化中起源古老且高度保守, 侧面显示BZR/BES家族蛋白可能具有能影响植物生存的重要作用。
2.3 甘蓝型油菜中BnaBZR1时空表达特征
为进一步了解在甘蓝型油菜中的功能, 对甘蓝型油菜不同时期, 不同组织中、和表达进行了分析, 三基因定量引物为 BZR1_A07-F: 5'-CTCTC ATCTCCAACTTCCAA-3', BZR_A07-R: 5'-GACTC ATCACACTCAGGTAT-3'; BES1_A06F-F: 5'-TTCTC CGACCTTCAACCTA-3', BES1_A06F-R: 5'-TCCTC CATAGCCACATCAT-3¢; BES1_A06R-F: 5'-GGCTACTATACCTGAATGTGA-3', BES1_A06R-R: 5'-GCT GTTGCTGTTGTGAGA-3'; 内参引物为BnUBC21- F: 5'-CCTCTGCAGCCTCCTCAAGT-3', BnUBC21-R: 5'-TATCTCCCCTGTCTTGAAATGC-3'。
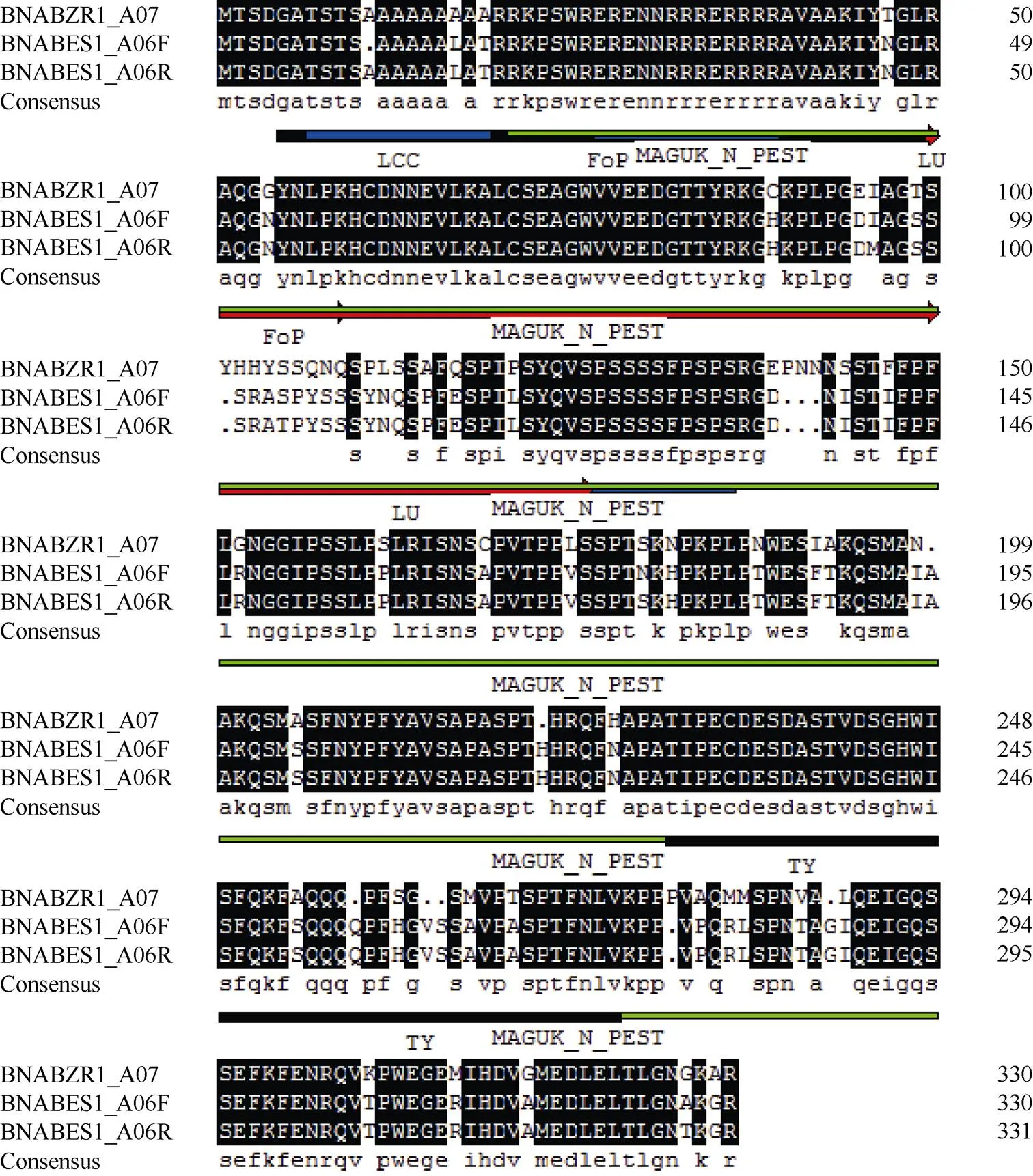
图3 BnaBZR氨基酸序列比对
由图5可知, 湘油15号中、和具有相似的表达模式, 基因表达均表现一定的组织差异和一定的生长发育时间差异, 在茎叶花果等地上部分表达比地下部分高, 苗期、花期表达比抽薹期和角果成熟期高。在湘油15号根中的表达在各个生育时期均相对较低且平稳, 在茎叶中表达水平较高, 但在角果成熟过程中茎叶中表达水平显著下降, 同时在花中可以发现高水平的表达。在湘油15号中相对于整体表现较低表达水平, 茎叶与根中表达水平的差异相对较小。在花期和表达水平达到峰值, 根中表达水平随发育时期表现较明显的差异。植物各组织均有油菜素内酯合成能力, 但地上部分组织中油菜素内酯含量高于地下部分, 尤其是花、种子和嫩叶中含量较高,、和在甘蓝型油菜中表现的组织差异与此具有相似规律, 这也与油菜素内酯一方面能通过一系列信号转导促进BZR/BES活化, 同时BZR/BES又能对油菜素内酯生物合成形成负反馈的规律吻合, 这一定程度上表明甘蓝型油菜中、和与油菜素内酯信号具有紧密的关联性。
3 讨论
湘油15号是我国第一个通过国家审定并大面积推广的双低油菜品种, 是油菜育种中应用非常广泛的重要种质资源, 但湘油15号未经重测序, 大量基因并未被克隆和研究, 一定程度上制约了种质资源的有效利用。本文从甘蓝型油菜湘油15号中克隆获得3个基因拷贝, 测序比对显示与油菜基因组测序品种中双11中序列一致, 各自编码的BnaBZR蛋白的生物信息学分析显示其具有典型的植物BZR/BES功能结构域, 亚细胞定位于细胞核, 具有多重转录因子功能的核心元件序列, 存在大量磷酸化, 酰基化功能位点, 且本身具有磷酸化酶活性域, 表明基因是重要的涉及磷酸化调控的转录因子, 这与拟南芥[11]、水稻[12]等植物中BZR1/BES1通过形成复合体并相互磷酸化, 介导与PIF4、SMOS1等转录因子的互作, 并调控下游基因表达一致。

图4 不同植物BZR/BES蛋白的进化树分析
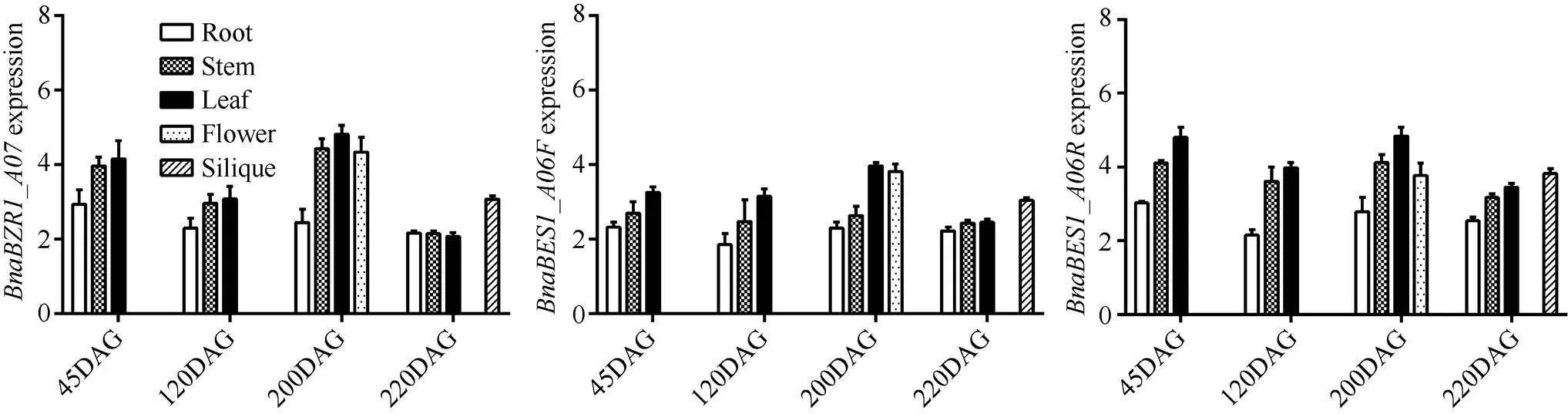
图5 不同发育时期不同组织中BnaBZR1_A07、BnaBES1_A06F和BnaBES1_A06R的表达量
DAG表示种子萌发后天数。DAG means days after seed germination.
聚类分析显示, BZR/BES蛋白在进化上具有较高的保守性, BnaBZR1_A07与白菜、甘蓝、拟南芥等植物中BZR1及相似蛋白具有高度同源性, BnaBES1_A06F/R则与白菜、甘蓝、拟南芥等植物中BZR2及相似蛋白高度同源, 同时, 十字花科各近缘种中BZR1与BZR2间分枝距离相对较远, 表明BZR1与BZR2的分化可能先于物种分化, 但进化上又高度保守。另对甘蓝型油菜BZR/BES相关基因染色体分布分析发现A、C亚基因组中BZR/BES出现较平衡, 这显示了甘蓝型油菜A、C亚基因组在进化关系上仍然具有较高的同源性, 也间接证实“禹氏三角”中白菜与甘蓝的近缘关系[13]。
油菜素内酯途径已在多种植物中被证明与植物抗病、种子形成等生物学过程关系密切[14], 油菜素内酯在油菜[15]、水稻[16]、番茄[17]等多种作物中被证实可以用来提高抗逆性, 并有油菜素内酯施用增加玉米[18]、番茄等茄科作物[19]产量的报告。因此阐明甘蓝型油菜中油菜素内酯信号的转化、传递对油菜育种和生产具有重要意义。BZR/BES是油菜素内酯信号与IAA、GA、光信号等多种信号途径互作的节点[20], 是油菜素内酯信号调控植物生长发育的关键转录因子, 对其克隆、表达研究对阐释油菜素内酯信号在油菜中的作用具有重要意义。本文克隆获得3个全长CDS, 分别定位在A07、A06、A06染色体上, 序列相似度较高, 这显示基因起源较早且保守, 在原始的芸薹属植物中即已存在且具有重要功能。其中和同处在A06染色体端部相距不足10 kb的区域内且二者序列相似性超过98%, 表明这很可能是一次基因重复造成。
对湘油15中、和表达研究显示, 三者具有基本一致的表达规律, 表明基因在甘蓝型油菜中可能具有相同或者相似的上游调控因子, 同时可能受到相同或者相似的环境因素、植物内环境等影响。对甘蓝型油菜中基因的表达调控、基因功能有待进一步研究。
4 结论
从甘蓝型油菜湘油15号cDNA中克隆到3个全长编码序列(coding sequence, CDS), 分别定位于A07、A06、A06号染色体, 分别命名为和。序列长分别为996、993、996 bp, 各自编码331、330和331个氨基酸。该基因编码蛋白与其他植物BZR/ BES具有较高相似性, 具有典型的BZR/BES结构域, 其起源较早, 但保守性较高。分析了和在湘油15号中的时空表达模式。
[1] Wang Z Y, Bai M Y, Oh E, Zhu J Y. Brassinosteroid signaling network and regulation of photomorphogenesis., 2012, 46: 701–724
[2] Gallego-Bartolomé J, Minguet E G, Grau-Enguix F, Abbas M, Locascio A, Thomas S G, Blázquez M A. Molecular mechanism for the interaction between gibberellin and brassinosteroid signaling pathways in., 2012, 109: 13446–13451
[3] Peleg Z, Blumwald E. Hormone balance and abiotic stress tolerance in crop plants., 2011, 14: 290–295
[4] Wang W, Bai M Y, Wang Z Y. The brassinosteroid signaling network: a paradigm of signal integration., 2014, 21: 147–153
[5] Sun Y, Fan X Y, Cao D M, Tang W, He K, Zhu J Y, Patil S. Integration of brassinosteroid signal transduction with the transcription network for plant growth regulation in., 2010, 19: 765–777
[6] Tong H, Chu C. Brassinosteroid signaling and application in rice., 2012, 39: 3–9
[7] Efimova M V, Savchuk A L, Hasan J A K, Litvinovskaya R P, Khripach V A, Kholodova V P, Kuznetsov V V. Physiological mechanisms of enhancing salt tolerance of oilseed rape plants with brassinosteroids., 2014, 61: 733–743
[8] Skoczowski A, Janeczko A, Gullner G, Tóbias I, Kornas A, Barna B. Response of brassinosteroid-treated oilseed rape cotyledons to infection with the wild type and HR-mutant ofor with P. fluorescence., 2011, 104: 131–139
[9] Pokotylo I V, Kretynin S V, Khripach V A, Ruelland E, Blume Y B, Kravets V S. Influence of 24-epibrassinolide on lipid signalling and metabolism in., 2014, 73: 9–17
[10] Sahni S, Prasad B D, Liu Q, Grbic V, Sharpe A, Singh S P, Krishna P. Overexpression of the brassinosteroid biosynthetic geneinsimultaneously increases seed yield and stress tolerance., 2016, 6: 28298
[11] Lachowiec J, Mason G A, Schultz K, Queitsch C. Redundancy, feedback, and robustness in theBZR/BEH gene family., 2016: 053447
[12] Qiao S, Sun S, Wang L, Wu Z, Li C, Li X, Wang X. The RLA1/SMOS1 transcription factor functions with OsBZR1 to regulate brassinosteroid signaling and rice architecture., 2017, 29: 292–309
[13] Surhone L M, Timpledon M T, Marseken S F. Rapeseed. Germany: Betascript Publishing, 2010. pp 6–8
[14] Hao J, Yin Y, Fei S. Brassinosteroid signaling network: implications on yield and stress tolerance., 2013, 32: 1017–1030
[15] 李玲, 李俊, 张春雷, 张树杰, 马霓, 李光明. 外源 ABA 和 BR 在提高油菜幼苗耐渍性中的作用.中国油料作物学报, 2012, 34: 489–495 Li L, Li J, Zhang C L, Zhang S J, Ma L, Li G M. Effects of exogenous ABA and BR on waterlogging resistance of juvenile rapeseed., 2012, 34: 489–495 (in Chinese with English abstract)
[16] Yang D L, Yang Y, He Z. Roles of plant hormones and their interplay in rice immunity., 2013, 6: 675–685
[17] Zheng Q, Liu J, Liu R, Wu H, Jiang C, Wang C, Guan Y. Temporal and spatial distributions of sodium and polyamines regulated by brassinosteroids in enhancing tomato salt resistance., 2016, 400: 147–164
[18] 王庆燕, 管大海, 潘海波, 李建民, 段留生, 张明才, 李召虎. 油菜素内酯对春玉米灌浆期叶片光合功能与产量的调控效应.作物学报, 2015, 41: 1557–1563 Wang Q Y, Guan D H, Pan H B, Li J M, Duan L S, Zhang M C, Li Z H. Effect of brassinolide on leaf photosynthetic function and yield in spring maize filling stage., 2015, 41: 1557–1563 (in Chinese with English abstract)
[19] Hayat S, Alyemeni M N, Hasan S A. Foliar spray of brassinosteroid enhances yield and quality ofunder cadmium stress., 2012, 19: 325–335
[20] Fridman Y, Savaldi-Goldstein S. Brassinosteroids in growth control: how, when and where., 2013, 209: 24–31
Cloning and Characterization of Brassinazole-resistant (BnaBZR1 and BnaBES1) CDS fromL.
FENG Tao and GUAN Chun-Yun*
College of Agronomy, Hunan Agricultural University / National Oilseed Crops Improvement Center in Hunan, Changsha 410128, Hunan, China
Brassinazole-resistant (BZR) is a key transcription factor in brassinosteroid signaling pathway of plants, containing brassinazole-resistant 1 (BZR1) and brassinazole-resistant 2 (BES1). In this study, three novel BZR1 coding sequences (CDSs) were isolated from cDNA ofL. cv. Xiangyou 15 leaves, which were mapped on the chromosomes A07, A06, and A06, and designated as,, and, respectively. These threeCDSs were 996, 993, and 996 bp in length and encoded predicted proteins with 331, 330, and 331 amino acid residues, respectively. BnaBZR proteins were predicted to be located on the cell nucleus and have a typical plant BZR/BES conserved domain. Multiple sequence alignments and phylogenetic analysis showed that the deduced amino acid sequences of BnaBZR were highly homologous to previously reported BZR/BES of,, and. And the similarity of BZR1 or BES1 among different related species was higher than the similarity between BZR1 and BES1 in the same species or related species, indicating that BZR1 and BES1 differentiation is an early evolutionary event. The expression patterns of,, andin Xiangyou 15 were similar, whit high expression level at the seedling stage and flowering stage, while slightly lower level at the stage of bolting and podgrain ripening. The expression level ofin the aerial parts such as leaves, stems, flowers and pods was higher than that in underground parts.
L.; BZR; gene clone; gene expression; bionformatic analysis
2018-05-03;
2018-08-20;
2018-09-19.
10.3724/SP.J.1006.2018.01793
通信作者(Corresponding author):官春云, E-mail: guancy2011@aliyun.com
E-mail: 812298771@qq.com
本研究由国家重点基础研究发展计划(973计划)项目(2015CB150206)资助。
This study was supported by the National Basic Research Program (973 Program) (2015CB150206).
URL:http://kns.cnki.net/kcms/detail/11.1809.s.20180917.1543.004.html
