链状亚历山大藻铜胺氧化酶基因的克隆和表达分析❋
2018-10-15商二磊隋正红刘昊昕
商二磊, 隋正红, 刘 源, 米 萍, 刘昊昕
(中国海洋大学海洋生物遗传学与育种教育部重点实验室,山东 青岛 266003)
多胺是一类广泛存在于植物中的脂肪族多聚阳离子化合物[1-3],为生物正常生长发育所必须,在植物的发育和生理过程中扮演着十分重要的角色,如调控细胞增殖、种子萌发和衰老[4-7]。细胞中多胺的平衡受包括铜胺氧化酶(Copper-containing amine oxidase, CuAO)在内的多胺氧化酶调控[1]。CuAO是一类重要的多胺氧化酶,存在于细菌、真菌、植物和动物细胞中[8],主要催化多胺伯氨基的氧化,生成对应的氨基醛、过氧化氢(H2O2)和NH3,如催化腐氨(putrescine, Put)生成4-氨基丁醛、H2O2和NH3[2, 9-10]。作为多胺的氧化产物之一,H2O2参与细胞的程序性死亡过程[2, 11-12]。铜胺氧化酶在植物的生长发育[1]、应对生物和非生物胁迫上都发挥着重要的作用[1-2]。
赤潮是一种有害的生态异常现象,在消亡阶段赤潮生物大量死亡,赤潮生物机体分解需要消耗水体的溶解氧,导致其它生物缺氧死亡,破坏海洋的生态平衡。另外,衰亡期的赤潮生物因死亡而释放的有毒代谢产物也影响海洋生态系统和人类的健康。所以赤潮消亡机理的研究有着重要的意义。很多研究认为,赤潮消亡是一种细胞程序性死亡的过程[13-16]。链状亚历山大藻(Alexandriumcatenella)是一种常见的甲藻,是引发中国近岸赤潮的种类之一,由其引起的赤潮也广泛分布于世界各海域。链状亚历山大藻能产生麻痹性贝毒,该毒素能被贝类积累,通过食物链最终影响人类的健康[17]。目前有关由链状亚历山大藻引发赤潮衰亡过程中调控机制的研究仍不清楚。
本研究的目的是通过链状亚历山大藻中铜胺氧化酶基因的克隆及目的基因在该藻不同生长时期的表达研究,初步探究铜胺氧化酶基因在链状亚历山大藻生长过程中的表达模式,为进一步研究该藻赤潮衰亡过程中的细胞生理变化与调控机制提供基础资料。
1 材料和方法
1.1 材料的培养
链状亚历山大藻,保存于中国海洋大学藻种室。在无菌f/2培养基中培养,温度(20±1) ℃,光暗周期12 h/12 h,光照强度30 μmol·m-2·s-1[18]。将培养至对数期的链状亚历山大藻进行同步化处理[19],即将藻置于黑暗下48 h,随后将处理过的藻细胞以2×106cells/L的密度接种至无菌f/2培养基中,在如上条件下培养。在延迟期(第2天)、对数期(第7天)和衰亡期(第23天)进行取样。
1.2 实验方法
1.2.1 核酸的提取和cDNA的制备 将含约5×106个细胞的藻液通过孔径为10 μm的无菌筛绢,然后用400 mL无菌f/2培养基冲洗,将藻细胞收集于1.5 mL EP管中,保存于-80 ℃冰箱备用。使用植物基因组DNA提取试剂盒(TIANGEN)和RNAiso Plus试剂盒(TaKaRa)按操作说明分别提取链状亚历山大藻细胞的DNA和总RNA。对提取的RNA,先用DNase I(TaKaRa)消化DNA,用1.5% 的琼脂糖凝胶电泳检测完整性,再用Nano Drop 2000检测其浓度和质量,选择A260/A230 在2.0~2.2,A260/A280在1.8~2.0之间的RNA用于cDNA合成。使用反转录试剂盒(TaKaRa)按照操作说明制备cDNA。
1.2.2 目的基因的克隆及生物信息学分析 根据实验室已有的链状亚历山大藻转录组数据(SRX368254)的注释结果,从中检索出两个编码CuAO的unigene基因,分别将其命名为cad-1(登录号KY930889)和cad-2(登录号KY930890)。基于转录组中cad-1和cad-2的序列,使用Primer Premier 5.0软件设计ORF全长扩增引物,引物信息见表1。以DNA和cDNA分别作模板进行PCR扩增,cad-1以cad-1fs/cad-1fa为引物,使用LA Taq DNA聚合酶(TaKaRa),反应程序:94 ℃ 10 min;94 ℃ 30 s,62 ℃ 30 s,72 ℃ 3 min,30个循环;72 ℃ 10 min。cad-2以cad-2fs/cad-2fa为引物,使用Gflex DNA聚合酶(TaKaRa),反应程序:94 ℃ 1 min;94 ℃ 10 s,60 ℃ 15 s,68 ℃ 3 min,30个循环;68 ℃ 10 min。PMD19-T作为连接载体,对目的条带进行亚克隆,挑取阳性菌落进行PCR验证后,送青岛擎科梓熙生物技术有限公司测序。
使用NCBI数据库ORF finder预测基因开放阅读框;使用在线工具Protparam预测蛋白的理化性质;使用Predictprotein软件预测蛋白的二级结构;使用在线分析工具NetPhos 2.0预测蛋白的磷酸化位点;使用TMHMM 2.0软件预测蛋白的跨膜结构域;使用Clustal W软件做氨基酸多序列比对;使用MEGA 6.0以邻位连接法(NJ)构建系统进化树。
1.2.3 荧光定量PCR 使用荧光定量PCR技术检测cad-1和cad-2在链状亚历山大藻不同生长时期的表达水平。cad-1和cad-2荧光定量PCR的扩增引物分别为qp-cad-1S/qp-cad-1A和qp-cad-2S/qp-cad-2A;gapdh和actin为内参基因,扩增引物分别为gapdhS/gapdhA和actinS/actinA。使用SYBR I试剂盒(Roche)在Light Cycler®480ⅡPCR荧光定量仪上进行扩增,扩增体系为20 μL,其中cDNA 1 μL,SYBR I 10 μL,正反引物的终浓度为0.5 μmol/L,剩余体积用无菌ddH2O补充,每个样品设置3个生物学重复和3个技术重复。反应程序为95 ℃ 5 min,1个循环;95 ℃ 10 s,60 ℃ 10 s,72 ℃ 20 s,40个循环。基因的相对表达量用2-ΔΔCT法[20]进行计算。
1.2.4 数据分析 本实验中每组处理均设置3个平行样。使用统计分析软件SPSS 21.0对实验数据进行单因素方差分析(One-way ANOVA),显著性差异水平P≤0.05。
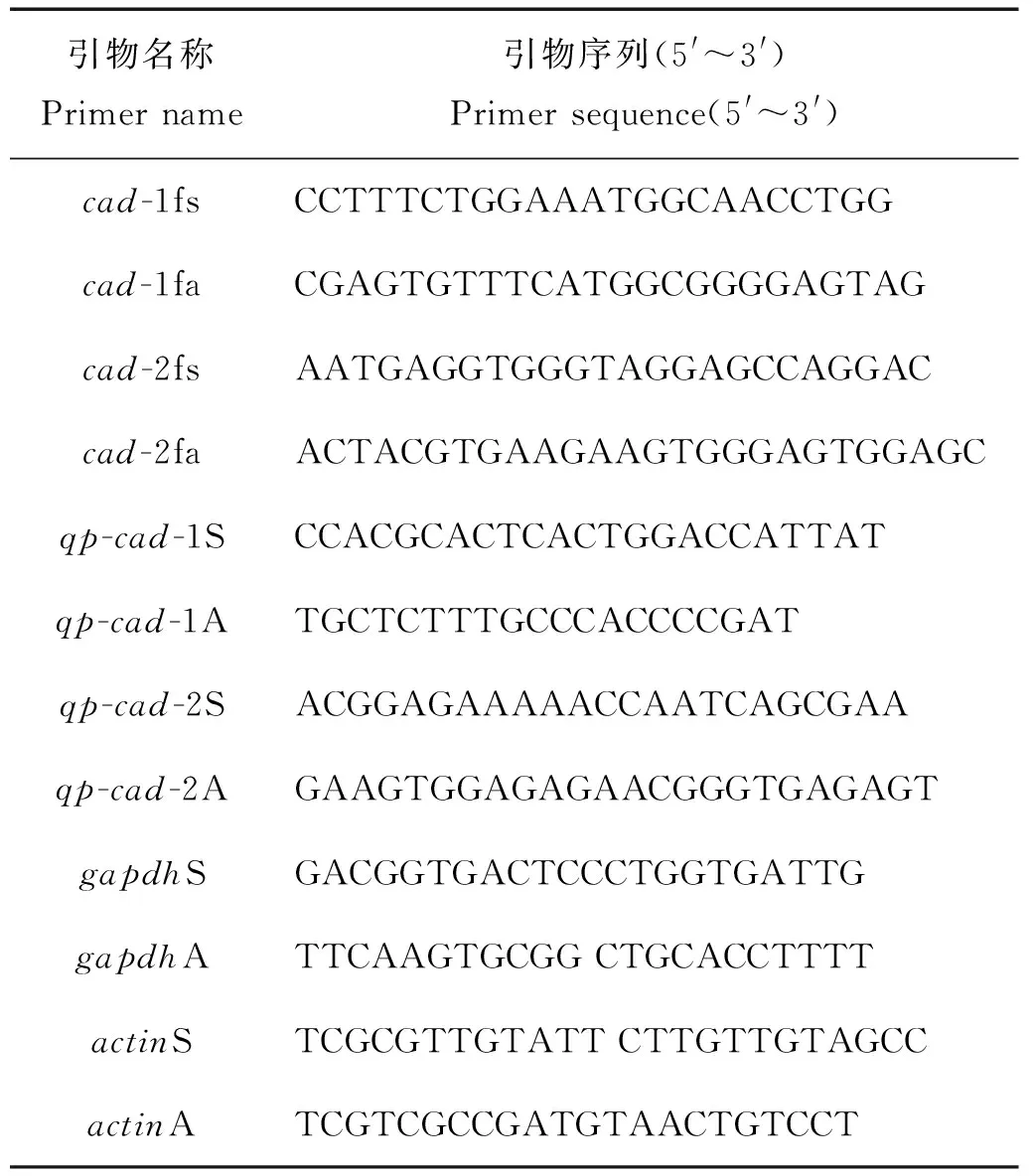
表1 PCR使用的引物信息
2 结果与分析
2.1 基因的克隆和测序
根据ORF finder的预测结果,设计引物进行PCR扩增、测序验证。电泳结果见图1。 以cDNA为模板对cad-1进行扩增,获得长度为1 068 bp序列,开放阅读框长度为1 023 bp;以DNA为模板扩增所得的序列与cDNA一致。以cDNA为模板对cad-2进行扩增,获得长度为1 802 bp序列,开放阅读框长度为1 800 bp;而以DNA为模板扩增所得序列长度为2 867 bp,比对分析发现,该序列上除含有开放阅读框外,还有一个1 065 bp的内含子。cad-2DNA和cDNA序列比较图2。cad-1和cad-2 ORF全长的比较见图3,它们有一个较为保守的重叠区,重叠区核酸的比对见图4,有11个核苷酸的差异。
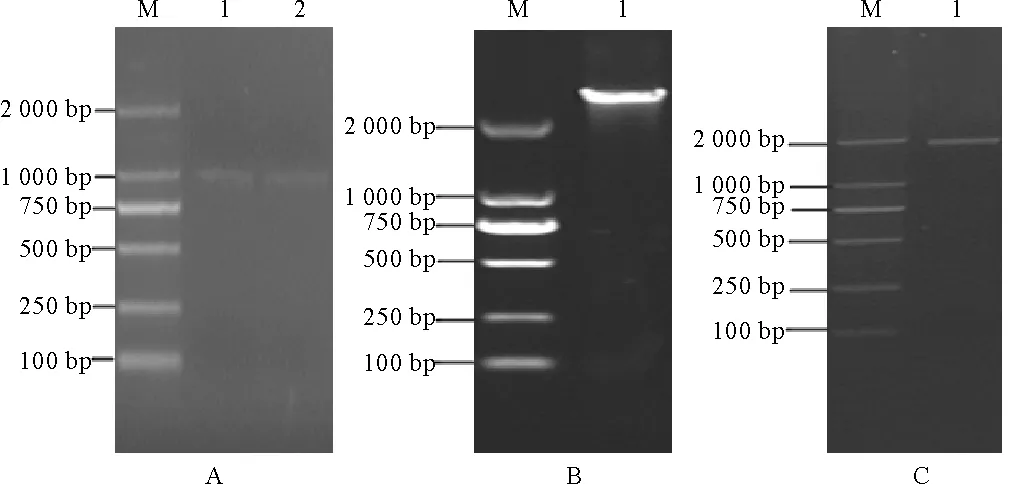
(M泳道:DL 2000 marker;A:泳道1和2是分别是以DNA和cDNA为模板扩增cad-1得到的条带;B:泳道1是以DNA为模板扩增cad-2得到的条带;C:泳道1是以cDNA为模板扩增cad-2得到的条带。M: DL 2000 marker; A: line1 and 2 mean bands that amplified from template DNA and cDNA forcad-1 respectively; B: line1 means band that amplified from template DNA forcad-2; C: line1 means band that amplified from template cDNA forcad-2.)
图1cad-1和cad-2全长序列扩增的电泳图
Fig.1 Amplification ofcad-1 andcad-2

图2 DNA和cDNA上cad-2开放阅读框全长的比较

图3 cad-1和cad-2 ORF全长序列的比较

图4 cad-1和cad-2重叠区核酸序列的比对
2.2 基因序列的生物信息学分析
序列分析发现,cad-1编码340个氨基酸;ProtParam软件预测发现,其编码蛋白质(Cad-1)的分子式为C1661H2589N487O499S9,分子量为37.7 kDa,理论等电点为5.96;在线软件NetPhos 2.0预测发现,蛋白质磷酸化位点共15个(丝氨酸位点8个、苏氨酸位点7个);Predictprotein预测蛋白质二级结构显示, Cad-1由α螺旋(3.82%)、β折叠(31.47%)、和无规则卷曲(64.71%)组成。TMHMM预测结果显示, Cad-1无跨膜域。
cad-2编码599个氨基酸,其编码的蛋白质(Cad-2)分子式为C2871H4451N801O848S22,分子量为64.5 kDa,理论等电点为5.75; Cad-2的磷酸化位点共29个(丝氨酸位点19个、苏氨酸位点7个、酪氨酸位点3个);蛋白质二级结构预测显示, Cad-2由α螺旋(12.69%)、β折叠(26.38%)和无规则卷曲(60.93%)组成。TMHMM预测结果显示, Cad-2的1~34位氨基酸在膜内,35~57位氨基酸形成一个跨膜螺旋,58~599位氨基酸位于膜外。
不同物种的CuAO氨基酸序列Clustal W比对结果见图5。不同物种CuAO氨基酸序列的相似性不高,但具有一个共同的保守结构域“NYDY”,由Asn-Tyr-Asp-Tyr四个氨基酸残基组成,且均含有3个组氨酸残基。NCBI blastp比对发现,Cad-1和Cad-2与颗石藻Emilianiahuxleyi(XP_005787067.1)铜胺氧化酶的Identities 分别为37%和34%,Positives分别为51%和49%。

(酸杆菌Acidobacteriabacterium(AMY12828.1);拟南芥Arabidopsisthaliana(NP_174452.2);水稻Oryzabrachyantha(XP_006659924.1);高粱Sorghumbicolor(XP_002461776.1);莱茵衣藻Chlamydomonasreinhardtii(XP_001693748.1);颗石藻Emilianiahuxleyi(XP_005787067.1。虚线方框为蛋白的保守域“NYDY”,实线方框为三个组氨酸保守位点,“*”表示完全保守的位点,“:”表示保守性较高的位点,“.”表示保守性较低的位点。Red square indicate the conserved active site “NYDY” of CuAO proteins, blue squares indicate the conservative sites of three histidine residues, “*” indicate full conserved site, “:” indicate high conserved site, “.” indicate low conserved site.)
图5 CuAO蛋白部分氨基酸序列比对结果
Fig.5 The result of amino acid sequence alignment of CuAO proteins
为了分析链状亚历山大藻的CuAO与其它物种相关蛋白的进化关系,选取14个物种CuAO的氨基酸序列,以亚硝化侏儒菌Nitrosopumilus为外类群,使用MEGA 6.0构建基于NJ法的系统进化树(见图6)。进化树分为2支,链状亚历山大藻中的Cad-1和Cad-2先与颗石藻和抑食金球藻聚为一小支,而后与圆柱拟脆杆藻和莱茵衣藻聚为一支,表明链状亚历山大藻与颗石藻和抑食金球藻铜胺氧化酶的亲缘关系较近;其它绿藻、红藻、蓝藻、微拟球藻和玉米聚为一支。
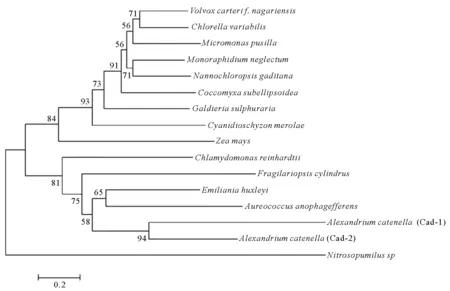
(团藻Volvoxcarterif.nagariensis(XP 002959097.1);小球藻Chlorellavariabilis(XP 005851907.1);微胞藻Micromonaspusilla(XP_003057454.1);单针藻Monoraphidiumneglectum(XP 013904686.1);微拟球藻Nannochloropsisgaditana(EWM22333.1);胶球藻Coccomyxasubellipsoidea(XP 005651923.1);温泉红藻Galdieriasulphuraria(EME27392.1);单细胞红藻Cyanidioschyzonmerolae(BAM83045.1);玉米Zeamays(XP 008663089.1);莱茵衣藻Chlamydomonasreinhardtii(XP 001693748.1);圆柱拟脆杆藻Fragilariopsiscylindrus(OEU12233.1);颗石藻Emilianiahuxleyi(XP 005787067.1);抑食金球藻Aureococcusanophagefferens(EGB06404.1);链状亚历山大藻Cad-1和Cad-2;亚硝化侏儒菌Nitrosopumilussp(WP_050555097.1))
图6 CuAO氨基酸序列的系统进化分析
Fig.6 The phylogenetic tree of amino acid sequence of CuAO
2.3 基因cad-1和cad-2的表达
因两目的基因间存在一段较为保守的重叠区,故将两对荧光定量PCR引物设计在非重叠区内。使用荧光定量PCR技术对不同生长时期链状亚历山大藻中cad-1和cad-2在RNA水平上的相对表达量进行分析,延迟期作为对照,计算其它取样点基因的相对表达量,结果见图7。在链状亚历山大藻中,对数期cad-1的表达量是延迟期的2.54倍;衰亡期的表达量是延迟期的8.43倍;cad-2的相对表达量和cad-1有着相似的变化,对数期相比延迟期下调2.98倍,衰亡期比延迟期上调14.92倍。
3 讨论
CuAO是细胞中一类重要的多胺氧化酶,存在于原核生物和真核生物中。研究表明,CuAO参与很多生命过程,如植物的生长、发育以及应对生物和非生物胁迫[1-2, 21-22]。Rea等研究发现[23],鹰嘴豆(Cicerarietinum)组织的损伤能引起CuAO基因表达水平的增加。An等和吴梦瑶等都发现,蚕豆(Viciafaba)保卫细胞中CuAO参与气孔关闭[24-25]。也有研究发现,在胁迫等条件下,多胺氧化酶催化多胺氧化产生的过量过氧化氢(H2O2)参与细胞程序性死亡[11-12, 26]。
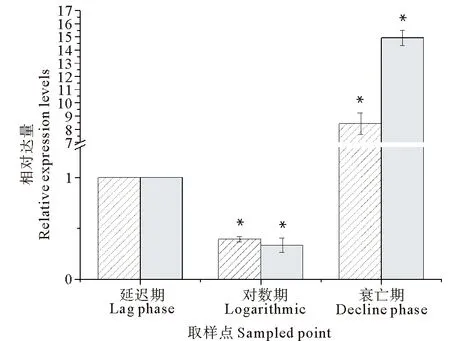
(*表示其它取样点相对延迟期有显著性差异。Asterisk indicate significant difference.)
图7 不同生长时期无菌链状亚历山大藻中基因的相对表达量
Fig.7 The gene relative expressions at different growth stages of sterileAlexandriumcatenella
本研究中,从链状亚历山大藻中克隆了两个铜胺氧化酶基因,分别为cad-1和cad-2。cad-1 ORF全长1 023 bp;cad-2 ORF全长1 800 bp,含一个1 065 bp的内含子。cad-1和cad-2有一段重叠区,但有11个核苷酸的差异,表明它们是两条不同的基因。Cad-1和Cad-2与其它物种铜胺氧化酶同源比对差异较大,是因为选择的物种遗传差异较大,且Cad-1和Cad-2同时进行比对。blastp比对发现,Cad-1和Cad-2与颗石藻E.huxleyi铜胺氧化酶的Identities 分别为37%和34%,Positives分别为51%和49%;Cad-1和Cad-2 均含有铜胺氧化酶的活性位点“NYD”[27]和可能与铜离子结合相关的3个组氨酸残基[8]。跨膜预测结果发现,Cad-1和Cad-2的“NYD”结构域均位于膜外,推测链状亚历山大藻铜胺氧化酶的功能域可能在膜外。序列的比对结果表明,cad-1和cad-2是从基因组的不同位置转录的,同一种催化酶对应着两条序列不同的编码基因,且结构分析表明两蛋白有不同的膜定位特征,两基因在细胞生长过程中的表达模式也有区别,这也印证了链状亚历山大藻基因组的复杂性,提示了基因表达调控的复杂性。系统发生分析结果显示, Cad-1和Cad-2先与颗石藻和抑食金球藻铜胺氧化酶聚为一小支,再与圆柱拟脆杆藻和莱茵衣藻铜胺氧化酶聚为一支,其它绿藻、红藻、蓝藻、微拟球藻和玉米聚为一支,表明CuAO的进化与物种进化关系基本一致。
Moschou等[26]研究发现,在多胺氧化酶过表达的烟草中,增加的H2O2引发了严重的细胞程序性死亡;而在多胺氧化酶下调表达的烟草中,植物体H2O2的含量较少,表现出很少的细胞程序性死亡。Hiroshi等[11]发现,植物中多胺氧化酶是参与细胞程序性死亡的关键酶。有研究表明,赤潮消亡是一种细胞程序性死亡的过程[13-16]。马金华等从细胞生理状态变化的角度研究发现,衰亡期的链状亚历山大藻可溶性蛋白质含量下降,超氧化物岐化酶活性显著增加,丙二醛、H2O2含量增加,且出现了明显的DNA ladder,表明链状亚历山大藻赤潮的衰亡是一种细胞程性序死亡的过程[28]。本文研究了铜胺氧化酶基因在链状亚历山大藻不同生长时期表达量的变化。结果显示,相比延迟期,cad-1的相对表达量在对数期显著下调,在衰亡期显著上调;cad-2在不同时期的相对表达量与cad-1一致。综合马金华等[28]的结果发现(对数期链状亚历山大藻细胞H2O2含量较低,衰亡期H2O2含量显著增加)和本文的实验结果可见,链状亚历山大藻铜胺氧化酶基因的表达量在对数期显著下降,细胞H2O2含量较低,衰亡期铜胺氧化酶基因的表达量显著增加,细胞H2O2含量也显著增加,这与Moschou等[26]的发现一致。本文实验结果提示,铜胺氧化酶可能参与了链状亚历山大藻衰亡过程的调控。
4 小结
本文从链状亚历山大藻中克隆了两个铜胺氧化酶的基因cad-1和cad-2,cad-1的开放阅读框全长为1 023 bp,编码340个氨基酸;cad-2开放阅读框全长为1 800 bp,编码599个氨基酸。两个基因编码的蛋白含有保守结构域“NYDY”。进化分析发现,链状亚历山大藻与颗石藻和抑食金球藻的CuAO亲缘关系较近。cad-1和cad-2在对数期和衰亡期分别下调和上调表达,依据铜胺氧化酶的功能和与链状亚历山大藻衰亡相关的生理研究,推测铜胺氧化酶可能参与链状亚历山大藻赤潮衰亡过程的调控。
