miR-133b通过靶向调控EGFR影响食管鳞癌的侵袭、转移
2018-07-04朱金峰孔德华
曾 薇, 朱金峰,孔德华,单 莉
食管鳞癌的侵袭、转移是多因素的综合作用,是多基因变化积累、叠加的结果。随着miRNA的生成及功能异常在多种肿瘤发生、发展中的作用被逐渐认识[1],miRNA在食管鳞癌侵袭、转移中的机制研究越来越受到人们的关注[2]。miRNA长约20~24 nt,主要通过转录后或翻译水平调控基因表达[3]。目前研究[4]显示,在食管鳞癌组织中低表达的miR-133b可能作为抑癌基因,参与食管鳞癌的演进,并预测 miR-133b 可能作为食管鳞癌治疗的潜在靶点。该研究将对miR-133b参与食管鳞癌侵袭、转移的具体作用及机制做进一步的研究讨论。
1 材料与方法
1.1材料
1.1.1细胞系 人食管鳞癌细胞系ECA109、KYSE150购自ATCC细胞库。所有细胞为90% RPMI 1640培养基加10%胎牛血清,于37 ℃、5% CO2、饱和湿度的培养箱中培养。DMEM 培养基用于Transwell 实验。
1.1.2组织标本 收集2008年1月~2010年12月新疆医科大学附属肿瘤医院胸外科手术切除的食管鳞癌组织标本,包括肿瘤及与其配对的正常食管黏膜组织。所有患者术前未接受化疗、放疗或其他任何相关的抗肿瘤治疗。根据UICC TNM (第七版)分期。所有临床标本经过新疆医科大学附属肿瘤医院病理科专家诊断证实。临床标本的切取均通过新疆医科大学附属肿瘤医院伦理委员会批准,并征得患者的同意签署知情同意书。
1.2方法
1.2.1运用生物信息学方法预测miR-133b的靶基因 运用MiRbase、Targetscans及PicTar数据库预测 miR-133b的候选靶基因。
1.2.2细胞转染 转染前1 d,细胞生长密度达70%~80%。转染当天,使用250 μl无血清Opti-MEM I培养基稀释miR-133b mimic/negative control 4.0 μg,轻轻混匀,室温下静置5 min。同时将LipofectamineTM2000 10 μl稀释于250 μl无血清Opti-MEM I培养基中,轻轻混匀,室温下静置5 min。分别混合LipofectamineTM2000和miR-133b mimic/miR-133b negative control的稀释液,室温下静置20 min。6孔板的每个孔中加入混合液500 μl。4 h后更换细胞培养液,于37 ℃、CO2培养箱中继续培养48 h,用于后续检测。
1.2.3荧光定量PCR技术(qRT-PCR) 查找基因序列,设计引物,见表1。引物由上海生工合成。使用ABI7500型定量PCR仪。TRIzol法提取组织RNA。逆转录反应体系10 μl为:5×PrimerScript RT Master Mix 2 μl、总RNA 500 ng、加无RNase酶的水至10 μl。反应条件为:37 ℃、15 min;85 ℃、5 s;4 ℃、5 min。qRT-PCR反应体系10 μl为:SYBR Premix Ex TaqII(2×)5 μl,上/下游引物各0.5 μl,cDNA 2 μl,RNase Free dH2O 2 μl。反应条件为:95 ℃、30 s;95 ℃、5 s;60 ℃、30 s。每个基因的相对表达量以2-ΔΔCt法计算。

表1 qRT-PCR 实验引物序列
1.2.4Western blot 收集细胞,提取总蛋白,BCA法测定蛋白质浓度,SDS-PAGE胶电泳,将蛋白电转移到PVDF膜上,5%BSA封闭,一抗(1 ∶500)4 ℃孵育过夜,二抗(1 ∶1 000)室温孵育1 h,ECL化学发光显像。
1.2.5荧光素酶报告基因实验 转染前将细胞按70%的密度接种至96孔板。按比例配制报告基因质粒和转染试剂即Firefly:Renilla:转染试剂=0.1 μg:0.01 μg:0.25 μl,并常温下孵育5 min。配制miR-133b mimic/negative control和转染试剂的稀释液:加入转染试剂0.25 μl使miR-133b mimic/negative control的终浓度为100 nmol/L,常温孵育5 min。将稀释好的报告基因质粒、miR-133b mimic/negative control混匀,常温孵育20 min,然后分别添加到每孔细胞样品中。转染6 h后,更换细胞培养基。48 h后弃去培养基,用100 μl 1×PBS洗1遍。每孔加50 μl稀释好的 1×PLB,置于摇床上,常温下15 min进行裂解。在白色不透光的96孔酶标板中每孔加入裂解液的上清液10 μl, 再加入100 μl预先混好的LAR Ⅱ,2 s后测数据,得到萤火虫荧光素酶反应强度。每孔再添加100 μl预先混好的Stop&Glo Reagent,静止2 s后测数据,得到内参海肾荧光素酶反应强度。
1.2.6细胞增殖实验 接种2×105个细胞至96孔板,48 h后加入新鲜配置的MTT溶液,37 ℃、5% CO2培养箱继续孵育4 h后终止培养,每孔加入150 μl二甲基亚砜,酶联免疫检测仪490 nm处测量各孔的光密度(optical density,OD)值。
1.2.7细胞划痕实验 接种2×106个细胞至6孔板,待细胞完全融合后垂直划出等宽的细胞划痕,继续培养48 h,并于相应时间段在显微镜下观察划痕宽度,同时拍照记录。
1.2.8Transwell 实验 ECA109、KYSE150细胞转染24 h后(具体转染步骤详见方法1.2.2),胰酶消化并制备单细胞悬液,以5×104个细胞密度接种于Transwell小室,24孔板下层加入750 μl含10%胎牛血清的DMEM培养基,70 h后计算细胞过膜率。

2 结果
2.1生物信息学分析预测miR-133b的靶基因本研究主要探索miR-133b调控其靶基因参与食管鳞癌侵袭、转移的机制。表皮生长因子受体(epidermal growth factor receptor,EGFR)所参与的信号通路,与肿瘤的侵袭、转移密切相关。故本研究锁定在3个miRNA数据库中miR-133b靶基因预测结果的交集-EGFR,作为miR-133b的预测靶基因进行下一步的研究。见表2。

表2 miR-133b与靶基因EGFR的结合序列
2.2双荧光素酶报告基因实验验证miR-133b的靶基因为证实miR-133b对其靶基因EGFR的调控作用,课题组构建了EGFR野生型及突变型质粒进行双荧光素酶报告基因实验。具体实验分组如下:MT组即插入了EGFR突变型质粒3′UTR片段的萤光素酶报告基因载体,WT组即插入了EGFR野生型质粒 3′UTR片段的萤光素酶报告基因载体,NC组即萤光素酶报告基因载体。在食管鳞癌细胞ECA109、KYSE150中,通过转染miR-133b mimics,检测各组的荧光素酶活性,判断miR-133b对EGFR的调控作用。结果显示:转染miR-133b mimics对MT组和NC组的荧光素酶活性影响不显著,但能使WT组的萤光素酶活性明显下降(ECA109:t=20.24vsNC组,P<0.01,KYSE150:t=14.07vsNC组,P<0.01),提示miR-133b可直接靶向调控EGFR,见图1。

图1 双荧光素酶报告基因实验检测转染miR-133bmimics对各实验组荧光素酶活性的影响
2.3miR-133b对食管鳞癌细胞中EGFR表达的影响采用qRT-PCR、Western blot实验检测miR-133b对EGFR表达的影响。实验分组如下:miR-133b mimics组即在食管鳞癌细胞中转染miR-133b mimics;negative control组即在食管鳞癌细胞中转染negative control。qRT-PCR结果显示,在食管鳞癌细胞ECA109、KYSE150中,与negative control组相比,miR-133b mimics组中miR-133b在转录水平的表达显著上调,提示转染有效(ECA109:t=9.28vsnegative control组,P<0.01; KYSE150:t=35.53vsnegative control组,P<0.01)。但与negative control组相比,miR-133b mimic组中EGFR在转录水平的表达无显著变化,见图2A~D。Western blot结果显示,在食管鳞癌细胞ECA109、KYSE150中,与negative control组相比,miR-133b mimics组中EGFR在蛋白水平的表达发生显著下调,见图2E。
2.4miR-133b抑制食管鳞癌细胞增殖采用MTT实验检测miR-133b对食管鳞癌细胞增殖的影响。实验分组同2.3中描述。结果显示,在食管鳞癌细胞ECA109、KYSE150中,与negative control组相比,72 h时miR-133b mimics组细胞的增殖能力显著下降(ECA109:t=4.18vsnegative control组,P<0.05,KYSE150:t=11.18vsnegative control组,P<0.01),提示miR-133b 能够抑制食管鳞癌细胞的增殖,对肿瘤的生长起抑制作用,见图3。
2.5miR-133b抑制食管鳞癌细胞迁移采用细胞划痕实验检测miR-133b对食管鳞癌细胞迁移能力的影响。实验分组同2.3中描述。结果显示,在食管鳞癌细胞ECA109中,negative control组、miR-133b mimics组细胞划痕闭合率分别为(42.00%±4.36%)、(14.00%±4.58%),差异有统计学意义(t=7.67vsnegative control组,P<0.01);食管鳞癌细胞KYSE150中,negative control组、miR-133b mimics组细胞划痕闭合率分别为(52.33%±5.86%)、(22.00%±4.36%),差异有统计学意义(t=7.19vsnegative control组,P<0.01);上述实验提示,上调miR-133b表达可以抑制两种食管鳞癌细胞的迁移,见图4。
2.6miR-133b抑制食管鳞癌细胞侵袭采用Transwell实验检测miR-133b对食管鳞癌细胞侵袭能力的影响。实验分组同2.3中描述。结果显示,在食管鳞癌细胞ECA109中,negative control组、miR-133b mimics组细胞过膜数分别为(78.67±7.02)、(19.67±3.06),差异有统计学意义(t=14.02vsnegative control组,P<0.01);在食管鳞癌细胞KYSE150中,negative control组、miR-133b mimics组细胞过膜数分别为(87.33±5.77)、(24.67±7.37),差异有统计学意义(t=11.59vsnegative control组,P<0.01);上述实验提示,上调miR-133b表达对两种食管鳞癌细胞的侵袭能力具有抑制作用,见图5。
2.7miR-133b及EGFR在食管鳞癌组织中的表达采用qRT-PCR 实验检测miR-133b与EGFR在33例食管鳞癌组织中的表达,并进行相关性分析。结果显示,miR-133b在食管鳞癌组织中的表达下调,EGFR在食管鳞癌组织中的表达上调,二者表达呈负相关性(P<0.01),见图6。将miR-133b的表达与食管鳞癌患者的临床病理特征进行相关性分析显示:miR-133b的表达与肿瘤的TNM分期、淋巴结转移相关,即miR-133b在Ⅰ/Ⅱ期食管鳞癌中的阳性表达率显著高于Ⅲ期 (P<0.05)、miR-133b在淋巴结阴性食管鳞癌中的阳性表达率显著高于淋巴结阳性食管鳞癌(P<0.05),见表3。
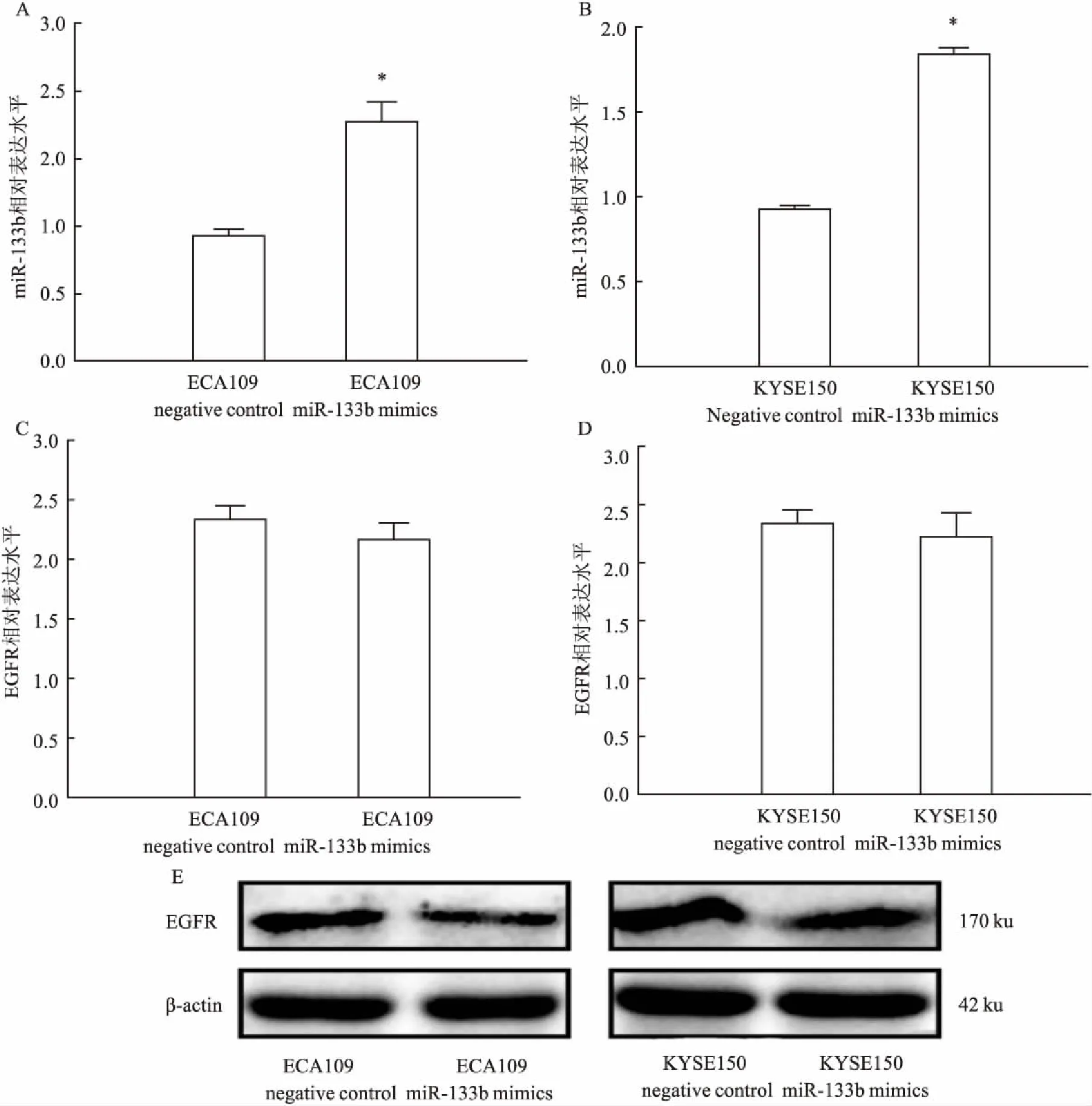
图2 miR-133b对EGFR在转录和蛋白水平表达的影响
A:ECA109细胞转染miR-133b mimics 检测miR-133b转录水平的表达;B:KYSE150细胞转染miR-133b mimics 检测miR-133b转录水平的表达;C:ECA109细胞转染miR-133b mimics 检测EGFR转录水平的表达;D:KYSE150细胞转染miR-133b mimics 检测EGFR转录水平的表达;E:ECA109、KYSE150细胞转染miR-133b mimics 检测EGFR蛋白水平的表达;与negative control组比较:*P<0.05
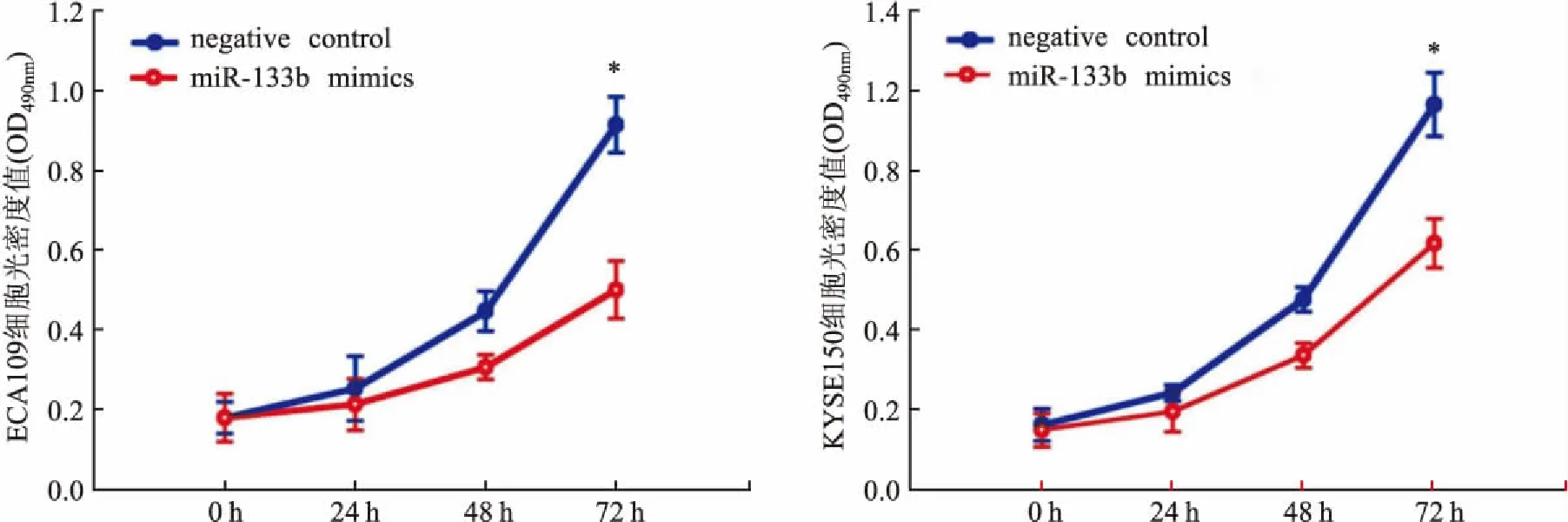
图3 miR-133b对两种食管鳞癌细胞增殖的影响
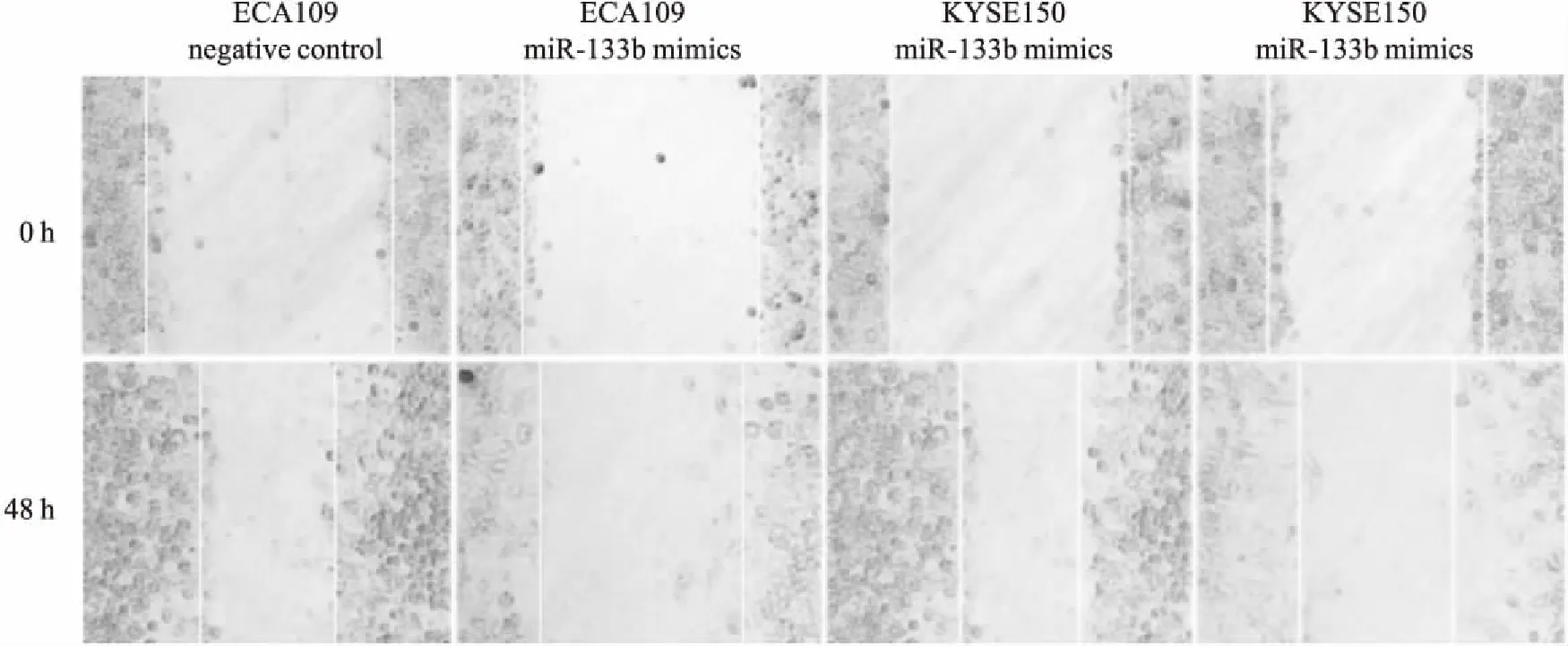
图4 miR-133b对两种食管鳞癌细胞迁移能力的影响 ×200

图5 miR-133b对两种食管鳞癌细胞侵袭能力的影响 ×200
A:ECA109 negative control组;B:ECA109 miR-133b mimics组;C:KYSE150 negative control组;D:KYSE150 miR-133b mimics组;与negative control组比较:*P<0.05

表3 miR-133b表达与食管鳞癌临床病理特征的关系
3 讨论

图6 食管鳞癌组织中miR-133b与EGFR的表达及相关性分析
食管癌位居全球恶性肿瘤发病率的前列[5]。中国是食管癌的高发区。Chen et al[6]2016年的统计分析指出,中国食管癌的发病率为477 900例,接近全球食管癌发病的50%。其中食管鳞癌占食管癌发病的90%以上,其恶性程度高,预后差,多数食管鳞癌在确诊时即分期较晚,丧失手术机会,并且一些食管鳞癌在早期既有较强的侵袭和转移趋势[7]。 在过去的几十年中,随着外科技术的提高以及新辅助化放疗技术的运用,食管鳞癌患者的预后有了一定程度的改善,但患者的5年生存率仍然较低[8]。
如果能寻找到与食管鳞癌侵袭、转移相关的分子生物学标志物,在食管鳞癌患者中发现侵袭、转移的高危人群并进行有针对性的治疗,将有望改善食管鳞癌患者的预后[9]。
目前越来越多的证据显示,miRNA的异常表达在肿瘤的侵袭、转移中发挥着重要的调控作用[10]。Fu et al[4]研究发现miR-133b在食管鳞癌中呈低表达,且miR-133b的差异表达与食管鳞癌的侵袭、转移相关。Kano et al[11]发现在食管鳞癌细胞中上调miR-133b,可以抑制细胞增殖、阻止肿瘤细胞侵袭,提示miR-133b可能作为抑癌基因参与食管鳞癌的发生、发展。Qin et al[12]研究发现,miR-133b在食管鳞癌组织和细胞中均呈低表达,上调食管鳞癌细胞中miR-133b的表达,可对食管鳞癌的上皮间充质转化起负调控作用,提示miR-133b在食管鳞癌的转移中发挥着重要的作用。
EGFR属于ErbB受体家族。研究[13]显示,在多种实体瘤中,EGFR的过表达与肿瘤细胞的增殖、侵袭、转移、凋亡以及血管生成有关,而EGFR基因的突变则有助于上皮细胞发生恶性转化。Jia et al[14]研究发现,在食管鳞癌组织中高表达的EGFR与肿瘤的淋巴结转移、浸润深度及TNM分期呈正相关。COX回归分析显示,EGFR可作为食管鳞癌患者的独立预后因素。Wang et al[15]采用免疫组化法对癌前病变、早期食管鳞癌、进展期食管鳞癌组织中EGFR的表达进行检测。结果显示,EGFR的阳性表达率随肿瘤的进展而增高。提示EGFR可能参与了食管鳞癌的发生、发展过程。Lin et al[16]研究发现,高表达的EGFR与肿瘤较差的分化程度相关,而EGFR的扩增则与肿瘤的分期、淋巴结的转移相关,并提示EGFR的拷贝数增加可作为食管鳞癌预后判定的指标。
关于miR-133b和EGFR在肿瘤中的表达调控关系,目前已有一些研究报道。Liu et al[17]研究发现,miR-133b可通过调控EGFR表达,影响其下游通路的活化,从而抑制非小细胞肺癌细胞的增殖和侵袭,促进细胞凋亡,提高EGFR-TKI药物治疗的敏感性。Tao et al[18]研究发现,在激素治疗不敏感的PC3 和DU145前列腺癌细胞中,miR-133b可通过调控EGFR表达,影响细胞的增殖、侵袭、迁移。但目前对于食管鳞癌中miR-133b与EGFR之间的靶向调控关系,尚无研究报道。而本研究则对这一调控机制进行了初步的探索和验证。在本研究中,课题组通过生物信息学预测EGFR为miR-133b的下游靶基因,并采用双荧光素酶报告系统证实miR-133b可直接靶向作用于EGFR。在食管鳞癌细胞中证实通过转染miR-133b mimic,上调miR-133b的表达,可以下调EGFR在蛋白水平的表达,并抑制食管鳞癌细胞的增殖、侵袭、迁移。在食管鳞癌组织中采用qRT-PCR实验证实miR-133b呈低表达,EGFR呈高表达,且二者表达呈负相关性。将miR-133b的表达与食管鳞癌的临床病理特征进行相关性分析显示,miR-133b的表达与肿瘤TNM分期、淋巴结转移密切相关。基于上述研究结果,课题组推测在正常食管黏膜组织中miR-133b可能通过与其靶基因EGFR的3′-UTR端结合,通过维持EGFR在蛋白水平的正常表达,从而使正常食管黏膜组织细胞的增殖与凋亡处于动态平衡。而在食管鳞癌组织中,由于某种或多种原因导致的miR-133b的生成及功能异常,致使miR-133b的表达下调、miR-133b对EGFR表达的调控作用减弱,使EGFR在蛋白水平的表达上调,导致细胞增殖过度,细胞的侵袭及迁移能力增强,从而促进了肿瘤的进展。
但是,目前对于miR-133b的上游调控机制,miR-133b靶向调控EGFR参与食管鳞癌的侵袭、转移过程的具体信号通路、以及下调食管鳞癌中EGFR的表达对miR-133b表达及食管鳞癌侵袭、转移的影响,目前尚不完全清楚,在今后的实验中课题组将针对上述问题进行进一步的探讨。
[1] D'Angelo B, Benedetti E, Cimini A, et al. MicroRNAs: a puzzling tool in cancer diagnostics and therapy[J]. Anticancer Res, 2016, 36(11):5571-5.
[2] Nariman-Saleh-Fam Z, Bastami M, Somi M H,et al. In Silico dissection of miRNA targetome polymorphisms and their role in regulating miRNA-mediated gene expression in esophageal cancer[J]. Cell Biochem Biophys,2016,74 (4):483-97.
[3] Lakshmipathy U, Hart R P. Concise review: MicroRNA expression in multipotent mesenchymal stromal cells[J]. Stem Cells,2008, 26(2):356-63.
[4] Fu H L, Wu D P, Wang X F, et al. Altered miRNA expression is associated with differentiation, invasion, and metastasis of esophageal squamous cell carcinoma (ESCC) in patients from Huaian, China[J]. Cell Biochem Biophys,2013,67(2):657-68.
[5] Di Pardo B J, Bronson N W, Diggs B S, et al. The global burden of esophageal cancer: A disability-adjusted life-year approach[J]. World J Surg,2016,40(2):395-401.
[6] Chen W,Zheng R,Baade P D,et al.Cancer statistics in China, 2015[J]. CA Cancer J Clin,2016,66(2):115-32.
[7] Hongo M, Nagasaki Y, Shoji T. Epidemiology of esophageal cancer: orient to occident. Effects of chronology, geography and ethnicity[J]. J Gastroenterol Hepatol,2009,24(5):729-35.
[8] Shapiro J, Biermann K, van Klaveren D, et al. Prognostic value of pretreatment pathological tumor extent in patients treated wth neoadjuvant chemoradiotherapy plus surgery for esophageal or junctional cancer[J]. Ann Surg,2017,265(2):356-62.
[9] Qi Y J, Chao W X, Chiu J F. An overview of esophageal squamous cell carcinoma proteomics[J]. J Proteomics,2012,75(11):3129-37.
[10] Zang W, Wang Y, Du Y, et al. Differential expression profiling of microRNAs and their potential involvement in esophageal squamous cell carcinoma[J]. Tumour Biol,2014,35(4):3295-304.
[11] Kano M, Seki N, Kikkawa N, et al. miR-145, miR-133a and miR-133b: Tumor-suppressive miRNAs target FSCN1 in esophageal squamous cell carcinoma[J]. Int J Cancer,2010,127(12):2804-14.
[12] Qin Y, Zhang Y, Tang Q, et al. SQLE induces epithelial-to-mesenchymal transition by regulating of miR-133b in esophageal squamous cell carcinoma[J]. Acta Biochim Biophys Sin (Shanghai),2017,49(2):138-48.
[13] Sakurai H. Non-canonical activation of receptor tyrosine kinases in cancer progression[J]. Yakugaku Zasshi, 2017,137(2):141-4.
[14] Jia W, Wang W, Ji C S, et al. Coexpression of periostin and EGFR in patients with esophageal squamous cell carcinoma and their prognostic significance[J]. Onco Targets Ther,2016,9:5133-42.
[15] Wang H, Jiang D, Song Q,et al. Prognostic impact and potential interaction of EGFR and c-Met in the progression of esophageal squamous cell carcinoma[J]. Tumour Biol,2016,37(7):9771-9.
[16] Lin G, Sun X J, Han Q B, et al. Epidermal growth factor receptor protein overexpression and gene amplification are associated with aggressive biological behaviors of esophageal squamous cell carcinoma[J]. Oncol Lett, 2015,10(2):901-6.
[17] Liu L,Shao X,Gao W,et al.MicroRNA-133b inhibits the growth of non-small-cell lung cancer by targeting the epidermal growth factor receptor[J]. FEBS J,2012,279(20):3800-12.
[18] Tao J, Wu D, Xu B, et al. microRNA-133 inhibits cell proliferation, migration and invasion in prostate cancer cells by targeting the epidermal growth factor receptor[J]. Oncol Rep,2012, 27 (6):1967-75.
