拟密码子适应指数方法的设计及应用
2018-06-02马冲,祖颖,朱平
马 冲,祖 颖,朱 平
(江南大学理学院,中国江苏无锡214122)
在生物体内,蛋白质承载了所有生物功能的遗传信息,在信息传递过程中起到了举足轻重的作用。氨基酸是蛋白质形成的最基本单位,并且与生物的生命活动息息相关。而碱基通过三联体密码子的形式来编码氨基酸,密码子决定着氨基酸的种类[1~4]。目前,常用的编码方法有氨基酸编码法[5]和拟氨基酸编码法[6]。在氨基酸编码法中,4个碱基通过三联体方式自由组合成64个密码子,其中tag、tga和taa为终止密码子,剩余61个密码子编码20种氨基酸。而朱平等[6]运用代数知识提出的拟氨基酸编码法中,将taa、tag、tac和tat标记为终止子,所有64个密码子编码成16种氨基酸。值得注意的是,氨基酸编码法和拟氨基酸编码法都采用atg为起始密码子。
生物体中,编码同一个氨基酸的两个或者两个以上的密码子叫做同义密码子[6]。研究表明,从原核生物到真核生物基因的编码过程中,各生物体内都普遍出现编码同一个氨基酸的同义密码子使用不均衡,即偏好性现象[7,8]。在研究基因的特异性表达时,密码子偏好性就引起了科学家的重视。事实上,目前研究密码子偏好性的方法有很多,比如层次聚类方法、图论方法等[9],这些都与生物体中基因的分子进化、翻译转录、遗传突变有着重要的关系[10]。通过对不同物种密码子使用情况的研究发现,不同物种基因的密码子使用都会出现偏好性。而同种生物中,不同功能的基因密码子使用也会出现偏好性[11]。因此,研究密码子偏好性对基因突变和物种进化分析有重要的作用。
目前,衡量密码子偏好性的分析方法包括同义密码子相对使用度(relative synonymous codon usage,RSCU)、密码子适应指数(codon adaptation index,CAI)、有效密码子数(effective number of codon,ENC)、最优密码子使用频率(frequency of optimal codons,FOP)、密码子偏爱指数(codon bias index,CBI)等[12,13]。其中,密码子适应指数主要反映编码区同义密码子与密码子之间的接近程度,其数值范围在0~1之间。密码子适应指数是衡量基因表达水平的重要参考指标,目前研究发现,单细胞生物适用于此方法,而哺乳动物暂时还不能用密码子适应指数来预测基因的表达水平[10],所以文中选取大肠杆菌、酵母菌、醋酸菌、链球菌、双歧杆菌、枯草杆菌6个单细胞物种进行研究。
由于密码子适应指数测量的是某个基因所用的密码子与高表达基因所用密码子的接近程度,但物种之间存在着巨大差异,所以研究偏好性的指标也层出不穷[14~16]。有些方法仅仅能运用于局限的物种或者某种特定的基因中,或者效果不明显[12,13],因此,不断开发与尝试新方法、新指标一直是偏好性研究的热点。本文将同义密码子相对使用度与拟氨基酸编码法进行对比,观察6个物种的密码子偏好性,得到密码子偏好性的结果;同时,在密码子适应指数的基础上提出新的方法——拟密码子适应指数方法(quasi-codon adaptation index,Q-CAI),并对这两个方法进行对比,进一步研究密码子偏好性的情况,这将对生物体的密码子偏好研究有重要意义,对解释基因突变和生物进化有重要参考价值。
1 理论基础与方法建立
1.1 密码子适应指数
Sharp等[17]提出同义密码子的相对使用度(RSCU)。石秀凡等[5]利用基于氨基酸编码下的同义密码子相对使用度,对78条人类基因进行了研究,并且得到了很好的结果。
引理1:同义密码子相对使用度是衡量密码子偏好性的重要指标,计算公式如下:

RSCUij是指第i个氨基酸的第j个密码子的同义密码子相对使用度值,xij是指编码第i个氨基酸的第j个密码子出现的次数,ni是指编码第i个氨基酸的同义密码子数量(n=1,2,…,6)[17]。
引理2:在RSCU的基础上,Sharp等[17]提出了密码子适应指数(CAI),Wij(the relative adaptiveness of a codon)表示密码子适应度,其计算公式如下:

式中RSCUimax,ximax分别表示编码第i个氨基酸的使用频率最高的密码子的RSCU值和x值。

L表示基因中所使用的密码子数,WK表示基因中每个密码子的适应度,CAI值在0~1之间,CAI值越大,表明密码子偏好性越强[17]。
1.2 拟密码子适应指数
朱平等[6]运用代数的相关知识,提出了拟氨基酸编码方法:令ZU-16={G,V,ED,A,WC,LF,-Y,S’,RS,MI,KN,T,R’,L’,QH,P},称 ZU 为拟氨基酸集,其中-Y为新终止子,但是(tgaєWC)不是终止子。拟氨基酸编码见表1。
同时,该团队提出了基于拟氨基酸编码方法下的相对同义密码子使用度(quasi relative synonymous codon usage,QRSCU),并且对78条人类基因进行了研究[6],不但得到了石秀凡等[5]的研究结果,而且这些特征更加明显,说明了拟氨基酸编码方法的科学性与合理性。
引理3:拟氨基酸编码方法下的同义密码子相对使用度,其计算公式如下:
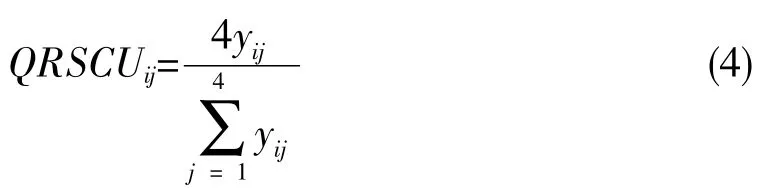
QRSCUij是第i个拟氨基酸的第j个密码子的同义密码子相对使用度值,yij是编码第i个拟氨基酸的第j个密码子出现次数[6]。
定义1:基于CAI提出了拟密码子适应指数(QCAI),Q Wij表示拟密码子适应度,其计算公式如下:
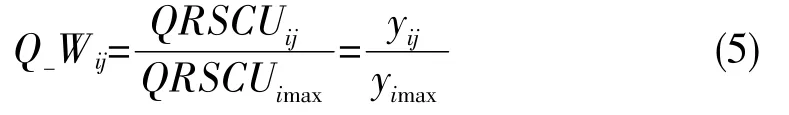
式中QRSCUimax,yimax分别表示编码第i个拟氨基酸的使用频率最高的密码子的QRSCU值和y值。

式中L是指基因中所使用的密码子数,Q WK表示基因中每个拟密码子的适应度,Q-CAI值在0~1之间,其值越大,表明密码子偏好性越强。
密码子偏好性一直被不断的研究与发展,作为人类遗传、进化的重要组成部分,衡量密码子偏好性的方法也就应运而生[18]。由于密码子在生物进化上具有复杂性和适应性,人们研究密码子偏好性方法的道路还很漫长。
便于密码子偏好性更加快速明显的表示,基于密码子适应指数,在拟氨基酸编码方法下,提出了拟密码子适应指数,其数值越高表示密码子的偏好性越强,基因的表达水平越高。
2 数据来源
选取单细胞生物作为研究材料,对CAI值预测基因的表达水平具有很好的适用性。从Gen-Bank/NCBI数据库中下载了大肠杆菌、酵母菌、醋酸菌、链球菌、双歧杆菌、枯草杆菌6个物种的90条mRNAs完整的全基因序列(序列信息见表2),通过计算RSCU值和QRSCU值,进一步计算出CAI值和Q-CAI值。
3 计算分析
3.1 6个物种的碱基分析
运用MATLAB软件计算所选90条序列的碱基组成(表3),包括A、T、G、C在密码子第3位结尾的数量(A3s,T3s,G3s,C3s),G/C的整体含量(GCs),G/C在密码子第3位的碱基含量(GC3s)。
1~15属于醋酸菌序列,16~30是大肠杆菌序列,31~45是酵母菌序列,46~60为枯草杆菌序列,61~75是链球菌序列,76~90条是双歧杆菌序列。由表3分析得出下面结论:
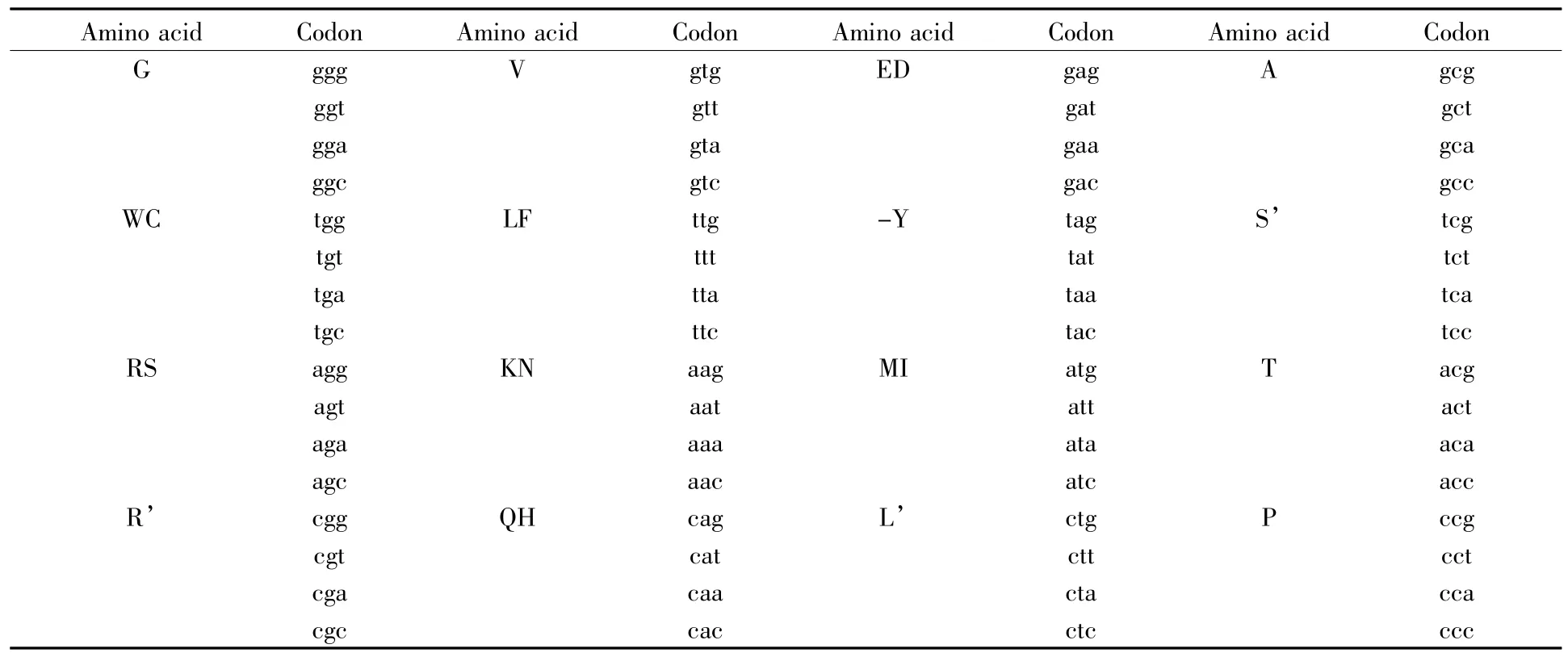
表1 拟氨基酸编码信息Table1 Quasi-amino acid coding information
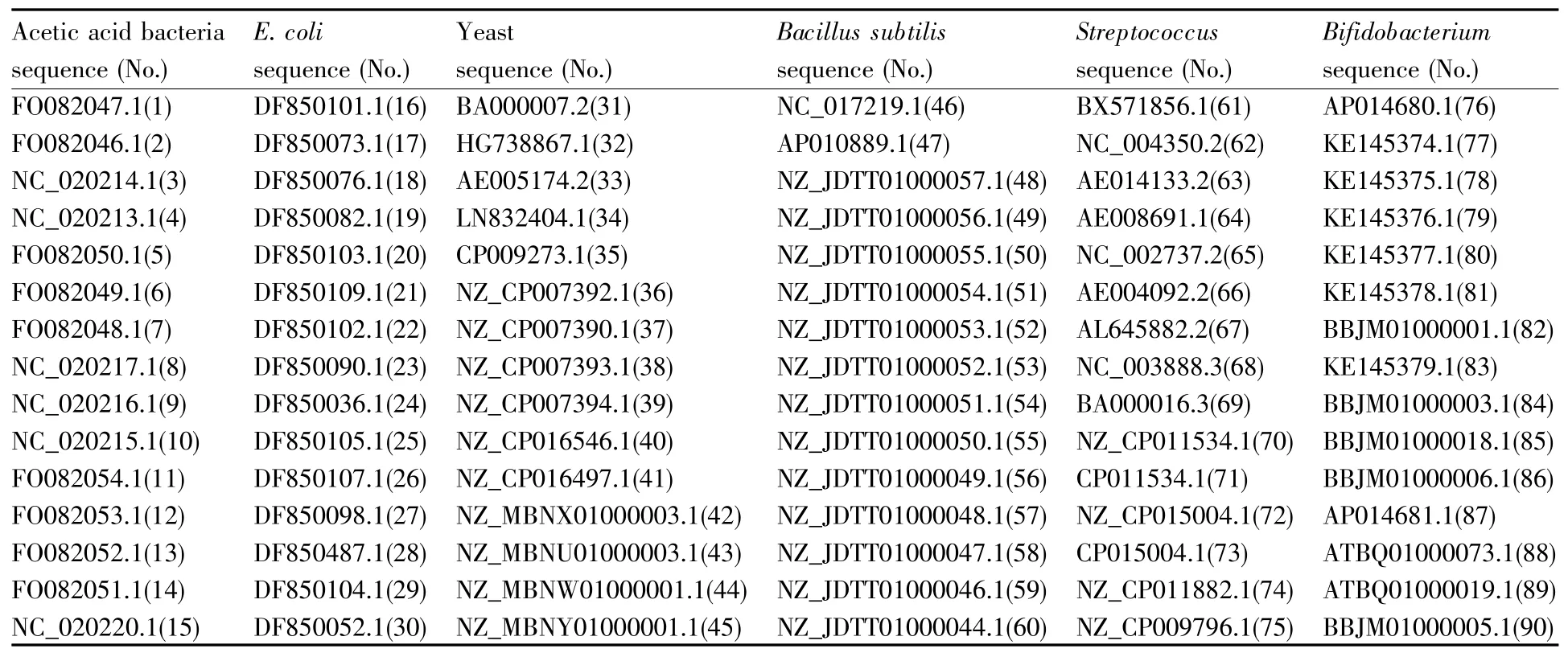
表2 数据的序列编号Table2 Serial number of data
1)醋酸菌序列偏好使用以g/c结尾的密码子,以g/c结尾的密码子明显多于以a/t结尾的密码子,并且GC含量是50%~57%,GC3s是54%~64%。
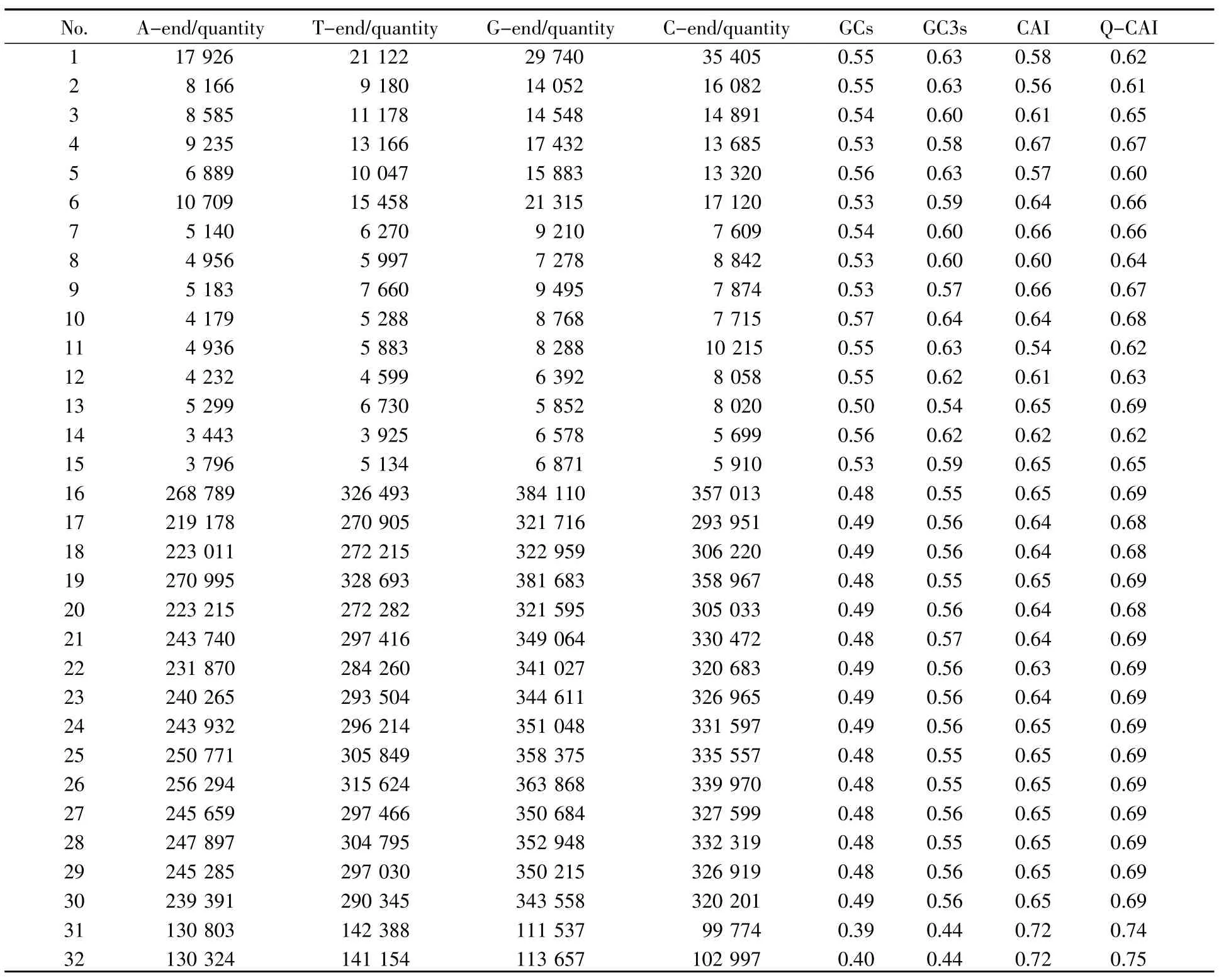
表3 6个单细胞物种90条mRNA序列的碱基组成Table3 Base composition of 90 mRNA sequences from 6 single-celled species

(接上表)
2)大肠杆菌序列明显偏好使用以g/c结尾的密码子,但GC整体含量不高,其中GC含量是48%~49%,GC3s是55%~57%。
3)酵母菌和枯草杆菌序列偏好使用以a/t结尾的密码子,GC含量低于AT含量,GC含量分别为39%~40%、28%~69%,而 GC3s分别为 44%~46%、23%~86%。
4)链球菌序列使用以a/t结尾的密码子比使用以g/c结尾的密码子的数量要高,且GC含量较低,为37%~50%,GC3s含量为36%~68%。
5)双歧杆菌序列明显偏好使用以g/c结尾的密码子,GC及GC3s的含量明显较高,分别为53%~63%和66%~80%。
3.2 基因CAI的偏好性分析
同义密码子的相对使用度能够分析密码子使用的偏好情况,密码子适应指数是在同义密码子相对使用度的基础上得出来的,它是衡量密码子偏好性的重要指标,其值越大,密码子的偏好性越强。表3给出了90条序列的CAI值和Q-CAI值运算结果,从CAI值的运算结果得出:
1)90条序列的CAI值分布于0.29~0.75,只有4条序列的CAI值低于0.4,其余序列CAI值普遍高于0.5,表明这6个物种的密码子偏好性很强。
2)酵母菌和大肠杆菌偏好性最强,醋酸菌、链球菌、枯草杆菌偏好性次之,双歧杆菌的偏好性最差。其中,15条酵母菌序列的CAI值都高于0.7,说明酵母菌的偏好性极强。15条大肠杆菌序列的CAI值都在0.6~0.7,表明大肠杆菌是仅次于酵母菌的高偏好性物种。
3)醋酸菌、链球菌、枯草杆菌的15条序列CAI值并不是均匀分布在某一固定区域,而是0.2~0.7之间均有分布,提示不同物种其密码子偏好性使用不同。
4)同一物种不同序列其CAI值分布也并不均匀,比如枯草杆菌的最高CAI值是0.74,最低值是0.29,即同一物种的不同序列其密码子偏好性并不相同。
3.3 基因Q-CAI的偏好性分析
拟密码子适应指数是在密码子适应指数的基础上,根据拟氨基酸编码方法提出的新方法,将Q-CAI与CAI方法的计算数据分析对比,从90条序列的Q-CAI值分析得出:
1)90条序列的Q-CAI值分布在0.42~0.76,且大部分序列都高于0.5,说明6个物种密码子偏好性很强,与CAI方法计算的结论相一致。
2)醋酸菌、大肠杆菌和酵母菌各自15条序列的Q-CAI值都比CAI值高,即在偏好性强的物种中,Q-CAI方法衡量密码子偏好性比CAI方法的数据结果更显著。
3)链球菌、枯草杆菌和双歧杆菌的Q-CAI值也普遍比CAI值要高,只有极少部分序列的密码子CAI值比Q-CAI值高,但极少部分序列的CAI值和Q-CAI值非常接近,即Q-CAI方法可以得到CAI方法的所有结论。
从上述分析可知,Q-CAI方法在衡量密码子偏好性时,能够得到CAI方法的所有结论,并且数据比前人提出的CAI值更高,即密码子的表达水平更高,能够更准确地得到密码子的偏好性强弱。因此,Q-CAI方法比CAI方法更加合理有效,可以作为研究基因表达以及遗传与变异的重要理论依据。
4 结论
在氨基酸编码法下,从90条序列的同义密码子相对使用度数据中分析得出:6个物种的同义密码子使用具有偏好性,醋酸菌、大肠杆菌和双歧杆菌的密码子偏好使用c/g结尾,GC整体含量也比较高。而酵母菌、枯草杆菌和链球菌的GC含量则相对较低。根据密码子适应指数的研究,进一步证明了6个物种密码子有较强的偏好性,其密码子表达水平较高。
根据拟密码子适应指数方法,对6个单细胞物种90条序列的数据进行分析,得到:所有数据的Q-CAI值普遍高于0.5,且偏好性较低的序列只有几条,6个物种的密码子同样具有较强的偏好性,与CAI方法得到的结果相一致,说明此方法具有合理性。
进一步与CAI运算方法对比,偏好性强的物种——醋酸菌、大肠杆菌和酵母菌所有序列的QCAI方法运算结果都比CAI方法运算结果数值更高、更明显。而且双歧杆菌、枯草杆菌和链球菌的Q-CAI方法运算结果也普遍比CAI方法运算结果数值高,说明拟密码子适应指数不仅能衡量密码子使用的偏好性,而且运算结果比CAI方法更加明显有效,因此,具有较好的适应性。这对密码子偏好性研究,以及基因突变和物种进化研究都具有重要的意义。
参考文献(References):
[1]Cristina J,Moreno P,Moratorio G,et al.Genome-wide analysis of codon usage bias inEbolavirus[J].Virus Research,2015,196:87-93.
[2]Camiolo S,Melito S,Porceddu A.New insights into the interplay between codon bias determinants in plants[J].DNA Research,2015,22(6):461-470.
[3]苏惠,李永光,谭文雍,等.大肠杆菌gdhA基因的密码子偏好性分析及优化[J].基因组学与应用生物学(Su Hui,Li Yongguang,Tan Wen-yong,et al.Analysis of codon usage bias and optimization of codon ofgdhAgene inEscherichia coli[J].Genomics and Applied Biology),2015,34(3):521-529.
[4]郭承恩,柴志欣,钟金城.人类1号、X、Y染色体基因密码子偏好性研究[J].生命科学研究(Guo Cheng-en,Chai Zhi-xin,Zhong Jin-cheng.Study on codon usage bias in the gene of human 1,X and Y chromosomes[J].Life Science Research),2014,18(5):387-394.
[5]石秀凡,黄京飞,梁宠荣,等.人类基因中同义密码子的偏好与密码子-反密码子间的结合强度密切相关吗?[J].科学通报(Shi Xiu-fan,Huang Jing-fei,Liang Chong-rong,et al.Is the preference for synonymous codons in human genes closely related to the binding strength between codon-anti-codons?[J].Chinese Science Bulletin),2000,45(23):2520-2525.
[6]朱平,高雷,徐振源.基于拟氨基酸编码方法的同义密码子的偏好性与结合强度的相关性研究[J].物理学报(Zhu Ping,Gao Lei,Xu Zhen-yuan.Correlation between the usage degree of the synonymous codon and the strength of combination based on the quasi-amino acid coding[J].Acta Physica Sinica),2009,6:4295-4300.
[7]Rudorf S,Lipowsky R.Protein synthesis inE.coli:dependence of codon-specific elongation on tRNA concentration and codon usage[J].PLoS One,2015,10(3):e0134994.
[8]吴彦庆,赵大球,王静,等.芍药查尔酮异构酶基因(CHI)克隆、密码子偏好性分析以及蛋白结构功能预测[J].华北农学报(Wu Yan-qing,Zhao Da-qiu,Wang Jing,et al.Cloning,codon usage bias and protein structure and function prediction of CHI gene inPaeonia lactiflora[J].Acta Agriculturae Boreali-Sinica),2016,31(2):71-80.
[9]张连顺,王吉华,姜万禄.用图论方法研究核酸序列的密码子使用与基因表达水平的关系[J].生物数学学报(Zhang Lianshun,Wang Ji-hua,Jiang Wan-lu.Study on the relation between codon usage of nucleic acid sequences and gene expression level by graph theory method[J].Journal of Biomathematics),1998,13(1):65-73.
[10]吴宪明,吴松锋,任大明,等.密码子偏性的分析方法及相关研究进展[J].遗传(Wu Xian-ming,Wu Song-feng,Ren Daming,et al.The analysis method and progress in the study of codon bias[J].Hereditas),2007,29(4):420-426.
[11]谈承杰,朱平.抑癌基因p53密码子偏好性分析及其突变致癌预测[J].计算机与应用化学(Tan Cheng-jie,Zhu Ping.Analysis of codon bias of tumor suppressor gene p53 with forecast to gene mutation[J].Computers and Applied Chemistry),2012,29(11):1299-1303.
[12]Šnajder M,Mihelic M,Turk D,et al.Codon optimisation is key for pernisine expression inEscherichia coli[J].PLoS One,2015,10(4):e0123288.
[13]Xiang H,Zhang R Z,Butler R R,et al.Comparative analysis of codon usage bias patterns in microsporidian genomes[J].PLoS One,2015,10(6):e0129223.
[14]招丽婵,邓雨修,王东东,等.不同PRRSV毒株间ORF1a基因密码子偏爱性差异分析[J].生命科学研究(Zhao Li-chan,Deng Yu-xiu,Wang Dong-dong,et al.Analysis of theORF1agene codon bias disparity in different PRRSV strains[J].Life Science Research),2009,13(5):422-429.
[15]Zhang D L,Chen D F,Cao L R.The effect of codon mismatch on theproteintranslationsystem[J].PLoSOne,2016,11(2):e0148302.
[16]吴艳,仇建邺,朱平.亚氨基酸编码方法及其应用[J].生物数学学报(Wu Yan,Qiu Jian-ye,Zhu Ping.Subtypes amino acids coding method and application[J].Journal of Biomathematics),2013,28(4):725-737.
[17]Sharp P M,Li W H.The codon adaptation index--a measure of directional synonymous codon usage bias,and its potential applications[J].Nucleic Acids Research,1987,15(3):1281-1295.
[18]孔娟娟,朱平.人类p53肿瘤蛋白的偏好性分析及其应用[J].计算机应用研究(Kong Juan-juan,Zhu Ping.Research and application of codon bias of human p53 tumor protein[J].Application Research of Computers),2011,28(8):2987-2990.
