基于生物多样性的流感暴发研究
2018-06-02周加兴唐旭清
周加兴,唐旭清,b*
(江南大学a.理学院;b.无锡市生物计算工程技术研究中心,中国江苏无锡214122)
甲型流感是由病毒引起的一种高致病率和高死亡率的传染病,每年造成大约500万人致病和50万人死亡[1]。20世纪,暴发了由不同基因型引起的3次大规模的流感大流行,分别是1918年的H1N1(“西班牙流感”)、1957年的H2N2(“亚洲流感”)和1968年的H3N2(“香港流感”)。21世纪初,一种新的甲型流感(“猪流感”)在北美洲暴发,并迅速传遍世界各地。从2009年4月至2010年8月,官方确认致死病例达18 500例,而且这个数字很可能只是死亡病例中的一部分[2,3]。每次流感病毒的暴发,对人类和畜类的生命造成严重的危害,对社会生产和生活带来诸多不便,所以对流感大流行建立模型进行预测分析迫在眉睫。
目前,已有许多学者在流感流行预测方面进行了研究。Banerjee等[4]利用碱基间的键能大小,由血凝素(hemagglutinin,HA)蛋白质发生抗原漂移的难易程度进而预测流感发生。Yang等[5]用贝叶斯推理结合流行病学模型对香港地区1998-2013年间的流感暴发情况进行了综合分析。杨冬红等[6]分析了流感暴发与厄尔尼诺现象的关系。Jacobs等[7]分析了流感暴发与感染人群年龄之间的关系。此外,Dugas等[8]基于谷歌流感模型,利用广义线性回归分析,结合流感的时序数据,对流感暴发进行了预测。
随着生物技术的发展,大量蛋白质序列通过实验被测定。而采用实验的方法去预测蛋白质的结构和功能会耗费大量的人力物力。近年来,随着理论方法的成熟,理论分析方法预测蛋白质结构和功能的应用越来越广泛。薛晓丽等[9]基于H1N1的HA蛋白质进化树揭示了H1N1病毒进化关系。靳佩轩等[10]基于流感病毒10种组成蛋白质的氨基酸序列,对未来流感病毒的变异和暴发进行了预测。Deng等[11]针对不同亚型的HA和NA,利用系统进化树对洞庭湖地区的禽流感进行了深入的分析。这些研究显示,采用HA蛋白质序列的相似性来研究流感病毒蛋白质功能和变异关系是一种有效可行的方法。
生物多样性是指在确定空间内生物的丰富程度及变异程度,生物多样性越大,就可能出现更优良、更有生命力的子代。国内部分科研人员在通过评价生物多样性来预测生物在环境中的生存能力和适应能力方面进行了较为深入的研究。彭丽潭等[12]通过分层聚类和信息融合分析了气候对丹顶鹤种群的影响。李桑等[13]通过DGGE技术研究了乡村沼气池污泥微生物的生物多样性对细菌微生物群落的影响。本文通过从生物多样性的角度解释病毒的变异、进化等关系,从而为流感暴发研究提供依据和支撑。
从NCBI下载了1902-2016年99 861条流感病毒的HA,进行40维蛋白质特征提取,将蛋白质序列转换成数值信息。同时,对每年的数据进行层次聚类,引入最优层次聚类评价指标,获取每一年数据的最优聚类数。最后,通过计算每一年的种群熵评价指标来度量每一年流感病毒的生物多样性,并计算种群熵变化率以衡量流感病毒的变异速率。
1 材料与方法
1.1 数据来源
本文所需数据均从NCBI(http://www.ncbi.nlm.nih.gov/gennomes/FLU/Database/nph-select.cgi)网站中Molecular Databases的Protein Sequence上下载。数据包括1902-2016年的99 861个流感病毒 HA蛋白质序列,其中 1903-1917、1920、1921、1922、1923、1924、1926、1928、1929、1932、1941、1944等年份HA蛋白质序列的数据缺失。
1.2 特征提取
特征提取是数据处理与序列比对的关键步骤,蛋白质序列比对现行的方法主要基于氨基酸序列的组成频率与位置关系。本文采用融合蛋白质组成、氨基酸理化属性及耦合信息的40维特征向量。如给定长度为n的蛋白质序列记为S=s1s2…si,si(i=1,2,…,n)表示20种基本氨基酸中的一种,对每一条蛋白质序列构造40维的特征向量V(C),即V(C)=(V1(G),V2(F),V3(H))[14]。其中,V1(G)表示HA蛋白质序列中各种氨基酸出现的频数构成的20维向量;V2(F)表示氨基酸的理化性质分成W1=(R,D,E,N,Q,K,H)非极性且亲水、W2=(L,I,V,A,M,F)非极性且疏水、W3=(S,T,Y,W)极性且亲水性、W4=(P,G,C)极性且疏水性等四类,每类氨基酸在HA蛋白质序列中出现的频数构成的4维向量;V3(H)表示HA蛋白质序列中按理化属性分为四类之后,氨基酸两两相连出现的频数构成的16维向量。
1.3 最优聚类评价指标
聚类分析旨在使得相似度较高的聚成同类,相似度较小的划为不同类,即同类之间的相似度高,不同类之间的相似度低。本文采用层次聚类处理流感病毒的HA蛋白质序列信息,定义蛋白质序列x1,x2,…,xm间的差异[15]为:
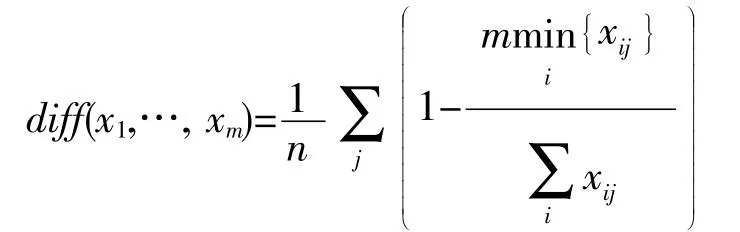
其中,xij为样本xi的第j个属性。Vm和Vn是HA蛋白质序列的两类,如果Vm和Vn的相似度最大,则将这两类合并。合并后新的聚类表示为:
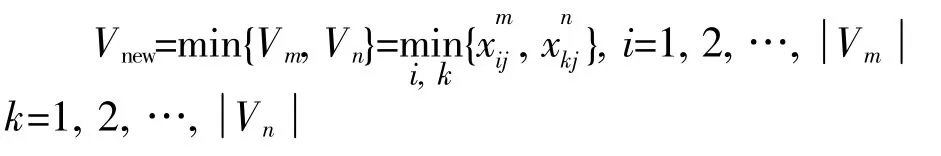
对于层次聚类建立的分层递阶结构,确定合适的粒度是关键问题[16,17]。假设d是可数集X={x1,x2,…,xn}的度量标准(如欧氏距离等)。存在一个粒度空间X(λ)єℵTd(X),记X(λ)={a1,a2,…,acλ},ak={xk1,聚类中心ak且为样本中心。引入类内偏差Sin(X(λ))和类间偏差Sbetween(X(λ)),如下:

最优层次评价指标(HEI)为:

聚类最优评价指标被用来确定合理的分类和粒度。当Sbetween(X(λ))<Sin(X(λ))时,HEI(X(λ))取最小值,取得合理的粒度,其目标优化模型如下:

1.4 生物多样性
种群熵值是评价生物多样性的指标之一。假设P(t)为第t代种群,种群中个体的数量为N,根据个体之间的差异可将种群划分为m个部分[18]:P1(t),P2(t),…,Pm(t)。显然,并且对于均有。设k1,k2,…,km分别为P1(t),P2(t),…,Pm(t)中个体数目,定义第t代种群的熵其中Pi=ki/N。
1.5 算法流程
Step 1 对流感病毒数据进行预处理,并剔除掉部分流感病毒。
Step 2 将蛋白质序列数值化转化成40维特征向量,并计算每一年与第一年数据的欧氏距离,然后绘出基于欧氏距离的流感病毒变异进化分布图。
Step 3 对所得40维特征向量进行基于欧氏距离的层次聚类,根据HEI(X(λ),确定每一年的最优聚类数。
Step 4 基于最优聚类数,计算每一年种群熵值和种群熵变化率并绘出种群熵的变化图。
2 结果与分析
首先对流感病毒的HA数据进行预处理,按年份对其进行40维特征向量提取。因为数据过少,不利于评价当年的生物多样性分析,所以剔除掉 1919、1925、1936、1937、1952、1955 等年份的数据。然后,利用算法计算每一年的种群熵值(表1),并绘出种群熵变化图(图1)。同时,计算每一年的数据与第一年(1902年)数据的欧氏距离并绘出病毒的变异进化分布图(图2)。最后,计算出20世纪种群熵变化率top10(表2)和21世纪种群熵变化率 top5(表3)。

表1 1902-2016年的种群熵值Table1 The population entropy from 1902 to 2016

表2 1902-2000年的种群熵变化率top10Table2 Top 10 years in change rate of the population entropy from 1902 to 2000

图1 1902-2016年种群熵变化图Fig.1 The population entropy variation from 1902 to 2016
从图1中历年信息熵值的变化可以宏观地看出,信息熵整体呈现上升趋势,表明了流感病毒生物多样性越来越丰富。这现象符合实际中生物发展的规律,因为病毒的不断变异和重组产生了新的基因型和亚种,使得流感病毒的生物多样性变得越来越丰富,即种群熵的值越来越大。20世纪以来,1918-1919年暴发了由H1N1引发的著名的“西班牙流感”,据初步统计,导致2 000~5 000万人死亡[19,20]。从表1可以看出,1918年流感病毒的种群熵值为0.702 9,明显比1902年与1927年等邻近年份的病毒种群熵值高。1955-1957年暴发了“亚洲流感”,导致全球大约280万人死亡[21]。表1显示,1956年的种群熵值为1.258 0,1957年的种群熵值为1.550 2,均也明显高于与它邻近的年份,同样表明1956年和1957年的流感病毒生物多样性丰富。1968年暴发了由甲型流感病毒H3N2所致的“香港流感”,1965年的种群熵为1.319 7,1968年的种群熵为1.305 1,也明显高于其他邻近的年份,说明流感病毒变异重组几年前就已经开始,一直处于潜伏期,直到1968年暴发[22]。1977年、1987年和1997年都发生了不同规模的流感暴发事件[23,24]。进入21世纪,2009年暴发了由新型甲型H1N1引发的“墨西哥流感”,迅速传遍全球导致至少18 500人死亡。从表1中可看出,2005-2009年的种群熵值分别为2.328 6、2.438 5、2.456 5、2.453 0、2.376 3,都高于邻近的年份,说明流感病毒从2005年开始出现较大的变异,直到2009年暴发成流感大流行[25]。同样2012-2014年的种群熵也高于邻近年份,查阅文献显示在2013-2014年暴发了H1N1流感季节性流行[26]。
上述分析显示,20世纪发生流感大流行的间隔时间约在10年左右,而进入21世纪后流感大流行发生的间隔时间缩短至4~5年左右。因为病毒从变异到暴发具有一定的潜伏期,本文选择对20世纪每一年的种群熵与5年前的种群熵值作差,21世纪每一年的种群熵与3年前的种群熵值作差。再将其与时间间隔的比值记为病毒变异的变化率。
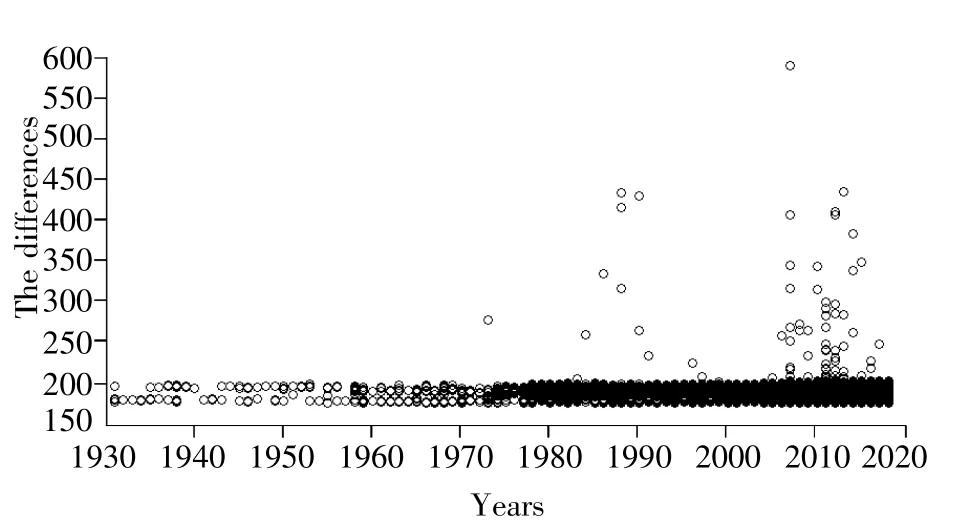
图2 流感病毒变异进化分布图Fig.2 Mutation distribution of influenza viruses

表3 2000-2016年的种群熵变化率top5Table3 Top 5 years in change rate of the population entropy from 2000 to 2016
表2显示在1956年、1963年、1976年和1998年等年份的种群熵变化率较大,即病毒的变异速率快,变异明显。相对,在病毒较明显的变异之后发生了1957年、1968年、1977年和1997年的流感大流行。表3显示在2006年、2007年和2014年等年份的种群熵变化率较大,即病毒变异速率快,变异明显。同样,在病毒较明显的变异之后发生了2009年和2014年流感的大流行。
计算出每一年的数据与第一年的数据的欧氏距离,并绘出相对第一年的变异进化图(图2),可以更容易看出流感病毒的变异情况。结合表1和图2发现,1902-1970年的种群熵值不高且变异程度也比较低,而1970-2016年的种群熵值较高且变异程度很活跃。但实际上,1918年、1957年以及1968年的3次流感大流行的波及范围广、死亡率高、破坏力强;相比之下,1977年、1997年以及2009年的大流行死亡率较低、破坏力也较弱。1918年“西班牙流感”的高死亡率主要集中在15~35岁的青年之间,原因在于1889-1890年暴发了由不同基因型引发的“俄罗斯流感”,导致这个年龄段的人失去了对由H1N1病毒引发的流感的免疫能力[27,28]。1957年由H2N2引发的“亚洲流感”病毒携带的HA抗原和NA抗原从未在人体中出现过,它是由人H1N1亚型与禽流感病毒的3个基因片段重组而来。从图3流感病毒系统发育树也可以看出,1918年和1957年的流感病毒同源性较低,所以1957年暴发流感时人类体内没有相关的抗体,从而导致全球各地280万人死亡[29]。而1968年由H3N2引发的“香港流感”,其流感病毒的N2亚型与1957年的H3N2一样,这样多数人体内存在N2表面蛋白抗体;此外,由于该病毒拥有与1889年大流行的H3N8病株同样的H3抗原,这样部分老年人也拥有对H3N2的免疫能力,从而使死亡人数降低,约750 000人死亡[22,30]。
对于1970年后暴发的流感基因型,如1977年的H1N1、1997年的H5N1、2009年的 H1N1以及2014年的H1N1等。综合前文分析及图3可知,由于前面流感的暴发使得人体内已经拥有部分重组或变异流感基因型的抗体,加上预测系统的成熟建立和医疗条件的改善,使得1970年后流感引起的死亡率和破坏力没有前面的几次高。
3 结束语
本文通过对99 861个流感病毒的HA蛋白质序列进行特征提取,采用层次聚类方法并引入最优层次评价指标计算出每一年的最优聚类数。利用每一年的种群熵值刻画流感病毒的生物多样性,从宏观角度能够较好吻合历史数据。进一步,通过变异进化分布图和种群熵变化率对流感病毒的变异进行深入分析。数据分析发现,种群熵值能很好地反应流感病毒的生物多样性,种群熵变化率也能很好地反应流感病毒的变异和不稳定情况。通过流感病毒种群熵值和种群熵变化率对发生流感大流行的时间进行粗略的估计,表明在21世纪流感病毒大流行的发生时间间隔会缩短且规模和破坏性都将降低。这些研究可为流感的预测提供依据和支撑。
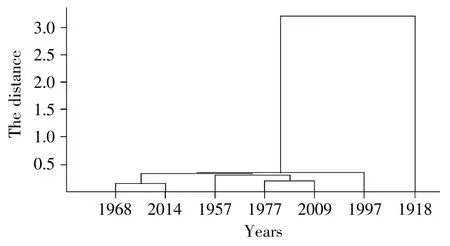
图3 流感大暴发年份的流感病毒系统发育树Fig.3 Phylogenetic tree of influenza viruses in epidemic years
参考文献(References):
[1]Xu R,Ekiert D C,Krause J C,et al.Structural basis of preexisting immunity to the 2009 H1N1 pandemic influenza virus[J].Science,2010,328(5976):357-360.
[2]Dawood F S,Iuliano A D,Reed C,et al.Preliminary estimates of global 2009 H1N1 influenza mortality[C]//Infectious Diseases Society of America 2011 Annual Meeting.Arlington:Infectious Diseases Society of America,2011.
[3]NovelSwine-OriginInfluenzaA(H1N1)VirusInvestigationTeam,Dawood F S,Jain S.Emergence of a novel swine-origin influenza a(H1N1)virus in humans[J].The New England Journal of Medicine,2009,360(25):2605-2615.
[4]Banerjee R,Roy A,Das S,et al.Similarity of currently circulating H1N1 virus with the 2009 pandemic clone:viability of an imminent pandemic[J].Infection,Genetics&Evolution,2015,32:107-112.
[5]Yang W,Cowling B J,Lau E H,et al.Forecasting influenza epidemics in Hong Kong[J].PLoS Computational Biology,2015,11(7):e1004383.
[6]杨冬红,杨学祥.流感世界大流行的气候特征[J].沙漠与绿洲气象(Yang Dong-hong,Yang Xue-xiang.The climatic characteristic of pandemic influenza[J].Desert and Oasis Meteorology,2007,1(3):1-8.
[7]Jacobs J H,Archer B N,Baker M G,et al.Searching for sharp drops in the incidence of pandemic A/H1N1 influenza by single year of age[J].PLoS One,2012,7(8):e42328.
[8]Dugas A F,Jalalpour M,Gel Y,et al.Influenza forecasting with Google flu trends[J].PLoS One,2013,8(2):e56176.
[9]薛晓丽,李阳,唐旭清.基于H1N1型禽流感病毒的HA蛋白序列进化树研究[J].计算机应用研究(Xue Xiao-li,Li Yang,Tang Xu-qing.Research on evolutionary tree for H1N1 flu virus based on HA protein sequences[J].Application Research of Computers),2015,32(9):2634-2638.
[10]靳佩轩,高洁.流感病毒组成蛋白质序列的分析与预测[J].食品与生物技术学报(Jin Pei-xuan,Gao Jie.Sequence analysis and prediction of the influenza virus protein[J].Journal of Food Science and Biotechnology),2016,35(4):393-398.
[11]Deng G,Tan D,Shi J,et al.Complex reassortment of multiple subtypes of avian influenza viruses in domestic ducks at the Dongting lake region of China[J].Journal of Virology,2013,87(17):9452-9462.
[12]彭丽潭,吴军,唐旭清.气候变化对丹顶鹤种群在繁殖地逗留时间的影响分析[J].生态与农村环境学报(Peng Li-tan,Wu Jun,Tang Xu-qing.Impact of climate change on stay of redcrowned cranes in their breeding habitat[J].Journal of Ecology and Rural Environment),2014,30(3):280-288.
[13]李桑,张琳,邱义兰,等.乡村沼气池污泥微生物多样性的研究[J].生命科学研究(Li Sang,Zhang Lin,Qiu Yi-lan,et al.Study on microbial diversities in rural biogas digesters[J].Life Science Research),2015,19(4):321-327.
[14]李巍巍,李阳,唐旭清.不同特征描述下H1N1病毒蛋白序列的比较[J].生命科学研究(Li Wei-wei,Li Yang,Tang Xuqing.Comparing the H1N1 flu virus protein sequences by different feature vectors[J].Life Science Research),2016,20(2):119-124.
[15]李阳,唐旭清.基于粗粒化的流感病毒蛋白进化树构建[J].模式识别与人工智能(Li Yang,Tang Xu-qing.Construction of phylogenetic treeof flu virus proteins based on coarse graining[J].Pattern Recognition and Artificial Intelligence),2016,29(10):936-942.
[16]Tang X Q,Zhu P.Hierarchical clustering problems and analysis of fuzzy proximity relation on granular space[J].IEEE Transactions on Fuzzy Systems,2013,21(5):814-824.
[17]Tang X Q,Zhu P,Cheng J X.The structural clustering and analysis of metric based on granular space[J].Pattern Recognition,2010,43(11):3768-3786.
[18]申元霞,张翠芳.一种新型保持种群多样性的遗传算法[J].系统仿真学报(Shen Yuan-xia,Zhang Cui-fang.A modified genetic algorithm with maintaining diversity[J].Journal of System Simulation),2005,17(5):1052-1053.
[19]周剑芳,杨磊,蓝雨,等.1918/1919年西班牙流感(H1N1)病原学概述[J].病毒学报(Zhou Jian-fang,Yang Lei,Lan Yu,et al.Epidemiological overview of 1918/1919 Spanish influenza[J].Chinese Journal of Virology),2009,25(suppl.):8-11.
[20]Worobey M,Han G Z,Rambaut A.Genesis and pathogenesis of the 1918 pandemic H1N1 influenza a virus[J].Proceedings of the National Academy of Sciences USA,2014,111(22):8107-8112.
[21]Duff F L.Pandemic influenza in 1957:review of international spread of new Asian strain[J].Journal of the American Medical Association,1958,166(10):1140-1148.
[22]袁帆,蓝雨,郭俊峰,等.1968年流感大流行的流行病学概述[J].病毒学报(Yuan Fan,Lan Yu,Guo Jun-feng,et al.Epidemiological overview of the 1968 influenza pandemic[J].Chinese Journal of Virology),2009,25(suppl.):33-35.
[23]Singh G,Oberoi M S,Kwatra M S,et al.Isolation of influenza virus from horses in the equine influenza outbreak of 1987[J].Current Science,1987,56:1285-1286.
[24]Rocchi G,De Felici A,Ragona G,et al.Influenza activity in metropolitan Rome,Italy,during the cold-weather months of 1976-1977[J].Developments in Biological Standardization,1977,39:425-428.
[25]Dawood F S,Iuliano A D,Reed C,et al.Estimated global mortality associated with the first 12 months of 2009 pandemic influenza a H1N1 virus circulation:a modelling study[J].The Lancet Infectious Diseases,2012,12(9):687-695.
[26]Linderman S L,Chambers B S,Zost S J,et al.Potential antigenic explanation for atypical H1N1 infections among middleaged adults during the 2013-2014 influenza season[J].Proceedings of the National Academy of Sciences USA,2014,111(44):15798-15803.
[27]Brownlee G G,Foder E.The predicted antigenicity of the haemagglutinin of the 1918 Spanish influenza pandemic sug gests an avian origin[J].Philosophical Transactions of the Royal Society B-Biological Sciences,2001,356(1416):1871-1876.
[28]Reid A H,Taubenberger J K,Fanning T G.Evidence of an absence:the genetic origins of the 1918 pandemic influenza virus[J].Nature Reviews Microbiology,2004,2(11):909-914.
[29]Kawaoka Y,Krauss S,Webster R G.Avian-to-human transmission of the PB1 gene of influenza a viruses in the 1957 and 1968 pandemics[J].Journal of Virology,1989,63(11):4603-4608.
[30]Cox N J,Subbarao K.Global epidemiology of influenza:past and present[J].Annual Review of Medicine,2000,51(1):407-421.
