2001-2016年广州市登革3型病毒E基因序列及系统进化树分析
2018-05-28,,,,,,
,,,,, ,
登革病毒(dengue virus,DENV)属于黄病毒科黄病毒属,是引起登革热(dengue fever,DF)和登革出血热(dengue haemorrhagic fever,DHF)的病原。在DENV的3种结构蛋白,衣壳蛋白(C蛋白)、膜蛋白(PrM蛋白和M蛋白)和包膜蛋白(E蛋白)中,由E基因编码的E蛋白构成病毒颗粒的表面凸起,具有血凝活性,在与宿主细胞融合和诱导产生抗体的过程中发挥重要作用。中和抗体需要通过识别病毒的E蛋白发挥中和作用[1]。
登革热在中国最早的记载是1873年发生于厦门。1978年登革热首次从广东佛山市传入广州市后,登革热在广州开始流行。据统计,在广东省2001-2011年报告的3 577例登革热病例中,70.90%(2 536例)出现在广州市。在广东省暴发历史上最大规模登革热流行的2014年,广州市病例占82.63%(37 340例)[2-3]。2001-2016年,广州市共发生登革热病例4万余例。因此,了解广州市登革热流行状况,对于登革热防控意义重大。
根据蚀斑减少中和试验(plaque reduction neutralization test,PRNT),可以将登革病毒分为4个血清型,血清型之间有交叉抗原反应。虽然在某些地区曾报道发现第5个血清型的登革病毒,但登革病毒的流行仍然以4个血清型为主[4]。同一血清型的登革病毒又可以根据E基因序列划分为不同的基因型。4种血清型的登革病毒在广州市登革热的流行中均有出现,并且以登革1型病毒为主[5]。自1980年首次分离到登革3型病毒(dengue virus serotype 3,DENV3)以来,DENV3在广州销声匿迹近30年,直到2009年,才再次被分离到[6]。自2009年以来,几乎每年都能分离到DENV3。在2010年和2012年,DENV3还引发了小范围的流行。因此,通过对DENV3在广州市的输入和流行的分析,并结合病毒E基因序列和系统进化树分析,从分子水平追踪其可能的传染来源,分析广州市DENV3流行特点,为正确了解广州市登革热流行状况提供重要线索,为预测未来流行趋势提供科学依据。
1 材料与方法
1.1血清标本 广州市确诊的登革热病例急性期血清标本。确诊病例诊断标准参照《登革热诊断标准》(WS216-2008),国家卫生和计划生育委员会发布的《登革热诊疗指南》(2014年第 2 版)和中国疾病预防控制中心下发的《登革热病例监测指南》。
1.2核酸检测及病毒分离 使用江苏硕士生物科技有限公司生产的登革3型病毒核酸检测试剂(荧光PCR法),对确诊病例标本进行荧光检测及分型,并将阳性标本进行病毒分离。病毒分离方法参考已发表文献[7]。
1.3E基因RT-PCR扩增及序列测定 使用Primer软件,参考DENV3标准株H87设计引物(见表1)。收集病变细胞的上清液,按照文献方法提取登革病毒RNA,进行RT-PCR扩增,挖胶回收后,送上海英潍捷基公司测序[7]。
表1 登革3型病毒E基因扩增引物序列
Tab.1 Primers used for amplification of E genes of dengue virus serotype 3
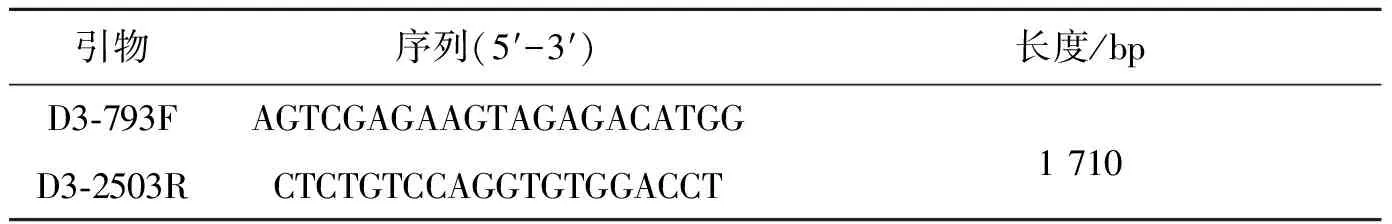
引物序列(5′-3′)长度/bpD3⁃793FAGTCGAGAAGTAGAGACATGG1710D3⁃2503RCTCTGTCCAGGTGTGGACCT
1.4E基因系统进化树分析 使用Mega 4.0软件,Kimura 2 parameter模型绘制基因系统进化树,步长设为1 000。
2 结 果
2.1登革病毒核酸检测结果 2001-2016年,用荧光PCR共检测到DENV3阳性60份。其中,2001-2008年确诊病例标本中,无DENV3核酸检测阳性标本。
2.2病毒分离结果 将60份荧光PCR检测DENV3阳性标本进行病毒培养,共分离到DENV3毒株24株,分离率为40.00%,2009年(7株),2010年(6株),2012年(9株),2015年(1株)和2016年(1株)。2009年和2010年分离到的毒株在以往发表的文章中已有提及[5]。分离到的24株DENV3毒株中,15株为本地病例标本中分离得到,占总毒株数的62.50%,9株为从输入标本中分离得到,占总毒株数的37.50%,输入国家如表2所示。
表2 2001-2016年登革3型病毒分离毒株按年统计表
Tab.2 Statistics of isolated dengue virus serotype 3 from 2001 to 2016

年份分离毒株数分离株中本地病例数分离株中输入病例数分离株输入国家参考文献2009743Vietnam,Thailand[5]2010624Tanzania,Thailand,Indonesia,Malaysia[5]2012981Malaysia本研究2015110None本研究2016101Malaysia本研究
2.4 病毒E基因特征
2.4.1核苷酸及氨基酸同源性比较 将分离到的24株DENV3的E基因进行扩增,测序后序列分析,核苷酸相似率为88.9%~100%,氨基酸相似率为92.2%~100%。分离株与其他地区分离到的DENV3如KT187290(马达加斯加2010年)、AB549332(坦桑尼亚2010年)、JF504679(中国浙江2009年)、KT452799(柬埔寨2010年)、AY858040(印度尼西亚2004年)、AB609590(菲律宾1956年)、以及JF968093(泰国2010年)有很高的相似性,核苷酸同源性为97.02%~99.80%,氨基酸同源性为92.56%~99.60%。每年的输入病例毒株与本地病例毒株的E基因序列有很高的相似率,如表3所示。
表3 输入病例毒株与本地病例毒株的E基因序列相似率
Tab.3 Similarity rate of E gene of local strains and imported strains
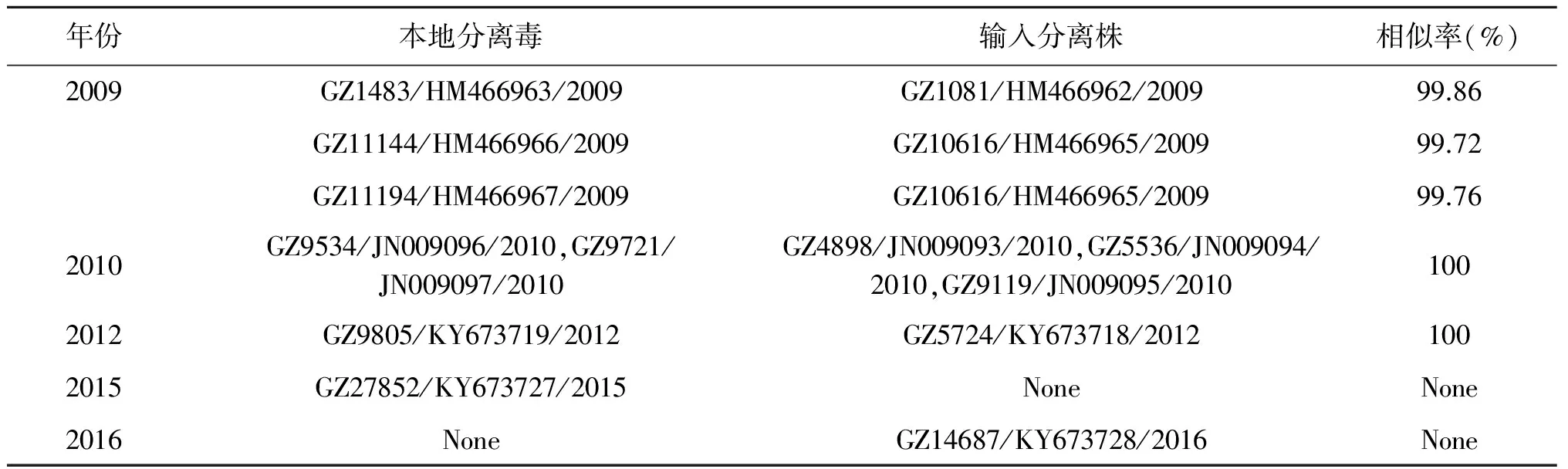
年份本地分离毒输入分离株相似率(%)2009GZ1483/HM466963/2009GZ1081/HM466962/200999.86GZ11144/HM466966/2009GZ10616/HM466965/200999.72GZ11194/HM466967/2009GZ10616/HM466965/200999.762010GZ9534/JN009096/2010,GZ9721/JN009097/2010GZ4898/JN009093/2010,GZ5536/JN009094/2010,GZ9119/JN009095/20101002012GZ9805/KY673719/2012GZ5724/KY673718/20121002015GZ27852/KY673727/2015NoneNone2016NoneGZ14687/KY673728/2016None
2.4.2E基因序列系统进化树分析 将分离到的24株病毒的E基因序列上传到National Center for Biotechnology Information (NCBI)。其中,2009-2010年分离到的13株毒株的E基因序列已在其他杂志发表[5](序列号为:HM466962-HM466968,JN009093-JN009098),其余2011-2016年分离的11株毒株的E基因序列为首次发表(序列号为:KY673718-KY673728)。将分离到的24株病毒与33条NCBI上的标准株序列及其他国家历年来获得的代表株序列的E基因进行分析比对,经Mega 4.0软件,构建系统进化树。按照基因亚型分类,DENV3可分为5个基因亚型[8],除基因亚型IV外,其余亚型在广州市2001-2016年均有发现,如图1所示。
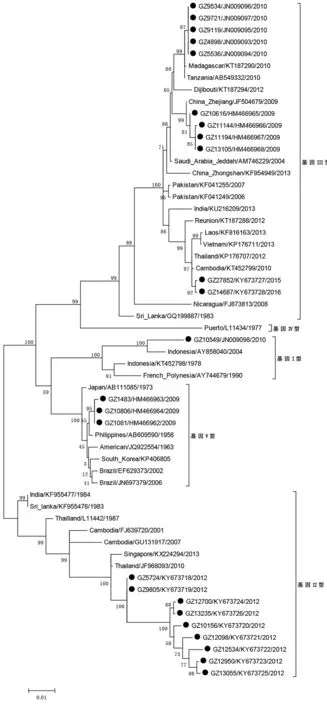
注:标注2001-2016年广州市分离的登革3型病毒图1 2001-2016年广州市登革3型病毒E基因系统进化树Fig.1 Phylogenetic tree of dengue virus serotype 3 in Guangzhou, 2001-2016
3 讨 论
与DENV1和DENV2相比,DENV3在广州市流行时间不长,流行年份也不多。2001-2008年,无DENV3核酸检测阳性和毒株分离。从1980年在广州市首次分离到DENV3,到2009年再次分离出DENV3,相隔近30年未分离到DENV3毒株,并且没有出现DENV3的流行[5, 7]。但在2009年之后,DENV3出现的周期明显缩短,并引发小范围的流行。DENV3的出现,使以DENV1流行为主的广州市登革热流行形势更加复杂和严峻。由于登革病毒存在抗体依赖的病毒感染增强作用(ADE,antibody-dependent enhancement),不同血清型登革病毒的二次感染更易引发重症登革热,进而可能导致广州市登革出血热或者登革休克综合征(DSS,dengue shock syndrome)患者数量的增加。因此,有必要加强对DENV3的监测和控制,防止DENV3的流行。
DENV3毒株输入的国家以东南亚为主。东南亚与广州贸易交往密切,东南亚旅游是近年来出国旅游热点,这为登革热的输入和传播提供了有利条件。除2015年外,每年本地DENV3病例出现之前,都有输入病例出现。并且通过流行病学调查证实,在2009年、2010年和2012年,输入病例引起二代本地病例,甚至在2010年和2012年引发本地DENV3小范围流行。对比输入病例毒株和本地病例毒株E基因,核苷酸的相似率达99.5%以上,进一步印证了输入病例引发本地病例的可能性。因此认为,广州市DENV3的流行仍以输入病例为主要来源,尚未观察到本地化的现象。这也与目前多数学者认为我国登革热流行方式属于输入病例引起本地暴发流行的观点相同[9]。自2009年来,DENV3的输入毒株和频率有增多的趋势。所以,增强出国人员的传染病防控意识,加强国境检验检疫,及时发现和诊断登革热病例,对登革热病例进行隔离和控制,对登革热的防控有重要意义。
DENV3可以根据E基因的核苷酸序列的同源性,进一步划分为5个基因亚型。基因亚型I-III主要与东南亚、印度次大陆、南太平洋、东非和美洲的登革热疫情有关;基因亚型IV和V基本无引起登革热疫情的报道,主要流行于美洲、南太平洋和亚洲[10]。从进化树上分析,在2001-2016年,广州市流行的DENV3的各种基因亚型交替出现,共计出现4个基因亚型。在引起本地DENV3小范围流行的2010年分离毒株以基因亚型III为主,2012年分离毒株以基因亚型II为主。总体来说,基因亚型III在流行年份和分离毒株数上稍占优势。基因亚型III由于分布的广泛性,被认为是一个全球型的基因亚型[10]。因此,基因亚型III在广州能否成为优势毒株,是否会本地化并引起流行,值得进一步的关注和研究。
2009年分离到的DENV3主要属于基因亚型III和基因亚型V,2010年出现基因亚型I,而2012年仅分离到基因亚型II毒株,2015年和2016年分离到的毒株又回归到基因亚型III。在不同的年份,广州流行的DENV3属于不同的基因亚型,揭示了DENV3可能有不同的输入来源。在一项关于DENV1抗体的研究中,79只小鼠感染基因亚型II毒株并产生抗体,结果仅有2只小鼠产生的抗体对DENV1的5个基因亚型都有保护作用[11]。同时有研究显示,DENV3流行株从基因亚型V向III的转换,很可能是导致1990年后印度登革热流行的原因[10]。这些结果与以往我们认为的,DENV感染产生的抗体对再次感染相同血清型的毒株有保护作用这一观点有所不同。感染DENV某一血清型的某一基因亚型所产生的抗体可能对同一血清型的不同基因亚型没有保护作用。这就为新基因亚型输入导致登革热的流行提供了可能性。同时基因亚型的增多,有可能使得不同毒株间发生基因交换,使本地DENV变异率增大,并导致自然选择出更有感染力的毒株[12]。因此,广州市DENV3基因亚型的增多和转换,使得广州市登革热流行传播更为复杂,流行风险进一步增高,防控压力增大。
参考文献:
[1] Li L, Lok SM, Yu IM, et al. The flavivirus precursor membrane-envelope protein complex: structure and maturation[J]. Science,2008,319(5871):1830-1834.DOI:10.1126/science.1153263
[2] 樊景春,林华亮,吴海霞,等. 广东省2006-2011年登革热时空分布特征[J]. 中国媒介生物学及控制杂志,2013,24(5):389-391.DOI:10.11853/j.issn.1003.4692.2013.05.003
[3] 郭汝宁,彭志强,宋铁,等. 广东省1990-2012年登革热和基孔肯雅热感染状况及流行风险分析[J]. 中华流行病学杂志,2014,35(2):167-169.DOI:10.3760/cma.j.issn.0254-6450.2014.02.014
[4] Mustafa MS, Rasotgi V, Jain S, et al. Discovery of fifth serotype of dengue virus (DENV-5): A new public health dilemma in dengue control[J]. Med J Armed Forces India,2015,71(1):67-70.DOI:10.1016/j.mjafi.2014.09.011
[5] Jiang L, Wu X, Wu Y, et al. Molecular epidemiological and virological study of dengue virus infections in Guangzhou, China, during 2001-2010[J]. Virol J,2013,10:4.DOI:10.1186/1743-422X-10-4
[6] Liang H, Luo L, Yang Z, et al. Re-emergence of dengue virus type 3 in Canton, China, 2009-2010, associated with multiple introductions through different geographical routes[J]. PLoS One,2013,8(2):e55353.DOI:10.1371/journal.pone.0055353
[7] 刘远,蒋力云,罗雷,等.广州市2001-2015年登革病毒2型E基因进化分析[J]. 中华流行病学杂志,2017,38(1):90-95.DOI:10.3760/cma.j.issn.0254-6450.2017.01.018
[8] Lanciotti RS, Lewis JG, Gubler DJ, et al. Molecular evolution and epidemiology of dengue-3 viruses[J]. J Gen Virol,1994,75 (1):65-75.DOI:10.1099/0022-1317-75-1-65
[9] 王金章,陈宏彬,张拥军,等. 2014年福建省南平市登革病毒E基因序列分析[J]. 中国人兽共患病学报,2016,32(3):281-285.DOI:10.3969/j.issn.1002-2694.2016.03014
[10] Sharma S, Dash PK, Agarwal S, et al. Comparative complete genome analysis of dengue virus type 3 circulating in India between 2003 and 2008[J]. J Gen Virol,2011,92(7):1595-1600.DOI:10.1099/vir.0.030437-0
[11] Shrestha B, Brien JD, Sukupolvi-Petty S, et al. The development of therapeutic antibodies that neutralize homologous and heterologous genotypes of dengue virus type 1: e1000823[J]. 2010,6(4).DOI:10.1371/journal.ppat.1000823
[12] Holmes EC, Burch SS. The causes and consequences of genetic variation in dengue virus[J]. Trends Microbiol,2000,8(2):74-77.DOI:http://dx.doi.org/10.1016/s0966-842X(99)01669-8
