两株布鲁菌MLST新序列型(ST)的鉴定
2018-05-28,,,,,,,,
,,, ,,,,,
布鲁菌病(以下简称布病)是由布鲁菌(Brucella)侵入机体引起的传染——变态反应性人兽共患传染病,在我国属于国家法定管理的乙类传染病,严重危害人类健康和畜牧业的发展。布鲁菌是一类兼性胞内寄生、革兰染色阴性的短小杆菌[1],以牛、羊、猪、犬等家畜和野生动物为主要储存宿主,也存在于海洋动物中,可经呼吸道、消化道、生殖系统黏膜以及皮肤等多种途径引起感染。人感染布鲁菌可累及全身多个组织器官,临床表现为发热、多汗、乏力、关节肌肉疼痛等[2-3],急性期可以治愈,但不易被确诊,常迁延为慢性感染,慢性期布病患者可有肝、脾及睾丸肿大,关节和脊柱强直,肌腱挛缩变硬等,很难治愈,如引起脑炎和心肌炎者可致命,严重影响人类健康[4]。动物感染后主要引起流产,造成畜牧业减产,进而导致极大的经济损失。布鲁菌易借助气溶胶的方式传播,呈高度传染性(仅吸入10-100个细菌即可导致人类患病[5]),因此布鲁菌被认为是一种潜在的生物战剂,已被列入《国际禁止生物武器公约》的核查清单,也是美国反生物恐怖袭击中的生物剂之一[1,6]。
近年来全球布病发病率上升,被WHO称为“再度肆虐”的传染病。我国自20世纪90年代中期以来,布病再度流行,疫情逐年上升,防控形势严峻。2000年以后,北京市布病疫情与全国一样,报告病例数呈逐年上升趋势,疫区人群感染率不断升高,并向普通人群扩散。此外,畜间疫情频发,并有局部暴发[7],防控形势非常严峻。了解布病病原特点和遗传特征是布病防控的基础,本项研究采用基于布鲁菌属外膜蛋白31基因的PCR(BCSP31-PCR)和基于插入序列IS711的牛、羊、绵羊附睾和猪种布鲁菌特异的PCR(AMOS-PCR)技术对2013年和2014年北京地区分离自布病患者血液的2株布鲁菌进行属和种(型)鉴定,采用多位点序列分型(Multiple locus sequence typing,MLST)技术对2株布鲁菌菌株的19个管家基因、1个外膜蛋白基因及1个基因间区的序列进行测定,将各个等位基因的序列与MLST数据库中的等位基因序列进行比对,确定菌株的等位基因谱及9位点序列型(sequence type,ST)和21位点序列型,分析其与布鲁菌不同ST型的遗传进化关系,为北京地区布病的防控提供科学依据。
1 材料与方法
1.1实验试剂 菌株培养基采用哥伦比亚血平板,购于英国OXOID公司,货号PB0123A。PCR试剂Platinum PCR SuperMix,购于美国Invitrogen公司,货号11306-016。核酸自动电泳用卡夹(QIAxcel DNA Screening Kit)与QX Alignment Marker 15bp/3Kb购自德国QIAGEN公司,货号分别为929004和929522。DL2,000bp DNA Marker购自宝生物工程(大连)有限公司,货号为D501A。引物由生工生物工程(上海)股份有限公司合成。
1.2实验菌株 实验菌株BJ3与BJ91为分离自北京地区2013年和2014年布病患者血液的分离株。
1.3菌株DNA制备 将分离菌株接种于哥伦比亚血平板,置于37 ℃生化培养箱,培养72 h后,刮取1~2接种环菌苔到150 μL去离子水中,振荡混匀制成菌悬液。再放入数控干浴器中,于100 ℃下煮沸10 min;12 000 r/min离心10 min,最后取上清液于-20 ℃冰箱中保存备用。
1.4BCSP31-PCR法鉴定布鲁菌属 采用布鲁菌属特异性基因BCSP31作为定属基因,按照参考文献[8-9]提供的B4和B5引物序列和参数进行布鲁菌属的鉴定。
1.5AMOS-PCR法鉴定布鲁菌种/型 采用参考文献[10-11]提供的以布鲁菌属IS711插入序列为基础建立的AMOS-PCR引物及参数进行布鲁菌种/型的鉴定。
1.6MLST分型 采用参考文献[12-13]提供的布鲁菌19个管家基因(gap、aroA、glk、dnaK、gyrB、trpE、cobQ、int-hyp、omp25、prpE、caiA、csdB、soxA、leuA、mviM、fumC、fbaA、ddlA、putA、mutL和acnA)、1个外膜蛋白基因(omp25)和1个基因间区int-hyp作为MLST的靶标基因,选取的靶标基因、引物序列及扩增长度见表1。PCR反应条件为:95 ℃预变性5 min;94 ℃变性30 s,63 ℃退火30 s,72 ℃延伸1 min,30个循环;72 ℃延伸10 min。ddlA基因扩增的退火温度为55 ℃,其他条件不变。PCR产物采用核酸自动电泳仪电泳检测,以DL 2 000 bp DNA Marker作为分子量标准。观察电泳结果判断PCR扩增是否成功。若PCR成功,直接将电泳检测后的样品委托生工生物工程(上海)股份有限公司进行纯化及测序。运用MLST在线工具(网址https://pubmlst.org/bigsdb?db=pubmlst_brucella_seqdef&page=sequenceQuery),分别将每株菌的21个特殊位点的测序结果与布鲁菌MLST标准菌株等位基因序列进行比对,明确序列是否与某个等位基因型的序列一致,如果一致,则定义为某个等位基因型,否则定义为一个新的等位基因型,从而获得每株菌的等位基因谱(gap/aroA/glk/dnaK/gyrB/trpE/cobQ/int-hyp/omp25/prpE/caiA/csdB/soxA/leuA/mviM/fumC/fbaA/ddlA/putA/mutL/acnA)。将得到的等位基因谱与布鲁菌MLST数据库标准菌株的等位基因谱进行比对,从而确定每株菌的ST型。
表1 用于扩增和测序分析的21对引物序列
Tab.1 21 Primer sets for amplificstion and sequencing
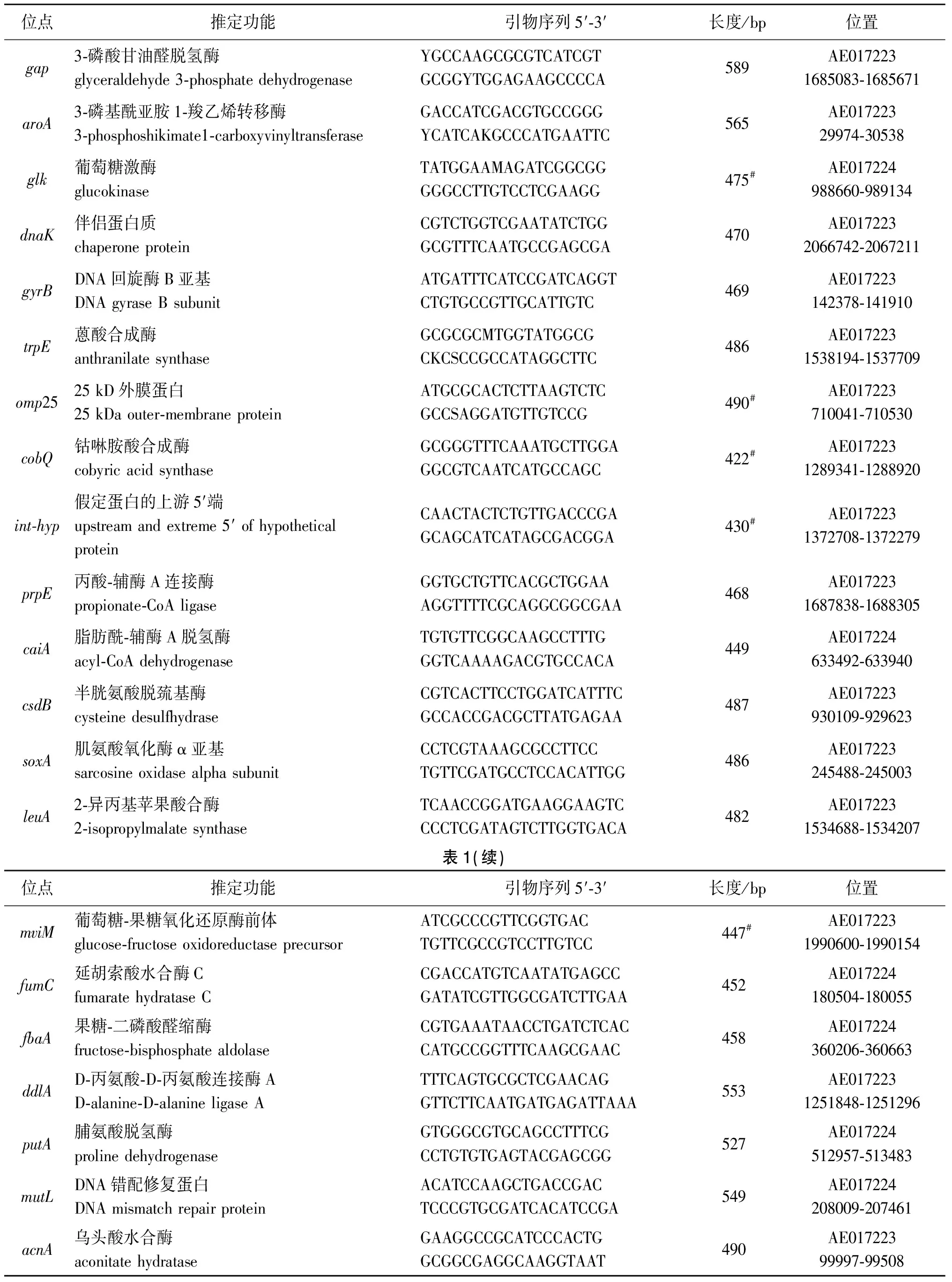
位点推定功能引物序列5′⁃3′长度/bp位置gap3⁃磷酸甘油醛脱氢酶glyceraldehyde3⁃phosphatedehydrogenaseYGCCAAGCGCGTCATCGTGCGGYTGGAGAAGCCCCA589AE0172231685083⁃1685671aroA3⁃磷基酰亚胺1⁃羧乙烯转移酶3⁃phosphoshikimate1⁃carboxyvinyltransferaseGACCATCGACGTGCCGGGYCATCAKGCCCATGAATTC565AE01722329974⁃30538glk葡萄糖激酶glucokinaseTATGGAAMAGATCGGCGGGGGCCTTGTCCTCGAAGG475#AE017224988660⁃989134dnaK伴侣蛋白质chaperoneproteinCGTCTGGTCGAATATCTGGGCGTTTCAATGCCGAGCGA470AE0172232066742⁃2067211gyrBDNA回旋酶B亚基DNAgyraseBsubunitATGATTTCATCCGATCAGGTCTGTGCCGTTGCATTGTC469AE017223142378⁃141910trpE蒽酸合成酶anthranilatesynthaseGCGCGCMTGGTATGGCGCKCSCCGCCATAGGCTTC486AE0172231538194⁃1537709omp2525kD外膜蛋白25kDaouter⁃membraneproteinATGCGCACTCTTAAGTCTCGCCSAGGATGTTGTCCG490#AE017223710041⁃710530cobQ钴啉胺酸合成酶cobyricacidsynthaseGCGGGTTTCAAATGCTTGGAGGCGTCAATCATGCCAGC422#AE0172231289341⁃1288920int⁃hyp假定蛋白的上游5′端upstreamandextreme5′ofhypotheticalproteinCAACTACTCTGTTGACCCGAGCAGCATCATAGCGACGGA430#AE0172231372708⁃1372279prpE丙酸⁃辅酶A连接酶propionate⁃CoAligaseGGTGCTGTTCACGCTGGAAAGGTTTTCGCAGGCGGCGAA468AE0172231687838⁃1688305caiA脂肪酰⁃辅酶A脱氢酶acyl⁃CoAdehydrogenaseTGTGTTCGGCAAGCCTTTGGGTCAAAAGACGTGCCACA449AE017224633492⁃633940csdB半胱氨酸脱巯基酶cysteinedesulfhydraseCGTCACTTCCTGGATCATTTCGCCACCGACGCTTATGAGAA487AE017223930109⁃929623soxA肌氨酸氧化酶α亚基sarcosineoxidasealphasubunitCCTCGTAAAGCGCCTTCCTGTTCGATGCCTCCACATTGG486AE017223245488⁃245003leuA2⁃异丙基苹果酸合酶2⁃isopropylmalatesynthaseTCAACCGGATGAAGGAAGTCCCCTCGATAGTCTTGGTGACA482AE0172231534688⁃1534207表1(续)位点推定功能引物序列5′⁃3′长度/bp位置mviM葡萄糖⁃果糖氧化还原酶前体glucose⁃fructoseoxidoreductaseprecursorATCGCCCGTTCGGTGACTGTTCGCCGTCCTTGTCC447#AE0172231990600⁃1990154fumC延胡索酸水合酶CfumaratehydrataseCCGACCATGTCAATATGAGCCGATATCGTTGGCGATCTTGAA452AE017224180504⁃180055fbaA果糖⁃二磷酸醛缩酶fructose⁃bisphosphatealdolaseCGTGAAATAACCTGATCTCACCATGCCGGTTTCAAGCGAAC458AE017224360206⁃360663ddlAD⁃丙氨酸⁃D⁃丙氨酸连接酶AD⁃alanine⁃D⁃alanineligaseATTTCAGTGCGCTCGAACAGGTTCTTCAATGATGAGATTAAA553AE0172231251848⁃1251296putA脯氨酸脱氢酶prolinedehydrogenaseGTGGGCGTGCAGCCTTTCGCCTGTGTGAGTACGAGCGG527AE017224512957⁃513483mutLDNA错配修复蛋白DNAmismatchrepairproteinACATCCAAGCTGACCGACTCCCGTGCGATCACATCCGA549AE017224208009⁃207461acnA乌头酸水合酶aconitatehydrataseGAAGGCCGCATCCCACTGGCGGCGAGGCAAGGTAAT490AE01722399997⁃99508
注:# 不同等位基因型有不同的片段长度
1.7聚类分析 应用eBURST软件将2株布鲁菌的ST型与数据库中的所有ST型进行聚类分析,探讨这两个ST型与已知ST型的进化关系。应用MegAlign软件对各ST型的核酸序列进行聚类分析,探讨各菌株间的进化关系。
2 结 果
2.1BCSP31-PCR鉴定 BCSP31-PCR方法对分离株和对照菌株核酸进行扩增,产物采用核酸自动电泳仪电泳进行检测,结果显示两株分离菌株及阳性对照菌株核酸均扩增出布鲁菌属特异的223 bp的条带,而阴性对照无条带出现。
2.2AMOS-PCR检测结果 AMOS-PCR方法对分离株和对照菌株核酸进行扩增,产物采用核酸自动电泳仪电泳进行检测,结果显示阳性羊种对照菌株核酸可扩增出178 bp的条带和羊种布鲁菌特异的731 bp的条带,2株分离菌株只有178 bp条带,此外无其他条带,而阴性对照无条带出现。
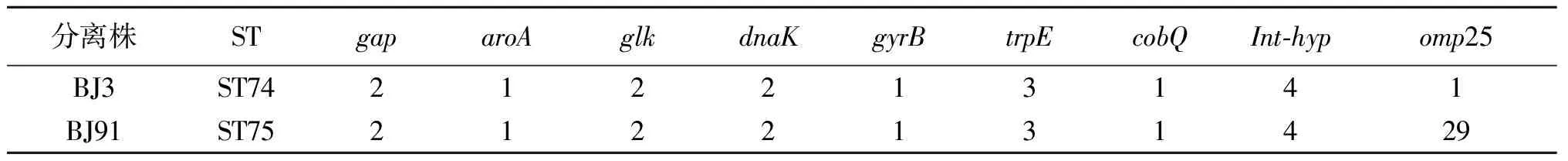
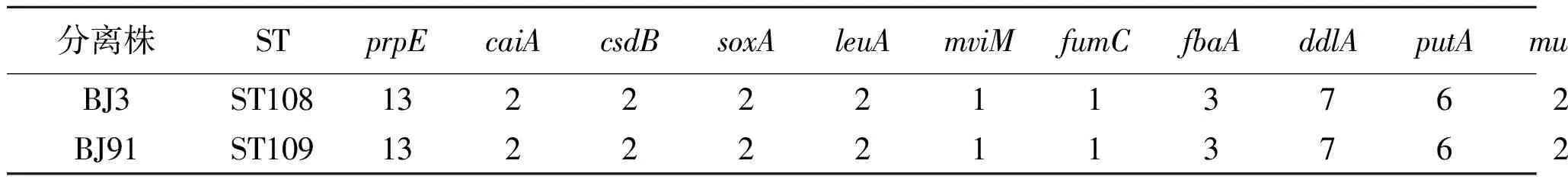
2.3MLST分型结果 2株菌的19个管家基因、1个外膜蛋白基因(omp25)和1个基因间区int-hyp的扩增片段测序结果与与布鲁菌MLST数据库等位基因信息比对,得到这2株分离株的21个位点(gap/aroA/glk/dnaK/gyrB/trpE/cobQ/int-hyp/omp25/prpE/caiA/csdB/soxA/leuA/mviM/fumC/fbaA/ddlA/putA/mutL/acnA)的等位基因序列号分别为2、1、2、2、1、3、1、4、1、13、2、2、2、2、1、1、3、7、6、2、3和2、1、2、2、1、3、1、4、29、13、2、2、2、2、1、1、3、7、6、2、3(表2,表3)。比对结果显示,这2株布鲁菌的9位点等位基因编号和21位点基因编号均为新组合,即新ST。
我们将这2株布鲁菌的资料及其各等位基因序列编号通过互联网录入布鲁菌MLST数据库,已被接收并确认为新ST,命名为ST74和ST75(9位点ST)与ST108和ST109(21位点ST)。
表2 2株分离株的等位基因谱(9位点ST)
Tab.2 Allele profile of 2 isolates (9 loci ST)
分离株STgaparoAglkdnaKgyrBtrpEcobQInt⁃hypomp25BJ3ST74212213141BJ91ST752122131429
表3 2株分离株的等位基因谱(21位点ST的其他12位点)
Tab.3 Allele profile of 2 isolates (addtional 12 loci of 21 loci ST)
分离株STprpEcaiAcsdBsoxAleuAmviMfumCfbaAddlAputAmutLacnABJ3ST1081322221137623BJ91ST1091322221137623
2.4聚类分析结果 聚类分析显示,迄今已发现的73个9位点MLST分型的ST型所包含的布鲁菌10个种呈现出相同种ST型菌株聚集较紧密,不同种之间遗传距离及亲缘关系较远的现象,这2株菌的9位点ST与其它几个均属于牛种布鲁菌的ST型聚在一起,与ST2高度同源,遗传关系较近,ST74与ST2只有1个等位基因int-hyp的差异,ST75与ST2有2个等位基因int-hyp、omp25的差异,提示ST74、ST75与ST2的亲缘关系较近,ST2可暂定为1个可行度较高的始祖ST型(图1)。运用MegAlign软件(By ClustalW Method)对各ST型9个位点的核酸序列进行聚类分析,结果显示ST74、ST75型与属于牛种布鲁菌的其他18个ST型聚在一起(图2),对各ST型21个位点的核酸序列进行聚类分析,结果显示ST108、ST109型与属于牛种布鲁菌的其他30个ST型聚在一起(图3),提示这2株菌为牛种布鲁菌,与AMOS-PCR结果吻合。
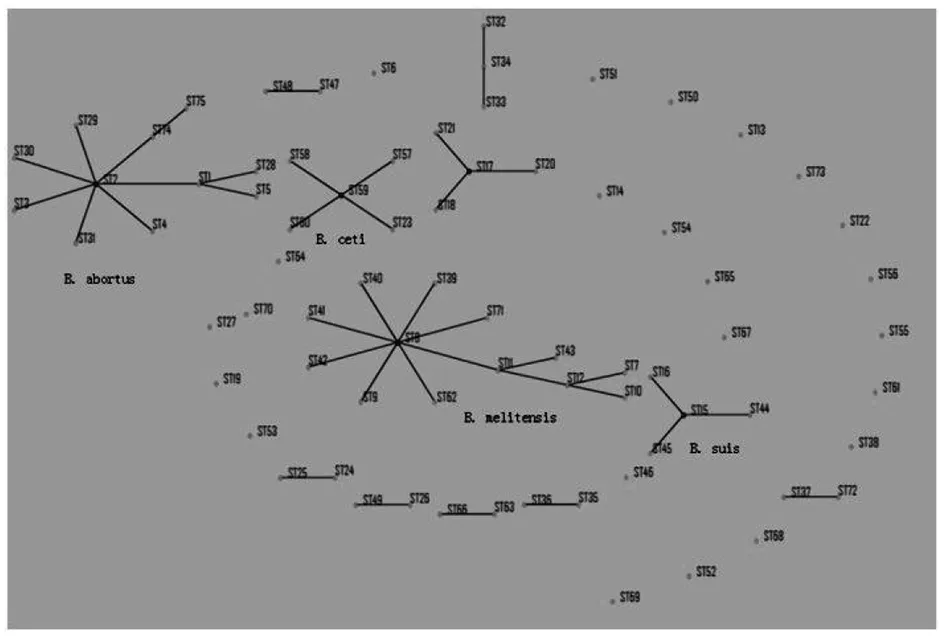
图1 2种新ST型与已知布鲁菌ST型的eBURST分析结果Fig.1 eBURST analysis of 2 novel STs and the known STs
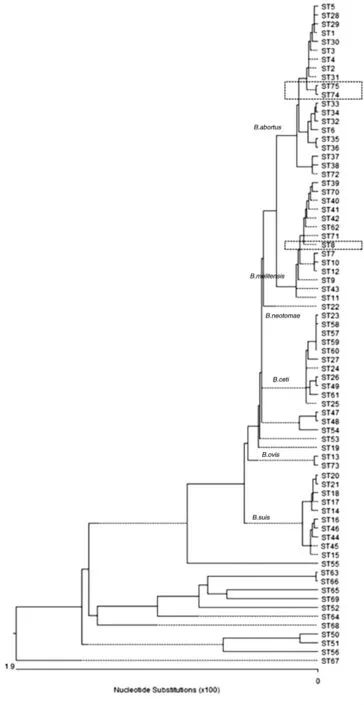
图2 2种新ST型与已知布鲁菌ST型间的进化关系(9位点ST)Fig.2 Phylogenetic tree of 2 novel STs and the known STs (9 loci ST)
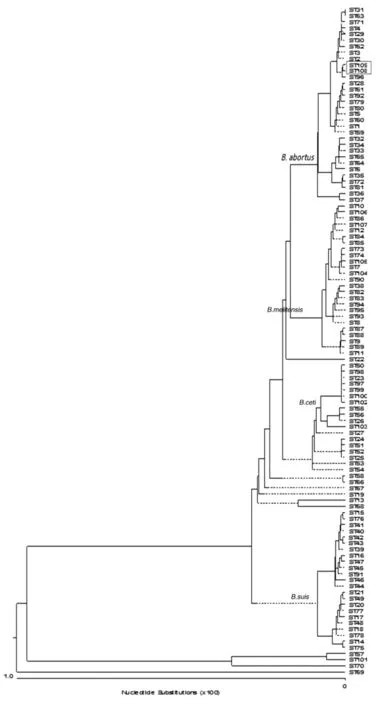
图3 2种新ST型与已知布鲁菌ST型间的进化关系(21位点ST)Fig.3 Phylogenetic tree of 2 novel STs and the known STs (21 loci ST)
3 讨 论
1985年WHO根据在细菌培养过程中的生化以及噬菌体裂解和血清凝集特性,将布鲁菌分为羊种布鲁菌、牛种布鲁菌、猪种布鲁菌、绵羊附睾种布鲁菌、沙林鼠种布鲁菌和犬种布鲁菌共6个种19个生物型,此为布鲁菌的经典分型。之后又陆续发现了4种新型布鲁菌。目前认为,布鲁菌有10个种,它们分别为马耳他布鲁菌(羊种布鲁菌B.melitensis,3个生物型)、流产布鲁菌(牛种布鲁菌B.abortus,8个生物型)、猪种布鲁菌(B.suis,5个生物型)、绵羊附睾种布鲁菌(B.ovis,1个生物型)、沙林鼠种布鲁菌(B.eotomae,1个生物型)、犬种布鲁菌(B.canis,1个生物型),从海洋哺乳动物如海豹、鲸、海豚等中分离出的鲸型布鲁菌(B.ceti)[14]和鳍型布鲁菌(B.pinnipedialis)[15],从野鼠中分离出的田鼠型布鲁菌(B.microti)[16],在人乳房植入体中发现的湖浪布鲁菌(B.inopinata)[17]。其中4个种(羊种B.melitensis,牛种B.abortus,猪种B.suis和犬种B.canis)对人致病,致病作用大小顺序一般是羊种大于牛种、猪种,大于犬种。布鲁菌因其生物种、型和菌株的不同,致病力各异,对人致病力最强的为羊种菌,其次为猪种菌,牛种菌对人致病力弱,但亦时有病例发生。因此,了解布病病原学特征对布病的有效预防和控制至关重要。本研究将来源于患者的2株菌株采用属特异性BCSP31-PCR进行鉴定,结果显示这两株分离的可疑菌株均为布鲁菌。AMOS-PCR结果只有178 bp条带而无其他大小的目的条带出现,提示分离菌株为为非羊种、非牛种1、2、4型、非猪种1型、非绵羊附睾种布鲁菌。
传统血清学方法鉴定只能将布鲁菌鉴定到种(型),无法对菌株进行溯源分析。随着分子生物学的发展,一些分子分型方法不断出现,如PCR产物限制性片段长度多态性(PCR-RFIP)、随机扩增多态性DNA(RAP-PCR)、扩增片断长度多态性分析(AFLP)、脉冲场凝胶电泳(pulsed-field gel electrophoresis,PFGE)、多位点可变数目串联重复序列分析(multi-locus variable-Humber tandem repeat analysis,MLVA-16)技术和多位点序列分型(multi-locus sequence typing,MLST)技术等,但是PCR-RFIP、RAP-PCR和AFLP分辨率较低,在分子流行病学中应用价值十分有限。由于布鲁菌属种间DNA同源性高达90%,需要高分辨力的分型方法用来提供疾病传播模式和分子流行病学信息。PFGE、MLVA和MLST在布病病的病原鉴定、传染源追踪与病原体的溯源等方面发挥了重要作用。但PFGE实验过程中活菌操作繁多,实验室感染风险较大。相比MLVA-16,MLST在反映菌株遗传特征方面更加敏感[18],分辨率高,重复性好,数据可实现网络共享,不同的实验室之间具有良好的可比性[19]。2007年Whatmore等[12]根据7个管家基因、1个外膜蛋白基因及1个基因间区设计引物,对160株布鲁菌进行MLST分析,试验将160株布氏菌分为27个序列型。为了提高分辨率,Whatmore[13]等又新增了12个靶标基因,加上之前的9个特殊位点,形成了BruMLSA21分型方案。对收集自全球的500多株布鲁菌进行MLST分析,结果分了101个序列型。此后不断有新的ST型被发现,数据库不断丰富,目前9位点序列型有73个ST型,21位点序列型有107个ST型。因此,本研究中我们采用布鲁菌MLST分子分型方法对2013年和2014年分离自北京地区布病患者的2株菌株进行分子分型分析并与已发现的ST型进行比对,结果发现2株菌株的等位基因谱与目前已有的ST型的等位基因谱不完全一致,为新的ST型。
MLST数据库(https://pubmlst.org/brucella/)是国际上公认的登载布鲁菌序列分型信息的重要数据库,注册登录后将发现的2种新ST型菌株的等位基因谱信息提交,被接受并予以命名,现该库关于Brucella的9位点ST更新为75种,21位点ST更新为109种。本研究中2株菌的ST分别被命名为ST74、ST75(9位点MLST)和ST108、ST109(21位点MLST)。ST74与ST2只有1个等位基因int-hyp的差异,ST75与ST2有2个等位基因int-hyp、omp25的差异,见表2。为了进一步分析新发现的ST型,利用eBURST工具分析目前布鲁菌数据库中所有的73个ST型和新发现的ST型,并绘制出各ST型之间的关系图。eBURST(Based Upon Related Sequence Type)是一种基于相关序列型的聚类分析方法。根据细菌的所有序列型的等位基因谱将细菌分为不同的组(group),计算每个序列型的单位点变异型SLV(Single Locus Variants)个数、双位点变异型DLV(Double Locus Variants)个数,鉴定可能的祖先序列型等。当2个ST型之间只有1个等位基因数字不同时,这2个ST型互为单位点变异型SLV;如果有2个等位基因数字不同,这2个ST型则互为双位点变异型DLV[20]。互为单位点变异型的序列型构成一组,若该组有足够多的ST型且其中的1个ST拥有最多数量的SLV,该ST型则可以暂定为1个可行度较高的始祖ST型。eBURST分析显示(图1):迄今已发现的73个ST型所包含的布鲁菌10个种呈现出相同种菌株的ST型聚集较紧密,而不同种之间遗传距离及亲缘关系较远的现象;以ST2、ST8、ST15、ST17、ST59各自为中心聚集在一起的ST型的亲缘关系较近,这样的菌株称为克隆群(或称克隆复合体clonal complexes),分别为牛种、羊种、猪种、猪种和鲸型。ST74、ST75,ST108、ST109与其它几个均属于牛种布鲁菌的ST型聚在一起,与ST2高度同源,遗传关系较近,从遗传学水平证实了这2株布鲁菌为两种新ST型牛种布鲁菌。ST2可暂定为1个可行度较高的始祖ST型。运用MegAlign软件(By Clustal W Method)对各ST型9个位点的核酸序列进行聚类分析,探讨不同ST型间的进化关系。结果显示(图2):羊种布鲁菌几乎所有的ST型(除ST70以外)均处于同一个进化分支上,表现的高度集中,说明羊种布鲁菌是一种高度保守的布鲁菌,不同ST型间变异少,而国内发现最多羊种ST型为ST8[21-22]。新发现的ST74、ST75型与ST2型遗传关系很近,处于同一个进化层次。ST74、75型与属于牛种布鲁菌的其他18个ST型聚在一起,21位点ST型的进化树分析显示ST108、109型与属于牛种布鲁菌的其他30个ST型聚在一起(图3),提示这2株分离株为牛种布鲁菌,与AMOS-PCR结果吻合。
近年来我国布病再度流行,防控形势依然严峻。2000年以后,北京市布病疫情与全国一样,报告病例数呈逐年上升,并由高危人群并向普通人群扩散。此外,畜间疫情频发,并有局部暴发[7],防控形势非常严峻。本研究结果显示MLST分型具有较好的分辨率,能够与其它方法互为补充,对布病的感染溯源具有重要作用,研究结果不仅充实了布鲁菌MLST数据库,也为北京地区布病的防控提供了科学依据,在保护人群健康和保障畜牧业发展方面具有重要意义。
参考文献:
[1] Doganay GD, Doganay M.Brucellaas a potential agent of bioterrorism[J]. Recent Pat Antiinfect Drug Discov, 2013, 8(1): 27-33. DOI: 10.2174/1574891X11308010006
[2] Troy SB, Rickman LS, Davis CE. Brucellosis in San Diego: epidemiology and species-related differences in acute clinical presentations[J]. Medicine (Baltimore), 2005, 84(3): 174-187.
[3] Hasanjani Roushan MR, Ebrahimpour S. Human brucellosis: An overview[J]. Caspian J Intern Med, 2015, 6(1): 46-47. DOI: 10.1097/01.md.0000165659.20988.25
[4] Franco MP, Mulder M, Gilman RH, et al. Human brucellosis[J]. Lancet Infect Dis, 2007,7(12): 775-786. DOI: 10.1016/S1473-3099(07)70286-4
[5] Bellamy RJ, Freedman AR. Bioterrorism[J]. Qjm, 2001, 94(4): 227-234.
[6] Santis RD, Ciammaruconi A, Pomponi A, et al.Brucella: Molecular diagnostic techniques in response to bioterrorism threat [J]. J Bioterr Biodef, 2011, S2: 004. DOI: 10.4172/2157-2526.S2-004
[7] 李锡太, 何战英, 陈艳伟,等. 运用集中度与圆形分布法分析北京市布鲁氏菌病时间分布特征[J]. 公共卫生与预防医学, 2016, 27(1):20-23.
[8] Baily GG, Krahn JB, Drasar BS et al. Detection ofBrucellamelitensisandBrucellaabortusby DNA amplification[J]. J Trop Med Hyg, 1992, 95(4): 271-275.
[9] Garshasbi M, Ramazani A, Sorouri R, et al. Molecular detection ofBrucellaspecies in patients suspicious of brucellosis from Zanjan, Iran[J]. Braz J Microbiol, 2014, 45(2): 533-538.
[10] Bricker BJ, Halling SM. Differentiation ofBrucellaabortusbv. 1, 2, and 4,Brucellamelitensis,Brucellaovis, andBrucellasuisbv. 1 by PCR[J]. J Clin Microbiol, 1994, 32(11): 2660-2666.
[11] Kang YX, Li XM, Piao DR, et al. Typing discrepancy between phenotypic and molecular characterization revealing an emerging biovar 9 variant of smooth phage-resistantB.abortusstrain 8416 in China[J]. Front Microbiol, 2015, 6(514): 1375. DOI: 10.3389/fmicb.2015.01375
[12] Whatmore AM, Perrett LL, MacMillan AP. Characterisation of the genetic diversity ofBrucellaby multilocus sequencing[J]. BMC Microbiol, 2007, 7(1): 34. DOI: 10.1186/1471-2180-7-34
[13] Whatmore AM, Koylass MS, Muchowski J, et al. Extended multilocus sequence analysis to describe the global population structure of the genusBrucella: phylogeography and relationship to biovars[J]. Front Microbiol, 2016, 7: 2049. DOI: 10.3389/fmicb.2016.02049
[14] Foster JT, Okinaka RT, Svensson R, et al. Real-time PCR assays of single-nucleotide polymorphisms defining the major Brucella clades[J]. J Clin Microbiol, 2008, 46(1): 296-301. DOI: 10.1128/JCM.01496-07
[15] Foster G, Osterman BS, Godfroid J, et al.Brucellaceti sp. nov. andBrucellapinnipedialissp. nov. forBrucellastrains with cetaceans and seals as their preferred hosts[J]. Int J Syst Evol Microbiol, 2007, 57(Pt 11): 2688-2693. DOI: 10.1099/ijs.0.65269-0
[16] Scholz HC, Hubalek Z, Sedlacek I, et al.Brucellamicrotisp. nov., isolated from the common vole Microtus arvalis[J]. Int J Syst Evol Microbiol, 2008, 58(Pt 2): 375-382. DOI: 10.1099/ijs.0.65356-0
[17] Scholz HC, Nockler K, Gollner C, et al.Brucellainopinatasp. nov., isolated from a breast implant infection[J]. Int J Syst Evol Microbiol, 2010, 60(Pt 4): 801-808. DOI: 10.1099/ijs.0.011148-0
[18] 李沛丽, 李世军, 刘英,等. 贵州省两株山羊源羊种布鲁菌MLST分析[J]. 中国人兽共患病学报, 2016, 32(6):547-552.DOI:10.3969/j.issn.1002-2694.2016.06.008
[19] Vázquez JA, Berrón S. Multilocus sequence typing: the molecular marker of the Internet era[J]. Enfermedades Infecciosas Y Microbiología Clínica, 2004, 22(2): 113-120. DOI: 10.1016/S0213-005X(04)73045-1
[20] 赵宏, 谌志强, 李君文.不同地理来源的食品中大肠埃希氏菌eBURST分析[J]. 解放军预防医学杂志, 2012, 30(5):325-328.
[21] Ke Y, Yuan X, Zhen Q, et al. Genome sequence ofBrucellamelitensisS66, an isolate of sequence type 8, prevalent in China[J]. J Bacteriol, 2012, 194(19): 5451. DOI: 10.1128/JB.01202-12
[22] Chen Y, Ke Y, Wang Y, et al. Changes of predominant species/biovars and sequence types ofBrucellaisolates, Inner Mongolia, China[J]. BMC Infect Dis, 2013, 13(1): 514. DOI: 10.1186/1471-2334-13-514
