鼠肠道病毒RT-PCR检测与基因序列分析
2018-05-28,,
,,
肠道病毒属(Enterovirus, EV)属于微小RNA病毒科(Picornaviridae),包括脊髓灰质炎病毒(Poliovirus,PV)、埃柯病毒(Echovirus,ECHO)、柯萨奇病毒(Coxsackie,CA)、鼻病毒(Rhinovirus, Rhi)、新型肠道病毒[1-3]等。近年来,由肠道病毒属病毒引的手足口病、疱疹性咽峡炎、无菌性脑膜炎等传染病在世界各地广泛散发或流行,带来了严重的公共卫生问题,引起了社会和公众的广泛关注[4-5]。
鼠类以储存宿主或中间宿主的形式传播各类疾病,是重要的医学媒介生物,已知全球由鼠类传播的疾病的病原体可以分为病毒、立克次体、螺旋体、细菌、寄生虫等5大类[6]。加强以鼠类等医学媒介生物及其携带病原体监测,已经成为口岸和地方卫生防疫部门的共识。但是目前尚未见到对鼠类携带肠道病毒属病毒及其传播风险的相关研究报道。
本研究选取GenBank公布的不同种肠道病毒属病毒全基因组多聚蛋白(genome polyprotein)氨基酸序列,用CODEHOP(consensus degenerate hybrid oligonucleotide primers)在线软件设计一对肠道病毒属一致-简并引物,以宁波口岸地区媒介监测捕获的165只鼠类样品为模板,提取鼠肠内容物RNA进行肠道病毒属病毒筛查,并对获得的PCR产物进行测序和遗传学分析。
1 材料与方法
1.1样品来源 共检测2016年从宁波港区捕获的鼠类样品165只,其中黑线姬鼠(Apodemusagrarius)80只、黄毛鼠(Rattuslosea)41只、褐家鼠(Rattusnorvegicus)16只、臭鼩(Suncusmurinus)13只、小家鼠(Musmusculus)9只,黄胸鼠(Rattusflavipetcus)6只。
1.2试剂 PrimeScriptTMOne Step RT-PCR Kit、100 bp DNA Ladder marker、PBS缓冲液、琼脂糖购自宝生物工程(大连)有限公司;总RNA 小量提取试剂盒(RNeasy mini kit)购自德国Qiagen公司。
1.3 方法
1.3.1病毒RNA提取 无菌条件下取得约50 mg鼠类肠道内容物样品,加入1 mL 1×PBS缓冲液,放入组织研磨仪进行研磨。取200 μL匀浆液,参照Qiagen公司RNA提取试剂盒使用说明书提取病毒RNA,用40 μL无RNA酶的超纯水溶解,于-80 ℃保存备用。
1.3.2引物合成 依据GenBank获得的不同肠道病毒属病毒代表株的多聚蛋白氨基酸序列,以CODEHOP程序设计并筛选出一对肠道病毒属一致-简并引物E4559/FE4937。依照Kiang D等人文章合成鼻病毒属通用引物序列DK001/DK004[7]。引物序列见表1。
表1 引物序列
Tab.1 Primers sequence

引物序列目的序列片段大小/bpE4559CAGATCCAGAT⁃CATTTTGATGG⁃ATAYRANCARCAEnterovirus2B378FE4937TGTTCTTCTATC⁃CATAAATTGAATTG⁃CYTTNCCRCANADK001CAAGCACTTCT⁃GTTTCCCHRV5′UTR390DK004CACGGACAC⁃CCAAAGTAGT
1.3.3一步RT-PCR反应体系 肠病毒一致-简并引物扩增采用50 μL反应体系: 5× One step RT-PCR Buffer 10 μL; dNTP Mixture (10 mmol/L) 2 μL,Enzyme Mix 2 μL,Ribonuclease Inhibitor 1 μL, 上游引物E4559 (20 μmol)/L 4 μL,下游引物FE4937(20 μmol)/L 2 μL,RNA模板10 μL,RNase Free Water 19 μL。反应程序为:60 ℃ 1 min,42 ℃ 10 min,50 ℃ 30 min,95 ℃ 15 min;94 ℃ 30 s,52 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 7 min,4 ℃保存。
鼻病毒通用引物引物扩增采用50 μL反应体系: 5× One step RT-PCR Buffer 10 μL; dNTP Mixture (10 mmol/L) 2 μL,Enzyme Mix 2 μL,Ribonuclease Inhibitor 1 μL, 上游引物DK001 (20 μmol)/L 3.75 μL,下游引物DK004(20 μmol)/L 3.75 μL,RNA模板10 μL,RNase Free Water 17.5 μL。反应程序为:60 ℃ 1 min,42 ℃ 10 min,50 ℃ 30 min,95 ℃ 15 min;94 ℃ 30 s,58 ℃ 30 s,72 ℃ 30 s,35个循环;72 ℃ 7 min,4 ℃保存。
1.3.4扩增产物序列分析 将扩增产物用1.5%的琼脂糖凝胶电泳后,凝胶成像系统观察扩增结果,将扩增产物送往上海英俊生物工程公司进行测序,将获得的基因片段测序与GenBank报道常见肠道病毒流行株进行同源性比较,并利用Mega5.2软件构建系统发生树。
2 结 果
2.1肠病毒属一致-简并引物RT-PCR检测 2份从黄毛鼠肠道内容物样品中提取的RNA检测结果为阳性,黄毛鼠病毒阳性感染率达到4.9%,其余样品的反应结果为阴性。扩增产物电泳后可见特异性扩增条带,片段大小约为378 bp与预期一致,见图1。
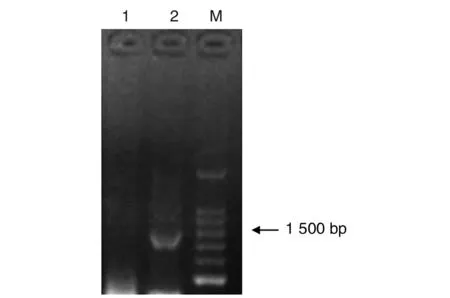
1:鼠肠道阳性样品RT-PCR扩增产物;2:阳性对照EV71病毒 RT-PCR扩增产物;M.100 bp DNA Ladder图1 肠道病毒一致-简并引物RT-PCR样品扩增电泳图Fig.1 Electrophoresis image of samples amplified by RT-PCR using enterovirus CODEHOP primers
2.2测序及遗传距离分析 2份阳性样品扩增产物进行基因测序,序列拼接校正后大小与预期结果一致,2份阳性样品基因序列相同,命名为DXEV1601。经比较与国内外23个不同肠道病毒流行株同源性相似度为50.3%~59.6%,其中与人鼻病毒C NAT001同源性为59.6%相对最为接近,见表2。
表2 DXEV1601与不同肠道病毒流行株部分基因序列的同源性比较
Tab.2 Homology comparison on partial nucleotide sequence of DXEV1601 to the different enterovirus strains
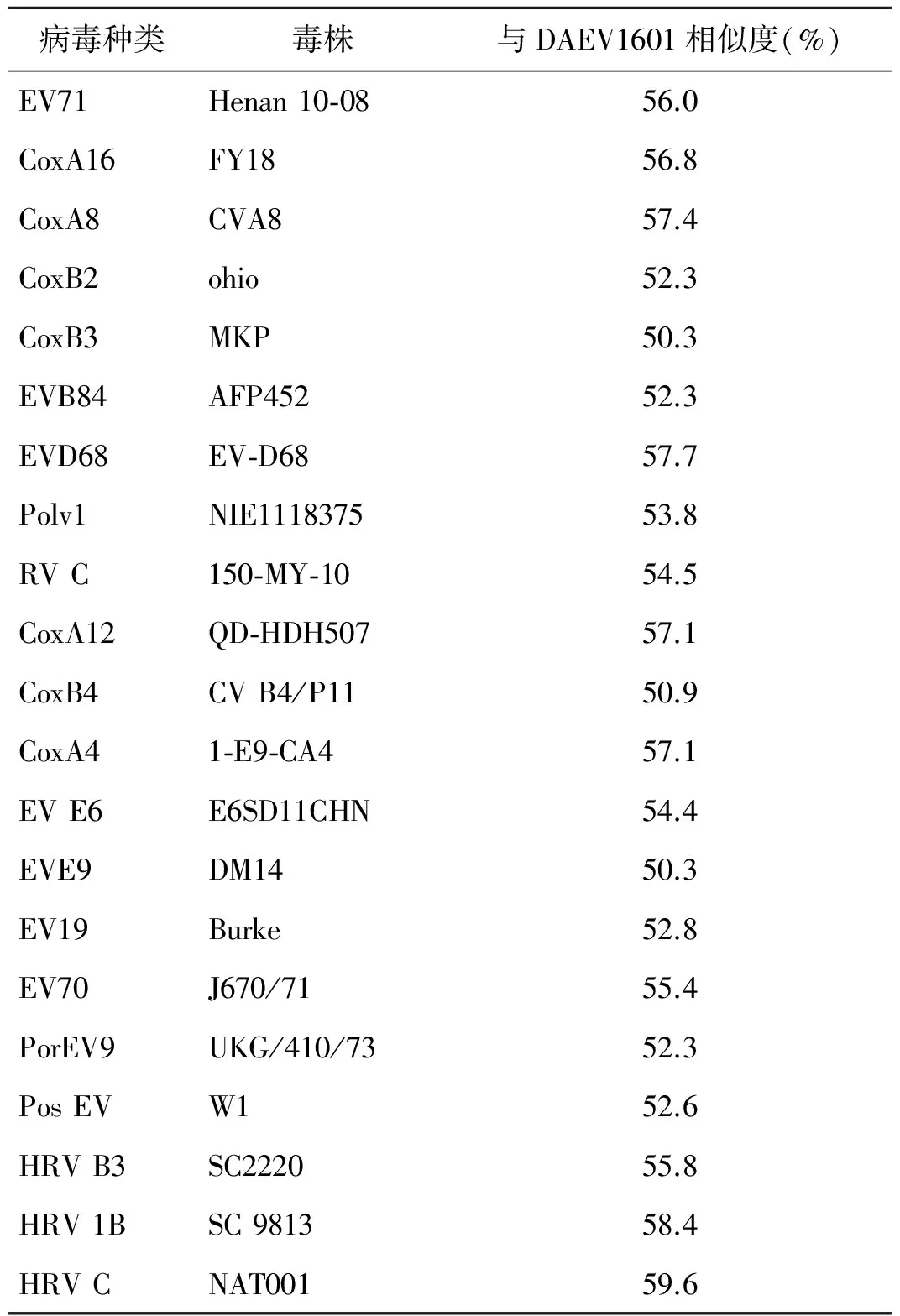
病毒种类毒株与DAEV1601相似度(%)EV71Henan10⁃0856.0CoxA16FY1856.8CoxA8CVA857.4CoxB2ohio52.3CoxB3MKP50.3EVB84AFP45252.3EVD68EV⁃D6857.7Polv1NIE111837553.8RVC150⁃MY⁃1054.5CoxA12QD⁃HDH50757.1CoxB4CVB4/P1150.9CoxA41⁃E9⁃CA457.1EVE6E6SD11CHN54.4EVE9DM1450.3EV19Burke52.8EV70J670/7155.4PorEV9UKG/410/7352.3PosEVW152.6HRVB3SC222055.8HRV1BSC981358.4HRVCNAT00159.6
2.3种系进化分析 DXEV1601使用肠病毒属CODEHOP引物扩增片段大小为378 bp,使用Mega5.0软件对其和其他肠病毒属病毒进行进化树分析,结果见图2。DXEV1601与人鼻病毒1B SC9813株在一个分支上,并所处鼻病毒分支上。
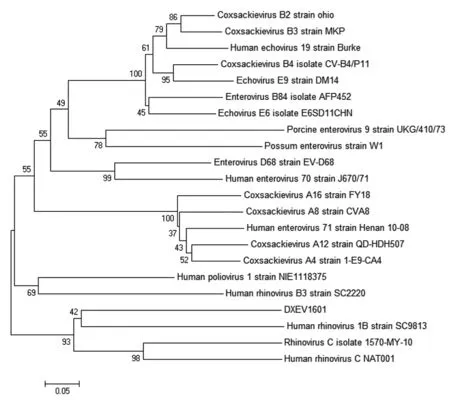
图2 肠道病毒部份基因序列构建的系统发生树Fig.2 Phylogenic tree of enterovirus partial nucleotide sequences
2.4鼻病毒通用引物RT-PCR检测 对DXEV1601样品使用鼻病毒通用引物扩增,扩增产物电泳后可见特异性扩增条带,片段大小约为349 bp与预期一致,而对照EV71病毒无特异性条带,与预期相符,见图2。

1:样品DXEV1601 RT-PCR扩增产物;2:EV71病毒 RT-PCR扩增产物;M:100 bp DNA Ladder图3 鼻病毒通用引物RT-PCR电泳图Fig.3 Electrophoresis image of samples amplified by RT-PCR using rhinovirus universal primers
3 讨 论
肠道病毒的基因组为7.4 kb的单股正链RNA,仅含一个开放读码框(open reading frame, ORF),两端为保守的非编码区(untranslated region,UTR)。ORF由编码病毒结构蛋白VP1、VP2、VP3、VP4的基因和非结构蛋白编码区2A、2B、2C、3A、3B、3C、3D组成,翻译产物为一个大分子的多聚蛋白[8-9]。本研究采用的一致-简并引物是依据随机GenBank公布的不同种肠道病毒的多聚蛋白氨基酸序列,通过CODEHOP程序设计,该引物可与GenBank公布肠道病毒流行株目的基因序列匹配,因此理论上本研究所设计的CODEHOP引物可用于肠道病毒属内各种病毒的检测。
传统上根据病毒在不同动物种类上的感染性、致病性和抗原性差异,将肠道病毒分为脊髓灰质炎病毒(Poliovirus, PV 1-3型)、柯萨奇病毒(Coxsackie virus, Cox)A组(CV A1-22和24型)和B组(CV B1-6型)、埃柯病毒(Echovirus, ECV 1-7、9、11-27、29-33)、鼻病毒(Rhinovirus, Rhi)[10]。随着PCR和基因测序技术等分子生物学技术的发展,目前已报道的肠道病毒基因型已超过百种[11]。基于肠道病毒VP1的简并引物RT-PCR和核苷酸序列分析,已成为肠道病毒基因分型的主要方法[12]。本研究采用了CODEHOP方法设计肠道病毒属的一致简并引物,与常规简并引物相比具有简并度低、退火温度高、特异性更强等优势[13]。另外,本研究所设计的引物位于肠道病毒非结构蛋白编码区2B区域,该区域的检测和分型未见有国内外的研究报道。本研究初步证实了宁波口岸分布中黄毛鼠中存在鼻病毒感染。
从不同肠道病毒流行株基因片段序列同源性比较可知,DXEV1601与其他肠道病毒属内病毒株的同源性相似度相对较低位于50.3%~59.6%,处于不同肠道病毒的同源性相似度(48.9%~90.3%)的范围之内。从肠道病毒部分基因序列构建的系统进化树看,鼠样品中检测到的病毒DXEV1601与鼻病毒1B SC9813株位于同一进化分支,遗传关系最为接近。同时使用鼻病毒通用引物进行验证,确证分离到的DXEV1601为鼻病毒。
参考文献:
[1] Fei Z, Fanrong K, Kenneth M, et al. Molecular identification and analysis of nonserotypeable human enteroviruses[J]. J Clin Microbiol, 2010, 48(4): 1276-1282. DOI: 10.1128/JCM.02384-09
[2] Tao Z, Cui N, Lin X, et al. Complete genome sequence analysis of an enterovirus 75 isolate from China[J]. Arc Virol, 2014,159(10): 2809-2813.DOI: 10.1007/s00705-014-2014-6
[3] Piralla A, Rovida F, Campanini G, et al. Clinical severity and molecular typing of human rhinovirus C strains during a fall outbreak affecting hospitalized patients[J]. J Clin Virol, 2009, 45(4): 311-317. DOI: https://doi.org/10.1016/j.jcv.2009.04.016
[4] Harris KG, Coyne CB. Enter at your own risk: How enteroviruses navigate the dangerous world of pattern recognition receptor signaling [J]. Cytokine, 2013, 63(3): 230-236. DOI:10.1016/j.cyto.2013.05.007
[5] Nathanson N, Kew OM. From emergence to eradication: The epidemiology of poliomyelitis deconstructed [J]. Am J Epidemiol, 2010, 172(11).1213-1229. DOI:10.1093/aje/kwq320
[6] Meerburg BG, Singleton GR, Kijlstra A. Rodent-borne diseases and their risks for public health[J]. Critical Rev in Microbiol, 2009, 35(3): 221-270.DOI:10.1080/10408410902989837
[7] Kiang D, Kalra I, Yagi S, et al. Assay for 5′ noncoding region analysis of all human rhinovirus prototype strains[J]. J Clin Microbiol, 2008, 46(11): 3736-3745. DOI:10.1128/JCM.00674-08
[8] Chen J, Fu Y, Ju L, et al. Detection and identification of viral pathogens in patients with hand, foot, and mouth disease by multilocus PCR, reverse-transcription PCR and electrospray ionization mass spectrometry[J]. J Clin Viro, 2014, 59(2): 115-119. DOI:10.1016/j.jcv.2013.11.007
[9] Ana A, Inmaculada C, Gloria T, et al. Molecular analysis of echovirus 13 isolates and aseptic meningitis, Spain[J]. Emerg Infect Dis, 2003, 9(8): 934-941. DOI:10.3201/eid0908.030080
[10] 严菊英,缪梓萍,吕华坤,等.浙江省2008-2012年肠道病毒相关病毒性脑炎病原谱及分子流行病学特征分析[J]. 中华流行病学杂志, 2013, 34(12): 1189-1193. DOI:10.3760/j.issn.0254-6450.2013.012.008
[11] Tapparel C, Junier T, Gerlach D, et al. New respiratory enterovirus and recombinant rhinoviruses among circulating picornaviruses[J]. Emerg Infect Dis, 2009, 15(5): 719.DOI: 10.3201/eid1505.081286
[12] Nix WA, Oberste MS, Pallansch MA. Sensitive, seminested PCR amplification of VP1 sequences for direct identification of all enterovirus serotypes from original clinical specimens[J]. J Clin Microbiol, 2006, 44(8): 2698-2704. DOI:10.1128/JCM.00542-06
[13] 胡群,马思杰,童淑梅. CODEHOP RT-PCR方法在汉坦病毒基因型别鉴定和分子溯源中的应用[J]. 中国人兽共患病学报, 2013, 29(9):854-857. DOI:10.3969/j.issn.1002 2694.2013.09.006
