微滴数字PCR技术在多拷贝木聚糖酶酿酒酵母工程菌筛选中的应用
2018-05-23张斯童王春凤
兰 雪,张斯童,李 哲,常 浩,孙 旸,王 刚,陈 欢,王春凤,陈 光,*
(1.吉林农业大学生命科学学院,吉林 长春 130118;2.吉林农业大学动物科学技术学院,吉林 长春 130118)
木聚糖酶是可将木聚糖降解成低聚木糖和木糖的水解酶,它不仅在能源工业上可发挥巨大作用,在饲料、造纸、食品等领域也有着广阔的应用前景[1-2]。而制约我国木聚糖酶生产与应用的主要限制性因素是缺乏能够与规模化生产相适应的高产菌株,因此,开发出具有我国自主知识产权的高产木聚糖酶新菌系迫在眉睫[3]。
酿酒酵母(Saccharomyces cerevisiae)是美国食品与药品管理局认定的一种安全菌株,是食品与饲用酶制剂表达的理想宿主[4]。酿酒酵母生物学和遗传学背景清楚,既具有原核生物生长繁殖快、遗传操作简单的特点,又具有真核生物对外源蛋白进行翻译后修饰加工的能力。近年来,在外源基因表达方面得到了深入研究和广泛应用[5-6]。rDNA序列是指细胞核中编码核糖体RNA的DNA序列,它是一段高度重复序列,其重复单位由转录区段和非转录区段组成。在酿酒酵母的XII染色体中,rDNA存在100~140 个重复单位[7]。如果以酿酒酵母rDNA序列为整合位点,从理论上说,可以得到100~140 个目的基因拷贝数。依赖于rDNA介导整合的外源基因导入酿酒酵母后,外源基因在酵母细胞内的表达会由于剂量效应而实现高效表达[8]。但研究发现,基因拷贝数在一定的范围内与蛋白表达量成正相关,通常在4~9 拷贝左右,超过这个范围,蛋白表达水平反而降低[9-10]。
微滴数字聚合酶链式反应(droplet digital polymerase chain reaction,ddPCR)是近年来兴起的一种新的基因绝对定量技术,通过极度稀释实现理论上的单分子扩增,然后用终点法PCR和泊松分布计算出样品的原始浓度,具有良好的准确度和重现性,可以实现真正意义上的绝对定量[11-12]。
本研究利用rDNA整合法构建多拷贝木聚糖酶酿酒酵母工程菌,并利用ddPCR技术对其拷贝数进行定量分析,并对不同拷贝数转化子产木聚糖酶活力进行测定,探讨基因拷贝数和酶活力之间的关系,以期为木聚糖酶生产菌株的构建和改造提供理论依据。
1 材料与方法
1.1 材料与试剂
大肠杆菌(Escherichaa coli)DH5α为吉林农业大学生物物理研究室保存菌种;黑曲霉CICC2462菌株中国工业微生物菌种保藏中心;酿酒酵母INVSc1(MATa、his3、leu2、trp1、ura3)、质粒pYES2(图1)中国工业微生物质粒菌种保藏中心;pMD19-T 日本TaKaRa公司。
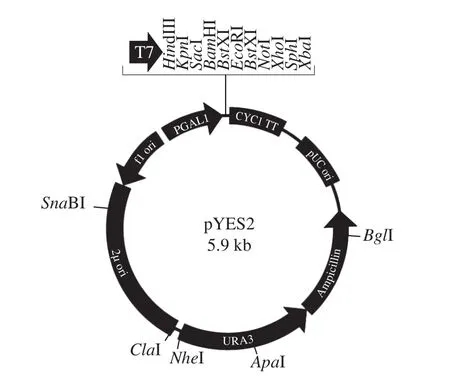
图1 pYES2质粒图谱Fig. 1 Map of plasmid pYES2
反转录试剂盒、去磷酸化试剂盒、限制性内切酶(NotI、XbaI、SnaBI等)、T4连接酶、SD-Ura培养基 日本TaKaRa公司;Salmon Sperm DNA 上海源叶生物科技有限公司;质粒提取试剂盒、PCR产物纯化试剂盒 生工生物工程(上海)股份有限责任公司;酵母基因组提取试剂盒 美国Omega公司;ddPCR EvaGreen Supermix、Droplet reader oil 美国Bio-Rad公司;总RNA提取试剂盒 美国Axygen公司;木聚糖美国Sigma公司;其他试剂均为国产分析纯。
1.2 仪器与设备
QX200型ddPCR仪、Universal hood III全能发光系统、电泳仪 美国Bio-Rad公司;PCR扩增仪 德国Eppendorf公司。
1.3 方法
1.3.1 xynB基因克隆
1.3.1.1 黑曲霉CICC2462总RNA提取
将黑曲霉CICC2462菌种经平板活化后,制备种子液,将种子液接种于液体发酵培养基,30 ℃、150 r/min培养72 h后,收集菌体,用总RNA提取试剂盒提取RNA[13]。
1.3.1.2 引物设计
根据美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)数据库中黑曲霉木聚糖酶基因序列,用Primer 5.0设计引物扩增xynB基因,并引入酶切位点NotI和XbaI。上游引物F1:5’-ATTTGCGGCCG CCTAGAGAGCATTTGCGATAGC-3’,下游引物R1:5’-TG CTCTAGAATGGTTCAGATCAAGGTAGCTG-3’。
1.3.1.3 xynB基因扩增
以提取的黑曲霉总RNA为模板,用TaKaRa一步法反转录试剂盒合成cDNA,以cDNA为模板扩增xynB基因,扩增条件见TaKaRa RNA PCR Kit(AMV)Ver.3.0说明书。琼脂糖凝胶电泳后,切胶回收纯化目的条带[14]。
1.3.1.4 克隆载体的连接、转化、鉴定
将回收产物与pMD19载体连接,转化大肠杆菌DH5α,接种到含有氨苄青霉素(ampicillin,Amp)的抗性平板上筛选。12 h后挑取单克隆进行菌落PCR鉴定,挑取阳性克隆于含有Amp抗性的LB液体培养基37 ℃、160 r/min培养。12 h后离心收集菌体,提取质粒并送测序,命名为pMD19-T-xynB。
1.3.2 rDNA序列扩增
1.3.2.1 酵母菌的培养以及DNA提取
用YPD固体培养基划线培养酿酒酵母INVSc1,挑取单克隆,YPD液体培养2 d后,收集菌体,用OMEGA酵母基因组提取试剂盒,提取DNA[15]。
1.3.2.2 引物设计及rDNA基因扩增
根据NCBI数据库中的信息,用Primer 5.0设计引物扩增2 300 bp的rDNA核心序列,并引入酶切位点SnaBI。上游引物F2:5’-ACGTACGTACAACGAACGAGACCTT AACCT-3’,下游引物R2:5’-ACGTACGTACGGAACCT CTAATCATTCGCT-3’。以酿酒酵母基因组为模板,扩增rDNA序列,扩增条件为:94 ℃预变性180 s;94 ℃变性10 s;55 ℃退火15 s;72 ℃延伸150 s;循环数为30,琼脂糖凝胶电泳后,切胶回收纯化目的条带。
1.3.2.3 克隆载体的连接、转化、鉴定
方法同1.3.1.4节,命名为pMD19-T-rDNA。
1.3.3 表达载体pYES2-xynB-rDNA的构建
用NotI和XbaI分别酶切载体pMD19-T-xynB和pYES2,琼脂糖凝胶电泳回收目的片段后,T4连接酶16 ℃连接过夜后转化DH5α,Amp抗性筛选后经xynB菌液PCR鉴定得到质粒pYES2-xynB,测序验证后,用SnaBI对载体pYES2-xynB和pMD19-T-rDNA进行酶切,对回收的pYES2-xynB片段进行去磷酸化后,T4连接酶16 ℃连接过夜后转化DH5α。12 h后挑取单克隆进行菌液PCR鉴定,挑取阳性克隆于含有Amp抗性的LB液体培养基37 ℃、160 r/min培养。12 h后离心收集菌体,提取质粒酶切鉴定,命名为pYES2-xynB-rDNA。
1.3.4 线性化pYES2-xynB-rDNA载体转化酿酒酵母
用SphI在rDNA位置对pYES2-xynB-rDNA进行单酶切,经琼脂糖凝胶回收后,用 LiAc/SS载体DNA/PEG化学转化法转化感受态酿酒酵母[16-17],转化体系为:感受态酿酒酵母,50 μL;50% PEG3350,240 μL;1 mol/L LiAc,36 μL;2 mg/mL boiled carrier DNA,50 μL;Plasmid DNA,34 μL。经42 ℃孵育后,将细胞重悬于1 mL YPD液体培养基中,30 ℃、150r/min培养1 h,收集菌体,用200 μL ddH2O重悬菌体后,将其涂布与SD-Ura平板,30 ℃培养3~5 d直至出现单菌落。对挑取的单菌落进行xynB的菌液PCR验证。
1.3.5 ddPCR鉴定转化子拷贝数
1.3.5.1 引物及探针
对得到的阳性转化子,接种于YPD培养基中,培养48 h后,收集菌体,用真菌总RNA提取试剂盒提取酿酒酵母RNA,经TaKaRa RNA PCR Kit (AMV) Ver.3.0反转录试剂盒合成cDNA,以cDNA为模板,ATC1基因为内参,利用ddPCR进行xynB基因拷贝数鉴定[18-19]。引物探针序列为:ATC1上游引物F3:5’-ATC1TATCCCCTGCATCCCTATCA-3’,下游引物R3:5’-CAGGCTTCGTTGCAGATACA-3’,探针:5’-HEXCATCTTCGTTAGCTTCATCCGACGCTA-BHQ-3’;xynB上游引物F4:5’-GCCGTGGACAGTTCTCTTTC-3’,下游引物R4:5’-TCATGACCCCGATGAGTGTA-3’,探针:5’-FAM-CCTGGTCAACTTTGCCCAGTCTAACAABHQ-3’。内参基因荧光标记基团为HEX,目的基因荧光标记基团为FAM。
1.3.5.2 ddPCR体系和条件
ddPCR包括配制体系、生成微滴、扩增循环和信号读取4 个步骤。ddPCR体系为20 μL,包含10 μL 2×ddPCR Master Mix,10 μmol/L 正向引物和反向引物各1 μL、探针0.5 μL,DNA模板2 μL。生成微滴需要使用专门的微滴生成卡和微滴生成仪,将40 μL PCR体系和70 μL微滴生成油(droplet generation oil)加入微滴生成卡,覆盖专用胶垫后置入微滴生成仪,生成微滴。ddPCR扩增使用两步法,设置程序如下:94 ℃预变性10 min;94 ℃变性15 s,60 ℃退火60 s,共45 个循环,扩增结束后进行98 ℃、10 min的热失活。每个模板重复3 个平行检测。扩增结束后,将96 孔板置入微滴读取仪中读取信号,并使用软件QuantaSoft V1.3.2.0分析分析实验数据,获得绝对定量结果[20-22]。
1.3.6 转化子发酵产酶实验
将1.3.5节鉴定得到的不同拷贝数的转化子接种至半乳糖培养基中,30 ℃、150 r/min培养72 h后,取上清液,用3,5-二硝基水杨酸(3,5-dinitrosalicylic acid,DNS)法测定木聚糖酶活力,具体方法见文献[23-24],木糖标准曲线回归方程为:y=0.127 5x-0.07,R2=0.993 5。
1.3.7 高酶活转化子产酶稳定性实验
将1.3.6节得到的产酶活力最高的转化子接种于半乳糖培养基中,进行传代,每次传代均在30 ℃、150 r/min培养72 h后,取上清液测定木聚糖酶活力,连续传代8 次。
2 结果与分析
2.1 xynB基因和rDNA序列的克隆
以提取的黑曲霉CICC2462总RNA为模板,经TaKaRa一步法反转录试剂盒得到cDNA,用引物F1和R1扩增得到预期目的片段,大小为984 bp。以酿酒酵母基因组为模板,用引物F2和R2扩增得到rDNA基因片段,大小约为2 300 bp。扩增产物大小均符合预期,如图2所示。
将上述基因扩增片段分别与pMD19-T载体连接,转化大肠杆菌DH5α,菌落PCR验证后,提取质粒,经NotI和XbaI酶切后,得到载体片段和xynB基因片段。用SnaBI酶切后,得到预期rDNA片段,如图3所示。将测序结果与GenBank(登录号:FJ_986225.1 和BK_006945.2)序列进行比对,相似度100%,表明xynB基因和rDNA未发生碱基突变。
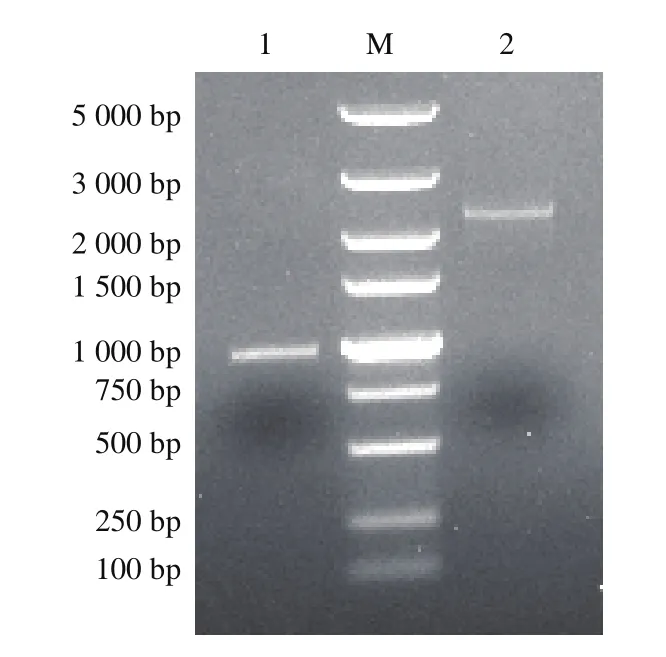
图2 xynB和rDNA片段的PCR扩增Fig. 2 PCR amplification of xynB and rDNA fragment
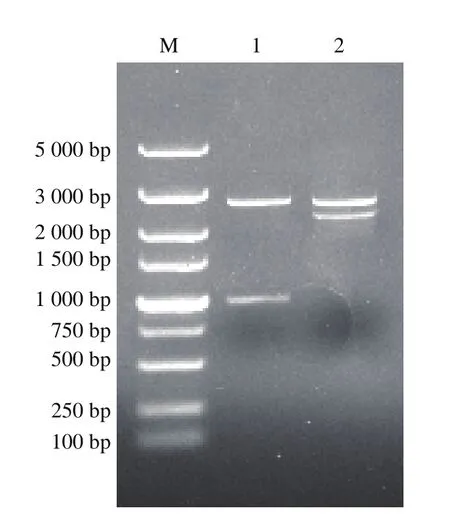
图3 克隆载体pMD19-T酶切鉴定Fig. 3 Restriction enzyme digestion of recombinant vector pMD19-T
2.2 表达载体pYES2-xynB-rDNA的构建
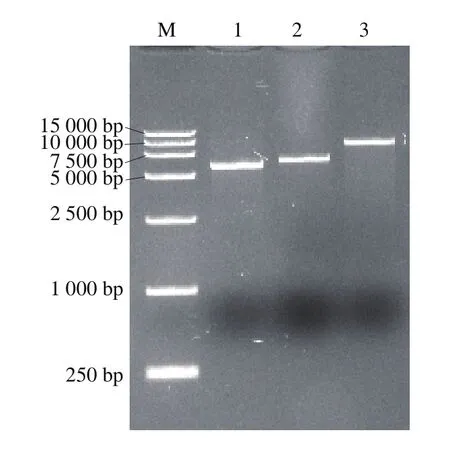
图4 表达载体的酶切鉴定Fig. 4 Restriction enzyme digestion of expression vector
用NotI和XbaI分别酶切载体pMD19-T-xynB和pYES2,回收目的片段后,T4连接酶16 ℃连接过夜后转化DH5α,经xynB菌液PCR鉴定得到质粒pYES2-xynB,测序验证后,用SnaBI对载体pYES2-xynB和pMD19-T-rDNA进行酶切,对回收的pYES2-xynB片段进行去磷酸化后,T4连接酶16 ℃连接过夜后转化DH5α,菌液PCR验证后,提取质粒酶切验证,结果如图4所示,pYES2载体大小为5 864 bp;pYES2-xynB载体大小为6 848 bp;pYES2-xynB-rDNA大小为9 148 bp。
2.3 转化子拷贝数的ddPCR检测结果
用SphI在rDNA位置对pYES2-xynB-rDNA进行线性化后,转化感受态酿酒酵母INVSc1,对经SD-Ura培养基筛选得到的阳性转化子提取RNA,反转录为cDNA后,使用ddPCR进行转化子拷贝数检测。图5显示的结果为在2 个检测通道下,检测的阳性微滴和阴性微滴的分布转框,图中阳性微滴和阴性微滴区分明显,系统可准确判读出阳性微滴和阴性微滴数目[25]。

图5 ddPCR测定转化子拷贝数Fig. 5 Detection of copy number of transformants by ddPCR
微滴生成是ddPCR的关键步骤,只有微滴数大于10 000时,DNA分子的分布才能符合泊松分布的统计学原理,系统才可以对阳性和阴性微滴进行计数。ddPCR扩增形成的微滴数如表1所示。由表1可知,反应中形成的微滴数处于11 411~15 859之间,均大于10 000,满足ddPCR微滴的分析要求[26-28]。根据软件读取的各转化子xynB基因和ATC1基因的浓度,得出目的基因xynB的拷贝数。ddPCR分析显示,利用rDNA整合法,成功获得了1、2、3、5、9、10、14、16、17和18共10 种不同拷贝数的转化子。
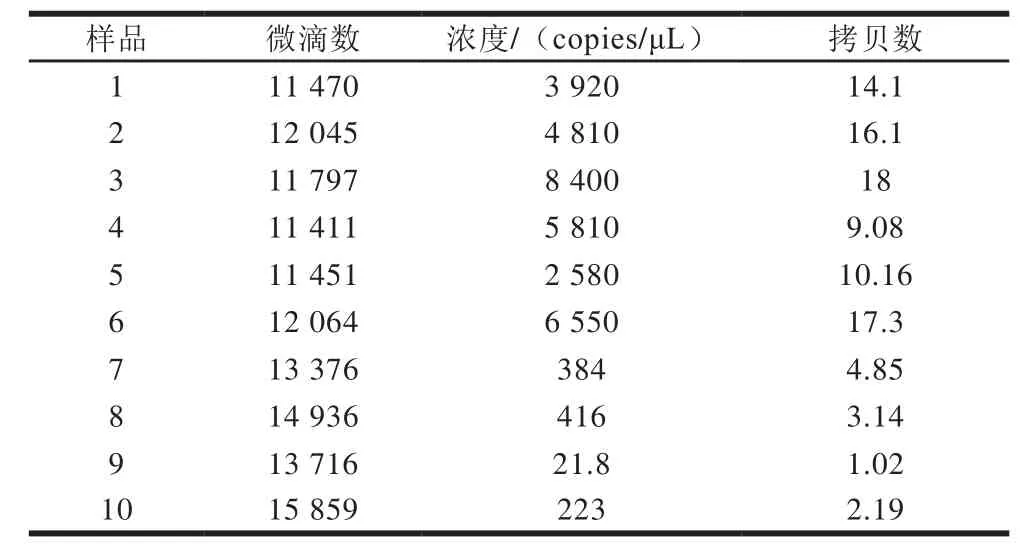
表1 ddPCR拷贝数分析结果Table 1 Estimated copy number of transformants by ddPCR
2.4 不同拷贝数转化子发酵产木聚糖酶活力分析
对10 株携带不同拷贝数的菌株,通过摇瓶发酵培养,经过半乳糖诱导72 h后,取发酵液进行木聚糖酶活力测定,不同拷贝数转化子木聚糖酶活力如图6所示,当xynB拷贝数小于9时,木聚糖酶活力随着拷贝数增加而增加,9 拷贝时木聚糖酶活力最高为308 U/mL,是单拷贝时的5.4 倍;超过9 拷贝后,木聚糖酶活力降低,到18 拷贝时,酶活力仅为9 拷贝时的44%。
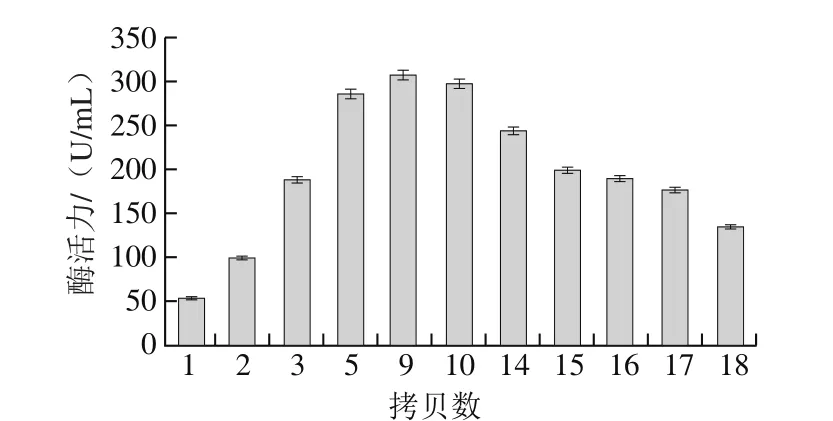
图6 拷贝数与酶活力之间的关系Fig. 6 Relationship between copy number and enzymatic activity
2.5 转化子产酶稳定性分析
对得到的酶活力最高的9 拷贝转化子,进行遗传稳定性研究,结果如图7所示,结果表明,用rDNA整合法获得的转化子产酶稳定性较好,经过8 次传代后,酶活力变化微小,第8次传代时酶活力为310 U/mL。
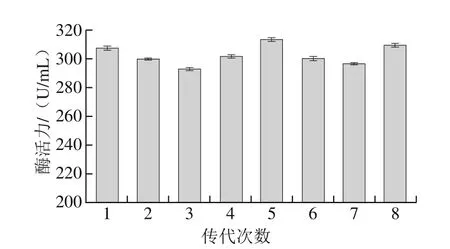
图7 传代次数与酶活力之间的关系Fig. 7 Relationship between passage number and enzymatic activity
3 讨论与结论
外源基因的拷贝数是影响外源蛋白高效表达的一个重要因素。大量研究表明,增加基因的拷贝数可以有效增加外源蛋白的表达[29-30]。近年来,随着现代分子生物学技术的发展,研究人员采取了多种策略来提高外源基因的拷贝数[31],如串联表达盒法等,但这种方式需要频繁地构建载体、酶切、纯化回收 DNA 片段,操作繁琐,且需要消耗大量的酶来制备线性化片段。本研究利用酿酒酵母12号染色体上rDNA多拷贝重组位点作为构建多拷贝工程菌的方法,高效而且简单方便,筛选得到最多18拷贝的转化子。但对于分泌表达来说,外源基因的剂量固然是一个重要的因素,但蛋白的翻译、内质网内蛋白折叠和加工、分泌途径同样会影响外源蛋白的表达,高剂量的外源基因的表达可能会对菌体本身的生长带来不利影响,因此,对于酿酒酵母的分泌型表达,并不是拷贝数越多越好,需要筛选得到最优拷贝数[32-33]。本研究利用rDNA整合法构建了酿酒酵母木聚糖酶工程菌,通过ddPCR技术鉴定得到了10 种不同拷贝数的菌株,对这些菌株产木聚糖酶的能力进行测定,发现当拷贝数小于9时,木聚糖酶活力与拷贝数呈正相关,在9 拷贝时达到最高,为308 U/mL;当拷贝数大于9时,木聚糖酶活力反而降低,到18 拷贝时,其产酶活力仅为9拷贝时的44%;对9 拷贝菌株的产酶稳定性进行研究,发现其在传代8 次后,仍能保持产酶稳定,说明用rDNA整合法得到的木聚糖酶酿酒酵母能稳定遗传。
本实验成功构建木聚糖酶rDNA整合表达载体,并整合到酿酒酵母基因组获得10 株不同拷贝数的菌株,对其酶活力进行测定,发现当拷贝数小于9时,产酶能力随拷贝数增加而增强,9 拷贝菌株产酶能力最强,酶活力为308 U/mL,超过9 拷贝,产酶能力降低。
参考文献:
[1]单志琼, 周峻岗, 周宇飞, 等. 产碱性木聚糖酶菌株的筛选及酶学性质[J]. 遗传, 2012, 34(3): 356-365. DOI:10.3724/SP.J.1005.2012.00356.
[2]孙振涛, 赵祥颖, 刘建军, 等. 微生物木聚糖酶及其应用[J]. 生物技术, 2007, 17(2): 93-97. DOI:10.3969/j.issn.1004-311X.2007.02.033.
[3]李杰, 张会, 张莹莹, 等. 食品级木聚糖酶黑曲霉工程菌的构建[J]. 东北农业大学学报, 2013, 44(11): 7-13. DOI:10.3969/j.issn.1005-9369.2013.11.002.
[4]LU Y, ZHONG H, TANG Q, et al. Construction and verification of CYP3A5 gene polymorphisms using a Saccharomyces cerevisiae expression system to predict drug metabolism[J]. Molecular Medicine Reports, 2017, 15(4): 1593-1600. DOI:10.3892/mmr.2017.6214.
[5]PAYNE T, HANFREY C, BISHOP A L, et al. Transcript-specific translational regulation in the unfolded protein response of Saccharomyces cerevisiae[J]. FEBS Letters, 2008, 582(4): 503-509.DOI:10.1016/j.febslet.2008.01.009.
[6]WANG H Y, XIAO D F, ZHOU C, et al. YLL056C, from Saccharomyces cerevisiae, encodes a novel protein with aldehyde reductase activity[J]. Applied Microbiology & Biotechnology, 2017,101(11): 4507-4520. DOI:10.1007/s00253-017-8209-5.
[7]KWAN E X, WANG X S, AMEMIYA H M, et al. rDNA Copy number variants are frequent passenger mutations in Saccharomyces cerevisiae deletion collections and de novo transformants[J]. G3 (Bethesda),2016, 6(9): 2829-2838. DOI:10.1534/g3.116.030296.
[8]JAMES S A, WEST C, DAVEY R P, et al. Prevalence and dynamics of ribosomal DNA micro-heterogeneity are linked to population history in two contrasting yeast species[J]. Scientific Reports, 2016, 6: 28555.DOI:10.1038/srep28555.
[9]武志强, 贾耐兵, 李娜, 等. 酵母整合型载体的构建及其功能分析[J]. 生物学通报, 2008, 43(5): 47-50. DOI:10.3969/j.issn.0006-3193.2008.05.020.
[10]程诚, 熊亮, 李勇昊, 等. 混合糖发酵重组酿酒酵母的菌株构建和菊芋秸秆同步糖化发酵研究[J]. 微生物学通报, 2016, 43(7): 1411-1418. DOI:10.13344/j.microbiol.china.150576.
[11]周巍, 李月华, 孙勇, 等. 微滴式数字PCR技术定量检测发酵乳中金黄色葡萄球菌[J]. 食品科学, 2017, 38(16): 287-291. DOI:10.7506/spkx1002-6630-201716046.
[12]詹成, 燕丽, 王琳, 等. 数字PCR技术的发展和应用[J]. 复旦学报(医学版), 2015, 42(6): 786-789. DOI:10.3969/j.issn.1672-8467.2015.06.017.
[13]王杰, 王全, 田娜, 等. 不同植物组织RNA提取方法的比较分析[J].北京农学院学报, 2015, 30(1): 76-80. DOI:10.13473/j.cnki.issn.1002-3186.2014.0093.
[14]周晨妍, 刘振华, 王丹丹, 等. 木聚糖酶Xyn43A基因在大肠杆菌及毕赤酵母中的表达比较[J]. 食品与发酵工业, 2016, 42(6): 15-19.DOI:10.13995/j.cnki.11-1802/ts.201606003.
[15]唐天乐, 高炳淼, 长孙东亭, 等. 高质量毕赤酵母基因组DNA提取方法比较[J]. 生物技术通报, 2010, 2010(1): 196-199. DOI:10.13560/j.cnki.biotech.bull.1985.2010.01.021.
[16]高丽丽, 王庆华, 梁会超, 等. 酿酒酵母羊毛甾醇合酶基因单倍体缺陷型突变株的构建[J]. 药学学报, 2014, 49(5): 742-746.DOI:10.16438/j.0513-4870.2014.05.001.
[17]GIETZ R D, WOODS R A. Yeast transformation by the LiAc/SS carrier DNA/PEG method[J]. Methods in Molecular Biology, 2006,313: 107-120. DOI:10.1007/978-1-4939-1363-3_1.
[18]SRISUTHAM S, SARALAMBA N, MALLERET B, et al. Four human Plasmodium species quantification using droplet digital PCR[J]. PLoS ONE, 2017, 12(4): e0175771. DOI:10.1371/journal.pone.0175771.
[19]CAVALLI M, DE NOVI L A, DELLA S I, et al. Comparative analysis between RQ-PCR and digital droplet PCR of BCL2/IGH gene rearrangement in the peripheral blood and bone marrow of early stage follicular lymphoma[J]. British Journal of Haematology, 2017, 177(4):588. DOI:10.1111/bjh.14616.
[20]ARVIA R, SOLLAI M, PIERUCCI F, et al. Droplet digital PCR(ddPCR) vs quantitative real-time PCR (qPCR) approach for detection and quantification of Merkel cell polyomavirus (MCPyV) DNA in formalin fixed paraffin embedded (FFPE) cutaneous biopsies[J].Journal of Virological Methods, 2017, 246: 15-20. DOI:10.1016/j.jviromet.2017.04.003.
[21]SUZAWA K, YAMAMOTO H, OHASHI K, et al. Optimal method for quantitative detection of plasma EGFR T790M mutation using droplet digital PCR system[J]. Oncology Reports, 2017, 37(5): 3100-3106.DOI:10.3892/or.2017.5567.
[22]张居作, 徐君飞, 陈汉忠, 等. BE-91菌株木聚糖酶活力测定条件的优化[J]. 湖北农业科学, 2010, 49(4): 950-953. DOI:10.14088/j.cnki.issn0439-8114.2010.04.003.
[23]史红玲, 汪俊卿, 邬敏辰, 等. 毕赤酵母产重组木聚糖酶发酵条件的优化及其酶学性质[J]. 中国生物制品学杂志, 2012, 25(10): 1362-1365. DOI:10.13200/j.cjb.2012.10.123.shihl.016.
[24]JIA P, PURCELL M K, PAN G, et al. Analytical validation of a reverse transcriptase droplet digital PCR (RT-ddPCR) for quantitative detection of infectious hematopoietic necrosis virus[J]. Journal of Virological Methods, 2017, 245: 73-80. DOI:10.1016/j.jviromet.2017.03.010.
[25]KLINE M C, DUEWER D L. Evaluating droplet digital PCR for the quantification of human genomic DNA: lifting the traceability fog[J].Analytical Chemistry, 2017, 89(8): 4648-4654. DOI:10.1021/acs.analchem.7b00240.
[26]姜羽, 胡佳莹, 杨立桃. 利用微滴数字PCR分析转基因生物外源基因拷贝数[J]. 农业生物技术学报, 2014, 22(10): 1298-1305.DOI:10.3969/j.issn.1674-7968.2014.10.013.
[27]于晓帆, 高宏伟, 孙敏, 等. 荧光PCR和数字PCR法检测转基因DAS-44406-6品系大豆[J]. 食品科学, 2016, 37(16): 235-241. DOI:10.7506/spkx1002-6630-201616038.
[28]BRUNETTI C, ANELLI L, ZAGARIA A, et al. Droplet digital PCR is a reliable tool for monitoring minimal residual disease in acute promyelocytic leukemia[J]. Journal of Molecular Diagnostics, 2017,19(3): 437-444. DOI:10.1016/j.jmoldx.2017.01.004.
[29]GITTINS J R, D’ANGELO C, OSWALD F, et al. Fluorescent protein-mediated colour polymorphism in reef corals: multicopy genes extend the adaptation/acclimatization potential to variable light environments[J]. Molecular Ecology, 2015, 24(2): 453-465.DOI:10.1111/mec.13041.
[30]黄贞杰, 陈玲, 张积森, 等. ScINO1基因克隆及酵母多基因多拷贝整合表达载体的构建[J]. 福建师大学报(自然科学版), 2012, 28(6):100-105. DOI:10.3969/j.issn.1000-5277.2012.06.019.
[31]LIN X Q, LIANG S L, HAN S Y, et al. Quantitative iTRAQ LCMS/MS proteomics reveals the cellular response to heterologous protein overexpression and the regulation of HAC1 in Pichia pastoris[J]. Journal of Proteomics, 2013, 91: 58-72. DOI:10.1016/j.jprot.2013.06.031.
[32]MARX H, MECKLENBRÄUKER A, GASSER B, et al. Directed gene copy number amplification in Pichia pastoris by vector integration into the ribosomal DNA locus[J]. FEMS Yeast Research, 2009, 9(8): 1260-1270. DOI:10.1111/j.1567-1364.2009.00561.x.
[33]吴志伟, 徐立新, 佟金, 等. 多拷贝策略在增强目的基因表达中的应用[J]. 生命科学研究, 2016, 20(2): 166-170. DOI:10.16605/j.cnki.1007-7847.2016.02.014.
