基于线粒体COI基因序列进行库蠓二囊亚属近似种的分子鉴定
2018-05-18,,,,
,,,,
蠓科(Ceratopogonidae)隶属于双翅目、长角亚目,是一类体型微小、种类繁多、分布甚广的昆虫,其中部分种类刺吸、叮咬人畜,称吸血蠓。库蠓(CulicoidesLatreille, 1809)是吸血蠓中分布最广、种类最多、与人畜关系最密切的属,体长一般1~3 mm,全世界有1 368种、我国有347种[1-2]。库蠓刺吸人、禽、畜等血液,不仅带来直接叮咬骚扰,而且是蓝舌病(BT)、乙型脑炎(JV)、丝虫病等多种疾病病原体的传播媒介[2-3]。因此,准确鉴定疾病媒介对蠓传疾病的监测、防治具有重要的意义。
库蠓属昆虫体型微小、形态学鉴定特征稀少(如翅斑形态、嗅觉器分布、感觉器有无等特征),近似种类容易混淆,且要求鉴定人员具有极高的专业能力。例如林岛库蠓C.gaponus和无害库蠓C.innoxius,其外部形态特征极其相似,仅有翅径端淡斑延伸至中1脉有区别。另外,用酒精保存蠓标本时翅上的斑纹很容易褪色,这必定会影响种类鉴定的准确性。因此,在蠓科昆虫分类研究中亟需能对近似种进行快速、准确鉴定的方法。
目前,以DNA序列分析为依据的分子鉴定已成为物种鉴定的常用技术手段,极大地弥补了传统分类学鉴定的缺陷。然而,蠓科昆虫的分子分类研究起步较迟,自1992年研究变翅库蠓(Culicoidesvariipennis)种群的基因差异开始[4],国外的蠓科分子生物学研究有了突飞猛进的进展,相比之下国内却鲜有报道。因此,本研究以库蠓属二囊亚属中近似的8个种类(林岛库蠓C.gaponus、标翅库蠓C.insignipennis、无害库蠓C.innoxius、连斑库蠓C.jacobsoni、南山库蠓C.lansangensis、新竹库蠓C.liui、长喙库蠓C.longirostris、异域库蠓C.peregrinus)为例,利用线粒体COI基因进行分子鉴定,以期更准确地鉴别不同地理种群库蠓近似种,从而为吸血蠓DNA分类研究提供科学依据。
1 材料与方法
1.1 材料
1.1.1蠓标本 研究中所用的9种、15个地理种群的32只库蠓标本,除无害库蠓C.innoxius是来自GenBank数据库外,其余8种均采自云南、贵州、四川、重庆、广西、海南等省份,详见表1。
1.1.2试剂 基因组提取试剂盒(QIAGEN, 德国)、PCR MasterMix(TIANGEN, 中国)、引物(上海捷瑞生物工程有限公司)、琼脂糖凝胶回收试剂盒(TIANGEN, 中国)、琼脂糖(Biowest Agarose, 西班牙)等。
1.1.3仪器设备 光学体式显微镜(德国Leica公司)、PCR扩增仪(美国BOI-RAD公司)、UVP凝胶成像系统(美国BOI-RAD公司)等。
表1 供试库蠓的相关信息
Tab.1 Information of the biting midges in this study

标本号种名雌雄采集地CQ-1荒川库蠓C.arakawai雄虫重庆北碚区CQ-2长喙库蠓C.longirostris雌虫重庆开州区CQ-3长喙库蠓C.longirostris雌虫重庆开州区GX-1荒川库蠓C.arakawai雌虫广西大瑶山自然保护区GX-2荒川库蠓C.arakawai雄虫广西大瑶山自然保护区GZ-1荒川库蠓C.arakawai雌虫贵州遵义市GZ-2荒川库蠓C.arakawai雄虫贵州遵义市GZ-3连斑库蠓C.jacobsoni雌虫贵州望谟县GZ-4南山库蠓C.lansangensis雌虫贵州望谟县GZ-5南山库蠓C.lansangensis雌虫贵州铜仁市GZ-6南山库蠓C.lansangensis雌虫贵州榕江县HN-1林岛库蠓C.gaponus雌虫海南鹦哥岭自然保护区HN-2连斑库蠓C.jacobsoni雌虫海南霸王岭自然保护区HN-3连斑库蠓C.jacobsoni雌虫海南鹦哥岭自然保护区HN-4连斑库蠓C.jacobsoni雌虫海南鹦哥岭自然保护区HN-5南山库蠓C.lansangensis雌虫海南鹦哥岭自然保护区HN-6南山库蠓C.lansangensis雌虫海南鹦哥岭自然保护区HN-7新竹库蠓C.liui雌虫海南鹦哥岭自然保护区SC-1荒川库蠓C.arakawai雌虫四川泸州市SC-2荒川库蠓C.arakawai雄虫四川泸州市SC-3长喙库蠓C.longirostris雌虫四川雅安市YN-1标翅库蠓C.insignipennis雌虫云南江城县YN-2连斑库蠓C.jacobsoni雌虫云南勐腊县YN-3南山库蠓C.lansangensis雌虫云南景洪市YN-4南山库蠓C.lansangensis雌虫云南江城县YN-5南山库蠓C.lansangensis雌虫云南勐腊县YN-6新竹库蠓C.liui雌虫云南江城县YN-7新竹库蠓C.liui雌虫云南勐腊县YN-8异域库蠓C.peregrinus雌虫云南江城县KJ909533无害库蠓C.innoxius雄虫印度KT307825无害库蠓C.innoxius雌虫印度KF145176无害库蠓C.innoxius雌虫印度
1.2 方法
1.2.1蠓采集 根据蠓的生活习性,选择网扫法和灯诱法在不同的生态环境(稻田、洼地、树林、牲畜厩舍、人群聚居地等)布置采样点进行蠓收集,并将其保存于无水乙醇中。
1.2.2标本鉴定 按照常规方法对蠓标本进行分选、解剖,除胸部外制成永久装片用于形态鉴定,胸部样本单独编号后进行分子实验。
待研究库蠓根据外部形态特征按照文献用显微镜镜检鉴定种类,并进行一侧翅的拍照(图1)。
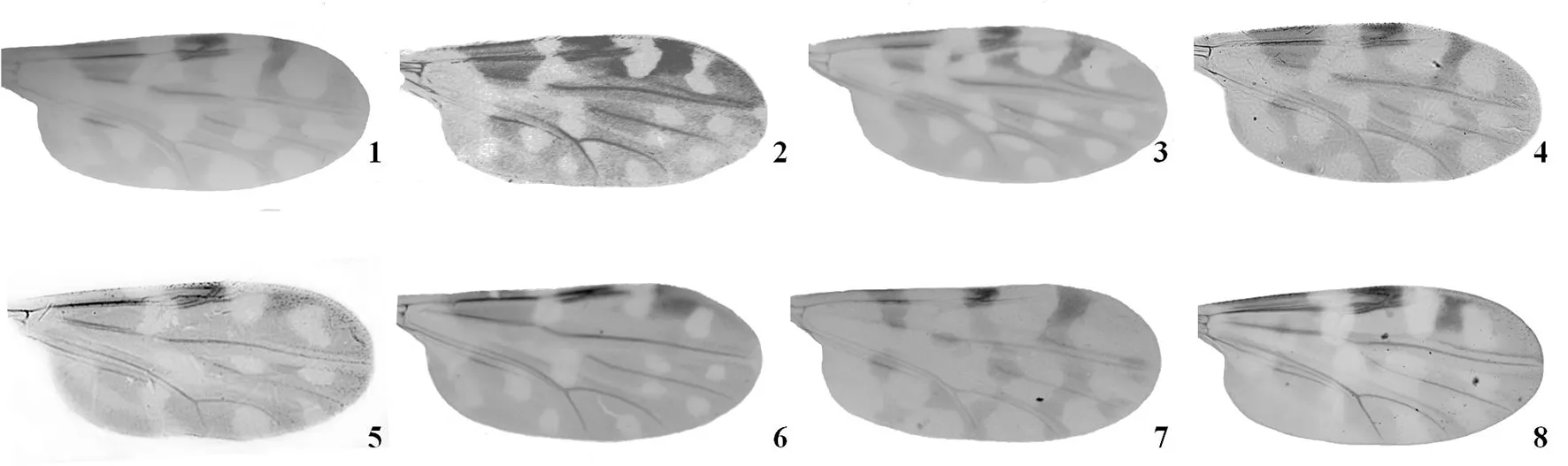
1:林岛库蠓C. gaponus;2:异域库蠓C. peregrinus;3:标翅库蠓C. insignipennis;4:无害库蠓C. innoxius;5:连斑库蠓C. jacobsoni;6:南山库蠓C. lansangensis;7:新竹库蠓C. liui;8:长喙库蠓C. longirostris图1 库蠓近似种一侧翅图Fig.1 Wing of one side of sibling species
1.2.3基因组DNA提取 将保存的库蠓胸部研磨后,按照QIAGEN DNeasy Blood & Tissue Kit说明书进行单只库蠓基因组DNA提取,并保存至-20 ℃。
1.2.4PCR扩增 PCR扩增引物包括上游引物LCO1490(5′-TAAACTTCAGGGTGACCAAA-AAATCA-3′)和下游引物HCO2198(5′-GGTCAACAAATCATAAAGATATTGG-3′),由上海捷瑞生物工程有限公司合成。PCR扩增体系及反应条件如下:DNA模板1.5 μL,上游引物(10 μmol/L) 1 μL,下游引物(10 μmol/L) 1 μL,2×PCR MasterMix 12.5 μL,ddH2O 9 μL,总体系25 μL,PCR反应条件为94 ℃预变性1 min;94 ℃ 5 min,55 ℃ 30 s,72 ℃ 1 min,循环35次;最后72 ℃延伸5 min。
1.2.5PCR产物鉴定与回收 PCR产物进行琼脂糖凝胶电泳,根据电泳条带判断是否为目的条带。按照琼脂糖凝胶DNA回收试剂盒说明书进行PCR产物切胶纯化,纯化后的产物送上海生工生物工程股份有限公司进行双向测序。
1.2.6COI序列分析 利用Clustal X、DNA MAN、MEGA 6.0等生物信息学软件对库蠓线粒体DNA COI序列进行比对、编辑、比较等分析,以Kimura双参数模型计算遗传距离矩阵。以荒川库蠓(C.arakawai)为外群,通过邻接法(NJ)、最大似然法(ML)构建分子系统树,并通过Bootstrap 1000次自举检验系统树中各结点的置信值。
2 结 果
2.1PCR扩增及序列测定 本研究通过对8种、13个地理种群的29只库蠓线粒体COI基因序列进行PCR扩增、纯化和双向测序,并经过序列比对及人工校对后,最终获得长度约650 bp的COI基因片段。
2.2序列比对 将不同种类库蠓的全部COI序列与NCBI的GenBank数据库中的同源序列进行比对,在数据库中搜索到荒川库蠓C.arakawai(登录号分别为KF528696、GU070920、KF528696、AB360975)、连斑库蠓C.jacobsoni(登录号分别为KT352455、KT297817)和异域库蠓(C.peregrinus)(登录号为KT307853)的同源序列,序列相似度皆大于99%,数据显示这3种库蠓的形态鉴定结果与COI基因序列比对结果一致,表明COI序列可用于库蠓种类鉴别。
2.3遗传距离 利用MEGA 6.0软件计算库蠓COI序列的遗传距离(表2),其中无害库蠓(C. innoxius)的COI序列来自GenBank(登录号分别为KJ909533、KT307825、KF145176)。结果表明,种内和种间遗传距离分别为0.000~0.019和0.214~0.302,两者差异显著(表3)。因此,通过COI序列遗传距离分析可以区分库蠓不同种类,即适用于种及以下水平的种类鉴别。
表2 库蠓线粒体COI序列的遗传距离矩阵
Tab.2 Genetic distance matrix of biting midges mitochondrial COI sequence
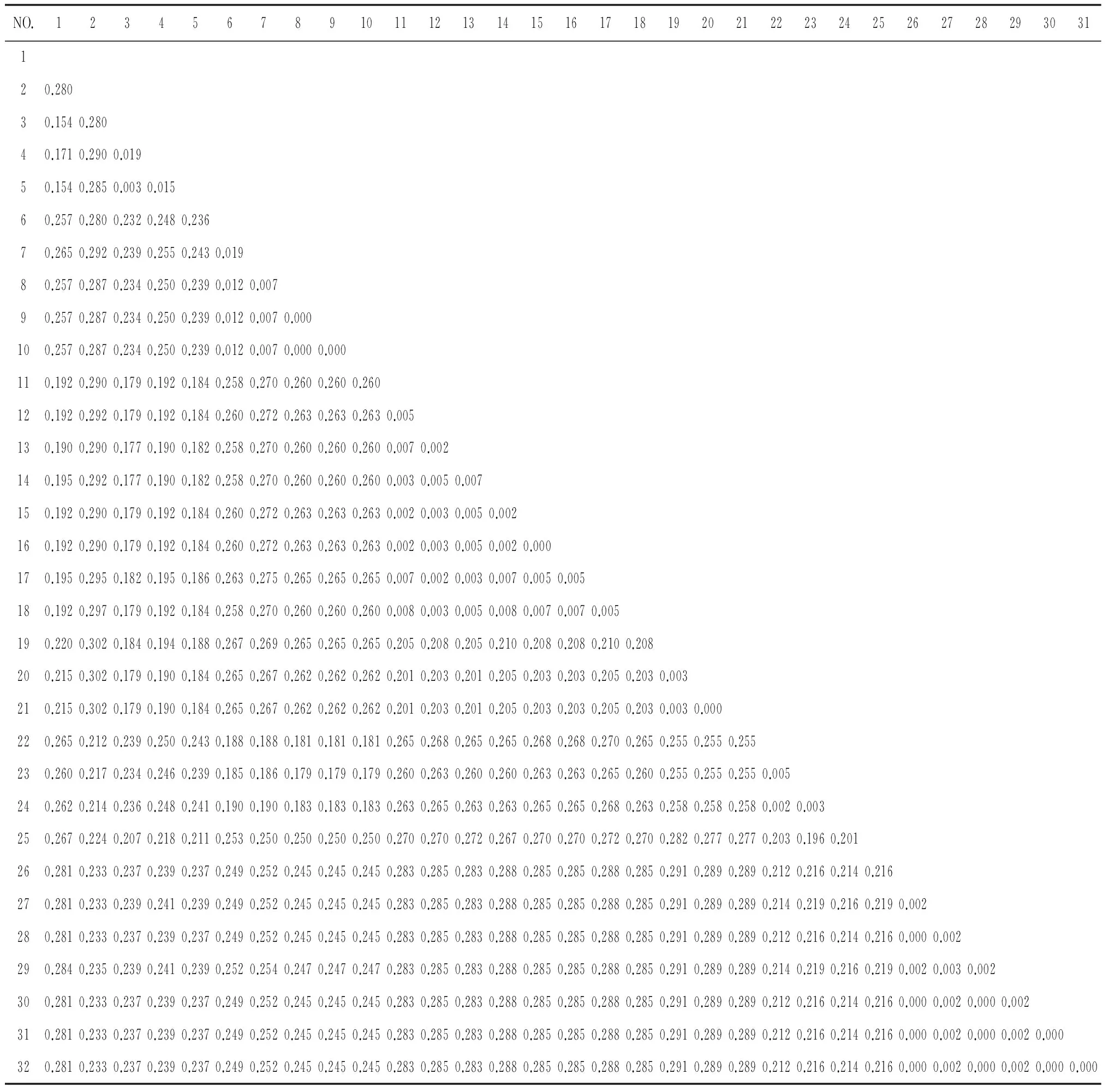
NO.12345678910111213141516171819202122232425262728293031120.28030.1540.28040.1710.2900.01950.1540.2850.0030.01560.2570.2800.2320.2480.23670.2650.2920.2390.2550.2430.01980.2570.2870.2340.2500.2390.0120.00790.2570.2870.2340.2500.2390.0120.0070.000100.2570.2870.2340.2500.2390.0120.0070.0000.000110.1920.2900.1790.1920.1840.2580.2700.2600.2600.260120.1920.2920.1790.1920.1840.2600.2720.2630.2630.2630.005130.1900.2900.1770.1900.1820.2580.2700.2600.2600.2600.0070.002140.1950.2920.1770.1900.1820.2580.2700.2600.2600.2600.0030.0050.007150.1920.2900.1790.1920.1840.2600.2720.2630.2630.2630.0020.0030.0050.002160.1920.2900.1790.1920.1840.2600.2720.2630.2630.2630.0020.0030.0050.0020.000170.1950.2950.1820.1950.1860.2630.2750.2650.2650.2650.0070.0020.0030.0070.0050.005180.1920.2970.1790.1920.1840.2580.2700.2600.2600.2600.0080.0030.0050.0080.0070.0070.005190.2200.3020.1840.1940.1880.2670.2690.2650.2650.2650.2050.2080.2050.2100.2080.2080.2100.208200.2150.3020.1790.1900.1840.2650.2670.2620.2620.2620.2010.2030.2010.2050.2030.2030.2050.2030.003210.2150.3020.1790.1900.1840.2650.2670.2620.2620.2620.2010.2030.2010.2050.2030.2030.2050.2030.0030.000220.2650.2120.2390.2500.2430.1880.1880.1810.1810.1810.2650.2680.2650.2650.2680.2680.2700.2650.2550.2550.255230.2600.2170.2340.2460.2390.1850.1860.1790.1790.1790.2600.2630.2600.2600.2630.2630.2650.2600.2550.2550.2550.005240.2620.2140.2360.2480.2410.1900.1900.1830.1830.1830.2630.2650.2630.2630.2650.2650.2680.2630.2580.2580.2580.0020.003250.2670.2240.2070.2180.2110.2530.2500.2500.2500.2500.2700.2700.2720.2670.2700.2700.2720.2700.2820.2770.2770.2030.1960.201260.2810.2330.2370.2390.2370.2490.2520.2450.2450.2450.2830.2850.2830.2880.2850.2850.2880.2850.2910.2890.2890.2120.2160.2140.216270.2810.2330.2390.2410.2390.2490.2520.2450.2450.2450.2830.2850.2830.2880.2850.2850.2880.2850.2910.2890.2890.2140.2190.2160.2190.002280.2810.2330.2370.2390.2370.2490.2520.2450.2450.2450.2830.2850.2830.2880.2850.2850.2880.2850.2910.2890.2890.2120.2160.2140.2160.0000.002290.2840.2350.2390.2410.2390.2520.2540.2470.2470.2470.2830.2850.2830.2880.2850.2850.2880.2850.2910.2890.2890.2140.2190.2160.2190.0020.0030.002300.2810.2330.2370.2390.2370.2490.2520.2450.2450.2450.2830.2850.2830.2880.2850.2850.2880.2850.2910.2890.2890.2120.2160.2140.2160.0000.0020.0000.002310.2810.2330.2370.2390.2370.2490.2520.2450.2450.2450.2830.2850.2830.2880.2850.2850.2880.2850.2910.2890.2890.2120.2160.2140.2160.0000.0020.0000.0020.000320.2810.2330.2370.2390.2370.2490.2520.2450.2450.2450.2830.2850.2830.2880.2850.2850.2880.2850.2910.2890.2890.2120.2160.2140.2160.0000.0020.0000.0020.0000.000
1:林岛库蠓C.gaponus(HN-1);2:标翅库蠓C.insignipennis(YN-1);3-5:无害库蠓C.innoxius(KJ909533.2/KF145176.1/KT307825.1);6-10:连斑库蠓C.jacobsoni(YN-2/GZ-3/HN-2/HN-3/HN-4);11-18:南山库蠓C.lansangensis(YN-3/GZ-4/GZ-5/YN-4/HN-5/HN-6/YN-5/GZ-6);19-21:新竹库蠓C.liui(HN-7/YN-6/YN-7);22-24:长喙库蠓C.longirostris(SC-3/CQ-2/CQ-3);25:异域库蠓C.peregrinus(YN-8);26-32:荒川库蠓C.arakawai(CQ-1/GX-1/GX-2/GZ-1/GZ-2/SC-1/SC-2)
表3 不同阶元库蠓线粒体COI序列遗传距离分析
Tab.3 Genetic distance analysis of different taxonomic levels Culicoides about the mitochondrial COI sequence
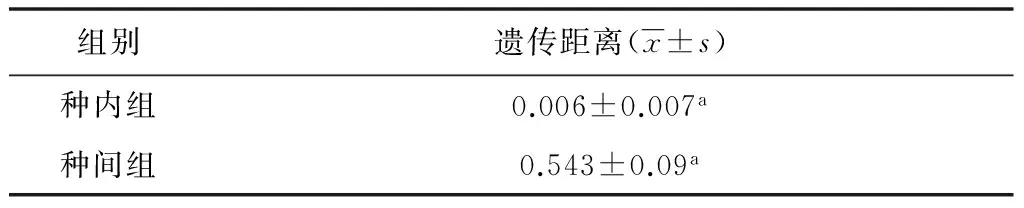
组别遗传距离(x±s)种内组0.006±0.007a种间组0.543±0.09a
注:与种内遗传距离相比,t=119.452,aP<0.05
2.4系统发育分析 以荒川库蠓C.arakawai为外群,采用邻接法(NJ)和最大似然法(ML)分别构建9种库蠓的系统发育树,得到基本一致的拓扑结构(图2)。系统发育树聚类分析表明:
①不同种类各自构成单系(群),同一种类不同地理种群各自聚为一支(如南山库蠓C.lansangensis、新竹库蠓C.liui、无害库蠓C.innoxius、林岛库蠓C.gaponus、连斑库蠓C.jacobsoni、长喙库蠓C.longirostris、异域库蠓C.peregrinus、标翅库蠓C.insignipennis、荒川库蠓C.arakawai),不同库蠓种类间无交叉,该结果与形态学鉴定结果一致。
②种类的亲缘关系明显,如无害库蠓C.innoxius与林岛库蠓C.gaponus首先聚在一起,亲缘关系最近并构成姐妹群,这与形态鉴定结果相一致。上述两近似种的形态特征十分相近(表4),其中翅斑分布除径端淡斑外几乎无异(图2)。
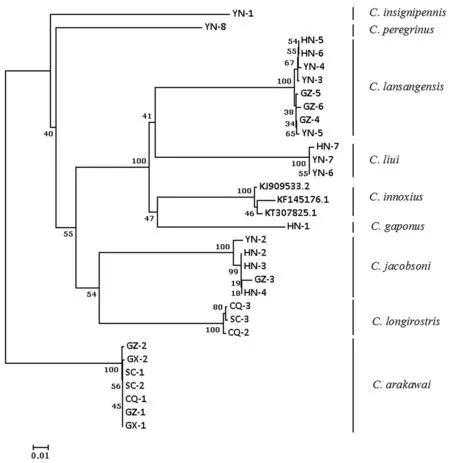
图2 基于线粒体COI序列构建的NJ树Fig.2 Neighbour-Joining tree constructed based on mitochondrial COI sequence
表4 林岛库蠓C. gaponus与无害库蠓C. innoxius形态特征对比
Tab.4 Comparison of morphological features of C. gaponus and C. innoxius

种名接眼式小眼面间柔毛感觉器窝翅径端淡斑延伸至中1脉翅臀室淡斑数目翅中4室基部前缘有淡色带翅径5室淡斑邻接端缘翅中1室淡斑和中2室淡斑连接受精囊形态林岛库蠓C.gaponus是无有是2是否是椭圆形无害库蠓C.innoxius是无有否2是否是近球形
3 讨 论
线粒体DNA(mtDNA)具多拷贝、分子量小、母系遗传、突变速度快等特点[5],能准确鉴别物种。COI是线粒体上约650 bp的DNA片段,结构较保守,具有较多系统发育信号,适用于解析亲缘关系密切的生物类群[6-8]。目前,COI基因已应用于蠓分类鉴定和系统发育分析等研究,且已有研究表明利用COI基因进行种类鉴定可鉴别95%的已知库蠓[9]。另外,COI基因很大程度上克服了传统形态学在鉴定蛹、幼虫及肢体残缺形态的成虫时难度大的缺陷[10]。因此,本研究基于形态学特征和COI分子标记数据对库蠓属二囊亚属的9个近似种进行分析,验证了线粒体COI基因序列可作为库蠓近似种的分类鉴定和系统发育分析的有效分子标记。
昆虫系统演化的研究是从分子水平上找出其种间DNA序列的特异性差异,以探索昆虫种、属间的相对亲缘关系,同时也为昆虫的分子鉴定奠定基础[11-12]。在库蠓近似种遗传距离的研究结果中,种间遗传距离 (0.154~0.302) 明显大于种内遗传距离(0.000~0.019),其中种内最大遗传距离0.019亦小于种间最小遗传距离0.154,说明种间遗传距离较大,足以区分不同物种,而种内个体变异在一定范围内,没有超出种的界限,进而可以准确区分二者,且种间是种内的平均遗传距离(0.258/0.0095)的27倍,完全符合种间差异大于种内差异10倍的物种鉴定规则[13-14]。同时,林岛库蠓C.gaponus与无害库蠓C.innoxius的种间遗传距离为最小(K-2P:0.154),这与其在形态学上为极度近似种的鉴定结果相符合。
本研究中库蠓属二囊亚属的9种近似种的NJ树和ML树结果基本一致,但仍存在部分近似种的种间和种内亲缘关系的微小差异,然而造成这种差异的原因在于两种建树方法的理论基础以及其建树的偏向性方面存在差异。从本研究的系统发育树聚类结果可以得出:①内群(二囊亚属)与外群(荒川库蠓C.arakawai)之间聚类分析明显各自形成两大支,同时其不同亚属间遗传距离也显著大于亚属间;②无害库蠓C.innoxius与林岛库蠓C.gaponus首先聚在一起,亲缘关系最近并构成姐妹群,这与K-2P遗传距离及形态学鉴定结果相一致,这进一步证实了COI分子鉴定的可靠性;③利用不同的方法构建的系统发育树中,荒川库蠓C.arakawai的四个不同地理种群(重庆北碚区、广西大瑶山自然保护区、贵州遵义市、四川泸州市)均并为一支,形成同一单系(群)且与采自同一地理生境(海南鹦哥岭自然保护区)下的林岛库蠓C.gaponus、连斑库蠓C.jacobsoni、南山库蠓C.lansangensis和新竹库蠓C.liui等4个不同种之间并无交叉,各自形成一单系(群),这充分表明以COI基因构建的系统发育树能有效区分不同地理种群的同一物种及同一地理生境下的不同物种。同时,近似种间各分支都有相对较高的置信值,说明近似种间的区分度好且可靠程度较高。然而,在种间和种内一些分支上亦存在相对较低的置信值(低于50),这可能是由于COI序列碱基组成及变异速度存在差异以及有效信息有限的短线粒体COI序列所致拓扑结构无法得到很好支持的原因[15],故在今后的研究中应从更丰富的样本数量以及更全面的有效序列信息等方面着手。
目前,已有多个研究表明在对蚤目、同翅目和双翅目(摇蚊科、麻蝇科)等昆虫进行系统发育分析时联合mtDNA、rDNA以及形态学特征综合分析得到的系统发育分析结果比单独使用线粒体COI基因分析得到的结果更为理想,有研究表明联合COI和Cytb基因进行某类群鉴定的成功率高达99%[16-18]。当然COI基因也并非在所有类群都适合进行分类,如腔肠类动物、马达加斯加蛙类等类群就不适合使用COI作为分子标记[9]。因此,联合多个互补基因序列以及形态学数据综合分析可得到更为准确的种类鉴定和系统发育分析结果。另外,研究者们也在不断探索发现新的分子标记并应用于昆虫系统发育研究中,如编码蛋白基因CAD序列,其在很大程度上更有利于分析昆虫的进化关系,且在库蠓的分子系统学研究中具有很大的价值和应用前景[19-22]。因此,在面对传统形态学鉴定和分子分类方法存在困难和局限时,应结合形态学特征和多个分子标记数据来对库蠓的物种鉴定和系统发育进行综合分析,并积极探寻数量更多、效果更好的分子标记来进行更为科学合理的实验研究。
参考文献:
[1] Borkent A. World species of biting midges (Diptera: Ceratopogonidae)[J]. Bam Mus Nat Hist, 2006, 233(233):1-257. DOI: 10.11646/zootaxa.3879.1.1
[2] 虞以新, 刘金华, 刘国平, 等. 中国蠓科昆虫[M]. 北京: 军事医学科学出版社, 2006: 1-42, 815-817.
[3] 王飞鹏, 黄恩炯, 蔡亨忠, 等. 吸血蠓及其传播的疾病[J]. 昆虫知识, 2010, 47 (6): 1270-1273.
[4] Henni HL, Sauvage F, Ninio C, et al. Wing geometry as a tool for discrimination ofObsoletusgroup (Diptera: Ceratopogonidae:Culicoides) in France[J].Infect Genet Evol, 2014, 21 (1): 110-117. DOI:10.1016/j.meegid.2013.10.008
[5] Simon C, Frati F, Beckenbach A, et al. Evolution, weighting, and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers[J]. Ann Entomol Soc Am, 1994, 87 (6): 651-701. DOI: 10.1093/aesa/87.6.651
[6] Hebert PDN, Cywinska A, Ball SL, et al. Biological identifications through DNA barcodes[J]. Proc R Soc Lond Ser B Biol Sci, 2003, 270 (7): 313-321. DOI:10.1098/rspb.2002.2218
[7] 岳巧云, 邱德义, 黄艺文, 等. 应用DNA条形码技术鉴定未知双翅目蛹[J]. 中国国境卫生检疫杂志, 2011, 34 (5): 343-347.DOI:10.16408/j.1004-9770.2011.05.029
[8] Taberlet P, Coissac E, Pompanon F, et al. Power and limitations of the chloroplast trnL (UAA) intron for plant DNA barcoding[J]. Nucleic Acids Res, 2007, 35 (3): e14. DOI:10.1093/nar/gkl938
[9] Ander M, Troell K, Chirico J. Barcoding of biting midges in the genus Culicoides: a tool for species determination[J]. Med Vet Entomol, 2013, 27 (3): 323-331. DOI:10.1111/j.1365-2915.2012.01050.x
[10] Nolan DV, Carpenter S, Barber J, et al. Rapid diagnostic PCR assays for members of theCulicoidesobsoletusandCulicoidespulicarisspecies complexes, implicated vectors of bluetongue virus in Europe[J]. Vet Microbiol, 2007, 124(1/2): 82-94. DOI:10.1016/j.vetmic.2007.03.019
[11] 叶军, 周国梁, 易建平, 等. 地中海实蝇幼虫分子鉴定[J]. 昆虫知识, 2007, 44 (4): 562-566.
[12] 岳巧云, 冯文军, 王章根, 等. DNA条形码技术在鉴定蛆症异蚤蝇中的应用[J]. 中国卫生检验杂志, 2010, 20 (6): 1400-1402.
[13] Hebert PDN, Stoeckle MY, Zemlak TS, et a1. Franci identification of birds through DNA barcodes[J]. Plos Biol, 2004, 2 (10): 1657-1663. DOI:10.1371/journal.pbio.0020312
[14] Hebert PDN, Penton EH, Burns JM, et a1. Ten species in one: DNA barcoding reveals cryptic species in the neotropical skipper butterflyAstraptesfulgerator[J]. Proc Natl Acad Sci USA, 2004, 101 (41): 14812-14817. DOI:10.1073/pnas.0406166101
[15] 李玮玮, 张修月, 陈伟才, 等. 小蔗螟Cytb基因序列的分析研究 (鳞翅目:螟蛾总科:草螟亚科)[J]. 四川动物, 2009, 28 (3): 353-357.
[16] Carew ME, Pettigrove VJ, Metzeling L, et a1. Environmental monitoring using next generation sequencing: rapid identification of macroinvertebrate bioindicator species[J]. Front Zool, 2013, 10 (1): 45-50. DOI:10.1186/1742-9994-10-45
[17] Guo Y, Cai J, Chang Y, et a1. Identification of forensically importantSarcophagidFlies(Diptera: Sarcophagidae) in China, based on COI and 16S rDNA gene sequences[J]. J Forensic Sci, 2011, 56 (6): 1534-1540. DOI: 10.1111/j.1556-4029.2011.01882.x
[18] 王德明, 林杨. 线粒体DNA基因序列在昆虫分子系统学研究中的应用[J]. 广东农业科学, 2010, 37 (6): 188-190.DOI: 10.3969/j.issn.1004-874X.2010.06.078
[19] Wiegmann BM, Trautwein MD, Kim JW, et al. Single-copy nuclear genes resolve the phylogeny of the holometabolous insects[J]. BMC Biol, 2009, 7(1): 34. DOI: 10.1186/1741-7007-7-34
[20] Moulton JK, Wiegmann BM. Evolution and phylogenetic utility of CAD (rudimentary) among Mesozoic-aged Eremoneuran Diptera (Insecta)[J]. Mol Phylogenet Evol, 2004, 31(1): 363-378. DOI: 10.1016/S1055-7903(03)00284-7
[21] Bellis GA, Dyce AL, Gopurenko D, et al. Revision of the immaculatus group ofCulicoidesLatreill (Diptera: Ceratopogonidae) from the Australasian Region with description of two new species[J]. Zootaxa, 2013, 3680 (1): 15-37. DOI: 10.11646/zootaxa.3680.1.4
[22] Bellis G, Dyce A, Gopurenko D, et al. Revision of theCulicoides(Avaritia)Imicolacomplex Khamala & Kettle (Diptera: Ceratopogonidae) from the Australasian region[J]. Zootaxa, 2014, 3768(4): 401-427. DOI: 10.11646/zootaxa.3768.4.1
