耐碳青霉烯类肠杆菌科细菌耐药基因分析
2018-04-16刘宇豪次仁曲珍周俊英武汉大学武汉43007
刘宇豪,陈 晨,次仁曲珍,周俊英,3 (.武汉大学,武汉 43007;
2.西藏山南妇幼保健院,西藏山南 856000;3.武汉大学中南医院,武汉 430071)
由于抗生素的不合理使用,细菌对抗生素的耐药率越来越高,耐碳青霉烯类肠杆菌科细菌(CRE)检出率也逐年递增,CRE耐药机制非常复杂,已在世界范围内引起广泛关注[1,2]。本研究通过对中南医院CRE耐药菌株分布及耐药基因的分析,为CRE的感染控制提供依据。
1 材料和方法
1.1菌株来源收集2015~2017年武汉大学中南医院检验科保存的53株CRE。
1.2主要试剂Taq酶(美国Fermentas公司),dNTP(湖北晶茂生物技术公司),DNA Marker 100bp(广州东盛生物科技),6×Loading Buffer(大连TaKaRa公司),细菌基因组DNA小量提取试剂盒(北京庄盟公司),Goldview核酸染料(北京赛百盛生物工程公司)。
1.3PCR引物合成KPC,IMP,OXA-48,VIM和NDM 5种引物由北京擎科公司合成,引物系列见表1。
表1

检测碳青霉烯酶基因所用的PCR引物
注:*Y=C or T。
1.4PCR反应条件blaKPC及blaIMP的反应条件为:94℃预变性10 min,94℃变性30 s,55℃ 退火40 s,72℃延伸50 s,共36个循环,最后72℃5 min。blaOXA,blaVIM及blaNDM的反应条件为:94℃预变性10 min;94℃30 s,58℃40 s,72℃50 s,36个循环,72℃延伸5 min。
1.5药敏试验按梅里埃药敏卡AST-13说明书进行。
1.6CRE表型删选实验采用改良Hodge试验。
1.7CRE菌株基因组DNA提取按照试剂说明书进行。
1.8PCR扩增体系总体积为25 μl,包含DNA模板2 μl,MgCl225 mmol/L 2.5 μl,dNTP(10 mmol/L)0.5 μl,上、下引物各为1 μl,10×PCR缓冲液2.5 μl,DNA聚合酶(5 U/μl)1 μl,双蒸水14.5 μl。
1.9耐药性分析采用WHONET5.6软件进行统计学分析。
2 结果
2.1CRE菌株的分布53株CRE菌株中,肺炎克雷伯菌34株占64.15%,大肠埃希菌12株占22.64%,阴沟肠杆菌7株占13.21%;科室分布为重症监护病房15株CRE占28.30%,呼吸内科病房9株占16.98%,干部病房6株占11.32%,其余科室23株占43.40%;标本类型为尿液标本20株CRE占37.74%,痰液16株占30.19%,引流液9株占16.98%,伤口分泌物4株占7.55%,血液2株占3.77%,脑脊液2株占3.77%。
2.2CRE的耐药率2016年送检标本中,检出肺炎克雷伯菌811株,CRE 69株,检出率为8.51%;大肠埃希菌1 581株,CRE 33株,检出率为2.09%;阴沟肠杆菌225株,CRE 15株,检出率为6.67%。耐碳青霉烯类的肺炎克雷伯菌和大肠埃希菌耐药情况见图1,图2。
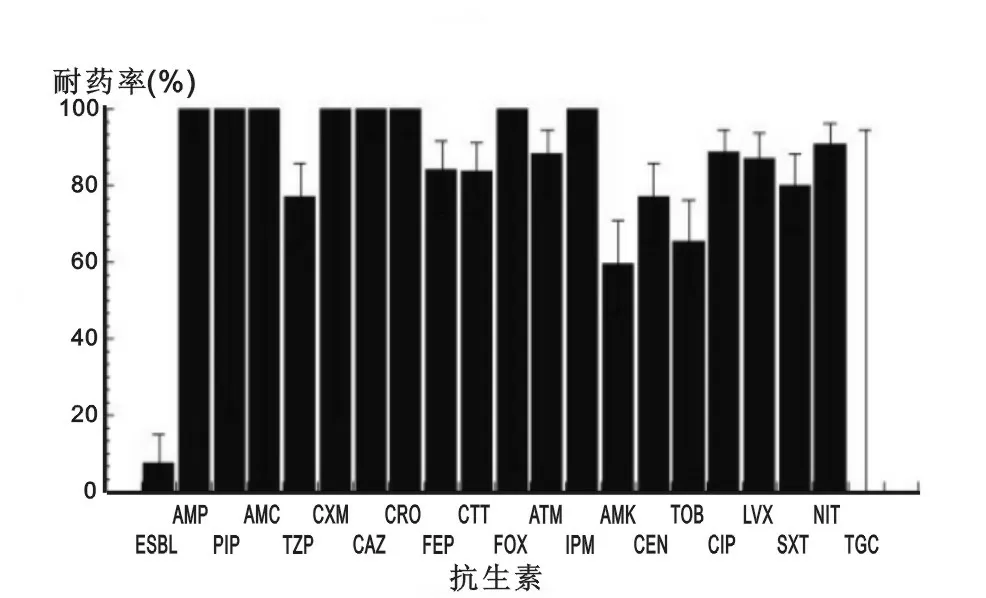
图1 肺炎克雷伯菌耐药率(%)
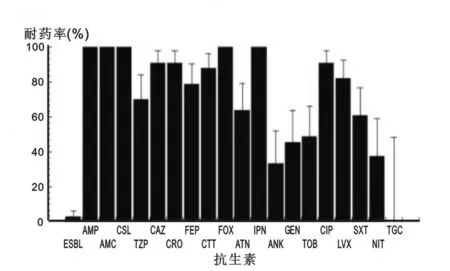
图2 大肠埃希菌耐药率(%)
2.3亚胺培南、美罗培南和厄他培南3种药敏纸片的改良Hodge试验53株CRE菌株中,改良Hodge试验阳性株有31株,占58.49%。其中亚胺培南、美罗培南和厄他培南3种药敏纸片的Hodge试验结果一致,见图3。
2.4CRE菌株耐药相关基因扩增结果31株CRE中,携带blaKPC(798bp)基因有29株,检出率96.67%,携带blaNDM (621bp)基因有9株,检出率29.03%,同时扩增出blaKPC和blaNDM基因有8株,检出率25.81%,未扩增出blaIMP,blaOXA,blaVIM基因,见图4,图5。

图3 改良Hodge试验结果示意图
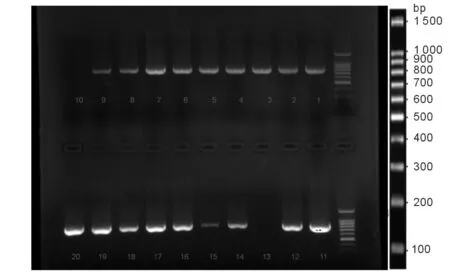
图4 blaKPC电泳结果示意图

图5 blaNDM基因型电泳结果示意图
3讨论药敏结果显示,CRE对碳青霉烯类、头孢菌素类、氨曲南和氟喹诺酮类等抗菌药物耐药率较高,对庆大霉素、妥布霉素、阿米卡星、复方新诺明、替加环素等抗菌药物耐药率较低。治疗CRE感染时,建议临床优先考虑替加环素,或替加环素与氨基糖苷类抗生素联合使用。
改良Hodge试验是CLSI推荐检测碳青霉烯酶表型的筛查方法,本实验53株CRE中,Hodge试验阳性31株,其阳性菌株少的原因可能与某些菌株产碳青霉烯酶量较低有关[3]。Hodge试验采用亚胺培南、美罗培南和厄他培南3种药敏纸片,其试验结果一致,但观察结果发现:厄他培南和美罗培南矢状箭头较为明显,亚胺培南不及它们明显,因此建议Hodge试验时,首选厄他培南或美罗培南纸片。
在药物选择压力下,CRE携带耐药基因的质粒、转座子等可发生水平传播[4],抗菌药物选择压力不仅能使耐药基因广泛传播,而且会促进耐药基因通过突变、插入序列等方式,使自身基因发生变化,进而使耐药性发生改变,引起耐药基因的传播[5]。本研究中发现1株不常见耐药基因型,推测该菌株可能携带一种新的耐碳青霉烯酶基因,因此建议扩大耐药基因检测范围,或者测序进一步确认该菌株的基因型。31株耐药基因分析中,CRE基因型主要是KPC,其次是NDM,这与目前国内及世界范围内,肠杆菌科细菌携带碳青霉烯酶基因为KPC-2和NDM-1一致[6]。重症医学科和呼吸内科病房检出的CRE主要是肺炎克雷伯菌,耐药基因型主要是KPC型,干部病房CRE主要是大肠埃希菌,耐药基因型主要是KPC和NDM的混合型。医院应重点加强对这些科室CRE的监管力度,严格控制临床医生合理使用抗生素,特别是碳青霉烯类药物的用药指征,防止CRE区域性传播。
参考文献:
[1]Yong D,Toleman MA,Giske CG,et al.Characterization of a new Metallo-beta-lactamase gene,bla(NDM-1),and a novel erythromycin esterase gene carried on a unique genetic structure inKlebsiellapneumoniaesequence type 14 from India[J].Antimicrob Agents Chemother,2009,53(12):5046-5054.
[2]张芳芳,王晓丽,瞿洪平,等.肠杆菌科细菌产碳青霉烯酶的主要类型和流行病学分析[J],中国感染与化疗杂志,2014,14(6):521-525.
Zhang FF,Wang XL,Qu HP,et al.Prevalence and genotypes of carbapenemase-producingEnterobacteriaceae[J].Chinese Journal of Infection and Chemotherapy,2014,14(6):521-525.
[3]Amjad A,Mirza I,Abbasis,et al.Modified hodge te-st:A simple and effective test for detection of carbapenemase production[J].Iran J Microbiol,2011,3(4):189-193.
[4]Schuurmans JM,Van Hijum SA,Piet JR,et al.Effect of growth rate and selection pressure on rates of transfer of an antibiotic resistance plasmid betweenE.colistrains[J].Plasmid,2014,72:1-8.
[5]Jacquier H,Marcade G,Raffoux E,et al.In vivo selection of a complex mutant TEM(CMT) from an inhibitor-resistant TEM(IRT)during ceftazidime therapy[J].J Antimicrob Chemother,2013,68(12):2792-2796.
[6]Li H,Zhang J,Liu Y,et al.Molecular characteristics of carbapenemase-producingEnterobacteriaceaein China from 2008 to 2011:predominance of KPC-2 enzyme[J].Diagn Microbiol Infect Dis,2014,78(1):63-65.
