柴达敏盐湖产胞外蛋白酶嗜盐古菌的筛选及其酶学性质
2018-04-12侯靖许文梅崔恒林
侯靖,许文梅,崔恒林
(江苏大学 食品与生物工程学院,江苏 镇江,212013)
蛋白酶能催化蛋白质肽键的水解,是用途最广泛的酶制剂之一,广泛应用于食品、洗涤、皮革等领域,在全球的销售额占酶制剂总份额的46%左右[1]。目前对蛋白酶的研究主要集中在寻求具有特殊性质的微生物酶来解决工业应用中酶蛋白有限稳定性的问题以及扩大其应用领域,这些特殊的性质包括耐热性、耐盐性、稳定性强等[2]。极端微生物是最适生活在极端环境中的微生物的总称,包括嗜热、嗜冷、嗜盐、嗜酸、嗜碱、嗜压等多种类型。极端微生物通常具有独特的生理机制及代谢产物,能够提供丰富的极端酶资源。
嗜盐古菌是一类通常生活在盐湖、晒盐场、腌制食品等高盐环境中的极端微生物,与大部分极端微生物相比,具有易培养、生长快等特点,在生物技术方面具有广泛的应用前景[3]。从自然环境中筛选具有优良酶学性质的嗜盐古菌胞外蛋白酶成为近年来国内外学者的研究热点。DAMMAK等人从突尼斯的斯法克斯晒盐场分离得到1株高产胞外蛋白酶的嗜盐古菌HalorubrumezzemoulenseETR14,并报道了其蛋白酶耐盐碱、热稳定的特征[4]。ELBANNA等人从位于埃及法尤姆美利斯湖的Emisal盐业公司的粗制盐中分离获得一株产蛋白酶菌株Halobacteriumsp. HP25,并发现其蛋白酶耐盐碱、耐热,并且能够耐受一定的有机溶剂和表面活性剂[5]。AKOLKAR等人从印度坎德拉盐田的卤水样品中分离获得Halobacteriumsp. SP1(1)菌株,研究了其胞外蛋白酶酶学性质[6],并将其用于改良传统鱼露发酵工艺,缩短鱼露发酵时间[7]。我国学者石万良等人对分离自湖北应城盐矿的Natrinemasp. R6-5菌株所产胞外蛋白酶进行了纯化以及酶学性质表征[8],高瑞昌等人研究了分离自江苏如东盐田的HalogranumrubrumRO2-11菌株的胞外蛋白酶性质[9]。然而,相对于丰富的高盐环境和嗜盐古菌资源,对其胞外蛋白酶的研究尚处于起步阶段,尤其是对反应条件要求不严格的蛋白酶资源仍有很大挖掘空间[5]。
本研究从分离自我国内蒙古柴达敏盐湖的嗜盐古菌中筛选高产胞外蛋白酶菌株,并对所产蛋白酶的酶学性质进行初步研究,为进一步分离纯化及工业应用提供理论基础。
1 材料与方法
1.1 材料与试剂
39株嗜盐古菌分离自内蒙古柴达敏盐湖。
酵母提取物、鱼蛋白胨:OXOID试剂公司;脱脂奶粉、酪蛋白、PCR扩增试剂盒:上海生工生物技术服务有限公司;其他试剂:国药集团化学试剂有限公司,均为分析纯。
NHM培养基成分(g/L):酵母提取物0.05,鱼蛋白胨0.25,丙酮酸钠1.0,KCl 5.4,CaCl20.29,NH4Cl 0.27,MgSO4·7H2O 26.8,MgCl2·6H2O 23.0,NaCl 184.0,K2HPO40.3,pH 7.0。AHM培养基成分(g/L):酵母提取物0.05,鱼蛋白胨0.25,丙酮酸钠1.0,KCl 5.4,NaNO30.5,CaCl20.1,MgSO4·7H2O 0.1,NaCl 184.0,pH 9.0。固体培养基添加20 g/L琼脂。
本研究使用的缓冲液包括50 mmol/L磷酸-氢氧化钠缓冲液(pH 6.0),50 mmol/L Tris-HCl缓冲液(pH 7.0、8.0、9.0),50 mmol/L硼砂-氢氧化钠缓冲液(pH 10.0、11.0),以及含有不同浓度NaCl的相应pH的缓冲液。
1.2 仪器与设备
电子天平:上海精科天平仪器厂;pHS-3TC型酸度计:上海天达仪器有限公司;全自动灭菌锅:上海三申医疗器械有限公司;超净工作台:苏州净化设备有限公司;LRH-250生化培养箱:上海一恒科技有限公司;HBA-1151PCR仪:MJ Research公司;DYY-6C型电泳仪:北京市六一仪器厂;HYG-C型多功能摇床:太仓实验设备厂;Avanti J-14离心机:Beckman公司;HH-S2数显恒温水浴锅:江苏省金坛市医疗仪器厂;DU800TM核酸蛋白分析仪:Beckman-Coulter公司。
1.3 方法
1.3.1产胞外蛋白酶菌株筛选
CDM1~CDM12这12株嗜盐古菌经NHM培养基活化,CDA1~CDA27这27株嗜盐古菌经AHM培养基活化。配制含5 g/L脱脂奶粉的NHM或AHM平板,取5 μL活化后的菌液接种到脱脂奶粉平板,于37 ℃培养4 d形成明显的菌苔。产胞外蛋白酶菌株的菌苔周围有透明圈,否则,没有透明圈。根据透明圈直径与菌苔直径之比初步评价产蛋白酶能力。
1.3.216S rRNA基因序列分析
取少量菌体加入30~50 μL无菌水,沸水浴10 min裂解细胞,作为PCR模板。引物0018F和1518R用于扩增16S rRNA基因[10]。PCR产物测序由北京诺赛基因组研究中心完成。NCBI BLAST用于基因序列分析。
1.3.3粗酶液的制备
CDM1~CDM12这12株嗜盐古菌经NHM培养基活化,CDA1~CDA27这27株嗜盐古菌经AHM培养基活化,以2%的比例接种于200 mL NHM或AHM培养基中,37 ℃,160 r/min培养至稳定期。于4 ℃,9 000×g离心15 min,上清液经0.22 μm微孔滤膜过滤,再采用截留分子量为10 kDa的超滤离心管(Millipore)浓缩10倍,即得粗酶液。
1.3.4蛋白酶活力的测定
参照福林酚法测定蛋白酶活力[11]。称取2.00 g酪蛋白,加入70 mL缓冲液和100 μL 10 mol/L NaOH溶液,沸水浴加热至酪蛋白完全溶解,定容至100 mL,调节pH值至缓冲液相应的值,即得酪蛋白溶液。
将25 μL酶液用缓冲液稀释10倍至250 μL后,加入250 μL相应缓冲液配制的酪蛋白溶液,一定温度下反应30 min,加入500 μL 100 g/L三氯乙酸溶液终止反应。37 ℃放置30 min后,于4 ℃,9 000×g离心10 min,向500 μL上清液中加入2.5 mL 0.4 mol/L Na2CO3溶液和0.5 mL 33%福林酚试剂,40 ℃显色20 min,于4 ℃,9 000×g离心10 min,于680 nm测定上清液的吸光度。空白对照反应体系即在酶液中先加入三氯乙酸溶液,后加入酪蛋白溶液。蛋白酶将酪蛋白水解成酪氨酸等物质,利用酪氨酸标准曲线,得到酪氨酸的量计算蛋白酶活力。1个酶活力单位(U)定义为特定反应条件下,每分钟产生1 μg酪氨酸的酶量。
1.3.5酶学性质的研究
1.3.5.1最适反应温度
将25 μL酶液用2 mol/L NaCl,pH 7.0缓冲液稀释10倍至250 μL后,分别在35、40、45、50、55、60、65、70、75、80 ℃下反应,测定酶活力。
1.3.5.2热稳定性
将25 μL酶液用2 mol/L NaCl,pH 7.0缓冲液稀释10倍至250 μL后,在30、50、70、90 ℃下分别放置0、20、40、60 min,在最适温度下测定酶活力。
1.3.5.3最适反应pH
分别用含2 mol/L NaCl的pH 6.0、7.0、8.0、9.0、10.0、11.0缓冲液将25 μL酶液稀释10倍至250 μL后,在最适温度下测定酶活力。
1.3.5.4最适反应NaCl浓度
分别用含有0、0.5、1.0、1.5、2.0、2.5和3.0 mol/L NaCl的缓冲液将25 μL酶液稀释10倍至250 μL后,在最适温度、最适pH下测定酶活力。
1.3.5.5NaCl浓度稳定性
分别用含有0、1.0、2.0、3.0 mol/L NaCl的缓冲液将25 μL酶液稀释10倍至250 μL后,在30 ℃放置0、20、40、60 min,随后分别与250 μL含3.0、2.0、1.0、0 mol/L NaCl的酪蛋白溶液在最适温度、最适pH下测定酶活力。
1.3.5.6抑制剂对蛋白酶活力的影响
用最适缓冲液将25 μL酶液稀释10倍至250 μL后,分别加入1 mmol/L苯甲基磺酰氟(phenylmethanesulfonyl fluoride,PMSF)、1 mmol/L对氯汞苯甲酸(p-chloromercuribenzoic acid,PCMB)、5 mmol/L乙二胺四乙酸(ethylenediaminetetraacetic acid,EDTA)、0.1 mmol/L胃蛋白酶抑制剂(pepstatin A),30 ℃放置20 min,在最适条件下测定酶活力。对照反应体系不加抑制剂。
1.4 数据分析
酶学性质研究所有试验均测定3次,以“平均值±标准差”表示。采用SPSS 18.0进行单因素方差分析(one-way ANOVA)和事后检验(Turkey法),p<0.05为差异有统计学意义。
2 结果与分析
2.1 产胞外蛋白酶菌株的筛选
从表1可以看出,分离自内蒙古柴达敏盐湖的39株嗜盐古菌中,8株能够在NHM或AHM-脱脂奶粉平板上产生水解圈。16S rRNA基因序列分析表明CDA5、CDA6、CDA14、CDA16和CDA17与HalovivaxcerinusIC35菌株的16S rRNA基因序列的相似性为95%~96%。CDM10与NatribaculumlongumTRM20345 的16S rRNA基因序列的相似性为93%。CDM12与HalovariusluteusDA50的16S rRNA基因序列的相似性为95%。CDA27与NatronorubrumtexcoconenseJCM 17497的16S rRNA基因序列的相似性为99%。通常16S rRNA基因序列相似性小于98.65%认为属于不同的种[12],相似性小于95%认为属于不同的属[13]。因此,除了CDA27外,其他7株菌可能为新的分类单元。
8株菌水解圈直径与菌苔直径的比值范围为2.29~4.72,其中CDM10和CDM12比值在4.0以上。将这8株菌进行摇瓶发酵,在2 mol/L NaCl、35 ℃、pH 7.0的条件下测定酶活,CDM10和CDM12的蛋白酶活力分别为11.1和14.3 U/mL,其他菌株的酶活力均低于10 U/mL。因此,CDM10和CDM12用作进一步研究对象。总的来说,水解圈直径与菌苔直径的比值和液体发酵蛋白酶活的高低大致呈正相关。

表1 产胞外蛋白酶菌株Table 1 Extracellular protease-producing haloarchaeal strains
2.2 CDM10和CDM12胞外蛋白酶酶学性质研究
2.2.1最适反应温度与热稳定性
从图1可以看出,CDM10和CDM12的蛋白酶在35~80 ℃内均有酶活,随着温度升高,酶活先增加后降低。CDM10的蛋白酶最适反应温度为70~75 ℃(p>0.05),在65~80 ℃内具有较高酶活(最高酶活的80%以上)。CDM12的蛋白酶最适反应温度为60~75 ℃(p>0.05),在55~75 ℃内具有较高酶活(最高酶活的80%以上)。因此,CDM10和CDM12的胞外蛋白酶均具有良好的耐热性,其中CDM10的蛋白酶在55~70 ℃内酶活仍然随着温度升高迅速增加,而CDM12的蛋白酶在此温度范围内酶活缓慢增加。

图1 CDM10和CDM12蛋白酶的最适反应温度Fig.1 The optimum temperature for protease activitiesof CDM10 and CDM12
从图2可以看出,CDM10的蛋白酶在30、50 ℃具有良好的稳定性(p>0.05),在70、90 ℃随着处理时间增加酶活略有降低(p<0.05)。70 ℃处理60 min后酶活降低13.2%,90 ℃处理60 min后酶活降低17.6%。CDM12的蛋白酶在30、50℃具有良好的稳定性(p>0.05),在70、90 ℃随着处理时间延长酶活略有降低(p<0.05)。70 ℃放置60 min后酶活降低19.0%,90 ℃放置60 min后酶活降低30.1%。因此,CDM10和CDM12的胞外蛋白酶具有较好的热稳定性,CDM10胞外蛋白酶的热稳定性优于CDM12。蛋白酶良好的热稳定性对于工业应用非常重要,因为很多工业过程均需在一定的温度维持较长时间。

图2 不同温度处理对CDM10(a)和CDM12(b)蛋白酶活力的影响Fig.2 Effect of pre-incubation at different temperatureson protease activities of CDM10 (a) and CDM12 (b)
2.2.2最适反应pH
从图3可以看出,CDM10和CDM12的蛋白酶在pH 6.0~11.0范围内均具有催化活性,随着pH升高,酶活先升高后降低。CDM10蛋白酶的最适反应pH为8.0~9.0(p>0.05)。pH 6.0时的酶活为最高酶活的43.5%;pH 11.0时的酶活最低,为最高酶活的16.5%。CDM12蛋白酶的最适反应pH为7.0~9.0(p>0.05)。pH 6.0时的酶活为最高酶活的60.1%;pH 11.0时的酶活最低,为最高酶活的42.4%。总的来说,CDM10的胞外蛋白酶比CDM12的蛋白酶对pH的变化更敏感。
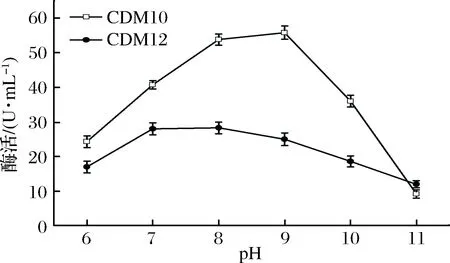
图3 CDM10和CDM12蛋白酶的最适反应pHFig.3 The optimum pH for protease activities ofCDM10 and CDM12
2.2.3最适反应NaCl浓度与盐度稳定性
从图4可以看出,CDM10和CDM12的蛋白酶在0.0~3.0 mol/L NaCl浓度范围内均具有催化活力,随着NaCl浓度升高,酶活先升高后下降。CDM10蛋白酶的最适反应NaCl浓度为1.5 mol/L,在1.0~2.0 mol/L NaCl浓度范围内具有较高活性(最高酶活的80%以上)。NaCl浓度为0.0 mol/L时的酶活为最高酶活的60.6%,NaCl浓度为3.0 mol/L时的活性为最高活性的48.0%。CDM12蛋白酶的最适反应NaCl浓度为1.5~2.5 mol/L(p>0.05),在1.0~3.0 mol/L NaCl浓度范围内均具有较高活性(最高酶活的80%以上)。NaCl浓度为0.0 mol/L时的酶活为最高酶活的45.4%。因此,CDM10和CDM12的蛋白酶具有较广的盐浓度适应性。
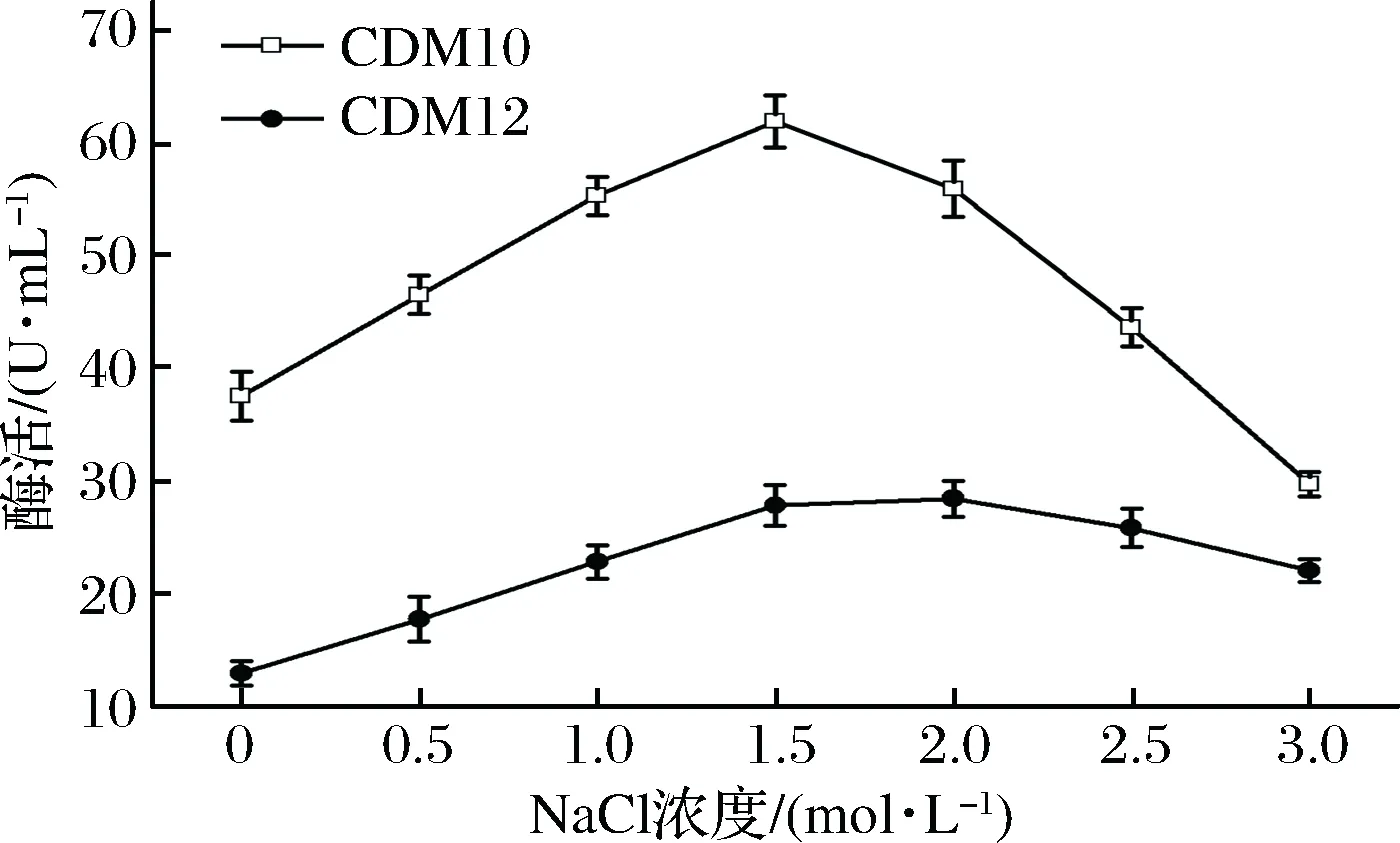
图4 CDM10和CDM12蛋白酶的最适反应NaCl浓度Fig.4 The optimum NaCl concentration for proteaseactivities of CDM10 and CDM12
从图5可以看出,CDM10和CDM12的胞外蛋白酶在0.0、1.0、2.0、3.0 mol/L NaCl中均具有良好的稳定性(p>0.05),暗示其对低盐和高盐浓度均有较好的耐受性。普通蛋白质在高盐环境中会发生盐析沉淀从而变性失活,嗜盐古菌蛋白质独特的结构特征使得能够在高盐环境中发挥活性,即主要通过增加表面酸性氨基酸残基的比例以形成负电屏蔽,从而在蛋白周围形成一层水化层来维持高盐环境中的活性和稳定性[14]。然而,目前报道的很多嗜盐古菌胞外蛋白酶,如HalogeometricumborinquenseTSS101[15]、Natronococcusoccultus[16]的胞外蛋白酶,仅在高盐浓度下具有活性,在低盐浓度下会失活。CDM10和CDM12蛋白酶广泛的盐浓度适应性将使其在高盐或低盐工业中均能发挥作用。

图5 不同NaCl浓度处理对CDM10(a)和CDM12(b)蛋白酶活力的影响Fig.5 Effect of pre-incubation at different NaCl concentrationson protease activities of CDM10 (a) and CDM12 (b)
2.2.4抑制剂对蛋白酶活性的影响
根据活性中心的功能基团,蛋白酶主要分为天冬氨酸蛋白酶、丝氨酸蛋白酶、金属蛋白酶和半胱氨酸蛋白酶[17]。为了探究CDM10和CDM12胞外蛋白酶种类,分别测定不同的蛋白酶抑制剂对胞外蛋白酶酶活的影响。从图6可以看出,丝氨酸蛋白酶抑制剂PMSF使CDM10和CDM12胞外蛋白酶活性分别降低了94.4%和90.7%。

图6 抑制剂对CDM10(a)和CDM12(b)蛋白酶活力的影响Fig.6 Effect of inhibitors on protease activities ofCDM10 (a) and CDM12 (b)
半胱氨酸蛋白酶抑制剂PCMB使CDM10和CDM12胞外蛋白酶活力分别降低了47.0%和65.9%。金属蛋白酶抑制剂EDTA可能通过螯合金属离子使酶活性中心失去辅因子从而使CDM10和CDM12胞外蛋白酶活力分别降低了33.0%和60.4%。天冬氨酸蛋白酶抑制剂pepstatin A对酶活没有显著影响(p>0.05)。这些结果表明CDM10和CDM12的胞外蛋白酶是丝氨酸蛋白酶,半胱氨酸和金属离子对于蛋白酶活性的发挥具有重要的作用。
3 结论
本研究从分离自内蒙古柴达敏盐湖的39株嗜盐古菌中筛选出8株产胞外蛋白酶菌株,16S rRNA基因序列分析表明其中7株可能为新的分类单元,暗示着我国高盐环境中蕴藏着丰富的嗜盐古菌蛋白酶资源。CDM10和CDM12产蛋白酶能力最强,其蛋白酶具有良好的热稳定性,对低盐和高盐浓度均有较好的耐受性,因而具有广泛的应用前景。嗜盐古菌蛋白酶酶学特性的研究能够为其在工业技术领域的应用奠定基础。
[1]秦艳梅, 刘春卯, 郑翔, 等. 微生物中性蛋白酶在食品工业的最新进展及应用前景[J]. 食品工业, 2017, 38(3), 210-213.
[2]NIGAM P S. Microbial enzymes with special characteristics for biotechnological applications[J]. Biomolecules, 2013, 3(3): 597-611.
[3]MARGESIN R, SCHINNER F. Potential of halotolerant and halophilic microorganisms for biotechnology[J]. Extremophiles, 2001, 5(2): 73-83.
[4]DAMMAK D F, SMAOUI S M, GHANMI F, et al. Characterization of halo-alkaline and thermostable protease fromHalorubrumezzemoulensestrain ETR14 isolated from Sfax solar saltern in Tunisia[J]. Journal of Basic Microbiology, 2016, 56(4): 337-346.
[5]ELBANNA K, IBRAHIM I M, REVOL-JUNELLES A. Purification and characterization of halo-alkali-thermophilic protease fromHalobacteriumsp. strain HP25 isolated from raw salt, Lake Qarun, Fayoum, Egypt[J]. Extremophiles, 2015, 19(4): 763-774.
[6]AKOLKAR A V, DESHPANDE G M, RAVAL K N, et al. Organic solvent tolerance ofHalobacteriumsp. SP1(1) and its extracellular protease[J]. Journal of Basic Microbiology, 2008, 48(5): 421-425.
[7]AKOLKAR A V, DURAI D, DESAI A J.Halobacteriumsp. SP1(1) as a starter culture for accelerating fish sauce fermentation[J]. Journal of Applied Microbiology, 2010, 109(1): 44-53.
[8]石万良, 钟传奇, 唐兵, 等. 极端嗜盐古生菌 (Natrinemasp.) R6-5胞外嗜盐蛋白酶的纯化和性质研究[J]. 微生物学报, 2007, 47(1): 161-163.
[9]高瑞昌, 刘向东, 陆文婷, 等. 嗜盐古生菌HalogranumrubrumRO2-11胞外蛋白酶酶学特性研究[J]. 现代食品科技, 2015, 31(2): 32-36,92.
[10]CUI Heng-lin, ZHOU Pei-jin, OREN A, et al. Intraspecific polymorphism of 16S rRNA genes in two halophilic archaeal genera,HaloarculaandHalomicrobium[J]. Extremophiles, 2009, 13, 31-37.
[11]中华人民共和国专业标准. ZB X 66030—87. 蛋白酶活力测定法[S].
[12]KIM M, OH H, PARK S, et al. Towards a taxonomic coherence between average nucleotide identity and 16S rRNA gene sequence similarity for species demarcation of prokaryotes[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(2): 346-351.
[13]TINDALL B J, ROSSELLó-MóRA R, BUSSE H, et al. Notes on the characterization of prokaryote strains for taxonomic purposes[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60: 249-266.
[14]REED C J, LEWIS H, TREJO E, et al. Protein adaptations in archaeal extremophiles[J]. Archaea, 2013(1 448): 373-275.
[15]VIDYASAGAR M, PRAKASH S, LITCHFIELD C, et al. Purification and characterization of a thermostable, haloalkaliphilic extracellular serine protease from the extreme halophilic archaeonHalogeometricumborinquensestrain TSS101[J]. Archaea, 2006, 2(1): 51-57.
[16]STUDDERT C A, SEITZ M K H, GIL M I P, et al. Purification and biochemical characterization of the haloalkaliphilic archaeonNatronococcusoccultusextracellular serine protease[J]. Journal of Basic Microbiology, 2002, 41(6): 375-383.
[17]ZHOU Ming-yang, CHEN Xiu-lan, ZHAO Hui-lin, et al. Diversity of both the cultivable protease-producing bacteria and their extracellular proteases in the sediments of the South China sea[J]. Microbial Ecology, 2009, 58: 582-590.
