优化反应体系提升Phanta DNA聚合酶对非处理标本的检测性能
2018-04-11陈振华熊晓辉许文玲郭晓博任顺利韩锦雄张海如
陈振华,蔡 甜,熊晓辉,许文玲,郭晓博,任顺利,韩锦雄,张海如,4
(1.佛山市南海区人民医院,广东 佛山 528000;2.南京工业大学 食品与轻工学院,江苏 南京 211800;3.南京诺唯赞生物科技有限公司,江苏 南京 210034; 4.武汉生物工程学院 生命科学与技术学院,湖北 武汉 430415)
20世纪80年代诞生的PCR技术奠定了近代分子生物学发展的基础,然而直到热稳定的DNA聚合酶出现,PCR技术才得以被大规模、高通量应用[1-2]。如今,PCR技术和热稳定DNA聚合酶已被广泛应用于疾病诊断与治疗、传染病检测、药物作用机理、食品检测和法医鉴定等各个领域[3-6]。在多方面的应用中,对DNA聚合酶和PCR技术也提出了更高要求,如更高灵敏度、更低检测下限、更高产量、更高保真度、更快的反应速度、更广的模板兼容范围、对杂质更高的耐受能力、更高GC兼容性以及更低的成本等[7-9]。PCR反应性能一般可从两个方面得以提高,一是使用不同的DNA聚合酶,二是对反应条件进行优化[10-14]。
应用较为广泛的Taq DNA聚合酶具有5′-3′ DNA聚合酶活性和5′-3′ DNA外切酶活性[15],扩增能力强,但保真度不高。PfuDNA聚合酶分离于Pyrococcusfuriosis菌中,是另一类应用较为广泛的DNA聚合酶[16-17],具有5′-3′ DNA聚合酶活性和3′-5′ DNA外切酶活性,扩增性能较弱,但保真度较高。Phanta DNA聚合酶是基于PfuDNA聚合酶改造而来的一类DNA聚合酶,保真度和行进性均得到大幅提升,并适用于广泛的模板类型,是食品检测、法医鉴定和疾病诊断等方面的优选用酶。
基于之前对于DNA聚合酶的研究,笔者对Phanta DNA聚合酶的反应条件进行优化,在反应中调节不同的组分以增强聚合酶性能和PCR反应效果,提高对血液、植物和食品等非处理标本的扩增能力。
1 材料与方法
1.1 材料
1.1.1DNA聚合酶、添加剂和试剂
Phanta Super-Fidelity DNA Polymerase、Taq DNA Polymerase、dNTP、DNA Marker/Ladder、质粒提取试剂盒、基因组DNA提取试剂盒、血液基因组提取试剂盒,诺唯赞生物科技有限公司;植物基因组提取试剂盒,天根生化科技有限公司;甘油、二甲基亚砜(DMSO)、二硫苏糖醇(DTT)、MgCl2,Sigma公司;PCR引物由金斯瑞生物科技有限公司合成;其他试剂均为市售分析纯。
1.1.2模板和抑制剂
大肠杆菌DH5α,诺唯赞生物科技有限公司;pEGFP质粒,TaKaRa公司;Hela细胞,保存于诺唯赞生物科技有限公司细胞培养实验室;全血,来自于佛山市南海区人民医院;水稻叶片,南京农业大学赠送;食品(玉米),来自于超市。
1.1.3培养基和裂解液
液体LB培养基(g/L):蛋白胨 10,NaCl 10,酵母粉 5。
固体LB培养基(g/L):蛋白胨 10,NaCl 10,酵母粉 5,琼脂粉 20。
裂解液:20 mmol/L Tris-HCl,100 mmol/L EDTA,0.1% 十二烷基磺酸钠(SDS),pH 8.0。
1.2 方法
1.2.1DNA模板制备
将pEGFP质粒转化入大肠杆菌DH5α感受态细胞中,在含有50 μg/mL卡那霉素的固体LB培养基平板上,37 ℃培养过夜,挑取单克隆作为PCR反应模板。
血液基因组、细胞基因组和植物基因组的提取使用提取试剂盒,按照说明书进行提取。
取植物或食品浸泡入100 μL裂解液中,捣碎,加入蛋白酶K至终质量浓度为200 μg/mL。60 ℃加热10 min,然后95 ℃加热10 min。充分混匀,室温离心后取上清即可作为裂解液模板使用。
1.2.2PCR反应
在50 μL PCR反应体系中,加入10 μL Phanta Super-Fidelity DNA Polymerase配套的5× SF Buffer(含有10 mmol/L MgSO4),1 μL dNTP Mix (10 mmol/L each),上下游引物(10 μmol/L)各2 μL和1 μL Phanta Super-Fidelity DNA Polymerase。根据不同的试验设计,分别加入不同浓度DMSO和MgCl2以及不同量的模板,用灭菌超纯水补至50 μL。反应条件设置为预变性95 ℃ 5 min;循环反应:变性95 ℃ 30 s;退火60 ℃ 30 s;延伸72 ℃ 30 s/kb;共35个循环;彻底延伸72 ℃ 10 min。反应结束后通过琼脂糖凝胶电泳、EB染色,观察反应结果。Taq DNA聚合酶的反应体系和程序参照说明书设置。
1.2.3添加剂及浓度优化
选取甘油、DMSO、DTT等3种常用添加剂,测试其对Phanta DNA聚合酶扩增性能的影响[11]。在正常PCR反应体系中,分别加入不同量的甘油、DMSO和DTT,使反应体系中甘油和DMSO的质量分数分别为1%、2.5%、5%、7.5%和10%;DTT的浓度为1、2.5、5、7.5和10 mmol/L。每个反应体系加入50 ng Hela细胞基因组DNA作为模板。反应结束后通过琼脂糖凝胶电泳、EB染色,观察反应结果,确定最适添加剂及浓度。
1.2.4Mg2+浓度优化
在PCR反应体系中,分别加入不同量的MgCl2,使反应体系中Mg2+终浓度分别为2.5、3、3.5、4和5 mmol/L。每个反应体系加入50 ng Hela细胞基因组DNA作为模板。反应结束后通过琼脂糖凝胶电泳、EB染色,观察反应结果,确定最适Mg2+浓度。
1.2.5扩增灵敏度测试
分别使用正常反应体系和加入5% DMSO和3 mmol/L Mg2+(终浓度)的反应体系,加入不同量Hela细胞基因组DNA,使得每个反应中基因组DNA量分别为0.05、0.1、0.5、1、10、25和50 ng,并以Taq DNA聚合酶作为对照。反应结束后通过琼脂糖凝胶电泳、EB染色,观察反应结果,确定可扩增的最低模板起始量。
1.2.6植物粗品扩增测试
分别使用正常反应体系和加入5% DMSO和3 mmol/L Mg2+(终浓度)的反应体系,加入不同植物(水稻、小麦、拟南芥)叶片粗提液进行扩增。并以Taq DNA聚合酶为对照。反应结束后通过琼脂糖凝胶电泳、EB染色,观察反应结果,确定对植物粗品的耐受能力。
1.2.7全血扩增测试
分别使用正常反应体系和加入终质量分数5%的DMSO和3 mmol/L Mg2+(终浓度)的反应体系,加入不同体积(1、5、10、15、20和25 μL)的全血进行扩增,并以Taq DNA聚合酶作为对照。反应结束后通过琼脂糖凝胶电泳、EB染色,观察反应结果,确定可扩增的最高全血体积。
1.2.8食品粗品检测测试
挑取1个含有pEGFP质粒的大肠杆菌单克隆加入到100 μL食品(玉米)裂解液中,涡旋混匀。以混合物为模板,分别使用正常反应体系和加入终质量分数5% DMSO和3 mmol/L Mg2+的反应体系,在体系中加入2、4、6、8和10 μL的混合液进行PCR反应。反应结束后通过琼脂糖凝胶电泳、EB染色,观察反应结果,确定对食品粗品的耐受能力。
2 结果与讨论
在分子诊断、食品检测、法医鉴定等应用中,很多情况需要对样品或样品粗处理品进行直接检测,有时即使提纯的DNA也会含有一些很难去除的杂质。这些杂质中可能含有的PCR抑制剂极有可能会造成检测失败或出现假阴性的结果,影响最终判定[7,18]。这就对耐受杂质、耐受PCR抑制剂的PCR反应体系提出了需求。普通DNA聚合酶对抑制剂耐受性较差,已有报道的和市售的耐抑制的酶有Klentaq、Phanta、Phusion等DNA聚合酶[19]。Klentaq虽有报道对血液具有一定耐受性,但其扩增性能较弱,灵敏度较低[20];Phanta DNA聚合酶对植物、血液等具有一定耐受能力,并具有高保真性,且保真度优于Phusion,因此,本研究中选用Phanta DNA聚合酶作为备选酶,旨在现有缓冲体系(Phanta Super-Fidelity DNA Polymerase配套的5× SF Buffer)的基础上,调整添加剂种类和浓度、Mg2+浓度、聚合酶量来进一步优化反应体系,使聚合酶扩增性能进一步提升,得以对植物粗品、血液和食品粗提液进行直接扩增。
2.1 添加剂种类和浓度对反应体系的影响
添加剂种类和浓度对PCR反应有重要影响,一般来说,添加剂的作用主要表现在保护酶、促进解链和退火等几个方面[20-21]。对于不同的聚合酶,所需的最适添加剂种类和浓度均不同,使用相同添加剂对不同聚合酶催化的PCR反应表现出的效果也不尽相同。笔者选取了甘油、DMSO和DTT 3种常见添加剂来优化Phanta DNA聚合酶的扩增性能,结果见图1。由图1可以看出,甘油和DTT对于Phanta DNA聚合酶的扩增性能影响不大,但DMSO可以有效提升Phanta DNA聚合酶的扩增性能,并且在质量分数为5%时效果最佳。

图1 添加剂种类和浓度优化结果Fig.1 Optimization of the additives and their concentrations
2.2 Mg2+浓度对反应体系的影响
Mg2+是DNA聚合酶作用必不可少的金属离子,可有效释放聚合酶活性,但浓度过高也会抑制酶活[14,22]。在PCR反应中,Mg2+还会与dNTP结合,影响PCR反应中游离Mg2+浓度。在不同的PCR反应中,对于Mg2+有不同浓度需求。因此,笔者在添加剂优化基础上,在含有5% DMSO的反应体系中加入不同浓度Mg2+,来寻找Phanta DNA聚合酶的最适Mg2+浓度,结果见图2。由图2可知,Mg2+浓度在3 mmol/L时,Phanta DNA聚合酶具有最高的活性。
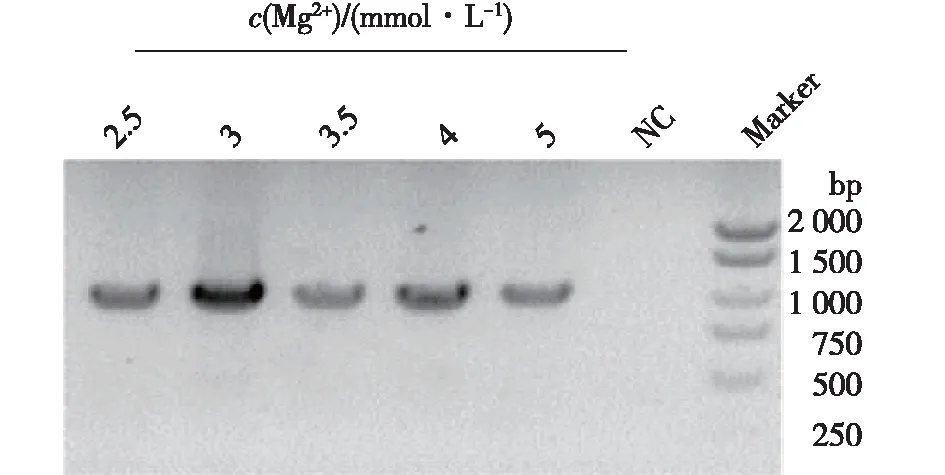
图2 Mg2+浓度优化结果Fig.2 Optimization of magnesium ion concentration
2.3 酶量对反应体系的影响
在以上优化添加剂种类、浓度和Mg2+浓度的基础上,笔者对聚合酶使用量也进行了优化。聚合酶在反应体系内使用量在一定范围内可达最优,酶量过低反应效率会降低,酶量过高则会可能引起非特异性扩增或抑制反应进行。在2.2中优化的反应体系中分别加入不同量的聚合酶,结果如图3所示。由图3可知:50 μL体系中,使用1 U的Phanta DNA聚合酶即可达到最优效果,酶量过低扩增效率过低,酶量过高会引起非特异性扩增。
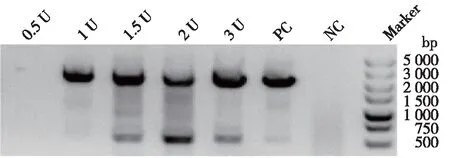
图3 酶量优化结果Fig.3 Optimization of polymerase amount
2.4 优化反应体系有利于提高扩增灵敏度
根据以上实验结果,笔者确定了在5× SF Buffer基础上改进的Phanta DNA聚合酶的优化反应条件,即在5×SF Buffer基础上加入5% DMSO、3 mmol/L Mg2+以及50 μL体系中使用1 U的Phanta DNA聚合酶。在此优化体系的基础上,考察Phanta DNA聚合酶的灵敏度是否得到提升。通过设立模板浓度梯度,考察了优化的反应体系对于反应灵敏度的影响,结果见图4。由图4可知:Taq DNA聚合酶可对1 ng以上基因组模板进行有效扩增,对于0.5 ng基因组扩增效果明显降低。对于未进行反应体系优化的Phanta DNA聚合酶,可对0.5 ng基因组进行有效扩增,而使用优化的反应体系,Phanta DNA聚合酶可对低至0.05 ng基因组进行有效扩增,灵敏度相比普通Taq DNA聚合酶提高10倍。Phanta DNA聚合酶相比于Taq DNA聚合酶,可以更好地结合双链DNA,因此具有较高的灵敏度。

图4 优化的反应条件提高Phanta DNA聚合酶的灵敏度Fig.4 Improved sensitivity of Phanta DNA polymerase with optimized reaction conditions
2.5 优化反应体系有助于提高对植物粗品的耐受度
植物育种过程需要大量鉴定基因型,提取大量植物DNA过程极为繁琐复杂[23-24],并且植物中含有多糖多酚、蛋白等大量PCR抑制剂[7],粗品直接进行扩增较为困难。通过在反应体系中加入不同体积的植物粗品作为模板,考察了加入5% DMSO、3 mmol/L Mg2+的优化反应体系中Phanta DNA聚合酶对于植物粗品的耐受程度,各实验均为50μL体系,酶量为1 U,结果见图5。由图5可知:Taq DNA聚合酶对于部分植物如水稻叶片裂解液可以扩增,但产量不高;但对于小麦和玉米等植物叶片裂解液则无法扩增。未优化条件的Phanta DNA聚合酶对于3种植物裂解液均可实现扩增,但扩增效果弱于直接使用DNA进行扩增的对照。经过添加剂、Mg2+以及酶量优化的反应体系中,Phanta DNA聚合酶对3种植物叶片裂解液均可实现有效扩增,扩增效果显著优于未经优化的体系。
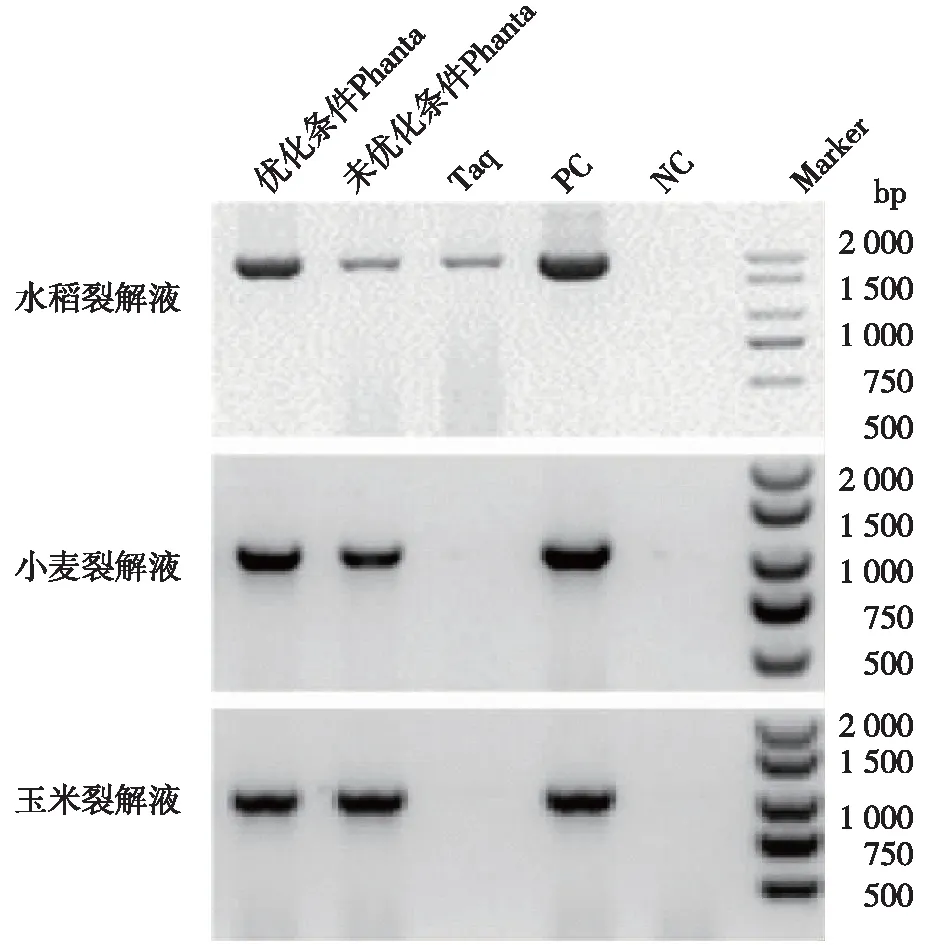
图5 植物叶片裂解液直扩结果Fig.5 Electrophoresis of direct PCR with leaf lysis products
2.6 优化反应体系有助于提高对全血的耐受度
全血中含有多种细胞和分泌因子,便于进行有效快捷的医疗诊断[25-27]。同时也由于其中含有血红素、血红蛋白等成分,会抑制PCR反应,对于分子诊断造成一定挑战[19]。通过设置反应中全血使用体积,考察了2.4中确立的优化反应体系中Phanta DNA聚合酶对于全血的耐受程度,结果见图6。由图6可知:Taq DNA聚合酶对于全血几乎无耐受,少量全血即可抑制PCR反应;Phanta DNA聚合酶对全血的耐受显著优于Taq DNA聚合酶,在体系中含有30%全血时,仍可实现扩增;优化反应体系后的Phanta DNA聚合酶在50 μL反应体系中可耐受的全血体积为20 μL,达到总反应体积的40%,相比未优化反应体系时,扩增产量和耐受血液体积显著提高。
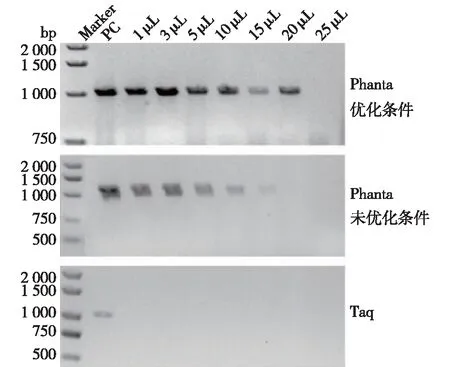
图6 优化反应条件有助于提高Phanta DNA 聚合酶对全血的耐受Fig.6 Improved food crude extracts tolerance of Phanta DNA polymerase with optimized reaction conditions
2.7 优化反应体系有助于提高对食物粗品的耐受度
食品安全关系重大,食品中有毒有害微生物的检测是食品安全检测的重要项目[27]。食品中微生物具有含量较低、成分复杂等特点,如使用PCR法进行检测,还需对食品中DNA进行提取[28]。如果能使用粗品进行直接PCR,将大大缩短食品检测的时间。在玉米裂解液中加入少量含pEGFP质粒的大肠杆菌[29],通过设置反应体系中加入的裂解液体积,考察了同2.4中确立的优化反应体系中Phanta DNA聚合酶对于食品粗品的耐受程度,结果见图7。由图7可知:Taq DNA聚合酶对于食品粗品在超过4μL粗品后即无法扩增;而优化反应体系后的Phanta DNA聚合酶在50 μL反应体系中可对加入的多达8μL的粗品耐受,并可有效扩增其中的微量pEGFP质粒。
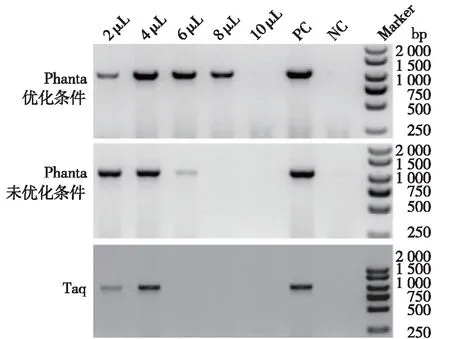
图7 食品粗提液直扩结果Fig.7 Electrophoresis of direct PCR with food crude extracts
3 结论
通过使用改良的Phanta DNA聚合酶,并对其反应体系进行优化,发现在SF Buffer中加入5% DMSO、3 mmol/L Mg2+以及50 μL体系中使用1 U聚合酶情况下,Phanta DNA聚合酶具有更优的扩增性能。在此优化条件下,Phanta DNA聚合酶的灵敏度相比Taq DNA聚合酶提高了10倍,并提高了对植物粗品、全血和食品粗品的耐受能力,可对粗品以及粗品中含有的微量微生物进行有效扩增,为植物直扩、分子诊断、食品快检等实际应用提供了原料和技术基础。
参考文献:
[1]EELES R A,WARREN W,STAMPS A.The PCR revolution[J].Eur J Cancer,1992,28(1):289-293.
[2]EBERLE J.Polymerase chain reaction (PCR)[J].Med Monatsschr Pharm,1995,18(6):152-158.
[3]KHOT P D,FREDRICKS D N.PCR-based diagnosis of human fungal infections[J].Expert Rev Anti Infect Ther,2009,7(10):1201-1221.
[4]ROMSOS E L,VALLONE P M.Rapid PCR of STR markers:applications to human identification[J].Forensic Sci Int Genet,2015,18:90-99.
[5]ZHANG K,LIN G,LI J.Quantitative nucleic acid amplification by digital PCRby clinical viral diagnostics[J].Clin Chem Lab Med,2016,54(9):1427-1433.
[6]马小军.PCR技术及其应用[J].植物杂志,1996 (5):30-31.
[7]SCHRADER C,SCHIELKE A,ELLERBROEK L,et al.PCR inhibitors:occurrence,properties and removal[J].J Appl Microbiol,2012,113(5):1014-1026.
[8]OPEL K L,CHUNG D,MCCORD B R.A study of PCR inhibition mechanisms using real time PCR[J].J Forensic Sci,2010,55(1):25-33.
[9]王梁燕,洪奇华,张耀洲.实时定量PCR技术及其应用[J].中国细胞生物学学报,2004,26(1):62-67.
[10]HUE-ROYE K,VEGE S.Principles of PCR-based assays[J].Immunohematology,2008,24(4):170-175.
[11]HUBÉ F,REVERDIAU P,IOCHMANN S,et al.Improved PCR method for amplification of GC-rich DNA sequences[J].Mol Biotechnol,2005,31(1):81-84.
[12]KANG J,LEE M S,GORENSTEIN D G.The enhancement of PCR amplification of a random sequence DNA library by DMSO and betaine:application to in vitro combinatorial selection of aptamers[J].J Biochem Biophys Methods,2005,64(2):147-151.
[13]GHADESSY F J,ONG J L,HOLLIGER P.Directed evolution of polymerase function by compartmentalized self-replication[J].Proc Natl Acad Sci USA,2001,98(8):4552-4557.
[14]WILLIAMS J F.Optimization strategies for the polymerase chain reaction[J].Biotechniques,1989,7(7):762-769.
[15]WADA M,KLEIN C,SCHELL J,et al.A functional assay for Taq DNA polymerase in PCR[J].Biotechniques,1994,16(1):26-28.
[16]PICARD V,ERSDAL-BADJU E,LU A,et al.A rapid and efficient one-tube PCR-based mutagenesis technique using Pfu DNA polymerase[J].Nucleic Acids Res,1994,22(13):2587-2591.
[17]LIU E P,WANG Y,HE X H,et al.Whole blood PCR amplification withPfuDNA polymerase and its application in single-nucleotide polymorphism analysis[J].Genet Test Mol Biomarkers,2015,19(11):610-616.
[18]SIDSTEDT M,HEDMAN J,ROMSOS E L,et al.Inhibition mechanisms of hemoglobin,immunoglobulin G,and whole blood in digital and real-time PCR[J].Anal Bioanal Chem,2018,doi:10.1007/s00216-018-0931-z.
[19]KERMEKCHIEV M B,KIRILOVA L I,VAIL E E,et al.Mutants of Taq DNA polymerase resistant to PCR inhibitors allow DNA amplification from whole blood and crude soil samples[J].Nucleic Acids Res,2009,37(5):e40.
[20]RALSER M,QUERFURTH R,WARNATZ H J,et al.An efficient and economic enhancer mix for PCR[J].Biochem Biophys Res Commun,2006,347(3):747-751.
[21]李红.通过优化PCR反应条件扩增富含GC的DNA片段[J].苏州大学学报(医学版),1999,19(7):788-789.
[22]HARRIS S,JONES D B.Optimisation of the polymerase chain reaction[J].Br J Biomed Sci,1997,54(3):166-173.
[23]HAYASHI K,YOSHIDA H,ASHIKAWA I.Development of PCR-based allele-specific and InDel marker sets for nine rice blast resistance genes[J].Theor Appl Genet,2006,113(2):251-260.
[24]MA J,GUAN S C,ZHANG Z,et al.Single- and double-SSR primer combined analyses in rice[J].Genet Mol Res,2012,11(2):1032-1038.
[26]胡兆平,姚萍,王保龙,等.荧光定量PCR技术在血液筛查中的应用及可行性分析[J].临床输血与检验,2002,4(1):7-9.
[27]WU V C.A review of microbial injury and recovery methods in food[J].Food Microbiol,2008,25(6):735-744.
[28]何小维,刘玉.PCR技术在食品检测中的应用[J].食品研究与开发,2006,27(5):107-109.
[29]郝江燕,胡文忠,冯叙桥,等.食品中大肠杆菌生物检测方法的研究进展[J].食品工业科技,2013,34(15):370-375.
