诺丽Actin基因片段克隆及实时荧光定量PCR方法的建立
2018-03-19蓝增全王华芳
吴 田,蓝增全,王华芳
(1.西南林业大学 园林学院,云南 昆明 650224;2.西南林业大学 环境科学与工程学院,云南 昆明 650224;3. 北京林业大学 生物科学与技术学院,北京 100083)
植物肌动蛋白(Actin)是植物中普遍存在的重要蛋白质,参与了细胞分裂、细胞形成、胞内信号传导、胞内物质运输、程序化死亡等重要生理功能[1-5]。研究者已经在许多高等植物中克隆得到了Actin基因,如茶树[6]、向日葵[7]、花生[8]、碱蓬[9]、木薯[10]、芍药[11]、海州香薷[12]、山药[13]等。随着一系列高等植物Actin基因的克隆,发现这些基因高度相似,氨基酸序列的同源性较高,一般在70%以上[14],具有组成型表达、易于扩增等优点,因此可以作为重要的内参基因用于定量、半定量PCR研究中,以比较不同来源的目的基因表达量的差异[15]。
诺丽Morinda citrifolia Linn.属茜草科,在国外主要分布于美国的夏威夷、澳大利亚、印度尼西亚、印度的热带、亚热带等地区,在我国可以生长于西双版纳、海南及台湾南部等地区[16]。诺丽被原产地的人们用作食材和药物已经有2000多年的时间[17],诺丽的叶可以止疼消炎[18],果实的营养丰富,含有丰富的维生素A、维生素C、烟酸、钾、锰、硒以及其他有效成分[19],具有较高的保健价值。近几年,国内外对诺丽的研究多专注于其药用成分、临床试验等,笔者没有检索到关于诺丽基因表达方面的研究,在一定程度上不利于诺丽应用机理的阐述。目前,在GenBank中尚未有诺丽Actin基因的报道,因此,获取诺丽Actin基因,分析其表达特性,以确定其是否能够作为诺丽内参基因使用,对后续诺丽功能基因的研究具有重要意义。本研究拟根据其他植物Actin基因的保守序列设计一对简并性引物,以诺丽果实总RNA反转录成cDNA后作为模板,进行诺丽Actin基因克隆和序列比对分析,同时结合实时荧光定 量 PCR(real-time fluorescence quantitative PCR,qRT-PCR)技术研究其在诺丽叶片、茎段、花以及不同发育时期的果实中的表达,旨在为诺丽内参基因的确定、诺丽生长发育中肌动蛋白的作用、其他基因在诺丽中的表达和调控奠定基础。
1 材料与方法
1.1 实验材料
将成熟度约80%的诺丽果实采收后,25 ℃下贮藏2 d后作为基因克隆的植物材料。用于qRT-PCR的植物材料分别为诺丽不同熟期的果实、诺丽叶片、诺丽全花、诺丽茎段等(表1)。诺丽样品采自于云南省玉溪市元江县诺丽种植基地,于保鲜盒中带回实验室,除1号样品外,其他样品用液氮速冻,暂存于-70 ℃冰箱待用;1号样品于25 ℃下贮藏2 d后用液氮速冻,暂存于-70 ℃冰箱待用。大肠杆菌DH5α菌株由本实验保存。
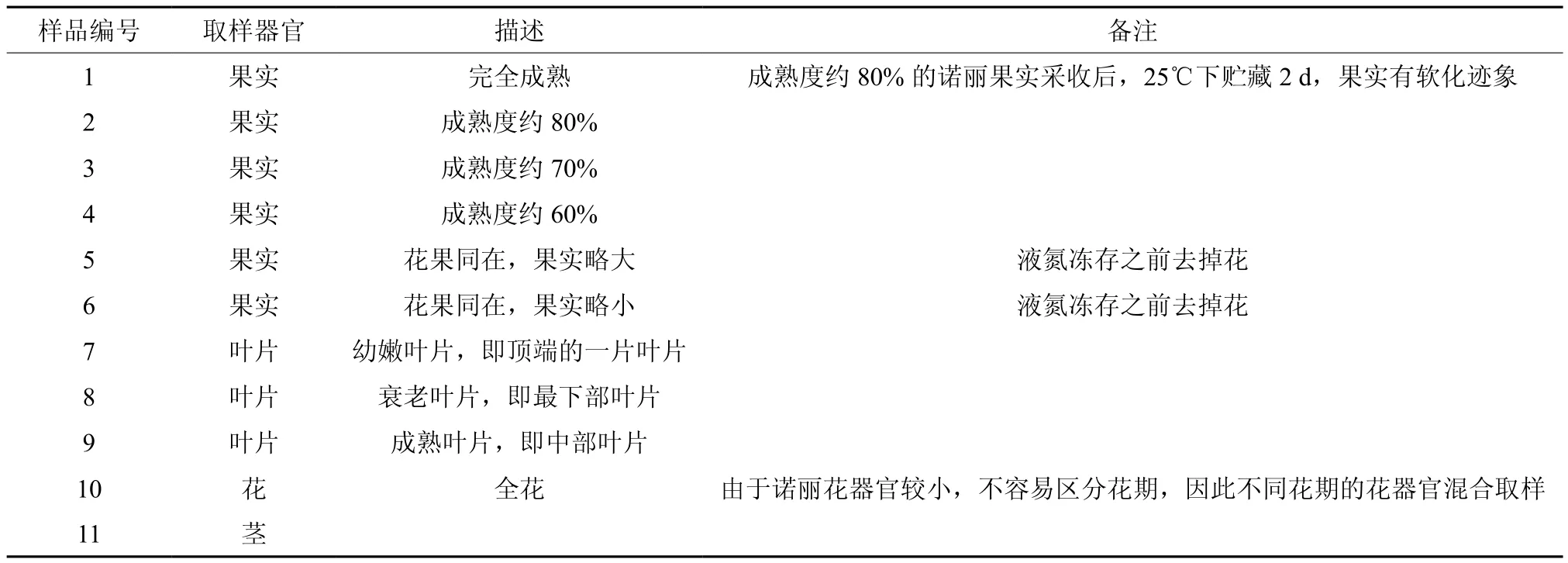
表 1 用于本研究的诺丽样品Table 1 Noni samples used in the research
1.2 主要试剂
柱式Trizol总RNA提取试剂盒(生工B511321)、M-MuLV First Strand cDNA Synthesis Kit M-MuLV第一链cDNA合成试剂盒(生工B532435)、第一链cDNA合成试剂盒(RevertAid Premium Reverse Transcriptase)、LA Taq(TaKaRa DRR02AG)、2×GC Buffer I、Marker(BBI B600032)、柱式DNA胶回收试剂盒(生工B518131)、定量PCR试剂SG Fast qPCR Master Mix(Roche 罗氏)、溴化乙锭、琼脂糖等均购自于上海生工。
1.3 实验方法
1.3.1 总RNA 的提取与反转录
总RNA的提取按照柱式Trizol总RNA提取试剂盒说明书进行。总RNA提取后通过电泳检测其完整性,通过吸光值法检测其浓度。cDNA第一链的合成按照试剂盒说明书进行。
1.3.2 引物的设计与合成
通 过 对 甜 橙(LOC102622338)、 玉 兰(AF281323)、 白 桦(EU588981)、 桑 树(DQ785808)、白杨(EF418792)等几种植物Actin基因的核苷酸序列进行同源性比较,找出高度保守的区段,根据同源性高和简并性低的原则,利用Primer Premier 5.0软件设计一对简并引物P1:5’-GGAACTGGAATGGTSAAGGCT-3’、P2:5’-CCACATCTGYTGGAAGGTGCT-3’, 其中:S=G/C,Y=C/T,用于扩增诺丽Actin 基因片段,预测目的片段的长度为400 bp。根据上述扩增的片段设计一对用于qRT-PCR的引物,上游引物P3:5’-TGTATGGCAACATCGTTCTCAGT-3’,下游引物P4:5’-CCACCTTAATCTTCATGCTGCT-3’,预计扩增长度111 bp。引物由上海生工合成。
1.3.3 诺丽Actin基因片段的扩增
PCR 反应体系为 25 µL,包括 ddH2O 6.3 µL,2×GC Buffer I 12.5 µL,简并引物 P1(10 µmol)、P2(10 µmol)各 0.5 µL,dNTPs(2.5 mM)4 µL,cDNA 1 µL,LA Taq 酶(5 U/µL)0.2 µL。PCR 反应条件为:95 ℃预变性3 min,94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸60 s,33个循环后72 ℃充分延伸7 min。PCR 扩增产物用1.5%琼脂糖凝胶、1×TAE电泳缓冲液进行检测,紫外透射光下观察并拍照。目的片段的回收和纯化按照试剂盒操作说明进行。
1.3.4 阳性克隆的筛选与鉴定
回收的PCR 产物连接到pMD-18T载体,热激法转化大肠杆菌DH5α感受态细胞,用 LB固体培养基(含有50 μg/mL氨苄青霉素)进行筛选,阳性克隆经菌落PCR 鉴定确认后,送至上海生工测序。
1.3.5 序列的生物信息学分析
序列的比较、翻译等在 DNAMAN 生物软件上进行,Blastx在NCBI网站上进行。
1.3.6 诺丽Actin基因的qRT-PCR
1.3.6.1 反应体系
采用SYBR Green染料法,在Light Cycler 480 Software Setup(Roche 罗氏)荧光定量 PCR 仪上进行扩增和数据分析。在本课题组进行前期qRT-PCR过程中确定的最佳扩增体系(表2)和反应条件进行实验,扩增条件为:95 ℃ 3 min充分变性后进入下述 48 个循环,95 ℃ 7 s,57 ℃ 10 s,75 ℃ 10 s。
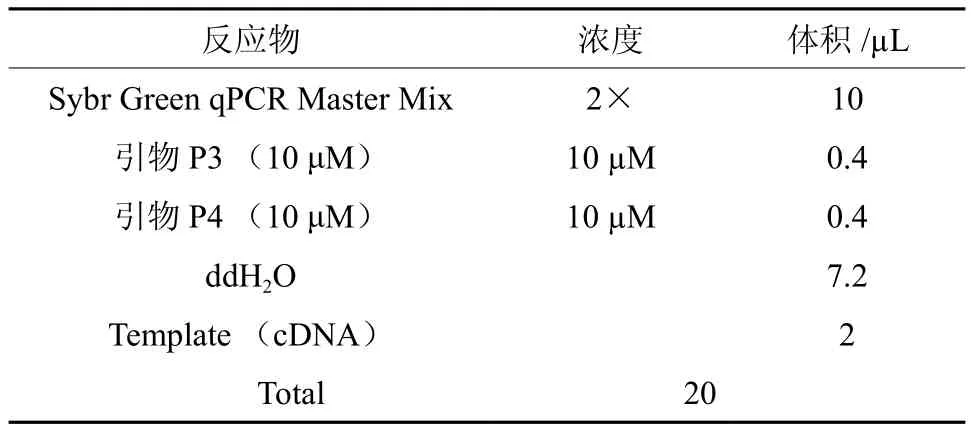
表 2 诺丽Actin基因的qRT-PCR 反应体系Table 2 The qRT-PCR reaction system of noni Actin
1.3.6.2 熔解曲线分析和标准曲线的建立
首先进行普通PCR反应,得到的PCR产物用1%琼脂糖凝胶电泳检测,以确定得到的产物是否为目的产物,再对qRT-PCR产物熔解峰进行分析。以其中一份阳性样品cDNA 进行1倍、10倍、100倍、1 000倍、10 000倍、100 000倍的梯度稀释作为模板,采用优化好的PCR反应条件进行qRTPCR,取6个点制作标准曲线。由方程E=(10-1/slope-1)×100%计算出引物的扩增效率,扩增效率在95%至110%即符合要求。
1.3.6.3 诺丽不同器官及不同发育时期果实的qRTPCR分析
将诺丽不同组织提取的RNA分别取200 ng进行反转录,然后进行qRT-PCR分析。
2 结果与分析
2.1 诺丽Actin基因片段的克隆
提取的总RNA经凝胶电泳检测,发现其完整性好、纯度高,可以用于诺丽Actin基因的克隆及qRT-PCR分析。以总 RNA反转录所得到的第一链cDNA为模板,P1、P2为引物进行 PCR 扩增。电泳检测发现,约在400 bp 处有一条PCR亮带,与预测片段大小一致(图1),推测是诺丽Actin基因片段。将该目的片段回收纯化,经PCR检测得到阳性克隆,片段大小约为400 bp,可以进行测序。

图1 诺丽Actin基因的RT-PCR的结果Fig. 1 RT-PCR of noni Actin
2.2 诺丽Actin基因的序列分析
测序得到一段393 bp的序列,编码131个氨基酸。将该片段在NCBI网站进行Blastx比较,结果表明其与actin 1(麦蓝菜,Vaccaria hispanica)有最高的一致性(identities)和相似性(positives),分别为96%和98%,与actin4(枇杷,Eriobotrya japonica)、actin 2(姜花,Hedychium coronarium)、actin( 鹅 掌 楸,Liriodendron tulipifera)、actin(枸杞,Lycium chinense)等均有较高的一致性和相似性(图2),进一步表明克隆得到的是诺丽Actin基因片段。

图2 诺丽Actin氨基酸序列与其他植物Actin氨基酸序列比对Fig.2 Homology comparison of Actin amino acid sequences between noni and other plants
2.3 诺丽Actin基因的定量分析
利用P3和P4引物进行普通 PCR 扩增,琼脂糖凝胶电泳结果显示扩增产物长度为111 bp,与预期长度一致,无引物二聚体和非特异条带(图3)。对荧光定量RT-PCR产物进行熔解峰分析(图4),诺丽Actin基因片段在Tm=82 ℃处显示特异性单峰,进一步说明扩增产物无引物二聚体及非特异性扩增,同时也说明引物设计的合理性和特异性,PCR反应条件得到了较好的优化。
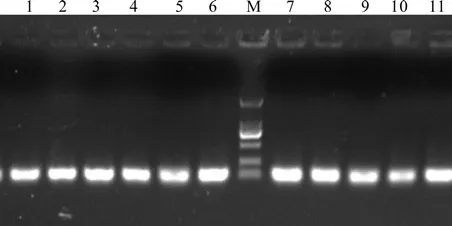
图3 引物P3和P4在诺丽cDNA中的扩增结果Fig.3 The ampli fi cation results of primers P3 and P4 in noni cDNA
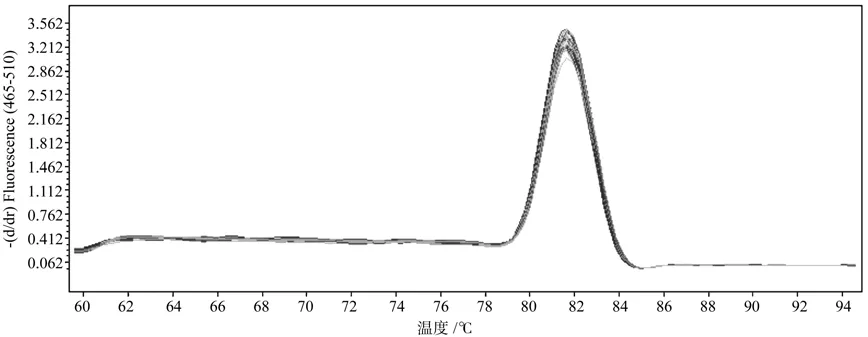
图4 诺丽Actin基因的熔解峰Fig. 4 Melting curve of noni Actin
2.4 诺丽Actin基因的qRT-PCR体系
以其中一份阳性 cDNA 样品梯度稀释后进行qRT-PCR 扩增,得到了扩增曲线(图5)和标准曲线(图6)。结果表明,以Ct值作为纵坐标(Y),以稀释倍数的对数为横坐标(X)建立的相对定量标准曲线,在所使用的浓度范围内具有良好的线性关系(R2=0.997 4),扩增效率为101.4%。

图5 诺丽Actin基因梯度稀释后的qRT-PCR扩增Fig.5 qRT-PCR Ampli fi cation of noni Actin after gradient dilution
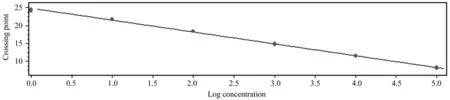
图6 诺丽Actin基因的标准曲线Fig.6 Standard curve of noni Actin
2.5 诺丽Actin基因在诺丽不同器官及不同发育时期果实中的表达分析
qRT-PCR的结果表明,诺丽Actin基因在不同熟期的诺丽果实(1-6号样品)中的Ct值分布在21.9~23.2,其中在完全成熟的诺丽果实中的Ct值最大,基因表达丰度最低;诺丽Actin基因在不同幼嫩程度的诺丽叶片(7-9号样品)中的Ct值分布在23.7~24.5,Ct值相近,说明该基因在不同幼嫩程度的诺丽叶片中表达量基本一致;诺丽Actin基因在全花和茎中的Ct值分别为21.2和21.7,表达量基本一致。由此可知,诺丽Actin基因在诺丽的各个组织、果实不同发育时期都有表达,因此推断诺丽Actin基因为组成型表达的肌动蛋白基因,适宜将其作为研究诺丽其他基因表达的内参基因。
3 讨 论
如果要进行基因表达量的研究,不论是半定量RT-PCR还是qRT-PCR,首先要找到相应的内参基因进行校正和标准化,主要是为了尽量避免不同样品在RNA质量、反转录效率和PCR反应条件上的差异[20-21]。在qRT-PCR分析中,可以有多种类型的看家基因作为内参基因,如甘油醛-3-磷酸脱氢酶基因(Glyceral-Dehyde-3-Phosphate Dehydrogenase,GAPDH)、 微 管 蛋 白 基 因(α-Tubulin)、肌动蛋白基因(Actin)、转录延伸因子基因(Elongation Factors 1 alpha,EF-lα)、泛素基因(Ubiquitin)、18S rRNA基因等[22-23]。但在大多植物的表达研究中,Actin基因家族是使用最频繁的看家基因,因为其在核苷酸和氨基酸水平均具有高度的保守性和同源性,且在各组织器官中表达恒定[24-25]。鉴于此,本研究首先考虑用诺丽Actin基因作为研究诺丽基因表达的内参基因。
本研究克隆了诺丽Actin基因,填补了诺丽内参基因研究领域的空白,丰富了高等植物Actin基因数据库,为进一步深入研究植物Actin的功能、保守结构域、变异性及其起源进化等提供了新的数据。其他研究者再进行诺丽基因表达研究时,可以直接使用P3、P4作为荧光定量PCR引物,也可以借鉴本研究测序得到的序列自行设计符合相应研究所需的qRT-PCR引物。本研究还考察了诺丽Actin基因的qRT-PCR表达模式,通常理想的内参基因除了能在各组织细胞及各种处理下稳定表达外,其表达丰度还不应过高,qRT-PCR过程中,内参基因的Ct值一般应在15~30之间,太大或太小都会降低定量的准确性[26],本研究在反转录RNA量为200 ng条件下,诺丽的Ct值均在21~24,表明表达丰度适宜,Actin基因适合做诺丽的内参基因。
然而,随着植物内参基因的研究深入,越来越多的研究表明,在同一植物中同一内参基因的稳定性在不同生理条件下通常并不恒定,且在不同物种中同源的内参基因也没有绝对的通用性,因此,在所有条件下都稳定表达的理想内参基因并不存在[27]。在qRT-PCR分析中,同时使用两个或多个内参基因有助于调整系统偏差,更有利于得到准确的基因表达结果[28]。因此,后续研究可以调取诺丽其他内参基因,如GAPDH基因、18S rRNA基因、α-tubulin基因、Ubipuitin基因等,同时利用BestKeeper、geNorm和NormFinder等内参基因分析软件,根据统计学方法进行筛选,为诺丽基因表达研究提供稳定的、可靠的和重复性高的最优内参基因。
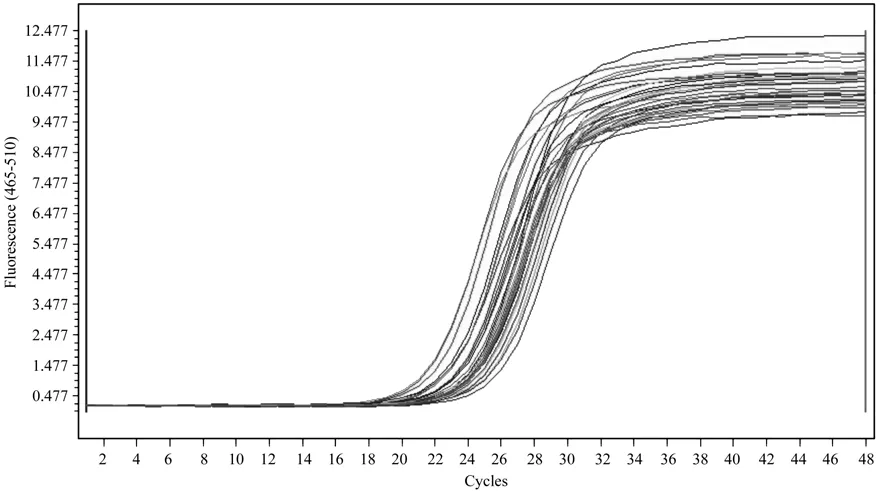
图7 诺丽Actin基因的扩增曲线Fig.7 Ampli fi cation curve of noni Actin
[1]贺淹才. 肌动蛋白和肌动蛋白基因的研究进展[J]. 生命的化学, 2002, 22(3): 248-250.
[2]陈 颖, 王 刚, 赵俊霞. 高等植物体内的肌动蛋白[J]. 生物学通报, 2003, 38(2): 13-15.
[3]王洪振, 程焉平. 细胞核内肌动蛋白参与基因转录的研究进展[J]. 吉林师范大学学报(自然科学版), 2005, 2: 34-36.
[4]Li J, Henty-Ridilla J L, Huang S, et al. Capping protein modulates the dynam is behavior of actin filamentsin response to phosphatidic acid in Arabidopsis [J]. Plant Cell, 2012, 24(9):3742-3754.
[5]Smertenko A, Franklin-Tong V E. Organisation and regulation of the cytoskeleton in plant programmed cell death [J]. Cell Death Differ., 2011, 18: 1263-1270.
[6]李远华, 陆建良, 范方媛, 等. 茶树根系Actin基因克隆及表达分析[J]. 茶叶科学, 2015, 35(4): 336-346.
[7]李园莉, 江元清. 向日葵肌动蛋白基因的cDNA克隆及进化分析[J]. 农业生物技术学报, 2002, 10(1): 67-71.
[8]杨丽霞, 李 玲. 花生肌动蛋白基因的克隆及序列分析[J]. 花生学报, 2007, 35(4): 6-9.
[9]马 清, 周向睿, 伍国强, 等. 盐生植物碱蓬Actin基因片段的克隆及序列分析[J]. 生物技术, 2009, 19(1): 1-3.
[10]许 娟, 罗兴录. 木薯Actin基因片段的克隆及序列分析[J].生物技术通报, 2011(6): 65-70.
[11]范丙友, 李 芳, 张文婷, 等. 芍药肌动蛋白基因的克隆及表达分析[J]. 中草药, 2013, 44(15): 2136-2142.
[12]蔡深文, 熊治廷, 刘 晨, 等. 海州香薷Actin基因片段克隆及表达分析[J]. 生物技术通报, 2015, 31(2): 111-115.
[13]龚明霞, 周芸伊, 王爱勤, 等. 山药Actin基因片段的克隆及表达分析[J]. 生物技术通报, 2016, 32(7): 73-80.
[14]Dominguez R, Holmes K C. Actin structure and function [J].Annu. Rev. Biophys., 2011, 40: 169-186.
[15]Daniel HM, Meyer W. Evaluation of ribosomal RNA and actin gene sequences for the identi fi cation of ascomycetous yeasts [J].Int. J. Food Microbiol., 2003, 86(12): 61-78.
[16]吴 田, 蓝增全. 诺丽(Morinda citrifolia)离体培养研究[J].山东农业大学学报(自然科学版), 2011, 42(2): 179-182.
[17]Chan-Blanco Y, Vaillant F, Perez A M, et al. The noni fruit(Morinda citrifolia L.) : A review of agricultural research,nutritional and therapeutic properties [J]. J. Food Compos. Anal.,2006, 19: 645-654.
[18]于纯淼, 李煦照, 于栋华, 等. 海巴戟果实诺丽(noni)生理功能研究进展[J]. 食品工业科技, 2011, 32(12) : 573-576.
[19]West B J, Deng S, Jen C J, et al. Nutrient and phytochemical analyses of processed noni puree [J]. Food Res. Int., 2011, 44(7):2295-2301.
[20]Brunner A M, Yakovlev I A, Strauss S H. Validating internal controls for quantitative plant gene expression studies[J]. BMC Plant Biol., 2004, 4(1): 14-20.
[21]Gutierrez L, Mauriat M, Guenin S, et al. The lack of a systematic validation of reference genes: A serious pitfall undervalued in reverse transcription-polymerase chain reaction (RT-PCR)analysis in plants [J]. Plant Bio. J., 2008, 6(6): 609-618.
[22]刘 圆, 王丽鸳, 韦 康, 等. 不同氮处理茶树实时定量PCR内参基因筛选和验证[J]. 茶叶科学, 2016, 36(1): 92-101.
[23]张玉芳, 赵丽娟, 曾幼玲. 基因表达研究中内参基因的选择与应用[J]. 植物生理学报, 2014, 50(8): 1119-1125.
[24]Wasteneys G O. The cytoskeleton and growth polarity [J]. Plant Biol., 2003, 3: 503-511.
[25]Liang W H, Tang C R, Wu N H. Cloning and characterization of a new actin gene from Oryza sativa [J]. Prog. Nat. Sci., 2004, 14:867-874.
[26]周晓馥, 王 晶, 史宏伟, 等. 18S rRNA作为植物实时荧光定量PCR内参基因的探究[J]. 吉林师范大学学报(自然科学版),2016, 37(2): 115-119.
[27]Kumar K, Muthamilarasan M, Prasad M. Reference genes for quantitative real-time PCR analysis in the model plant foxtail millet (Setaria italica L.) subjected to abiotic stress conditions[J].Plant Cell Tissue & Organ Culture, 2013, 115(1): 13-22.
[28]Luo H L, Luo K C, Luo L P, et al. Evaluation of candidate reference genes for gene expression studies in Cymbidium kanran[J]. Sci. Hortic., 2014, 167: 43-48.
