基于线粒体DNA D—loop序列分析刀鲚与短颌鲚的遗传多样性
2017-11-30张燕萍贺刚王生吴斌周辉明傅培峰方春林
张燕萍+贺刚+王生+吴斌+周辉明+傅培峰+方春林
摘要:以线粒体DNA D-loop 全序列为分子标记,研究4个刀鲚群体(长江口刀鲚、鄱阳湖湖口刀鲚、鄱阳湖湖区刀鲚、鄱阳湖湖口刀鲚幼鱼)及鄱阳湖短颌鲚群体的遗传多样性。结果显示:刀鲚的D-loop 序列长1 212~1 293 bp,短颌鲚的序列长 1 210~1 252 bp;在5个群体的 55尾个体中,共检测到变异位点72 个,其中单一多态位点50个,简约信息位点22个;共检测到 45 个不同的单倍型,单倍型多态性为 0.989,核苷酸多样性为 0.009 1,平均核苷酸差异数(K)为10.079。5个群体的遗传多样性丰富程度排序为鄱阳湖刀鲚幼鱼>鄱阳湖湖口刀鲚>鄱阳湖短颌鲚>鄱阳湖刀鲚>长江口刀鲚;不同单倍型间的遗传距离为 0.014~0.728,平均遗传距离为0.188。此外,系统发育树也表明,刀鲚、短颌鲚共同构成1个单系群,为2种生态型种群,尚未达到种或亚种的分化。
关键词:刀鲚;短颌鲚;线粒体DNA;D-loop 序列;遗传多样性;遗传结构差异
中图分类号: S917 文献标志码: A 文章编号:1002-1302(2017)20-0059-05
鲚属(Coilia)隶属于鲱形目(Cluperiformes)鳀科(Engraulidae),是分布于印度洋-太平洋地區、具有极高经济价值的中小型鱼类。世界上的鲚属鱼类有13种,其中中国分布有4种:七丝鲚(Coiiia.grayi)、凤鲚(Coiiia.mystus)、刀鲚(Coiiia. nasus)和短颌鲚(Coiiia.brachynathus),它们主要分布于中国沿海及长江中下游地区。刀鲚肉质细嫩鲜美,被列为“长江三鲜”之一,深受广大群众的喜爱,具有重要的经济价值,也是主要的捕捞对象之一[1]。短颌鲚属华中区特有种,是德国人Kreyenberg、Pappenheim于1908年依据洞庭湖的标本确立的一个物种。长期以来,由于其上颌骨较短与长江中下游及其附属大型湖泊等淡水性分布的特征,短颌鲚一直被视为有别于刀鲚的有效物种。但是随着人们在巢湖、太湖等通江湖泊发现刀鲚淡水定居性种群,短颌鲚与刀鲚在生态学上的差异变得模糊,两者的鉴别主要依据上颌骨长度等界限不甚清晰的形态特征,从而引发对短颌鲚物种有效性问题的讨论。鲚属鱼类外形十分相似,该属刀鲚和短颌鲚的分类问题一直存在较大争议。
短颌鲚在20世纪70年代也是鄱阳湖的主要捕捞对象。近些年来,由于过度捕捞和生态环境破坏等因素,自20世纪90年代起,其种群数量逐年下降。鄱阳湖鲚属鱼类在1983年产量约为1 100 t,1995年产量为1 385 t,1998年下降至550 t[2]。从鄱阳湖渔获物种群(组成)分析看,1974年鲚属鱼类占鄱阳湖渔获物质量的10%~15%[3],1984年占09%,2001年占1.5%。江西省水产科学研究所近10年对鄱阳湖调查表明,鲚属鱼类占鄱阳湖渔获物质量分数不足10%。目前在鄱阳湖鲚属鱼类鱼汛比20世纪70年代晚1~2个月,汛期也由20 d左右缩减为3~5 d,其资源状况不容乐观。由于每年鄱阳湖野生鲚属鱼类的资源量都有明显下降,其种群结构是否也在发生变化有待研究,因此对鲚属幼体的遗传多样性和物种判别研究十分必要。
线粒体DNA具有母系遗传、分子结构稳定、较快的进化速率等特点[4],成为研究物种起源、系统进化等的有效分子标记。线粒体DNA控制区序列,由于不编码蛋白质,比线粒体其他部分的基因具有更快的进化速率,因此广泛应用于亲缘关系近的物种或种群间的遗传结构多样性及种群历史等方面的研究。本研究通过对线粒体DNA控制区全序列进行分析,研究长江口、鄱阳湖刀鲚群体和短颌鲚群体在遗传结构方面的差异,以期为长江流域刀鲚和短颌鲚合理的保护、管理、利用提供科学依据。
1 材料与方法
1.1 材料
本研究所用的刀鲚样本于2014年4—11月采集于上海长江口(13个样本)、鄱阳湖湖口(10个样本)及鄱阳湖湖区(14个样本)3个不同的采样点的4个群体;鄱阳湖刀鲚幼鱼9尾采自全湖;短颌鲚样本来自鄱阳湖湖区。每尾鱼剪取少许肌肉分别放入Eppendorf管内,于95%乙醇中保存备用。
1.2 方法
1.2.1 DNA提取 取100 mg乙醇保存肌肉样品剪碎,分别放入1.5 mL Eppendorf管内,加入400 L SET缓冲液,再依次加入40 μL 10%十二烷基硫酸钠(SDS)、10 μL( 20 mg/mL)蛋白酶K,混合后在55℃水浴中消化过夜,再分别用平衡酚、三氯甲烷/异戊醇抽提除蛋白,最后用预冷的无水乙醇沉淀DNA,烘干后用TE溶解。所得基因组DNA样品用紫外分光光度计测量DNA样品的浓度、纯度,同时用琼脂糖凝胶电泳检测基因组DNA的完整性并估测分子量,将DNA原样稀释至 50 ng/μL,4℃保存备用。
1.2.2 PCR扩增及测序 使用2个通用引物DF1、DR2扩增D-loop区段基因,引物序列DF1: 5′-CTAACTCCCAAA GCTAGAATTCT-3′,DR2: 5′-ATCTTAGCATCTTCAGTG-3′[5]。扩增反应混合物总体积 50 μL,包括2.0 μL DNA模板、5 μL 10×reaction buffer、2 μL 2.5 mmol/L dNTP、各2 μL引物(浓度 10 mmol/L)、0.25μL Taq酶(5 U),用无菌ddH2O补至50 μL。PCR扩增程序:94 ℃ 4 min;94 ℃ 30 s,54 ℃ 45 s,72 ℃ 1 min,35个循环;72 ℃10 min。PCR产物用1.0%琼脂糖凝胶电泳检测,并拍照记录。目标扩增产物用试剂盒回收纯化后,由上海博尚生物有限公司进行双向测序,在本研究中,除用引物 DF1、DR2由2端向中部测序外,还根据所得序列再设计1对引物(F: 5′-GCGGATTCTCCGTAGACAAC-3′,R: 5′-GTAAAATTGTCTGGGTCTCC-3′)由序列中部向2端进行测序,以提高测序的准确性。endprint
1.3 数据分析
序列用Clustal W1.83软件[6]分析比对;利用DnaSP(version 4.0)软件[7]统计单倍型及变异位点(S),计算单倍型多样性(Hd)、核苷酸多样性(π)及平均核苷酸差异数(K)。采用MEGA(version 4.0)軟件[8]中的Kimura 双参数法计算各单倍型间的遗传距离;采用邻接(Neighbor-Joining,简称NJ)法,Bootstrap置信值估算重复1 000次,构建系统发育树。
利用Ariequin3.11软件[9]计算两两群体间的分化指数(Fst),进行分子变异分析(analysis of molecular variance,简称AMOVA)。利用DnaSP5.10软件[10]、Arlequin3.11软件分别进行核甘酸不配对分析和Fus Fs中性检验,来推断4个刀鲚群体、1个短颌鲚群体在进化过程中是否经历过种群扩张。
2 结果与分析
2.1 PCR扩增结果及序列特征分析
所有刀鲚、短颌鲚的D-loop片段均能被清晰稳定地扩增,PCR产物经琼脂糖凝胶电泳检测显示为1条清晰明亮的条带,大小为1 300 bp左右。双向测序后的序列经过拼接,获得1 210~1 293 bp的控制区全序列,其中长度为1 252 bp的序列最多,有41条,占74.505%。刀鲚的D-loop序列长 1 212~1 293 bp,短颌鲚的序列长1 210~1 252 bp,在刀鲚和短颌鲚控制区序列中发现了3~7个拷贝不等的38 bp的重复片段,此外还存在1~3 bp小片段的插入或缺失。
序列分析结果(表1)表明,在4个刀鲚群体、1个短颌鲚群体 D-loop区段基因片段中,A、T、G、C碱基占比分别为332%、33.2%、19.3%、14.3%。其中,A+T碱基组成比例为664%,大于C+G的组成比例33.6%。从表1可以看出,D-loop表现出很强的碱基组成偏向性,即在 A、C、G、T这4种碱基中,C的组成比例明显低于其他3种碱基。
2.2 群体遗传多样性和遗传差异性的分析
通过序列比对,在5个群体 55尾个体中,共检测到变异位点(S)72个(图1),其中单一多态位点50个,简约信息位点22个。本研究中共检测到45种不同的单倍型,每种单倍型对应的样本数为1~5个不等,长江口刀鲚、鄱阳湖湖口刀鲚、鄱阳湖刀鲚、鄱阳湖刀鲚幼鱼、鄱阳湖短颌鲚群体中单倍型的分布数量分别为13、9、13、9、7种。
由表2可以看出,在所有45种单倍型中,仅有Hap9、Hap14、Hap22、Hap35、Hap37、Hap43在2个及2个以上的个体中存在,其他39种单倍型仅在1个个体中存在,Hap9含有最多的5个个体。Hap9在长江口刀鲚、鄱阳湖湖口刀鲚、鄱阳湖刀鲚群体中同时出现,Hap14同时出现在长江口刀鲚、鄱阳湖刀鲚幼鱼群体中,Hap22同时出现在鄱阳湖湖口刀鲚和鄱阳湖刀鲚群体中,Hap35同时出现在长江口刀鲚和鄱阳湖湖口刀鲚群体中,Hap37同时出现在鄱阳湖刀鲚和鄱阳湖刀鲚幼鱼群体中。在45种单倍型中,长江口刀鲚群体独有单倍型有10个(Hap1、Hap3、Hap10、Hap20、Hap21、Hap23、Hap26、Hap27、Hap28、Hap30),鄱阳湖湖口刀鲚群体独有的有6个(Hap5、Hap6、Hap15、Hap16、Hap19、Hap 24),鄱阳湖刀鲚群体独有的有8个(Hap2、Hap4、Hap17、Hap18、Hap25、Hap29、Hap31、Hap33、Hap34),鄱阳湖刀鲚幼鱼独有单倍型7个(Hap7、Hap8、Hap11、Hap12、Hap13、Hap32、Hap36),鄱阳湖短颌鲚群体的单倍型均为独有的(Hap38~Hap45)。由此看出,各群体间拥有非常少的共享单倍型,独有的单倍型在群体内占有量非常丰富,而丰富的独有单倍型的存在说明长江口刀鲚、鄱阳湖湖口刀鲚、鄱阳湖刀鲚、鄱阳湖刀鲚幼鱼及鄱阳湖短颌鲚群体间存在一定程度的分化。
群体内的遗传多样性可以通过单倍型多样性、核苷酸多样性和平均核苷酸差异数等数据来体现。由表3可见,55个个体的单倍型多态性(H)为 0.989±0.007;核苷酸多样性(π)为0.009 1,平均核苷酸差异数(K)为10.079。根据单倍型多样性、核苷酸多样性和平均核苷酸差异数等数据的大小可以看出,在4个刀鲚群体和1个短颌鲚群体中,遗传多样性丰富程度排序为鄱阳湖刀鲚幼鱼>鄱阳湖湖口刀鲚>鄱阳湖短颌鲚>鄱阳湖刀鲚>长江口刀鲚。
经计算,各单倍型间的遗传距离在 0.014~0728,平均遗传距离为0.188。
2.3 群体间遗传分化与分子系统树
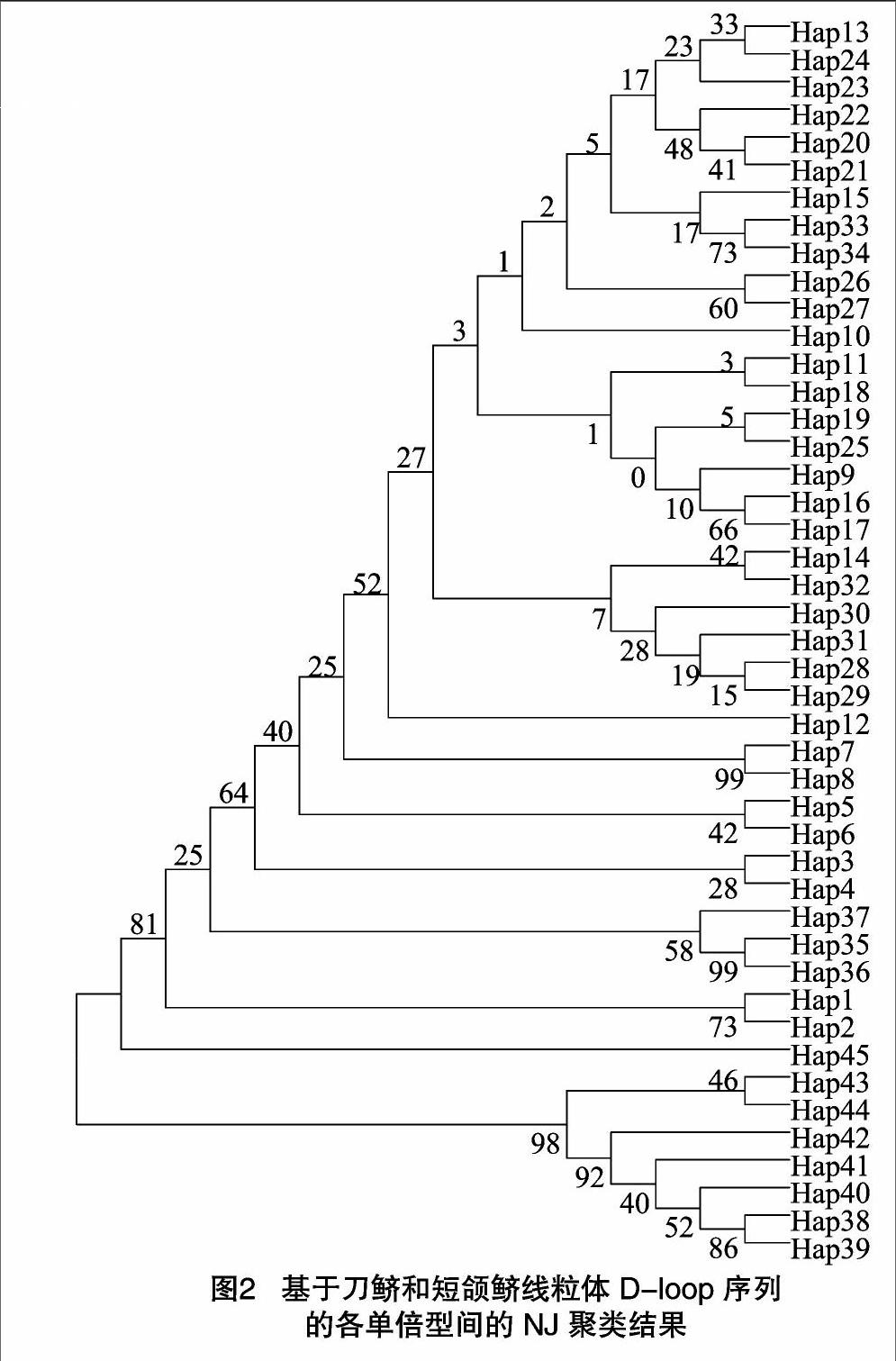
根据邻接法构建的单倍型间系统进化树显示:来源于鄱阳湖湖区短颌鲚样本的7种单倍型明显单独成为1系。与其他群体单倍型具有显著的分化(图2)。另外,采集自长江口刀鲚、鄱阳湖湖口刀鲚、鄱阳湖湖区刀鲚及鄱阳湖湖区刀鲚幼鱼样本的38种单倍型聚为1支。
从表4可看出,刀鲚群体之间的遗传分化指数较低,而短颌鲚群体(DH)与刀鲚群体之间存在相对较高的分化指数(0.039 9~0.052 34)。
对刀鲚、短颌鲚群体的AMOVA分析结果显示,将4个刀鲚群体分为1组(group 1:CD、HD、RD、DY),短颌鲚群体分为1组(group 2:DH)。在组间变异、组内群体间变异、群体内变异3个水平上进行分子变异方差分析,表5显示,6561%变异发生在群体内部。对不同种类分组上的AMOVA分析表明,群体间、组内变异分别占总变异的6071%、39.53%(P<0.001)。
2.4 种群历史动态
利用Ailequin 3.11软件对刀鲚、短颌鲚群体进行Tajimas D、Fu and Lis中性检验。表3结果表明:所有群体Tajimas D均为负值(-1.541 59~-0.317 70),但未达到显著水平。Fu and Lis在所有种群中均为负值(-1.654 44~-0.021 03),未达到显著水平。Tajimas D、Fu and Lis检验结果均表明,历史上刀鲚、短颌鲚未经历种群扩张。endprint
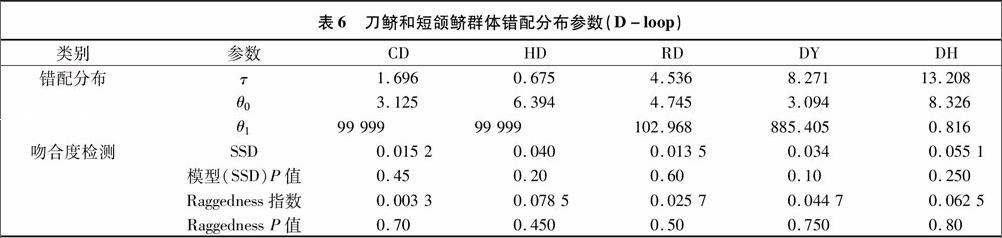
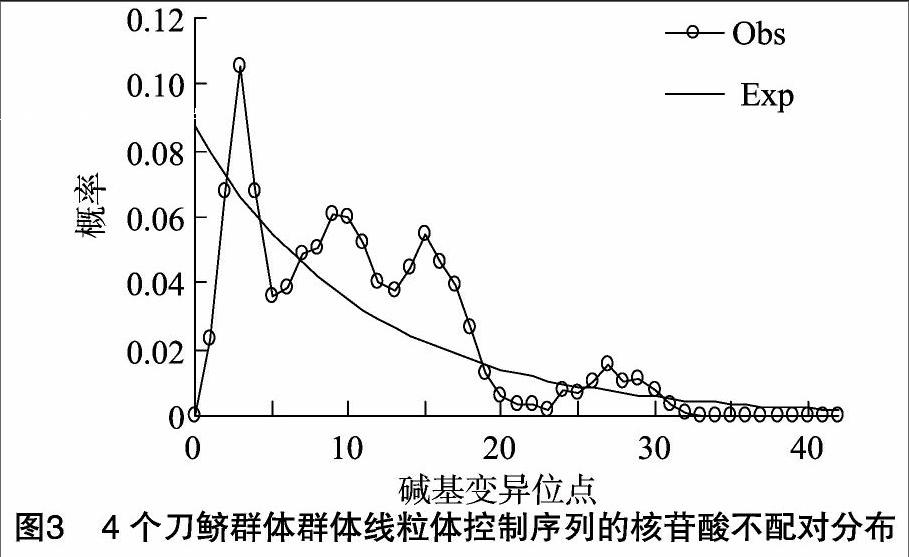
对刀鲚4个群体D-loop序列的错配分析发现,碱基歧点分布的期望值呈典型的多峰分布,说明长江流域刀鲚种群历史上未经历种群扩张(图3)。4个群体的Raggedness统计量(Rg)为0.45~0.80,均未达到显著水平,进一步支持刀鱭和短颌鲚种群未经历过种群扩张,观测分布和期望分布之间的偏离总方差(SSD)在4个刀鲚群体和1个短颌鲚群体中均未达到显著水平(表6),提示刀鲚和短颌鲚种群未经历过扩张时期。
3 讨论
3.1 刀鲚和短颌鲚种群控制区序列的碱基组成
所测定的46尾刀鲚mtDNA D-loop区序列中,碱基A、T、G、C的平均含量分别为 33.2%、33.2%、19.3%、14.3%;9尾短颌鲚mtDNA D-loop区序列的碱基A、T、G、C的平均含量分别为33.1%、33.1%、19.5%、14.2%;从刀鲚、短颌鲚碱基组成可以看出,D-loop区段表现出很强的碱基组成偏向性,即在A、T、G、C4种碱基中,C的含量明显低于其他3 种碱基的含量;此外,刀鲚和短颌鲚mtDNA D-loop区中A+T碱基的含量(66.4%、66.2%)高于G+C碱基含量(33.6%、338%),符合脊椎动物mtDNA D-loop区碱基组成的特点[11]。
线粒体控制区(D-loop)为非编码区,受进化压力较小,在D-loop处可见较高的突变积累而形成多态性[12]。核苷酸替换和长度变异在该处很常见[13-15]。在本研究中,发现刀鲚和短颌鲚控制区序列中存在38 bp的重复片段,拷贝数从 3~7个不等,此外还存在1~3 bp小片段的插入或缺失。
3.2 遗传变异及遗传多样性
本试验中测定的刀鲚、短颌鲚5个群体的D-loop序列中共检测出72个变异位点,占全部序列的5.75%,共定义了45个单倍型,显示出较为丰富的遗传多样性,与程起群等的研究结果一致[16-18]。从D-loop区序列变异得到的NJ系统进化树可以看出,来源于鄱阳湖的短颌鲚形成1个单系,显示与其他群体单倍型具有一定的分化,另外,采自长江口,鄱阳湖湖口和鄱阳湖湖区样本的38个单倍型聚为1支,刀鲚群体没有明显的地理分化格局。
群体间的遗传分化指数用来表示群体间的遗传分化程度,Fst值越大,表明2个群体间的分化程度越高;反之,分化程度越低[19]。群体间的遗传分化指数、K2-P遗传距离以及AMOVA分子变异分析均表明,长江流域4个刀鲚群体间存在着广泛的基因交流,没有形成明显的遗传分化,仍是个随机交配的群体。鄱阳湖为通江湖泊,每年4—6月,长江流域刀鲚洄游至鄱阳湖产卵,由于不同地理种群的刀鲚可能会进入同一地点或相对较近的区域产卵和受精,因此增加了群体外
交配的可能性,同时,这种繁殖方式增加了长江流域刀鲚各群体之间的基因交流机会, 由此导致长江流域刀鲚群体之间的遗传分化程度相对较低,而群体内个体之间的遗传变异较大。短颌鲚与刀鲚存在一定的遗传分化,唐文乔等过对mtDNA的D-loop区进行分析指出,短颌鲚和湖鲚是刀鲚的淡水生态型种群,并非有效种[5]。许志强等综合分析刀鲚和短颌鲚的颌骨长度和线粒体Cytb序列,得出短颌鲚属于刀鲚的1个淡水生态型种群而并非独立物种的结论[20]。本研究通过对长江口刀鲚、鄱阳湖湖口刀鲚、鄱阳湖刀鲚、鄱阳湖刀鲚幼鱼以及鄱阳湖短颌鲚5个群体的遗传多样性及系统发育树研究也表明,鄱阳湖的刀鲚和短颌鲚共同构成1个单系群,为2种生态型种群,尚未达到种或亚种的分化。
参考文献:
[1]袁传宓,秦安黔,刘仁华,等. 关于长江中下游及东南沿海各省的鲚属鱼类种下分类的探讨[J]. 南京大学学报(自然科学版),1980(3):67-82.
[2]江西省农业局水产资源调查队,江西省水产科学研究所.鄱阳湖水产资源调查报告[R]. 1974.
[3]长江流域刀鲚资源调查协作组.长江流域刀鲚资源调查报告[R]. 1977.
[4]张敏莹,徐东坡,刘 凯,等. 长江下游刀鲚生物学及最大持续产量研究[J]. 长江流域资源与环境,2005,14(6):694-698.
[5]唐文乔,胡雪莲,杨金权. 从线粒体控制区全序列变异看短颌鲚和湖鲚的物种有效性[J]. 生物多样性,2007,15(3):224-231.
[6]Thompson J D,Gibson T J,Plewniak F,et al. The CLUSTAL_X Windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research,1997,25(24):4876-4882.
[7]Rozas J,Sánchez-Delbarrio J C,Messeguer X,et al. DnaSP,DNA polymorphism analyses by the coalescent and other methods[J]. Bioinformatics,2003,19(18):2496-2497.
[8]Tamura K,Dudley J,Nei M and Kumar S. MEGA4:Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0[J]. Mol Biol Evol,2007,24(8):1596-1599.
[9]Excoffier L,Laval G,Schneider S. Arlequin (version 3.0):an integrated software package for population genetics data analysis[J]. Evolutionary Bioinformatics Online,2005,1(4A):47-50.endprint
[10]Librado P,Rozas J. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics,2009,25(11):1451-1452.
[11]Broughton R E,Milam J E,Roe B A. The complete sequence of the zebrafish (Danio rerio) mitochondrial genome and evolutionary patterns in vertebrate mitochondrial DNA[J]. Genome Research,2001,11(11):1958-1967.
[12]谢振宇,杜继曾,陈学群,等. 线粒体控制区在鱼类种内遗传分化中的意义[J]. 遗传,2006,28(3):362-368.
[13]Harrison R G. Animal mitochondrial DNA as a genetic marker in population and evolutionary biology[J]. Trends in Ecology & Evolution,1989,4(1):6-11.
[14]Billington N,Hebert P D N. Mitochondrail DNA diversity in fishes and its implications for introduetions[J]. Can J Fish Aquat Sci,1991,48(S1):80-94.
[15]Rand D M. Endotherms,ectotherms,and mitochondrial genome-size variation[J]. Journal of Molecular Evolution,1993,37(3):281-295.
[16]程起群,溫俊娥,王云龙,等. 刀鲚与湖鲚线粒体细胞色素b基因片段多态性及遗传关系[J]. 湖泊科学,2006,18(4):425-430.
[17]葛家春,曹 廷,陈婵娟,等. 利用扩增片断长度多态性技术分析长江刀鲚的遗传多样性[J]. 南京大学学报(自然科学),2008,14(3):332-338.
[18]马春艳,刘 敏,马凌波,等. 长江口刀鲚遗传多样性的随机扩增多态DNA(RAPD)分析[J]. 海洋水产研究,2004,25(5):19-24.
[19]杨金权,胡雪莲,唐文乔. 长江及其南部邻近水域刀鲚的种群遗传结构及种群历史[J]. 上海水产大学学报,2008,17(5):513-519.
[20]许志强,葛家春,黄 成,等. 基于颌骨长度和线粒体Cytb序列变异探讨短颌鲚的分类地位[J]. 大连水产学院学报,2009,24(3):242-246.endprint
