H2AFZ、MLF1IP和NEK2在乳腺癌不同亚型中的表达差异与预后相关性分析
2017-10-13孙天一寿成超北京大学临床肿瘤学院生化与分子生物学研究室消化肿瘤内科北京004
孙天一,章 程,,寿成超(北京大学临床肿瘤学院:生化与分子生物学研究室,消化肿瘤内科,北京004)
·论著·
H2AFZ、MLF1IP和NEK2在乳腺癌不同亚型中的表达差异与预后相关性分析
孙天一1,章 程1,2,寿成超1(北京大学临床肿瘤学院:1生化与分子生物学研究室,2消化肿瘤内科,北京100142)
目的:寻找与乳腺癌分型和预后相关的全新分子标志物,为乳腺癌的诊疗提供依据.方法:本研究借助生物信息学手段和统计学方法对多个来自肿瘤公共数据库的大样本数据进行了深度分析.结果:H2AFZ、MLF1IP和NEK2为乳腺癌不利预后因子,三者皆与乳腺癌患者ER/PR/HER2表达水平具有显著的相关性.H2AFZ、MLF1IP和NEK2的mRNA水平在ER和PR阳性乳腺癌中低表达,而在HER2阳性乳腺癌中高表达.此外,H2AFZ、MLF1IP和NEK2在乳腺癌Luminal A亚型中的表达水平较低,而且与预后负相关,而在其他亚型中水平较高,与预后相关性不大.结论:作为不利预后因子,H2AFZ、MLF1IP和NEK2在乳腺癌患者不同分型人群中的表达差异可能是造成乳腺癌各分型恶性程度和预后差异的原因之一.
乳腺癌;大样本数据分析;分子分型;预后相关基因
0 引言
乳腺癌是全球范围内女性最常见、死亡人数最多的肿瘤之一[1].由于早期诊断以及有效治疗方法的发展,乳腺癌的发病率和死亡率在二十余年来逐步降低,患者的生存期也在逐渐延长[2].然而,乳腺癌仍是导致女性因患肿瘤死亡的主要原因,防治任务艰巨.
目前被广泛接受的乳腺癌治疗标准依托分子病理分型,根据雌激素表达情况、肿瘤大小、淋巴结节、年龄和肿瘤分期等临床和病理因素来判定患者能否适用激素治疗,或是采用化学疗法[2].乳腺癌早已不再被视为单一的疾病,而是一个多方面、复杂表征的赘生物和肿瘤细胞集合,由来自不同亚型的细胞群组成,呈现出多种多样的临床、病理和分子特征以及不同的预后预期,因此也需要据此选择适当的治疗方案[3].乳腺癌的一大特征是雌激素受体(estrogen receptor, ER)、孕激素受体(progesterone receptor,PR)和人表皮生长因子受体 2(human epidermal growth factor receptor 2,HER2)的表达.根据现行的临床标准,除与正常乳腺癌相似的正常乳腺样型(Normal⁃like)外,乳腺癌可被分为4种分子亚型:管腔A 型(Luminal A,ER+和/或 PR+、HER2-)、管腔B 型(Luminal B,ER+和/或 PR+、HER2+)、HER2 过表达型(ER-和/或 PR-、HER2+)和基底细胞样亚型(Basal⁃like,简称 Basal,ER-、PR-、HER2-)[3-4].雌激素受体和孕激素受体是乳腺癌最重要的预后指标之一,对辅助、新辅助治疗及晚期乳腺癌激素治疗的效果都有重要指示作用.雌激素受体阳性(ER+)的乳腺癌主要分为Luminal A和Luminal B型,其中Lu⁃minal A型较B型的ER相关基因表达较高,但是增殖性基因表达较低.
雌激素受体阴性(ER-)的乳腺癌主要有HER2+和Basal型,此两种亚型比Luminal A的恶性程度高,更可能进入3期(grade 3);而Luminal A较HER2+型和 Basal型有更好的预后,然而复发率较高[5-6].
近年来,随着以二代测序为代表的高通量生物学研究手段和生物信息学手段的进步,肿瘤相关领域的研究进展迅速,大量肿瘤标志物和潜在靶点基因进入人们的视野[7].虽然关于各类肿瘤最终的预后及预测分析的共识尚未达成,但是利用生物标志物对肿瘤进行预测和鉴定,并建立严格可重复的的鉴定体系和治疗方法这一概念已经初露峥嵘,并有望成为未来肿瘤防治的重要方向[6,8-9].本研究旨在通过对乳腺癌患者大样本数据的挖掘,寻找与ER/PR/HER2表达情况和乳腺癌分子分型相关的全新肿瘤标志物,为乳腺癌的防治提供新思路.
1 材料和方法
1.1 数据来源 本研究中采用的数据集皆来自于GEO (Gene Expression Omnibus, https://www.ncbi.nlm.nih.gov/geo/) 和 NKI (The Netherlands Cancer Institute, http://www.nki.nl) 公开数据库,数据的使用遵循相关数据库的流程和隐私保护规定.此外,本研究还使用了 Kmplotter分析工具(http://kmplot.com/analysis/)进行 Kaplan⁃Meier生存分析[10].
1.2 数据集的处理和目的基因的挑选 各个数据集中的患者根据免疫组化结果和临床分子病理分型结果进行区分,其基因表达谱数据和临床信息借助Excel软件整合匹配,并使用 SPSS17.0和 GraphPad Prism 5.0进行统计学分析和成图.对于每个基因,取其所有探针的均值作为该基因的表达值.根据目的基因的表达值,依照中位数原理将患者划分为高表达组(高于中位数)和低表达(低于中位数)组,随后对各组进行分析.
1.3 统计学处理 分组之间表达差异的统计采用student⁃t test, 由 SPSS17.0 完成.目的基因之间相关性的分析采用Pearson Correlation分析.某因素与预后关联的判断采用 Kaplan⁃Meier分析,匹配 Log⁃rank检验.对于所有的统计学处理,P<0.05被视为差异具有统计学意义.
2 结果
2.1 ER/PR/HER2表达差异及不同分型的乳腺癌预后比较 现有的诸多报道已经提示,乳腺癌中雌激素受体、孕激素受体和人表皮生长因子受体2的表达水平与患者预后及临床预期表征有着密切的联系.同时,不同乳腺癌亚型的预后水平具有显著的差异,其中HER2型和Basal型与其他亚型相比预后较差.为验证使用的数据集能否反映真实的临床情况,我们对来自GEO的大样本乳腺癌 GSE47561(n=1750)[11]数据和来自NKI的大样本乳腺癌数据集(n=295)[7]按照患者分子分型标准进行了 Kaplan⁃Meier生存分析和Log⁃rank检验.结果显示,Luminal A型患者具有最好的预后,包括无病生存(disease⁃free survival, DFS),无远端转移生存(distant metasta⁃sis free survival, DMFS)和总生存(overall survival,OS),与临床共识相吻合(图1A).
此外,本研究采用合并的多个大样本乳腺癌数据库数据(包括 E⁃MTAB⁃365,E⁃TABM⁃43, GSE11121,GSE12093,GSE12276,GSE1456,GSE16391,GSE16446,GSE16716,GSE17705, GSE17907, GSE18728, GSE19615,GSE20194, GSE20271, GSE2034, GSE20685, GSE20711,GSE21653, GSE2603, GSE26971, GSE2990, GSE31448,GSE31519, GSE32646, GSE3494, GSE37946, GSE41998,GSE42568, GSE45255, GSE4611, GSE3527, GSE6532,GSE7390,GSE9195, n = 3951, http://kmplot.com),对乳腺癌ER/PR/HER2表达水平差异的患者预后进行了Kaplan⁃Meier生存分析和 Log⁃Rank 检验,预后指标包括 OS、DMFS和无复发生存(recurrence⁃free survival,RFS).ER阳性的患者和PR阳性的患者具有较好的预后,而HER2阳性的患者预后较差,这一系列结果与临床共识和文献报道相吻合,证明使用的数据能够真实可靠地反映临床情况[4-5](图 1B).

图1 数据检验:ER/PR/HER2表达差异及不同分型的乳腺癌预后比较
2.2 H2AFZ、MLF1IP和NEK2的表达水平与乳腺癌ER、PR和HER2水平相关 本研究对乳腺癌数据集GSE47561进行了表达谱数据和患者临床信息的匹配,并借助 SPSS17.0 和 GraphPad Prism 5.0 进行统计学分析,寻找不同临床表征患者之间表达差异的基因.其中,H2AFZ(H2A histone family, member Z)、MLF1IP(myeloid leukemia factor 1⁃interacting protein)和NEK2(NIMA related kinase 2)三个分子在雌激素受体阴性(ER-)时表达显著高于雌激素受体阳性组(ER+);在孕激素受体阴性组(PR-)表达显著高于孕激素受体阳性组(PR+);在人表皮生长因子受体2阳性组(HER2+)表达显著高于其阴性组(HER2-)(图2A).同时,在该数据集中对这三种分子在五种不同分子分型的乳腺癌中表达情况的分析发现,在预后较好的 Normal⁃like和 Luminal A亚型中,H2AFZ、MLF1IP和NEK2三个分子的表达水平较低,在预后较差的三个亚型中三个分子的表达水平较高(图 2B).
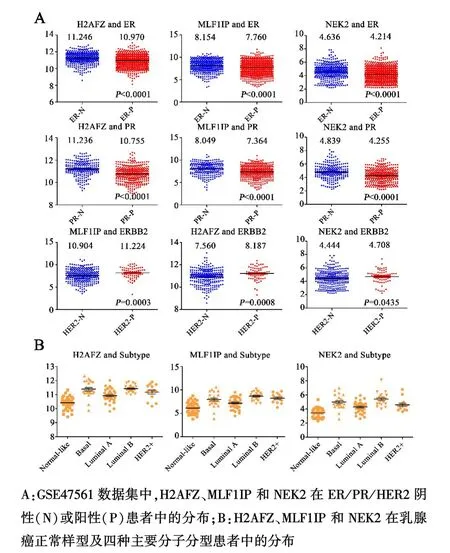
图2 H2AFZ、MLF1IP和NEK2的表达水平在不同乳腺癌亚型中的分布
2.3 H2AFZ、MLF1IP 和 NEK2 与 ER、PR 和HER2表达水平线性相关 为进一步验证H2AFZ、MLF1IP和NEK2在乳腺癌中表达水平的趋势,本研究在GSE47561数据集中对此三个分子和ER/PR/HER2表达水平的关系进行了Pearson Correlation分析.分析结果显示,H2AFZ、MLF1IP和 NEK2的mRNA 水平皆与 ER(P<0.0001)和 PR (P=0.0036,P=0.0074,P<0.0001) 水平呈显著负向相关,而与HER2 水平 (P<0.0001,P=0.0035,P=0.0022) 显著正向相关,提示 H2AFZ、MLF1IP和 NEK2的表达水平可能与 ER/PR表达水平存在拮抗作用,而与HER2表达水平存在促进作用(图3).

图3 GSE47561数据集中,H2AFZ、MLF1IP和 NEK2与ER(ESR1)、PR(PGR)和 HER2 (ERBB2) 表达水平的相关性分析
2.4 H2AFZ、MLF1IP和NEK2为乳腺癌潜在的不利预后因子 本研究在合并多个乳腺癌数据后的大样本人群中 (n=3951)对 H2AFZ、MLF1IP和 NEK2与患者预后(OS, DMFS)进行 Kaplan⁃Meier生存分析和Log⁃Rank检验,发现这三个分子的高表达与OS和DMFS呈显著负相关(图4A).
进一步根据 ER/PR/HER2表达阴阳性(-/+)情况分层分析发现,这三个分子在ER+患者中均与OS和DMFS显著负相关,而在 ER-患者中与 OS和DMFS的相关性并不显著(图4B);H2AFZ和MLF1IP分子在PR+患者中与OS负相关、与DMFS不相关,而在PR-患者中与OS不相关,同时NEK2也仅在PR+患者中体现出与预后(主要为DMFS)负相关的趋势(图4C);在HER2-患者中三个分子与OS显著负相关,同时 H2AFZ、NEK2与 DMFS显著负相关,而在HER2+的患者中只有 MLF1IP与DMFS负相关(图4D).这些结果提示,H2AFZ、MLF1IP和 NEK2皆为乳腺癌不利预后因子,同时它们对肿瘤恶性程度的促进和患者预后的不利影响很可能受到较高ER/PR表达的拮抗和较高HER2表达的促进.
2.5 H2AFZ、MLF1IP和NEK2对乳腺癌预后的影响限于中低分级患者 本研究在合并多个乳癌数据后的大样本人群中对H2AFZ、MLF1IP和NEK2与淋巴结(LN)转移和分级(grade)的关系进行了研究,结果表明该三个分子的表达无论是在LN-还是在LN+的乳腺癌患者中均与DMFS负相关,而与OS的负相关性限于在LN-的患者中(图5A).

图4 H2AFZ、MLF1IP和NEK2与不同分子亚型乳腺癌的预后分析
随后,本研究对不同分期的患者进行了生存分析(OS,DMFS).在 grade1患者中,MLF1IP 的表达与 OS和DMFS皆负相关,而尽管P值略大,H2AFZ也展现出了与预后负相关的趋势;在grade2患者中,这三个分子的表达均与 DMFS负相关,同时 MLF1IP和NEK2展现出与预后的负相关趋势;在grade3患者中,三个分子的水平与OS皆不相关,仅有MLF1IP的表达与DMFS负相关.考虑到grade1患者较少的数量影响了统计结果的显著性,这三个分子对乳腺癌预后的不利影响主要限于中低分级(grade2及以下)的患者(图5B).

图5 H2AFZ、MLF1IP和NEK2与乳腺癌淋巴结转移 (LN)及分级 (grade)的关系
2.6 H2AFZ、MLF1IP 和 NEK2在 Luminal A 亚型中与预后负相关,在其他亚型中与预后的相关性较低
将合并多个乳癌数据后的大样本人群按照乳腺癌分子分型标准划分为 Basal/HER2+/Luminal A 和Luminal B型,随后分析 H2AFZ、MLF1IP和 NEK2在各个亚型中与预后的关系.Luminal A亚型的乳腺癌中,H2AFZ、MLF1IP和NEK2均与OS和DMFS负相关,而在其他情况下只有NEK2在Luminal B亚型的乳腺癌中与DMFS负相关,其余情况下三者与其余亚型的预后关联不大(图6).

图6 在合并多个乳癌数据后的大样本人群中(如前所述,来自http://kmplot.com,n=3951)对H2AFZ、MLF1IP和NEK2与乳腺癌多种亚型进行的生存分析
在前面的分析中,本研究已经发现 H2AFZ、MLF1IP 和 NEK2仅在 ER+/PR+/HER2-或中低分化的患者中与预后相关(图4、5),该类型的患者对应Luminal A型.根据前期分析,由于 H2AFZ、MLF1IP和NEK2与ER/PR水平呈负相关,与HER2+水平呈正相关,故Luminal A型患者本身表达这三个分子的水平较低(图2),因此Luminal A亚型患者较好的预后可能与H2AFZ、MLF1IP和NEK2的表达量低有关.若H2AFZ、MLF1IP和NEK2中的一个或多个水平升高,便可能打破平衡,导致相关肿瘤信号通路激活,Luminal A患者预后变差.而在Luminal B、Basal或HER2+型患者中,H2AFZ、MLF1IP和NEK2表达维持在较高水平,由此可能促进肿瘤恶性进展,故此几类患者的预后明显劣于Luminal A型.
3 讨论
长期以来,乳腺癌ER、PR和HER2的表达区别是影响患者预后最主要的原因之一[3].根据分子及病理指标进行分型,是乳腺癌诊断和治疗中必不可少的关键环节.本研究借助生物信息分析手段,对大样本乳腺癌表达谱数据和临床信息进行了分析,挖掘得到与预后负相关的H2AFZ、MLF1IP和NEK2基因.作为不利预后因子,H2AFZ、MLF1IP和NEK2与ER/PR的表达水平相反,与HER2的水平升高一致,同时在Luminal A型乳腺癌中表达量最低,在其余亚型中表达量较高,这些表达差异很可能与乳腺癌不同亚型患者的预后密切相关.我们猜想,H2AFZ、MLF1IP和NEK2在Luminal A型患者中的特异性低表达可能为该种亚型预后较好的因素之一.
H2AFZ为细胞周期相关基因,参与维持异染色质和常染色质水平,与哺乳动物干细胞的生成和分化有关[12],常受到甲基化、乙酰化、泛素化等修饰的调控[12].Járez⁃Velázquez等[13]指出其高表达可能预示着儿童急性淋巴母细胞白血病(acute lymphoblastic leukemia)较短的预后和较高的复发率,而Baptista等[14]发现其在前列腺癌中受到 sirtuin 1的调控.H2AFZ亦可通过加速细胞周期进程以及调节EMT相关蛋白来参与肝癌的发生[15],同时,一些组学研究筛查也发现H2AFZ是鼻咽癌等肿瘤潜在的分子靶向标志物[16-17],但其与乳腺癌的关系研究较少.我们知道,ER作为一种配体依赖的转录因子,可以协同辅调节因子来调节靶基因的转录激活[18].有研究表明,H2AFZ可以作为核小体的组成部分参与到ERɑ靶基因的转录激活过程中[19].近期研究也表明,H2AFZ是通过使eRNA与增强子结合并招募RNA聚合酶2来建立并维持稳定的染色质环境,这对提高ERɑ靶基因的增强子活性至关重要[20].我们猜想或许是H2AFZ在ER阳性的乳腺癌患者中充当不利预后因子的原因之一.
MLF1IP又名CENPU,于2003年被发现,是潜在的转录抑制基因[21],其高表达可能与急性粒细胞白血病(AML)、恶性胶质瘤的进展相关[22].近期研究显示,该分子与着丝粒和微管的连接及染色体的分离相关[23],且能够促进小鼠红细胞的增殖加速[24],为肺癌和前列腺癌中潜在的肿瘤相关基因[25-26],但其与肿瘤,尤其是乳腺癌的相关性至今仍报道较少.
NEK2为有丝分裂相关的丝/苏氨酸激酶,在有丝分裂S期表达水平升高,并在G2期达到峰值[27].NEK2定位于中心体,在许多乳腺癌细胞中高表达,同时在侵袭性乳腺癌中上调,可与CDK4协同促进HER2+乳腺癌的细胞核和中心体扩大,导致细胞多核型[28-29].在预后较差的乳腺癌 Basal型和 HER2 型中,均存在由中心体扩大引起的染色体不稳定现象[30],该现象可引起肿瘤细胞的增殖增加、易侵袭、转移等表现变化[31-32].另外,NEK2 在乳腺癌 IDC(invasive ductal carcinoma)及 DCIS(concomitant duc⁃tal carcinoma)的细胞浆中表达升高,其浆表达与组织学分级(histological grade)和肿瘤大小有关[33].在经它莫西芬治疗的患者中,NEK2的表达与无复发和无远端转移生存负相关,其表达往往预示着较差的预后[33].
考虑到H2AFZ、MLF1IP和NEK2皆在细胞生命活动,尤其是在有丝分裂的调控中具有重要的作用,它们三者对乳腺癌进展的推动很可能来源于对细胞增殖的促进和对细胞周期的影响.细胞周期调控异常会引起HER2依赖的乳腺癌的耐药现象[34],这很可能是H2AFZ,MLF1IP和NEK2作为乳腺癌不利预后因子的原因之一.另外,MLF1IP和NEK2都可引起染色体不稳定现象[23,29],因而可以认为这两个分子会促进乳腺癌的恶性表型.实验结果也显示,H2AFZ、MLF1IP和NEK2的表达水平可能与ER/PR表达水平存在拮抗作用,而与HER2表达水平存在促进作用.又因为普遍认为ER/PR是乳腺癌的有利因子,而HER2的表达与预后负相关[5],那么此三者的高表达就会对肿瘤恶性起到持续的贡献.所以我们猜想,正是由于H2AFZ、MLF1IP和NEK2在不同分子病理特征的乳腺癌患者中存在表达差异,导致了相应亚型的患者预后出现区别.然而,H2AFZ、MLF1IP和NEK2在ER、PR阳性或HER2阴性患者中体现出的低表达水平究竟源于何种机理,还有待于进一步的研究.同时,H2AFZ、MLF1IP和NEK2的表达水平差异可以作为乳腺癌患者病理检测的重要依据,为患者激素治疗/Herceptin靶向治疗方案的选择和预后/疗效评估提供指导.
[1]Chu DZ.Re: Breast cancer incidence,1980-2006: combined roles of menopausal hormone therapy, screening mammography, and estrogen receptor status[J].J Natl Cancer Inst,2008,100(8):596-597.
[2]Ravdin PM, Cronin KA, Howlader N, et al.The decrease in breast⁃cancer incidence in 2003 in the United States[J].N Engl J Med,2007,356(16):1670-1674.
[3]Brenton JD, Carey LA, Ahmed AA, et al.Molecular classification and molecular forecasting of breast cancer: ready for clinical application[J].J Clin Oncol,2005,23(29):7350-7360.
[4]Sørlie T, Tibshirani R, Parker J, et al.Repeated observation of breast tumor subtypes in independent gene expression data sets[J].Proc Natl Acad Sci U S A,2003,100(14):8418-8423.
[5]Perou CM, Sørlie T, Eisen MB, et al.Molecular portraits of human breast tumours[J].Nature,2000,406(6797):747-752.
[6]Sotiriou C,Neo SY,McShane LM,et al.Breast cancer classification and prognosis based on gene expression profiles from a population⁃based study[J].Proc Natl Acad Sci U S A,2003,100(18):10393-10398.
[7]van de Vijver MJ, He YD, van't Veer LJ, et al.A gene⁃expression signature as a predictor of survival in breast cancer[J].N Engl J Med,2002,347(25):1999-2009.
[8]Lauss M, Kriegner A, Vierlinger K, et al.Consensus genes of the literature to predict breast cancer recurrence[J].Breast Cancer Res Treat,2008,110(2):235-244.
[9]Sørlie T, Perou CM, Fan C, et al.Gene expression profiles do not consistently predict the clinical treatment response in locally advanced breast cancer[J].Mol Cancer Ther,2006,5(11):2914-2918.
[10]Györffy B, Lanczky A, Eklund AC, et al.An online survival analysis tool to rapidly assess the effect of 22,277 genes on breast cancerprognosis using microarray data of 1,809 patients[J].Breast Cancer Res Treat,2010,123(3):725-731.
[11]Ur⁃Rehman S, Gao Q, Mitsopoulos C, et al.ROCK: a resource for integrative breast cancer data analysis[J].Breast Cancer Res Treat,2013,139(3):907-921.
[12]Faast R, Thonglairoam V, Schulz TC, et al.Histone variant H2A.Z is required for early mammalian development[J].Curr Biol,2001,11(15):1183-1187.
[13]Juárez⁃Velázquez R, Reyes⁃León A, Salas⁃Labadia C, et al.Signifi⁃cance of CASP8AP2 and H2AFZ expression in survival and risk of relapse in children with acute lymphoblastic leukemia[J].Leuk Lymphoma,2014,55(10):2305-2311.
[14]Baptista T, Graca I, Sousa EJ, et al.Regulation of histone H2A.Z expression is mediated by sirtuin 1 in prostate cancer[J].Oncotarget,2013,4(10):1673-1685.
[15]Yang HD, Kim PJ, Eun JW, et al.Oncogenic potential of histone⁃variant H2A.Z.1 and its regulatory role in cell cycle and epithelial⁃mesenchymal transition in liver cancer[ J].Oncotarget, 2016,7(10):11412-11423.
[16]Xiong S, Wang Q, Zheng L, et al.Identification of candidate molec⁃ular markers of nasopharyngeal Carcinoma by tissue microarray and in situ hybridization[J].Med Oncol,2011,28 Suppl 1:S341-S348.
[17]Rhodes DR, Yu J, Shanker K, et al.Large⁃scale meta⁃analysis of cancer microarray data identifies common transcriptional profiles of neoplastic transformation and progression[J].Proc Natl Acad Sci U S A,2004,101(25):9309-9314.
[18]Green KA, Carroll JS.Oestrogen⁃receptor⁃mediated transcription and the influence of co⁃factors and chromatin state[J].Nat Rev Cancer,2007,7(9):713-722.
[19]Gévry N, Hardy S, Jacques PE, et al.Histone H2A.Z is essential for estrogen receptor signaling[J].Genes Dev,2009,23(13):1522-1533.
[20]Brunelle M, Nordell Markovits A, Rodrigue S, et al.The histone variant H2A.Z is an important regulator of enhancer activity[J].Nucleic Acids Res,2015,43(20):9742-9756.
[21]Hanissian SH, Akbar U, Teng B, et al.cDNA cloning and charac⁃terization of a novel gene encoding the MLF1⁃interacting protein MLF1IP[J].Oncogene,2004,23(20):3700-3707.
[22]Hanissian SH, Teng B,Akbar U,et al.Regulation of myeloid leukemia factor⁃1 interacting protein (MLF1IP) expression in glioblastoma[J].Brain Res,2005,1047(1):56-64.
[23]Minoshima Y, Hori T, Okada M, et al.The constitutive centromere component CENP⁃50 is required for recovery from spindle damage[J].Mol Cell Biol,2005,25(23):10315-10328.
[24]Zhang T, Ma X, Li B, et al.A novel erythroid regulator MLF1IP promotes proliferation in mice by regulating cell cycle[J].Blood,2015,126(23):2835.
[25]Zhang L, Ji G, Shao Y, et al.MLF1 interacting protein: a potential gene therapy target for human prostate cancer[J].Med Oncol,2015,32(2):454.
[26]Lou Y, Li R, Liu J, et al.HSG⁃MLF1IP axis as potential targets for lung adenocarcinoma treatment[J].J Clin Oncol,2015,33(15 suppl):e13591-e.
[27]Fry AM,Meraldi P,Nigg EA.A centrosomal function for the human Nek2 protein kinase, a member of the NIMA family of cell cycle reg⁃ulators[J].Embo J,1998,17(2):470-481.
[28]Harrison Pitner MK, Saavedra HI.Cdk4 and nek2 signal binucleation and centrosome amplification in a her2+ breast cancer model[J].PLoS One,2013,8(6):e65971.
[29]Wang S, Li W, Lv S, et al.Abnormal expression of Nek2 and β⁃catenin in breast carcinoma: clinicopathological correlations[J].Histopathology,2011,59(4):631-642.
[30]Fukasawa K.Oncogenes and tumour suppressors take on centrosomes[J].Nat Rev Cancer,2007,7(12):911-924.
[31]Roylance R, Endesfelder D, Gorman P, et al.Relationship of extreme chromosomal instability with long⁃term survival in a retro⁃spective analysis of primary breast cancer[J].Cancer Epidemiol Biomarkers Prev,2011,20(10):2183-2194.
[32]Pannu V, Mittal K, Cantuaria G, et al.Rampant centrosome ampli⁃fication underlies more aggressive disease course of triple negative breast cancers[J].Oncotarget,2015, 6(12):10487-10497.
[33]Marina M, Saavedra HI.Nek2 and Plk4: prognostic markers, drivers of breast tumorigenesis and drug resistance[J].Front Biosci(Land⁃mark Ed),2014,19:352-365.
[34]Harari D, Yarden Y.Molecular mechanisms underlying ErbB2/HER2 action in breast cancer[J].Oncogene,2000,19(53):6102-6114.
Correlation analysis between expression levels of H2AFZ, MLF1IP and NEK2 in cross⁃sub⁃type diversity of breast cancer and prognosis
SUN Tian⁃Yi, ZHANG Cheng, SHOU Cheng⁃Chao
1Department of Biochemistry and Molecular Biology,2Department of Digestive Oncology, Peking University Cancer Hospital & Insti⁃tute, Beijing 100142, China
AIM: To investigate potential biomarkers associated with breast cancer molecular subtyping and prognosis,and provide basis for diagnosis and treatment of breast cancer.METHODS:Large⁃sample data of breast cancer sets extracted from public online databases were analyzed by bioinformatics and statistics.RESULTS: H2AFZ, MLF1IP and NEK2 were negative prognostic factors of breast cancer,and they were significantly correlated with the expression level of ER/PR/HER2 in breast cancer patients.The mRNA of H2AFZ,MLF1IP,NEK2 showed low levels in ER and PR⁃positive breast cancer, while were highly expressed in HER2⁃positive breast cancer.The expression levels of H2AFZ,MLF1IP, NEK2 in Luminal A were lower, and were negatively correlatied with prognosis,meanwhile the expression levels of H2AFZ, MLF1IP, NEK2 in the other subtypes were higher, and the correlation between H2AFZ, MLF1IP, NEK2 and prognosis was not significant.CONCLUSION: As adverse⁃prognostic factors,the different expression of these molecules might be one of the reasons causing the malignant degree and prognostic differences among breast cancer subtyping.
breast cancer; large⁃sample data mining; molecular subtyping; prognosis⁃related genes
Q279
A
2095⁃6894(2017)09⁃23⁃09
2017-04-29;接受日期:2017-05-13
科技部973项目(2015CB553906)
孙天一.E⁃mail:styfirst@ 163.com
寿成超.博士,教授,博导.E⁃mail:Chengchaoshou@ outlook.com
