草鱼野生与选育群体线粒体DNA控制区D-loop遗传变异分析
2017-09-12王沈同党云飞李立森黄文吉沈玉帮李家乐
王沈同 张 猛 党云飞 李立森 黄文吉 方 圆 王 丰 沈玉帮 李家乐
(上海海洋大学省部共建水产种质资源发掘与利用教育部重点实验室, 上海 201306)
草鱼野生与选育群体线粒体DNA控制区D-loop遗传变异分析
王沈同 张 猛 党云飞 李立森 黄文吉 方 圆 王 丰 沈玉帮 李家乐
(上海海洋大学省部共建水产种质资源发掘与利用教育部重点实验室, 上海 201306)
为探究经过2个选育世代后选育群体遗传多样性和遗传结构的变化, 研究对4个野生群体(邗江、九江、石首和吴江)和2个选育世代(F1和F2)进行了线粒体DNA控制区(D-loop)序列的遗传变异分析。实验结果表明, 4个野生群体在单倍型数目(H)、单倍型多样性(Hd)、核苷酸多样性(π)、平均核苷酸差异数(K)水平上均高于2个选育世代, 在2个选育世代内表现为F1代群体的核苷酸多样性(π)和平均核苷酸差异数(K)大于F2代群体, 但单倍型多样性(Hd)小于F2代群体; 单倍型分析结果表明, 6个群体间无共享单倍型, 4个野生群体间共发现2种共享单倍型(Hap1和Hap3), 石首群体和2个选育世代共享1种单倍型(Hap15); 遗传分化指数(Fst)分析结果表明,邗江、九江、吴江3个野生群体和2个选育世代间存在较大的遗传分化(Fst范围为0.41475—0.55128), 石首群体与F1代群体之间存在较小的遗传分化, 与F2代群体之间存在中等水平的遗传分化, 同时F1代群体与F2代群体之间存在较小的遗传分化; 基于6个群体276个个体构建的邻接(Neighbor-Joining, NJ)进化树和基于27种单倍型构建的单倍型网络图也得到了相似的结论, 即邗江、九江、吴江3个野生群体和2个选育世代间的亲缘关系较远, 石首群体和2个选育世代两两之间的亲缘关系较近。以上结果表明, 经过2个世代的选择育种, 选育群体的遗传结构已发生了变化, 并且随着选育的进行, 选育世代的遗传多样性下降的较为明显, 这警示着我们在今后的育种工作中应适当改变现有的选育方案, 并实时监测选育群体的遗传多样性, 以便为今后进一步的选育工作打下坚实的基础。
草鱼; 选育群体; 野生群体; mt DNA; D-loop; 遗传变异
草鱼(Ctenopharyngodon idella)是我国重要的淡水养殖鱼类之一, 主要分布于长江、珠江、黑龙江三大水系, 自1958年草鱼人工繁育成功以来, 年产量逐年增长[1], 据FAO统计, 草鱼年产量在2012年位于世界水品种产量的第三位[2], 但是近几年草鱼的种质资源日益衰减, 主要表现为性成熟年龄提前,生长速度变慢, 繁殖率低, 疾病发生率变高等, 主要原因归结为草鱼种质资源的退化, 人工繁殖管理的不规范导致草鱼的近交系数变高, 群体的遗传多样性下降[3]。鉴于上述现象, 开展草鱼选择育种的工作迫在眉睫。
在开展遗传育种工作过程中群体遗传多样性的高低显得尤为重要, 较高水平的遗传多样性能确保每一代在育种过程中可以获得更可观的遗传进展、提高育种的效率, 并能提高群体的适应能力和抗病能力[4,5], 然而在一些物种的育种过程中其后代的遗传多样性表现为明显的下降[6,7], 这将不利于后续育种工作的进行, 因此在开展育种工作过程中, 有必要对选育群体进行遗传多样性和遗传结构评估, 以便为育种工作提供一些指导性的意见、提高育种的效率, 进而加快育种工作的进展。
线粒体DNA具有严格的母系遗传特点, 同时具有拷贝数高、DNA分子量小、缺乏重组、结构和组织简单而高度保守、DNA突变率高等一系列优点[8]。控制区(D-loop)是线粒体DNA上的一段非编码区, 受进化压力小、遗传变异程度高[9,10], 并且进化速率快, 是其他区段的5倍[10,11], 常被用于群体水平的遗传变异分析。由于草鱼个体大、性成熟周期长、繁殖和测量性状不易操作等特点, 至今仍没有选育出新的品种, 而且对于草鱼选育过程中选育群体遗传多样性评价及比较的工作相对较少。本研究采用PCR扩增和DNA测序技术对草鱼4个野生群体和2个选育世代的线粒体D-loop区域部分片段进行了遗传变异检测, 初步分析草鱼在经过2个世代的选育工作后, 其选育群体遗传多样性和遗传结构的变化, 为今后制定出更加合理有效的选育方案提供参考。
1 材料与方法
1.1 实验材料与DNA提取
本研究所用的F1代和F2代长江水系选育群体和4个野生群体(邗江、九江、石首和吴江)从苏州市申航生态科技发展股份有限公司采集(表 1)。2007—2008年收集了4个长江水系野生群体(邗江、九江、石首和吴江), 经过遗传分析后构建了长江水系基础群体。2010年从基础群体中随机选取88尾亲本采用随机交配和定向交配(个体的遗传距离设计)结合的方式繁殖产生了F1代[12]。经过5年的培育, 2015年选取来自于不同家系的106尾亲本采用随机交配的方法繁殖产生了F2代。剪取6个群体的草鱼胸鳍组织, 保存于无水乙醇中以便于之后的DNA提取, 本实验中所有样品采用海洋动物组织基因组DNA提取试剂盒(天根生化科技有限公司)提取基因组DNA, 通过1%的琼脂糖凝胶电泳检测其完整性, 经NanoDrop 2000C分光光度计(赛默飞世尔科技有限公司)检测其纯度及浓度, 并将DNA样品稀释成20 ng/μL, 于–20℃保存备用。
1.2 PCR扩增及测序
根据草鱼现有线粒体D-loop区域区段引物[13]完成PCR扩增, 上游引物和下游引物序列分别为: DLF: 5′-CCTAGCGCCCAGAAAAGGGAGATT-3′; DLR: 5′-GCGGGGGATTGAGGGCATACTC-3′, PCR反应体系总体积为50 μL: Taq PCR Mastermix 13 μL [成分为0.1 U Taq Polymerase/μL、500 μmol/L dNTP each、20 mmol/L Tris-HCl (pH 8.3)、100 mmol/ L KCl、3 mmol/L MgCl2、其他稳定剂和增强剂],上下游引物各1 μL (10 μmol/L), 模板DNA 40 ng, ddH2O 33 μL。该试剂购自天根生化科技(北京)有限公司。PCR扩增程序为: 94℃预变性3min; 94℃变性30s, 50℃复性30s, 72℃延伸2min, 扩增35个循环; 72℃延伸10min。扩增反应在Eppendorf梯度PCR仪上完成, 反应产物经1%琼脂糖凝胶电泳检测, 合格的PCR产物由上海生工生物工程股份有限公司进行测序。
1.3 序列整理与数据分析
采用Mega5.1[14]进行序列间的同源对比和序列编辑, 统计各群体的突变位点数、突变类型和核苷酸组成, 并基于Kimura双参数模型(Kimura 2-Parameter, K2P)构建6个群体276个个体的邻接(Neighbor-Joining, NJ)进化树, 系统发育树中节点的自举置信水平应用自引导估计, 循环次数为1000次。采用DNASP 5.0软件[15]统计各单倍型分布情况、单倍型数(H)、单倍型多样性(Hd)、核苷酸多样性(π)、平均核苷酸差异数(K)、Tajima’s D值。采用Arlequin3.5软件进行6个群体的分子方差分析(AMOVA), 并计算6个群体两两之间的遗传分化指数(Fst)。采用Network软件构建单倍型网络图, 用以检测单倍型之间的进化关系。
2 结果
2.1 线粒体控制区(D-loop)序列分析
对野生群体和选育世代线粒体控制区(D-loop)序列进行同源对比后, 得到894 bp的同源序列。6个群体位点变异情况如表 1所示, 除石首群体存在2个颠换类型, 其他群体碱基突变类型均为转换类型。碱基组成分析表示各群体碱基组成大致相同, T、C、A、G 4种碱基平均值分别为32.17%、21.03%、34.45%和12.36%, A+T (66.62%)所占比值明显高于G+C (33.38%), 表现出明显的AT偏好, 这与其他脊椎动物线粒体控制区(D-loop)核苷酸的组成特点相一致[16]。
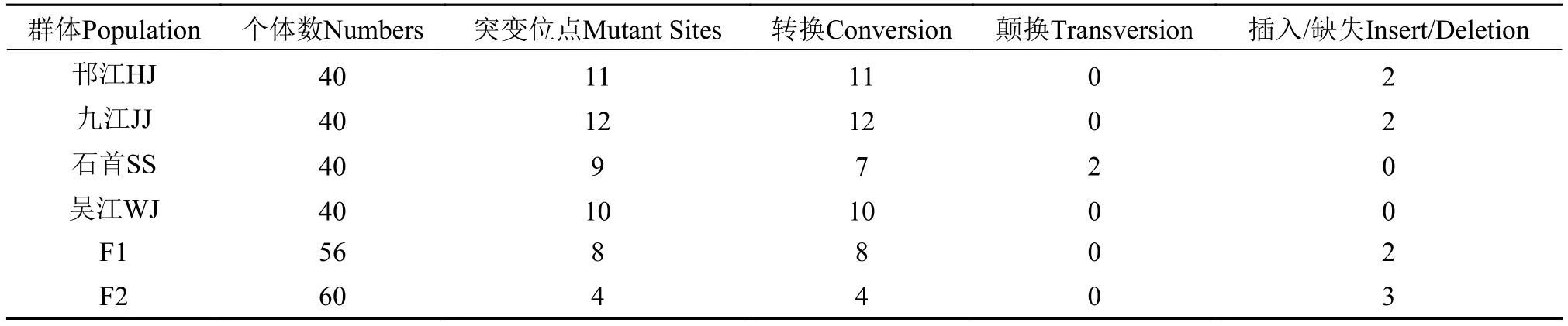
表 1 草鱼线粒体DNA控制区(D-loop)序列突变位点数目Tab. 1 The number of mutation sites of mt DNA D-loop region in grass carp
2.2 草鱼6个群体的遗传多样性及单倍型分析
6个群体线粒体DNA控制区(D-loop)遗传多样性参数如表 2所示, 无论是在单倍型数目(H)、单倍型多样性(Hd)、核苷酸多样性(π)还是平均核苷酸差异数(K)水平上, 野生群体的遗传多样性均比2个选育世代的要高。对4个野生群体而言, 石首群体的单倍型多样性(Hd)最高(0.637), 吴江群体表现为最低(0.506); 邗江群体的核苷酸多样性(π)和平均核苷酸差异数(K)最高(π=0.00232, K=2.063), 吴江群体表现为最低(π=0.00142, K=1.271); 2个选育世代相互比较发现, 两者单倍型数目(H)一致, F1代选育群体的核苷酸多样性(π)和平均核苷酸差异数(K)均高于F2代选育群体, 但单倍型多样性(Hd)表现为前者小于后者; 草鱼6个群体的Tajima’s D 值为负值,且均未显著偏离中性(P>0.1)。
其单倍型分布如表 3所示, 6个群体共检测出27种单倍型, 没有发现共同享有的单倍型, 其中Hap15、Hap1为优势单倍型, 分别占6个群体总个体数的39%和32%。4个野生群体间存在2种共享单倍型Hap1和Hap3, 2个选育群体间存在1种共享单倍型Hap15。
2.3 草鱼6个群体的遗传结构
对草鱼6个群体两两之间遗传分化情况进行分析, 分析结果如表 4所示, F1代和F2代群体间的遗传分化指数(Fst)为0.05741(P<0.01), 结果表明2个世代间遗传分化程度较低, 邗江、九江、吴江3个野生群体与2个选育世代之间的遗传分化指数(Fst)均大于0.25(P值均小于0.01), 说明这3个野生群体与2个选育世代间存在着较大的遗传分化, 并发现石首群体与F1代群体的遗传分化指数Fst值为0.04861,与F2代群体的遗传分化指数Fst值为0.13205, 结果表明石首群体和F1代群体间几乎不存在着遗传分化, 而和F2代群体之间存在着中等水平的分化程度。
以遗传分化指数(Fst)为依据进一步对草鱼6个群体的核苷酸序列变异做分子方差分析, 6个群体分为2组(1组为HJ+JJ+WJ; 2组为SS+F1+F2), 总的方差剖分为组间方差(Va)、组内群体间方差(Vb)和群体内方差(Vc)3个部分, 并进行显著性检验。结果如表 5所示, 组间方差(Va)占总变异的38.11%, 组内群体间的方差组分(Vb)占总变异的2.21%, 群体内的方差组分(Vc)占总变异的59.69%, Fst=0.40312 (P<0.01)。
2.4 草鱼6个群体的系统发育树和单倍型简约网络图
根据草鱼4个野生群体和2个选育世代共276个个体构建的NJ系统发育树发现6个群体在系统发育树上基本被分成两大分支, 3个野生群体(邗江、九江、吴江)构成一大分支, 2个选育世代(F1和F2)和石首群体构成另外一个分支, 不同分支内个体间存在相互交叉, 其结果表明F1代、F2代和石首群体三者之间的亲缘关系较近, 而这3个群体和另外3个野生群体(邗江、九江、吴江)的亲缘关系较远。基于6个群体27种单倍型的单倍型网络图如图 2所示, F1代、F2代和石首群体3个群体间的亲缘关系较近, 而2个选育群体和邗江、九江、吴江3个野生群体的亲缘关系较远, 与NJ系统发育树的分析的结果基本一致, 并且从图中可以看出享有Hap15和Hap1单倍型的个体数在种群中所占比例明显高于其他单倍型所占比例。
3 讨论
3.1 草鱼野生与选育群体的遗传多样性分析
核苷酸多样性(π)和单倍型多样性(Hd)是衡量一个群体mt DNA变异程度的重要指标。其中核苷酸多样性(π)表示各种mt DNA单倍型在群体中所占的比例[17], 是评价群体遗传多样性的灵敏指标。本研究中6个群体的核苷酸多样性(0.00065—0.00232), 平均核苷酸差异数(0.578—2.063)在群体间的变化趋势一致, 均为野生群体>F1>F2, 以上数据表明野生群体的遗传多样性高于2个选育世代,并且随着选育的进行, F2代选育群体的遗传多样性低于F1代选育群体, 2个选育世代的遗传多样性较野生群体下降的较为明显, 这类现象在其他研究中也曾发生过, Eric等[18]发现在大西洋鲑(Salmo salar)选育过程中, 其第一代选育群体的遗传多样性下降的较为明显, 其推测原因可能为参与繁育的亲本数目较少所导致的, 颉晓勇等[19]对吉富品系尼罗罗非鱼(Oreochromis niloticus)不同世代选育群体D-loop序列进行测定和分析, 发现随着选育的发展,选育群体遗传多样性逐步降低, 基础群体和选育群体的遗传距离逐步增大, 赵广泰等[20]利用微卫星标记对大黄鱼(Larimichthys crocea)4个选育群体进行了遗传变异分析, 结果表明, 连续四代的选育, 选育群体的遗传基础逐步纯化, 遗传多样性逐步下降。本文通过2代选育群体遗传变异的分析, 发现其遗传多样性下降地较为明显, 推测原因可能有以下几点:
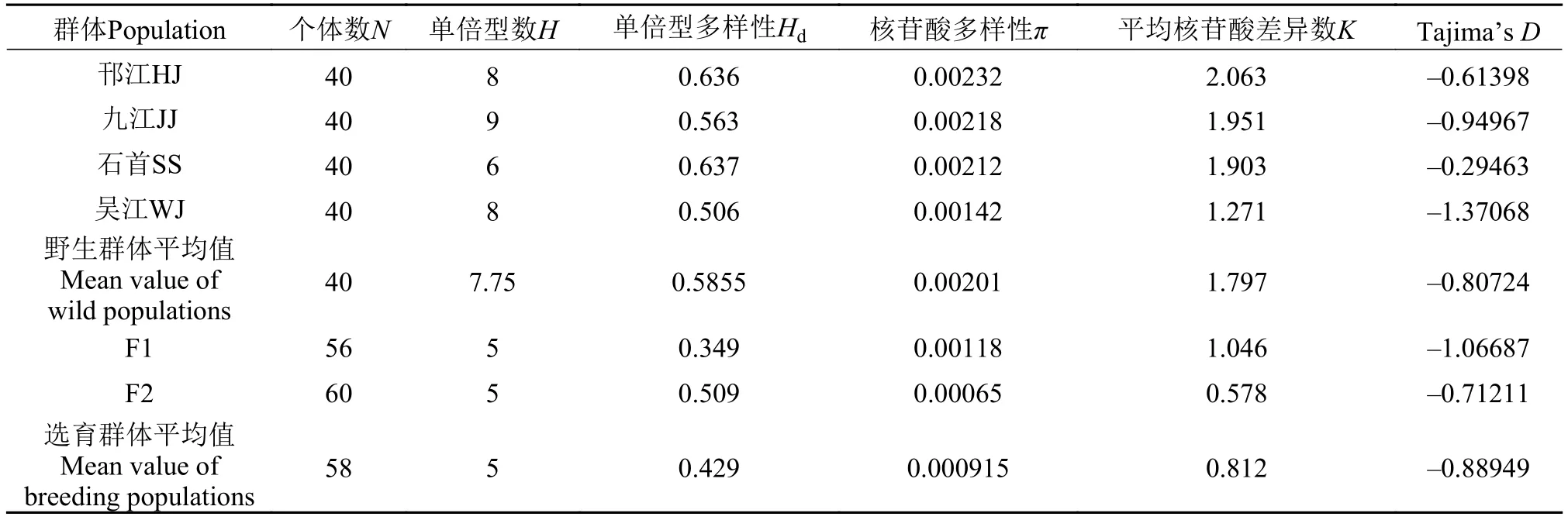
表 2 草鱼线粒体DNA控制区(D-loop)遗传多样性参数Tab. 2 Genetic diversity parameters of mt DNA D-loop region in grass carp

表 3 草鱼6个群体单倍型分布情况Tab. 3 Distribution of the haplotypes among six grass carp populations
首先, 选育方案中各世代参与的亲本数较少,有研究表明, 当群体有效亲本数量小于100时, 会导致遗传漂变的加剧, 降低群体的遗传多样性[21—23];其二, 有学者指出, 后代群体遗传多样性的下降不仅和有限的亲本数有关, 亲本随机交配这种交配方案本身会造成后代各家系数目的不均衡[24], Fu等[12]也发现草鱼存在后代各家系数目比例失衡的问题,究其原因, Dewoody和Avise[25]认为雄性之间在繁育时具有竞争性会导致个别雄性后代个数的增加, 而且研究表明个头越大, 体重越重, 肥满度越高的个体在竞争时更具有优势, 往往后代越多[26—28], 以上观点可以从逻辑上解释在一个密闭环境进行随机交配会发现后代各家系数目不均衡的现象, 并且随机交配的交配方案对于后代各家系数目不均衡的这种现象在其他物种中也曾发现过[24,28,29]。在本实验中, 我们推测在亲本繁殖过程中, 由于随机交配的交配方式, 亲本群体的有效群体数目下降得较为明显, 导致遗传漂变的发生, 遗传漂变的发生进而会导致后代群体遗传多样性的下降[30], 并且随着选育的进程这种现象越发明显; 其三, 有研究发现,较强的人工选择会导致后代遗传多样性的下降[31],本选育方案中只有较理想的个体(体重较高)会被选择当做候选亲本, 其他不理想的个体将会被淘汰掉,而这种较高强度的选择压力可能会影响后代群体的遗传多样性; 其四, 本实验繁育的子代是在养殖桶内随机采集的样品, 有研究表明不同的饲养环境对于后代群体的遗传多样性也具有影响[32], 在养殖桶内开始养殖由于缺乏养殖经验, 鱼苗早期成活率不高, 推测可能由于养殖桶内早期恶劣的生存环境导致群体经历了瓶颈效应, 只有适应力强的家系和个体保存了下来, 以此降低了后代群体的遗传多样性; 其五, 线粒体DNA具有母系无性遗传特点, 对于遗传漂变的影响会更为敏感, 因此不排除利用线粒体DNA部分片断分析群体遗传漂变现象过分夸大的可能性[33], 其他学者也指出, 当对一个物种运用不同的标记进行遗传多样性分析时, 可能会产生不同的结果, 因此我们认为有必要在今后运用不同的分子标记对后代群体的遗传多样性进行进一步的分析, 以检验当前线粒体DNA部分片断分析时得出的结果[34]。
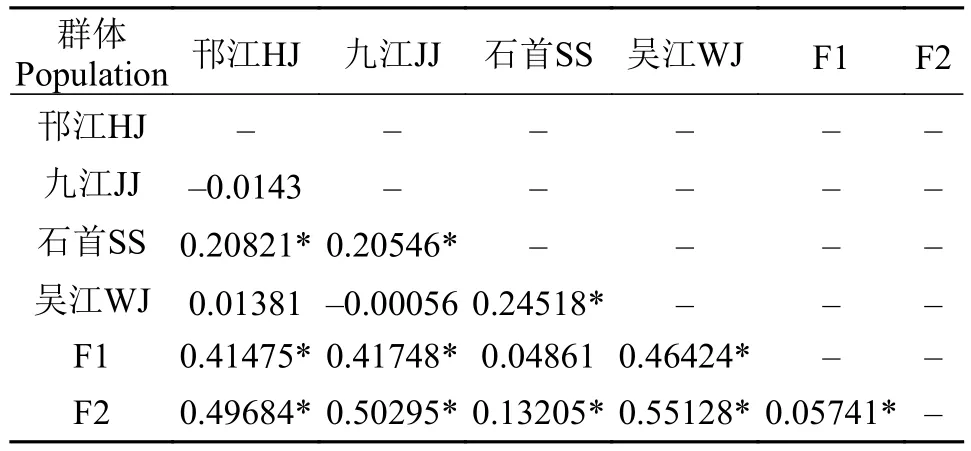
表 4 草鱼6个群体间遗传分化Fst值Tab. 4 Genetic differentiation Fstvalues among six populations of grass carp

表 5 草鱼6个群体间遗传差异的分子方差分析(AMOVA)Tab. 5 Analysis of molecular variance (AMOVA) of six populations of grass carp
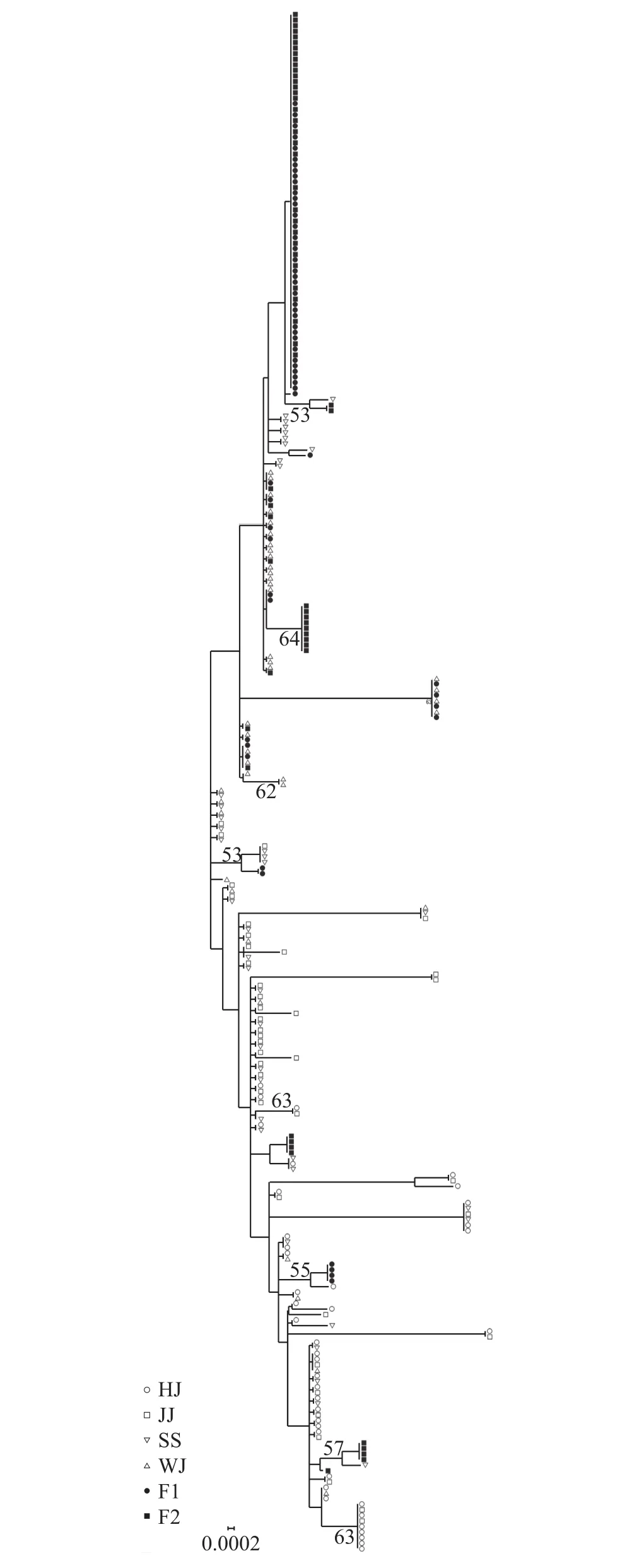
图 1 基于Kimura 2-Parameter模型构建的线粒体控制区序列的NJ聚类Fig. 1 The NJ clustering diagram of the mitochondrial control region sequences constructed based on Kimura 2-Parameter model
本研究发现单倍型多样性(Hd)参数在群体间的变化趋势与上述遗传多样性参数变化趋势有所不同, 表现为野生群体>F2>F1, F2代群体的核苷酸多样性(π)和平均核苷酸差异数(K)虽小于F1代群体,但单倍型多样性(Hd)高于F1代群体, 这种现象在其余物种中也曾发现过[35,36]。这可能是因为极少量的碱基突变就会导致单倍型数目的增长, 而核苷酸多样性的增长是需要时间的积累所影响的[37]。本研究发现F2代选育群体的单倍型多样性(Hd)大于F1代选育群体, 但由于各个单倍型之间的序列差异不大, 所以其核苷酸多样性(π)会相对较小, 那么就会导致单倍型多样性(Hd)和其他遗传多样性参数变化趋势不一致的现象, 但终归来看, 我们可以认为随着选育的进程, 选育世代(F1代和F2代)的遗传多样性下降的较为明显, 且F2代群体的遗传多样性低于F1代。
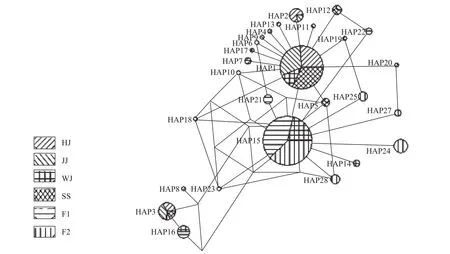
图 2 基于mt DNA控制区构建的草鱼单倍型网络关系图Fig. 2 The haploid types network diagram constructed based on mt DNA D-loop of grass carp
3.2 草鱼野生与选育群体的遗传结构分析
根据Wright[38]关于遗传分化程度的理论认为Fst值在0—0.05表示低度遗传分化, Fst值在0.05—0.15表示中度遗传分化, Fst在0.15—0.25表示遗传分化比较大。遗传分化分析发现F1代与F2代群体之间Fst值为0.05741, 结果表明选育群体之间存在较小的遗传分化, 邗江、九江和吴江3个野生群体与2个选育世代间的Fst在0.41475—0.55128, 表明这3个野生群体和2个选育世代间已发生了明显的遗传分化, 而石首群体与F1代群体的遗传分化指数Fst值为0.04861, 与F2代群体的遗传分化指数Fst值为0.13205, 结果表明石首群体和F1代选育群体间几乎不存在明显的遗传分化, 而和F2代选育群体之间存在着中等水平的分化程度, 进一步对6个群体进行分子方差分析(AMOVA), 其中组间方差组分(Va)占总变异的38.11%, 组内群体间的方差组分(Vb)占总变异的2.21%, 群体内的方差组分(Vc)占总变异的59.69%, 遗传分化指数(Fst)为0.40312, 且差异极显著(P<0.01), 结果表明石首群体、F1代、F2代群体和其余3个野生群体已发生了较高程度的遗传分化, 而石首群体, F1代和F2代之间的遗传分化不太明显, 符合分析遗传分化指数(Fst)得出的结果。
从单倍型分布角度来看, 邗江、九江和吴江3个野生群体与2个选育世代之间不存在共享单倍型, 石首群体与2个选育群体之间共享单倍型为一种(Hap15), 其结果也能从侧面说明2个选育世代和邗江、九江、吴江这3个野生群体之间亲缘关系较远, 而和石首群体的亲缘关系较近, 符合上述分析得出的结论。
从6个群体276个个体构建的NJ系统发育树角度来看, 系统发育树基本被分成2大分支, 2个选育世代(F1代和F2代)与石首群体为一大分支, 邗江、九江、吴江群体为另一分支, 石首群体与2个选育世代(F1代和F2代)在系统发育树上表现为相互交叉; 27种单倍型的单倍型网络图分析结果显示, 4个野生群体以Hap1单倍型为中心呈发散状态势, 2个选育世代及石首群体以Hap15单倍型为中心呈发散状态势, 以上数据都能说明邗江、九江、吴江3个野生群体和2个选育世代(F1代和F2代)之间的亲缘关系较远, 而石首群体和2个选育世代之间的亲缘关系较近, 符合上述分析得出的结果。
因此从总体来看, 2个选育世代与邗江、九江、吴江3个野生群体之间产生了较明显的遗传分化, 而石首群体和2个选育世代(F1代和F2代)两两之间存在着较小或中等水平的遗传分化, 我们推测原因如下: 其一, 在亲本交配繁殖过程中由于草鱼本身存在后代各家系数目不均等问题[12], 因此可能发生少数亲本的子代占据了大部分后代数目的现象, 这种遗传漂变现象可能会导致2个选育世代与邗江、九江、吴江3个野生群体之间产生较明显的遗传分化, 而石首群体可能占据了这少数亲本中的大多数, 因此在检测遗传结构时, 石首群体和2个选育世代(F1代和F2代)之间并没有存在较大的遗传分化; 其二, 在养殖过程中可能由于管理不善, 恶劣的池塘环境导致后代经历了瓶颈效应, 群体数目的大量减少, 强行的加大了后代群体遗传结构的分化; 其三, 人工选择在一定程度上可能改变了选育群体的遗传结构, 使之往一个方向变化, 并逐步趋向稳定, 而这种现象正是我们选择育种想要看到的结果; 其四, 有研究发现, mtDNA D-loop序列分析技术在检测遗传差异水平上可能具有更高的灵敏性[39], 因此不排除用mtDNA D-loop序列分析技术分析得出的遗传分化指数(Fst)有过分夸大的可能性。
综上所述, 经过2个世代的人工选择, 无论是从遗传分化指数(Fst), NJ系统发育树还是单倍型分析的角度来看, 其结果都表明选育群体的遗传结构已发生了改变, 说明我们的选育方案是有成效的, 但从另一个角度来看, 随着选育的进行, 其选育群体的遗传多样性下降地较为明显, 因此这警示着我们在今后的育种过程中, 应尽量保持较大的亲本繁殖数量, 尽量避免随机交配这种交配方案, 减少遗传漂变的发生, 或者增加交配亲本的组数, 分配多组亲本在不同的孵化环道封闭繁殖, 这样既能保证让不同的亲本都参与到交配当中去, 又能减少亲本定向交配时对于鱼体的物理损伤, 同时在幼苗培育时每组取相同的幼体合并培养, 尽可能的使选育群体保持较高的遗传多样性[40]。本实验也反映了在育种过程中实时检测和监控后代遗传多样性的重要性, 启示着我们在今后的选育过程中应及时运用不同的分子标记检验和监控选育群体遗传多样性和遗传结构的变化, 方可有利于今后进一步的育种工作, 为接下来的选育工作打下坚实的基础。
[1]Shen Y B, Zhang J B, Li J L. Advances in studies on genetic resources of grass carp [J]. Chinese Agricultural Science Bulletin, 2011, 27(7): 369—373 [沈玉帮, 张俊彬,李家乐. 草鱼种质资源研究进展. 中国农学通报, 2011, 27(7): 369—373]
[2]FAO. The State of World Fisheries and Aquaculture 2012[M]. Rome, 2012, 209
[3]Liao Y M, Liu J L, Tang X L. The reason of commercial traits degeneration of four major Chinese carps and advice for genetic conservation [J]. Fisheries Science & Technology Information, 1994, 21(2): 62—63 [廖亚明,刘金炉, 汤学林. 浅析“四大家鱼”性状退化的原因及重视种质保护的建议. 水产科技情报, 1994, 21(2): 62—63]
[4]Waples R S. Genetic interactions between hatchery and wild salmonids: lessons from the pacific northwest [J]. Canadian Journal of Fisheries & Aquatic Sciences, 1991, 48(48): 124—133
[5]Ferguson A, Mcginnity P, Stone C, et al. The genetic impact of escaped farm Atlantic salmon on natural populations [J]. Aquaculture, 1995, 137(1): 55—56
[6]Cruz P, Ibarra A M, Mejia-Ruiz H, et al. Genetic variability assessed by microsatellites in a breeding program of Pacific white shrimp (Litopenaeus vannamei) [J]. Marine Biotechnology, 2004, 6(2): 157—164
[7]Wolfus G M, Garcia D K, Alcivar-Warren A. Application of the microsatellite technique for analyzing genetic diversity in shrimp breeding programs [J]. Aquaculture, 1997, 152(1—4): 35—47
[8]Wolstenholme D R. Animal mitochondrial DNA: structure and evolution [J]. International Review of Cytology, 1992, 141(6): 173—216
[9]Rosel P E, Dizon A E, Haygood M G. Variability of the mitochondrial control region in populations of the harbour porpoise, Phocoena, on interoceanic and regional scales [J]. Canadian Journal of Fisheries & Aquatic Sciences, 2011, 52(6): 1210—1219
[10]Rosel P E, Haygood M G, Perrin W F. Phylogenetic relationships among the true porpoises (Cetacea: Phocoenidae) [J]. Molecular Phylogenetics & Evolution, 1995, 4(4): 463—474
[11]Cann R L, Brown W M, Wilson A C. Polymorphic sites and the mechanism of evolution in human mitochondrial DNA [J]. Genetics, 1984, 106(3): 479—499
[12]Fu J J, Shen Y B, Xu X Y, et al. Genetic parameter estimates for growth of grass carp, Ctenopharyngodon idella, at 10 and 18 months of age [J]. Aquaculture, 2016, 450: 342—348
[13]Fu J J, Wang R Q, Shen Y B, et al. Genetic variation analysis based on D-Loop sequences of wild populations of grass carp (Ctenopharyngodon idella) in China [J]. Acta Hydrobiologica Sinica, 2015, 39(2): 349—357 [傅建军,王荣泉, 沈玉帮, 等. 我国草鱼野生群体D-Loop序列遗传变异分析. 水生生物学报, 2015, 39(2): 349—357]
[14]Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods [J]. Molecular Biology & Evolution, 2011, 28(10): 2731—2739
[15]Rozas J, Sanchez-Delbarrio J C, Messeguer X, et al. DnaSP, DNA polymorphism analyses by coalescent and other methods [J]. Bioinformatics, 2004, 19(18): 2496—2497
[16]Brown G G, Gadaltta G, Pepe G, et al. Structural conservation and variation in the D-loop-containing region of vertebrate mitochondrial DNA [J]. Journal of Molecular Biology, 1986, 192(3): 503—511
[17]Liu Y H, Zhang M H. Population genetic diversity of roe deer (Capreolus pygargus) in mountains of Heilongjiang Province [J]. Zoological Research, 2009, 30(2): 113—120
[18]Verspoor E. Reduced Genetic variability in first-generation hatchery populations of Atlantic salmon (Salmo salar) [J]. Canadian Journal of Fisheries & Aquatic Sciences, 1988, 45: 1686—1690
[19]Jie X Y, Li S F, Cai W Q. Genetic variation analysis of Oreochromis niloticus breeding population based on DLoop sequence [J]. Journal of Shanghai Ocean University, 2011, 20(3): 336—341 [颉晓勇, 李思发, 蔡完其.基于D-Loop序列的罗非鱼选育群体遗传变异分析. 上海海洋大学学报, 2011, 20(3): 336—341]
[20]Zhao G T, Liu X D, Wang Z Y, et al. Genetic structure and genetic diversity analysis of four consecutive breeding generations of large yellow croaker (Pseudosciaena crocea) using microsatellite markers [J]. Journal of Fisheries of China, 2010, 34(4): 500—507 [赵广泰, 刘贤德,王志勇, 等. 大黄鱼连续4代选育群体遗传多样性与遗传结构的微卫星分析. 水产学报, 2010, 34(4): 500—507]
[21]Sekino M, Hara M, Taniguchi N. Loss of microsatellite and mitochondrial DNA variation in hatchery strains of Japanese flounder Paralichthys olivaceus [J]. Aquaculture, 2002, 213(1—4):101—122
[22]WinansG A. Genetic variability in Chinook salmon stocks from the Columbia river basin [J]. North American Journal of Fisheries Management, 1989, 9(9): 47—52
[23]Li Q, Park C, Endo T, et al. Loss of genetic variation at microsatellite loci in hatchery strains of the Pacific abalone (Haliotis discus hannai) [J]. Aquaculture, 2004, 235(1-4): 207—222
[24]Lind C E, Evans B S, Knauer J, et al. Decreased genetic diversity and a reduced effective population size in cultured silver-lipped pearl oysters (Pinctada maxima) [J]. Aquaculture, 2009, 286(1—2): 12—19
[25]Dewoody J A, Avise J C. Genetic perspectives on the natural history of fish mating systems [J]. Journal of Heredity, 2001, 92(2): 167—172
[26]Bekkevold D, Hansen M M, Loeschcke V. Male reproductive competition in spawning aggregations of cod (Gadus morhua, L.) [J]. Molecular Ecology, 2002, 11(1): 91—102
[27]Fessehaye Y, El-Bialy Z, Rezk M A, et al. Mating systems and male reproductive success in Nile tilapia (Oreochromis niloticus) in breeding hapas: A microsatellite analysis [J]. Aquaculture, 2006, 256(1—4): 148—158
[28]Frost L A, Evans B S, Jerry D R. Loss of genetic diversity due to hatchery culture practices in barramundi (Lates calcarifer) [J]. Aquaculture, 2007, 272(2): S272
[29]Sekino M, Saitoh K, Yamada T, et al. Microsatellitebased pedigree tracing in a Japanese flounder Paralichthys olivaceus, hatchery strain: implications for hatchery management related to stock enhancement program [J]. Aquaculture, 2003, 221(1—4): 255—263
[30]Ferguson M. The role of molecular genetic markers in the management of cultured fishes [J]. Reviews in Fish Biology & Fisheries, 1994, 4(3): 351—373
[31]Doebley J F, Gaut B S, Smith B D. The molecular genetics of crop domestication [J]. Cell, 2006, 127(7): 1309—1321
[32]Crossman J A, Scribner K T, Yen D T, et al. Gamete and larval collection methods and hatchery rearing environments affect levels of genetic diversity in early life stages of lake sturgeon (Acipenser fulvescens) [J]. Aquaculture, 2011, 310(3—4): 312—324
[33]Pitcher T J, Cingolani N, Bembo D G, et al. Stock discrimination among European anchovy, Engraulis encrasicolus, by means of PCR-amplified mitochondrial DNA analysis [J]. Oceanographic Literature Review, 1995, 94(1): 31—40
[34]Karl S A, Avise J C. Balancing selection at allozyme loci in oysters: implications from nuclear RFLPs [J]. Science, 1992, 256(5053): 100—102
[35]Peng S M, Shi Z H, Hou J L. Comparative analysis on the genetic diversity of cultured and wild silver pomfret populations based on mt D-loop and COI gene [J]. Journal of Fisheries of China, 2010, 34(1): 19—25 [彭士明, 施兆鸿, 侯俊利. 基于线粒体D-loop区与COI基因序列比较分析养殖与野生银鲳群体遗传多样性. 水产学报, 2010, 34(1): 19—25]
[36]Xiao M S, Cui F, Kang J, et al. Genetic structure and variation of wild Chinese catfish from huaihe river based on cytochrome b gene sequences [J]. Chinese Journal of Zoology, 2013, 48(1): 75—86 [肖明松, 崔峰, 康健,等. 淮河野生鲇鱼线粒体Cytb基因的序列变异与遗传结构分析. 动物学杂志, 2013, 48(1): 75—86]
[37]Arbogast B S. Phylogeography: The History and Formation of Species[J]. Integrative & Comparative Biology, 2000, 41(1):134—135
[38]Wright S. The gentical structure of population [J]. Nature, 1950, 166(4215): 247—249
[39]Xiao W, Wang T, Li D Y, et al. Genetic variation of mitochondrial DNA D-loop region in different generations of Egyptian strain of Oreochromis niloticus [J]. South China Fisheries Science, 2015, (3): 29—34 [肖炜, 王腾,李大宇, 等. 埃及品系尼罗罗非鱼不同选育世代 mtDNA D-loop 区遗传多样性分析. 南方水产科学, 2015, (3): 29—34]
[40]Domingos J A, Smith-Keune C, Jerry D R. Fate of genetic diversity within and between generations and implications for DNA parentage analysis in selective breeding of mass spawners: A case study of commercially farmed barramundi (Lates calcarifer) [J]. Aquaculture, 2014, 424—425(2): 174—182
GENETIC VARIATION OF MITOCHONDRIAL DNA D-LOOP REGION IN WILD AND BREEDING POPULATIONS OF GRASS CARP
WANG Shen-Tong, ZHANG Meng, DANG Yun-Fei, LI Li-Sen, HUANG Wen-Ji, FANG Yuan, WANG Feng, SHEN Yu-Bang and LI Jia-Le
(Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources, Ministry of Education, Shanghai Ocean University, Shanghai 201306, China)
To evaluate the genetic diversity and genetic structure of grass carp (Ctenopharyngodon idella), we analyzed the genetic variation of mitochondrial DNA D-loop region among four wild populations (Hanjiang, Jiujiang, Shishou, Wujiang) and two domesticated populations after two generations (F1 and F2) of selective breeding. The results showed that the wild populations had higher number of haplotype (H), haplotype diversity (Hd), nucleotide diversity (π) and average number of nucleotide differences (K) compared with the domesticated populations. Between the bred populations, F1 generation was higher than F2 generation in the aspects of nucleotide diversity (π) and average number of nucleotide differences (K), but haplotype diversity (Hd) in F1 generation was lower than F2 generation. Haplotype analysis showed that all six populations did not share a haplotype. By contrast, the four wild populations shared two haplotypes (Hap1 and Hap3). The Shishou population and two domesticated populations shared one haplotype (Hap15). Genetic differentiation index (Fst) analysis showed that there was great genetic differences between wild (Hanjiang, Jiujiang, Wujiang) and domesticated populations (range Fstfrom were 0.41475 to 0.55128). Genetically, Shishou population was closely related with F1 population, but its relationship with F2 was only moderate. Moreover, the genetic differentiation level between two breeding populations was small (Fst=0.05741). The analysis of neighbor-joining phylogenetic trees based on 276 individuals among six populations and haplotype network graph based on 27 haplotypes indicated the wild populations (Hanjiang, Jiujiang, Wujiang) had a distant relationship with those domesticated populations. Although Shishou population and two breeding populations were more closely related. The above results showed that among the two domesticated populations, the genetic structure has already changed after two generations of selective breeding, and the genetic diversity also decreased with the development of the breeding program. The results also urge us that the current breeding strategy should be improved in order to maintain the genetic diversity of the breeding populations and reduce the risk of inbreeding.
Grass carp; Breeding population; Wild population; mt DNA; D-loop; Genetic variation
Q349+.1
A
1000-3207(2017)05-0947-09
10.7541/2017.118
2016-11-07;
2017-04-25
现代农业产业技术体系(CARS-46-04); 上海市工程中心提升项目(16DZ2281200); 通威产学研项目(TW2014F003)资助[Supported by the the China’s Agricultural Research System (CARS-46-04); the Project of Shanghai Engineering and Technology Center for Promoting Ability (16DZ2281200); Tongwei Industry-university-research Project (TW2014F003)]
王沈同(1993—), 男, 山东青岛人; 硕士; 研究方向为水产动物种质资源与遗传育种。E-mail: 2317011197@qq.com
李家乐, 教授, 博士生导师; 研究方向为水产动物种质资源与遗传育种。E-mail: jlli2009@126.com
