大肠杆菌生产莽草酸的基因工程菌构建
2017-09-12蔡灵芝李燕军谢希贤
韩 超,蔡灵芝,毛 倩,李燕军,谢希贤
(1.代谢控制发酵技术国家地方联合工程实验室,天津 300457;2.天津科技大学 生物工程学院,天津 300457)
大肠杆菌生产莽草酸的基因工程菌构建
韩 超1,2,蔡灵芝1,2,毛 倩1,2,李燕军1,2,谢希贤1,2
(1.代谢控制发酵技术国家地方联合工程实验室,天津 300457;2.天津科技大学 生物工程学院,天津 300457)
莽草酸是芳香族氨基酸合成中的重要中间代谢物,可用于多种药物的化学合成.在代谢工程理论的指导下,构建积累莽草酸的高产菌株.通过构建敲除编码莽草酸激酶的基因aroL与aroK的菌株阻断莽草酸的代谢;通过构建敲除编码奎尼酸/莽草酸脱氢酶的基因ydiB的菌株减少副产物的合成;同时通过构建人工操纵子过表达编码DAHP合酶、莽草酸脱氢酶、脱氢奎尼酸脱水酶和三脱氢奎尼酸合酶的基因aroG,aroE,aroD和aroB以强化莽草酸合成途径.最终得到E.coliSHIKΔaroLΔydiB菌株,摇瓶发酵能够积累莽草酸5.5 g/L.
莽草酸;莽草酸激酶;大肠杆菌;aroL;ydiB
莽草酸被广泛应用于生物碱、吲哚衍生物和手性药物等物质的化学合成.近年来,莽草酸因被用于合成抗禽流感药物达菲而成为研究热点[1-3].莽草酸市场价格高居不下,前景良好[4].目前,众多的研究者投身于达菲合成路线的改良,旨在提高合成效率、缓解供求紧张.
莽草酸的生产方式可分为植物直接提取法、化学合成法和微生物发酵法.对于微生物发酵法来说,微生物体内已经存在天然的莽草酸合成途径,只是产物积累较慢,且能积累莽草酸的物种有限,因而必须通过基因工程手段对微生物进行改造,提高莽草酸的积累量.Knop等[5]针对大肠杆菌开展了持续多年的系统研究,首先将莽草酸激酶aroL和aroK全部失活,再将aroB基因整合替换基因组上的serA基因位点,最后过表达aroFFBR,tktA,aroE和serA基因,莽草酸质量浓度可达52 g/L,转化率为18%.2010年,Escalante等[6]采用aroL,aroK双突变菌同时使PTS系统(phosphotransferase system)缺失,发酵产莽草酸的质量浓度为7 g/L;2014年,肖梦榕等[7]敲除大肠杆菌W3110的aroL基因,改良PTS系统并以运动假单胞菌的glf,glk基因替代PTS系统,再强化表达了aroGfbr,aroB和aroE基因,使得菌株摇瓶生产莽草酸的质量浓度为1 024 mg/L,6 L发酵罐中莽草酸的质量浓度为15.12 g/L.莽草酸基因工程菌无论是产量、转化率还是工艺研究远未达到令人满意的水平,到目前为止,国内尚无利用代谢工程菌大规模发酵生产莽草酸的相关报道.笔者以具有积累莽草酸潜力的色氨酸生产菌EscherichiacoliSHIK为出发菌株,按照代谢工程改造思路分两部分进行改造:首先将与莽草酸代谢相关的基因敲除以阻断莽草酸代谢途径,同时敲除与副产物合成相关的基因,保证碳架主要流向莽草酸;其次通过构建人工操纵子,强化莽草酸合成途径,进一步增加莽草酸的积累.
1 材料与方法
1.1 菌 种
以天津科技大学工业微生物菌种保藏室提供的色氨酸工程菌E.coliSHIK作为出发菌株.
1.2 培养基及培养条件
斜面培养基:蛋白胨10 g/L,酵母粉5 g/L,牛肉膏10 g/L,NaCl 2.5 g/L,琼脂粉30 g/L;pH 7.0,121 ℃灭菌20 min,37 ℃培养12 h为一代.
摇瓶种子培养基:葡萄糖40 g/L,酵母粉2 g/L,蛋白胨2 g/L,MgSO4·7H2O 1.6 g/L,K2HPO4·3H2O 5.6 g/L,FeSO4·7H2O 2.8 mg/L,MnSO4·H2O 1.2 mg/L,VB11.3 mg/L,pH 7.0,115 ℃灭菌15 min,37 ℃,200 r/min培养10 h.
摇瓶发酵培养基:葡萄糖8 g/L,酵母粉1.0 g/L,柠檬酸2 g/L,MgSO4·7H2O 2 g/L,K2HPO4·3H2O 7.5 g/L,FeSO4·7H2O 1 mg/L,微量元素混合溶液(钼酸铵0.28 mg/L,硼酸5 mg/L,CoCl2·6H2O 1.4 mg/L,MnSO4·H2O 0.5 mg/L,CuSO4·7H2O 0.5 mg/L,ZnSO4·7H2O 0.6 mg/L)1 mL/L,pH 7.0,115 ℃灭菌15 min,36 ℃,200 r/min培养26 h.
1.3 质粒构建方法
采用限制性内切酶分别对目的片段和目的载体进行酶切处理,利用OMEGA公司的切胶回收试剂盒进行切胶回收.用核酸分析仪检测目的片段和目的载体的浓度,从而确定目的片段和目的载体的添加量,使摩尔比为3∶1~9∶1,一共5 μL体系.在体系中加入5 μL solution I,混匀.将其置于16 ℃水浴加热1 h.将连接体系用CaCl2介导转化法转入E.coliDH5α感受态细胞中,然后将其涂布于含有相应抗生素的抗性平板中,置于37 ℃恒温培养箱中倒置培养14~16 h.挑取单菌落接种至含有相应抗生素的LB摇管中,37 ℃,200 r/min培养12 h,提取质粒进行酶切验证,从而获得目的质粒.
1.4 检测方法
发酵液中葡萄糖浓度使用SBA-40C多功能葡萄糖-谷氨酸分析仪测定.菌体量通过测定波长600 nm下的OD值来确定.莽草酸、奎尼酸和乙酸等代谢物使用HPLC测定,色谱条件为:Shodex Asahipak NH2色谱柱(5 μm,4.6 mm×250 mm);流动相V(乙腈)∶V(2% H3PO4)=80∶20;流速0.8 mL/min;测定波长213 nm,柱温40 ℃.
2 结 果
2.1 敲除aroK和aroL基因对E.coliSHIK积累莽草酸的影响
以E.coliSHIK作为出发菌株,利用RED重组[8]实现对aroK和aroL基因的敲除,将获得的菌株分别命名为E.coliSHIKΔaroK和E.coliSHIKΔaroL.以缺失aroK基因的重组子E.coliSHIKΔaroK作为出发菌株,再次利用RED重组敲除aroL基因,得到的重组子即为同时缺失aroK和aroL基因的菌株,命名为E.coliSHIKΔaroKΔaroL.
摇瓶发酵结果表明:基因aroK和aroL的缺失总体上有利于莽草酸的积累,但不同菌株的莽草酸积累量和菌体生长情况不同,结果见图1.
首先,出发菌株E.coliSHIK也能积累一定量的莽草酸(33.6 mg/L),且较野生型菌株高,适合作为本研究的出发菌.比较E.coliSHIKΔaroK和E.coliSHIKΔaroL两株单敲除菌株,由于aroL所编码的莽草酸激酶II拥有更好的底物亲和力,在莽草酸激酶的催化过程中占有绝对的领导地位,敲除aroL基因,使得莽草酸的积累量达到所有菌株的最高值(475.1 mg/L);另一方面,敲除aroK基因对莽草酸积累并无正面影响,质量浓度仅16.4 mg/L,较出发菌株低.而E.coliSHIKΔaroKΔaroL双基因敲除菌株,由于完全阻断了莽草酸的下游代谢,严重影响到下游各代谢物的合成,尤其是菌体生长所必需的3种芳香族氨基酸的合成,导致菌体生长缓慢且最终菌体量较低,虽然保持了一定的莽草酸积累(98.9 mg/L),且拥有较高的比生产速率,但由于不能实现菌体正常生长,所以并不能够成为莽草酸生产菌的优选菌株.
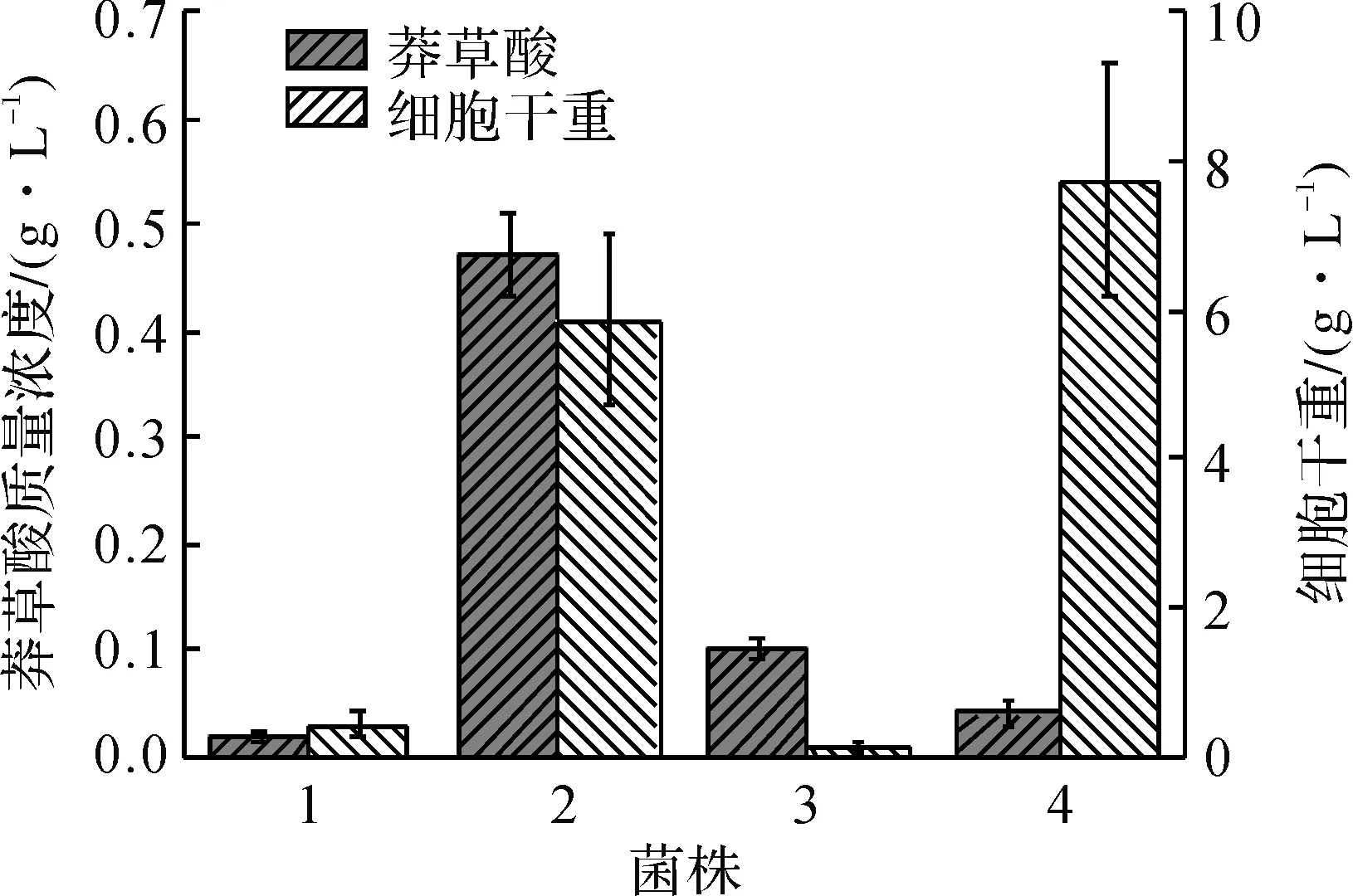
1—E. coli SHIKΔaroK;2—E. coli SHIKΔaroL;3—E. coli SHIKΔaroLΔaroK;4—E. coli SHIK图1 aroL与aroK基因敲除对莽草酸发酵的影响 Fig.1 Effects of aroL and aroK gene knockout on shikimate fermentation
2.2 过表达DAHP合成酶对积累莽草酸的影响
为了获得高效表达DAHP合成酶载体,利用拥有强启动子pTrc的常用质粒pTrc99A过表达DAHP合成酶的同工酶基因aroG,将获得的质粒命名为pTrc99A-aroG.将质粒pTrc99A-aroG转化至E.coliSHIKΔaroK,E.coliSHIKΔaroL,E.coliSHIKΔaroLΔaroK和E.coliSHIK中,以未导入质粒的菌株作为对照进行摇瓶发酵,结果见图2.
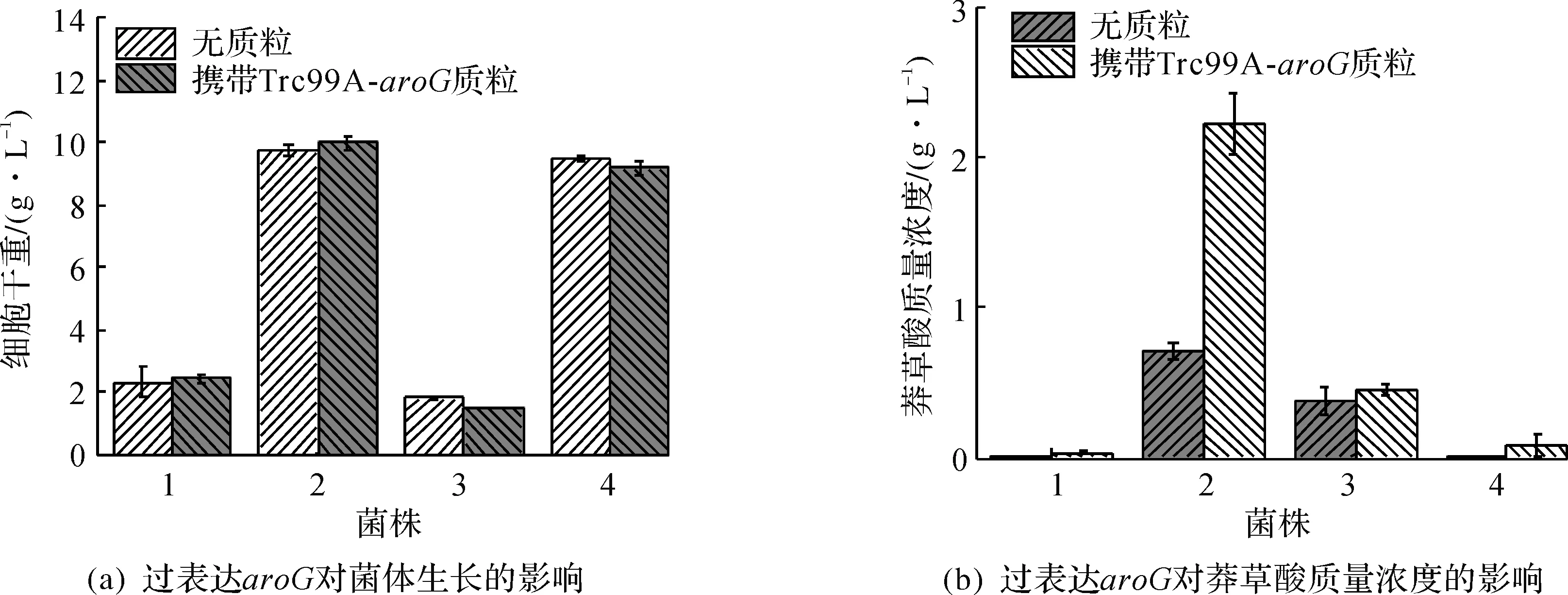
1—菌株E. coli SHIKΔaroK;2—菌株E. coli SHIKΔaroL;3—菌株E. coli SHIKΔaroLΔaroK;4—菌株E. coli SHIK 图2 E. coli SHIKΔaroK/pTrc99A-aroG,E. coli SHIKΔaroL/pTrc99A-aroG,E. coli SHIKΔaroLΔaroK/pTrc99A-aroG和E. coli SHIK/pTrc99A-aroG的发酵结果 Fig.2 Fermentation results of E. coli SHIKΔaroK/pTrc99A-aroG, E. coli SHIKΔaroL/pTrc99A-aroG, E. coli SHIKΔaroLΔaroK/pTrc99A-aroG and E. coli SHIK/pTrc99A-aroG
图2结果表明:过表达基因aroG,就生物量而言,除E.coliSHIKΔaroLΔaroK有所减少外,其他各菌株都有所提升或是保持稳定,对生物量总体影响不大;在莽草酸积累上,E.coliSHIKΔaroK,E.coliSHIKΔaroL,E.coliSHIKΔaroLΔaroK和E.coliSHIK过表达DAHP合成酶之后的产量分别较之前提高了25.73,2.67,0.39,81.25倍.因为aroG是编码DAHP合酶的基因,而DAHP合酶是莽草酸合成的前体物磷酸烯醇式丙酮酸和赤藓糖-4-磷酸合成DAHP的关键酶,所以过表达aroG基因,进一步将碳流从中枢代谢拉向莽草酸途径,所以莽草酸产量会有大幅度提高.而E.coliSHIKΔaroL/pTrc99A-aroG质量浓度达到最大2.60 g/L.
2.3 敲除ydiB基因对积累莽草酸的影响
选择敲除奎尼酸/莽草酸脱氢酶基因,是因为它的产物虽然同时具备奎尼酸脱氢酶和莽草酸脱氢酶的功能,但它对3-脱氢奎尼的亲和力更强,导致代谢流被更多地分配到奎尼酸合成途径,造成碳架的浪费.笔者在敲除aroL基因的基础上继续强化莽草酸途径,阻断支路途径消耗.利用RED重组,以E.coliSHIKΔaroL为出发菌,敲除ydiB基因,将获得的菌株命名为E.coliSHIKΔaroLΔydiB.将质粒pTrc99A-aroG转化至E.coliSHIKΔaroLΔydiB中,以E.coliSHIKΔaroL/pTrc99A-aroG和不含pTrc99A-aroG质粒的两株菌作为对照,摇瓶发酵结果见图3.
从生物量来看,菌体生长并没有受到ydiB基因缺失的影响.从莽草酸产量上,缺失ydiB基因并没有使莽草酸积累得到提高,但奎尼酸产量明显下降,甚至消失.因为ydiB基因编码的奎尼酸脱氢酶使三脱氢奎尼酸转化成奎尼酸是一个双向的过程,敲除了ydiB基因后阻断了三脱氢奎尼酸到奎尼酸的流向,同时也阻断了奎尼酸到三脱氢奎尼酸的流向,所以莽草酸产量并没有提高.导入pTrc99A-aroG质粒后,两株菌株的奎尼酸积累明显提高.

1—菌株E. coli SHIKΔaroL;2—菌株E. coli SHIKΔaroLΔydiB图3 E. coli SHIKΔaroL和E. coli SHIKΔaroLΔydiB菌株的发酵对比Fig.3 Fermentation comparison of E. coli SHIKΔaroL and E. coli SHIKΔaroLΔydiB
2.4 强化莽草酸合成途径对E.coliSHIKΔaroLΔydiB菌株积累莽草酸的影响
为了进一步强化莽草酸合成途径,笔者通过构建人工操纵子,在编码DAHP合酶的aroG基因后继续串联表达编码莽草酸脱氢酶、三脱氢奎尼酸脱水酶和三脱氢奎尼酸合酶的aroE,aroD和aroB相关基因.以质粒pTrc99A-aroG为基础,依次串联aroE,aroD和aroB基因,得到质粒pTrc99A-aroGE,pTrc99A-aroGED和pTrc99A-aroGEDB.将重组质粒转化至E.coliSHIKΔaroLΔydiB菌株,摇瓶发酵结果见图4.

YG—E. coli SHIKΔaroLΔydiB菌株携带pTrc99A-aroG质粒;YGE—E. coli SHIKΔaroLΔydiB菌株携带pTrc99A-aroGE质粒;YGED—E. coli SHIKΔaroLΔydiB菌株携带pTrc99A-aroGED质粒;YGEDB—E. coli SHIKΔaroLΔydiB菌株携带pTrc99A-aroGEDB质粒 图4 E. coli SHIKΔaroLΔydiB菌株表达重组质粒的发酵结果 Fig.4 Fermentation results of E. coli SHIKΔaroLΔydiB strains express the recombinant plasmids
从生物量来看,质粒负担对菌体的影响并不大,各菌株生长情况没有明显差异.在莽草酸积累方面,产量随着过表达基因数目的增加而增加,分别达到1.14,2.33,2.88,5.51 g/L.说明在阻断支路代谢的基础上,进一步强化莽草酸代谢途径确实有利于莽草酸的积累.
3 结 论
根据代谢工程原理,初步构建了L-莽草酸基因工程菌株,并具备一定的产酸能力.通过敲除莽草酸激酶编码基因aroK,aroL与奎尼酸/莽草酸脱氢酶编码基因ydiB,成功构建重组菌株E.coliSHIKΔaroLΔydiB,一定程度上减少奎尼酸积累,但不能彻底解除奎尼酸的积累,菌体内还存在其他酶能够催化3-脱氢奎尼酸转化成奎尼酸.笔者采用构建人工操纵子的方式,对莽草酸合成途径的4个关键基因进行组合,确定了最佳组合方案,证实强化莽草酸途径代谢有助于莽草酸的积累.后续研究可尝试进一步对莽草酸途径进行强化,如穿插启动子进行模块化改造,避免操纵子结构过长带来的影响.
DAHP合成酶编码基因无论是aroG还是aroF都可通过定点突变解除代谢反馈抑制,提高酶活.另外,将其他菌株的DAHP合成酶编码基因进行异源表达也是可行方案.
[1] ROHLOFF J C, KENT K M, POSTICH M J, et al. Practical total synthesis of the anti-influenza drug GS-4104[J].The journal of organic chemistry, 1998, 63(13):4545-4550.
[2] FEDERSPIEL M, FISCHER R, HENNIG M, et al. Industrial synthesis of the keyprecursor in the synthesis of the anti-influenza drug oseltamivir phosphate (Ro 64-0796/002GS-4104-02: ethyl (3R,4S,5S)-4, 5-epoxy-3-(1-ethyl-propoxy)-cyclohex-1-ene-1-carboxylate[J]. Organic process research & development, 1999, 3(4):266-274.
[3] BECKER M W, CHAPMAN H H, KELLY D E, et al. Manufacture of cyclohexenecarboxy-late esters and intermediates: WO9955664[P]. 1999-11-14.
[4] 聂良邓.以莽草酸为起始原料合成磷酸奥司他韦(达菲)及其类似物[D].上海:华东理工大学,2012.
[5] KNOP D R, DRATHS K M, CHANDRAN S S, et al. Hydroaromatic equilibration during biosynthesis of shikimic acid[J]. Journal of the American chemical society, 2001, 123(42):10173-10182.
[6] ESCALANTE A, CALDERN R, VALDIVIA A, et al. Metabolic engineering for the production of shikimic acid in an evolvedEscherichiacolistrain lacking the phosphoenolpyruvate: carbohydrate phosphotransferase system[J]. Microbial cell factories, 2010, 9(1):21.
[7] 肖梦榕,张梁,石贵阳.改进莽草酸合成代谢的大肠杆菌工程化研究新进展[J].微生物学报,2014,54(1):5-13.
[8] 张雪,温廷益.Red重组系统用于大肠杆菌基因修饰研究进展[J].中国生物工程杂志,2008,28(12):89-93.
(责任编辑:朱小惠)
Construction of genetic engineering strains for production of shikimic acid byEscherichiacoli
HAN Chao1,2, CAI Lingzhi1,2, MAO Qian1,2, LI Yanjun1,2, XIE Xixian1,2
(1.National and Local United Engineering Lab of Metabolic Control Fermentation Technology, Tianjin 300457, China; 2.College of Biotechnology, Tianjin University of Science and Technology, Tianjin 300457, China)
Shikimic acid is an important intermediate for aromatic amino acids. It can be used as building block for the chemical synthesis of various drugs. In this study, a high-yield shikimic acid producing strain was constructed through metabolic engineering. The genesaroLandaroKencoding the shikimate kinase were knocked out to block the shikimic acid catabolism and the geneydiBencoding the quinic/shikimate dehydrogenase was deleted to reduce the synthesis of byproduct. ThearoG,aroE,aroDandaroBgenes encoding DAHP synthase, shikimic acid dehydrogenase, dehydroquinic acid dehydratase and dehydroquinic acid synthase were overexpressed with introduction of constructed operons, to strengthen the shikimic acid synthesis pathway. Finally, the obtainedE.coliSHIKΔaroLΔydiBengineering strain resulted in a yield of shikimic acid of 5.5 g/L by shake flask fermentation.
shikimate; shikimate kinase;Escherichiacoli;aroL;ydiB
2017-05-03
韩 超(1993—),男,山东潍坊人,硕士研究生,研究方向为用谷氨酸棒杆菌代谢改造莽草酸途径,E-mail:871407470@qq.com.
Q78
A
1674-2214(2017)03-0143-04
