新疆哈萨克族15个常染色体STR位点检测及40个中国人群遗传关系*
2017-06-07王文菲吕自力钟明霞贾馨怡曾昭书
王文菲,吕自力,张 勇,钟明霞,贾馨怡,曾昭书
1)安徽省阜阳市公安局刑科所 安徽阜阳 236000 2)郑州大学基础医学院法医学教研室 郑州 450001 3)成都中医药大学第二附属医院生殖医学中心 成都 610075 4)乌鲁木齐市妇幼保健院检验科 乌鲁木齐 830001 5)山西运城护理职业学院 山西运城 044000 6)郑州大学法学院 郑州 450001
新疆哈萨克族15个常染色体STR位点检测及40个中国人群遗传关系*
王文菲1,2),吕自力3,4),张 勇4),钟明霞2,5),贾馨怡6),曾昭书2)#
1)安徽省阜阳市公安局刑科所 安徽阜阳 236000 2)郑州大学基础医学院法医学教研室 郑州 450001 3)成都中医药大学第二附属医院生殖医学中心 成都 610075 4)乌鲁木齐市妇幼保健院检验科 乌鲁木齐 830001 5)山西运城护理职业学院 山西运城 044000 6)郑州大学法学院 郑州 450001
#通信作者,男,1973年5月生,博士,教授,研究方向:法医分子遗传学,E-mail:zzs@zzu.edu.cn
法医学;常染色体;STR;遗传距离;中国人群
目的:对新疆哈萨克族进行群体遗传学数据调查,并将其结果用于40个中国人群间的遗传距离和遗传关系分析。方法:检测新疆哈萨克族人群血样中15个常染色体STR位点;收集公开发表的中国多省份、多民族、多人群相应的15个常染色体STR频率数据,以Phylip 3.69及Arlequin 3.5统计软件包计算40个族群之间的Nei's遗传距离(Rst值),并以Neighbor-Joining法构建40个族群之间的系统发生树。结果:在新疆哈萨克族中,除TPOX以外,其他14个STR的He、PIC均在0.7以上。全部40个人群之间的Rst均值为0.055 1,其中18个汉族人群之间的Rst均值为0.044 9,22个少数民族间的Rst均值为0.057 6。从系统发生树中发现汉族分支多且愈来愈多样化,我国境内一些距离稍远的少数民族之间有一定的聚合。结论:法医学常用的15个常染色体STR在新疆哈萨克族中具有高度遗传多态性。
短串联重复序列(short tandem repeats,STR)也被称为微卫星DNA(microsatellite DNA),属于第2代遗传标记,具有分布广、信息量大、高度多态性以及遵循孟德尔共显性遗传规律等特点。目前法医学界广泛应用以下15个常染色体STR位点进行个体识别和亲权鉴定:TH01、D7S820、CSF1PO、D5S818、TPOX、D13S5317、D8S1179、D16S539、D18S51、D19S433、VWA、D13S1358、D2S1338、FGA、D21S11[1-3]。该研究对新疆哈萨克族人群这15个STR位点进行了检测,并收集了全国其他多人群的相关数据,进行了人群间遗传距离和遗传关系的推断。
1 资料与方法
1.1 新疆哈萨克族数据来源 在新疆境内居住3代或以上的哈萨克族人群中随机选取健康无关个体100人(男30人,女70人),每例采取静脉血2 mL,枸橼酸钠抗凝。
1.2 其他人群数据来源 通过检索近年来期刊上公开发表的频率资料,收集中国境内各人群文献资料51份,每份至少含有9个STR位点资料、样本量至少有90人。对包括该研究哈萨克族在内的52个人群频率资料[4-50]构建系统发生树,对系统发生树中相距较近的同一来源分支进行合并,最终确定40个代表人群用于遗传关系分析。同一地区同一人群多份文献资料的合并方法为:对多个人群的基因频率数据用加权平均法进行合并作为该地区人群的最终数据,合并公式为pi=∑pijnj/∑nj,其中,pi为合并后人群的第i个等位基因的频率,pij为第j个相同人群的第i个等位基因的频率,nj为第j个相同人群的样本数。40个人群资料信息见表1。

表1 40个人群资料信息
1.3 STR 扩增及分型 对新疆哈萨克族100人的血样样品采用AmpFLSTR Identifiler PCR直接扩增试剂盒[51],在AB 9700型扩增仪上对15个STR进行复合扩增,在AB 3130遗传分析仪上进行分型。使用Data Collection 1.0软件收集数据,Gene Mapper ID 3.2软件进行基因型分析,以Modified-powerstats软件对分型结果进行处理,获得各个位点的等位基因频率、杂合度(He)、个体识别率(PD)、匹配概率(MP)、非父排除率(PE)、多态信息含量(PIC)、父权指数(PI)。
1.4 遗传距离分析及系统进化树构建 选择各人群数据中均包含的9个位点(D13S5317、D18S51、D21S11、D3S1358、D5S818、D7S820、D8S1179、FGA、vWA),用以比较40个族群间的分布特征。使用Phylip 3.69及Arlequin 3.5统计软件包计算40个群体之间的Nei's遗传距离(Rst值)[52],采用Neighbor-Joining法构建40个族群之间的系统发生树[53]。
2 结果
2.1 新疆哈萨克族个体的15个常染色体STR的基因多态性 无关新疆哈萨克族个体共100人的15个常染色体STR等位基因频率及He、PD、MP、PE、PIC、PI等见表2。
2.2 遗传距离分析及系统进化树 全部40个人群之间的Rst均值为0.055 1;最大值为0.257 0,为广西壮族和内蒙古达斡尔族之间;最小值为0.003 2,为福建汉族和湖南土家族之间。其中18个汉族人群之间的Rst均值为0.044 9,22个少数民族间的Rst均值为0.057 6。以9个STR位点为标尺时,长江以南汉族(含湖北汉族、江苏汉族、江西汉族、浙江汉族、重庆汉族、四川汉族、福建汉族、广东汉族、广西汉族、海南汉族)之间的Rst均值为0.030 6,长江以北汉族(含辽宁汉族、天津汉族、内蒙古汉族、河北汉族、山东汉族、陕西汉族、河南汉族、安徽汉族)之间的Rst均值为0.049 1。40个族群之间的系统发生树见图1。

表2 新疆哈萨克族人群15个常染色体STR等位基因频率及多态性信息
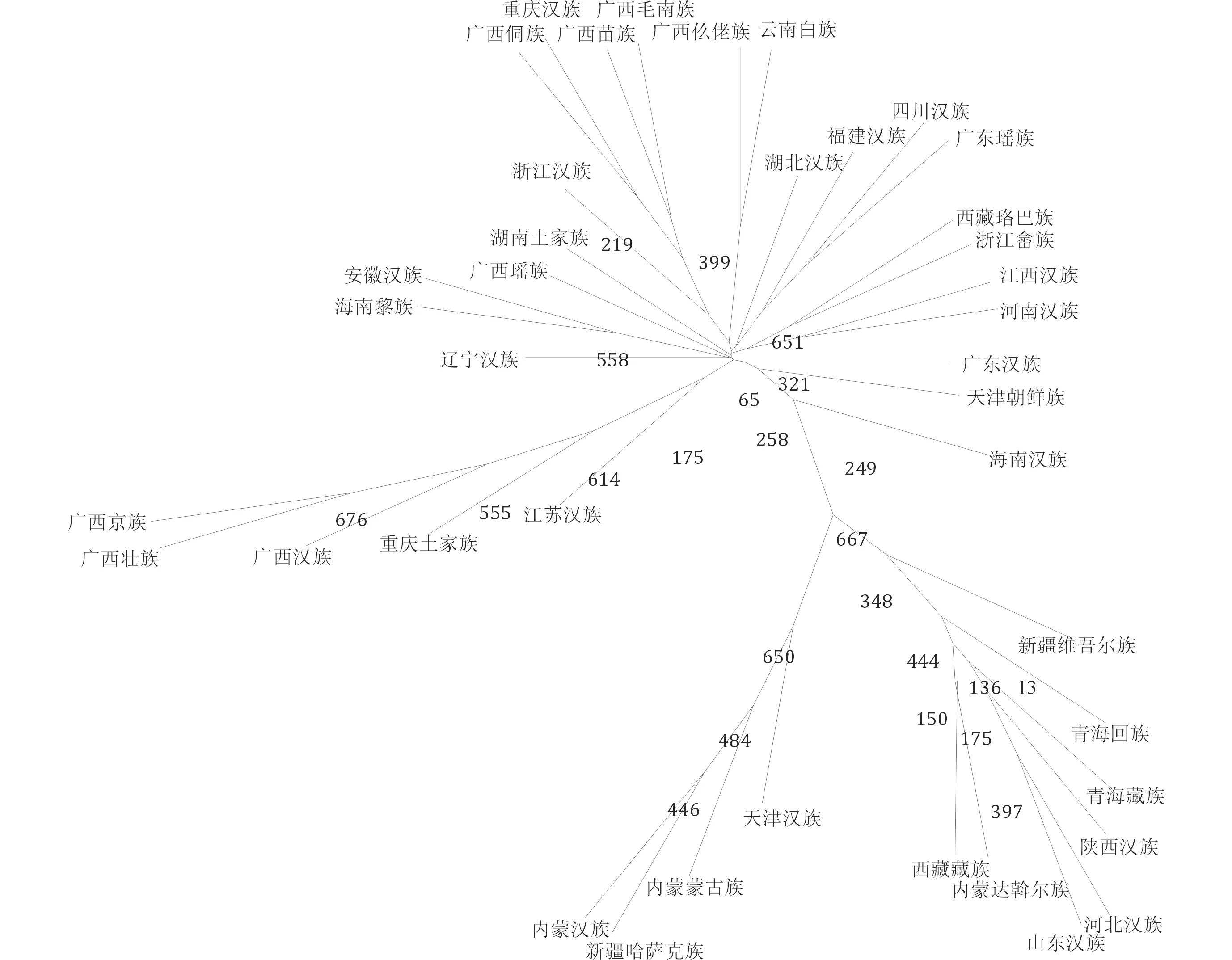
图1 通过Neighbor-Joining法构建的40个中国人群遗传距离系统发生树
3 讨论
我国是一个多民族的国家,研究各民族间的遗传关系历来是一个重要课题,应用STR遗传标记一直是该领域的重要方法。一般认为STR是用于研究关系较近群体的理想遗传标记[54]。通过研究STR多态性,变异速率以及比较序列间差异、人群间差异,分析不同人群间的遗传距离,可从分子生物学角度揭示人类的起源、迁徙、进化等历史进程。俞建昆等[55]曾利用30个常染色体微卫星位点研究了6个人群的遗传结构,发现纳西族、撒拉族和土族聚成一簇,汉族、白族聚成一簇,畲族单独为一枝。但是由单个课题组收集多个人群的样品,进行分型检测时所需花费多于投入,研究费力费时。因此,通过收集已发表的相关数据进行分析可使研究省时省力。
遗传距离为表示群体间或物种间遗传差异或遗传分化的最重要参数。研究群体间遗传差异程度以及族群间遗传分化程度,最常使用两个指标:Fst或Rst[56]。Fst一般多用于基因间等位基因状态的相关性,而Rst则多用于基因间等位基因大小的相关性。另外,Rst假设等位基因的突变符合渐变模式,而Fst未有相关假设。在基因流动比较高的情况下Fst更具有优势,而Rst在低基因流动的情况下能够发挥更好的作用。因此针对STR位点而言,作者选取Rst为遗传距离的指标更为合适。
该研究结果显示,18个汉族人群之间的Rst均值为0.044 9,小于22个少数民族间的Rst均值0.057 6,也小于全部40个人群之间的Rst均值0.055 1,说明汉族虽然分为南北汉族,但汉族之间的遗传距离总体小于汉族和其他民族之间的遗传距离。以该套STR位点为标尺时,长江以南汉族(含湖北汉族、江苏汉族、江西汉族、浙江汉族、重庆汉族、四川汉族、福建汉族、广东汉族、广西汉族、海南汉族)之间的Rst均值为0.030 6,长江以北汉族(含辽宁汉族、天津汉族、内蒙古汉族、河北汉族、山东汉族、陕西汉族、河南汉族、安徽汉族)之间的Rst均值为0.049 1,说明该研究取样范围内北方汉族间的遗传距离要大于南方汉族之间的遗传距离,也说明华南汉族之间较为一致。2007年龙友国等[57]以3个STR基因座对17个汉族的遗传关系进行了研究,发现河南、江西、安徽、台湾和深圳汉族等聚为一大类,荆州、成都等汉族为一类,而广西汉族单独为一类,这些结论基本与该研究该研究结果一致。Huang等[58]对汉族的Y-SNPs单倍型进行检测后发现,O型及其各亚型在南方汉族中频率均值为76.79%,在北方汉族中频率均值为61.80%,这也说明南方汉族比北方汉族的组分更为一致。该研究的结果与其有一致之处。Wen等[59]认为,汉文化的扩散源于汉族从北向南的人口扩张;几乎所有的汉族群体的Y 染色体单倍群分布都极为相似,Y 染色体主成分分析也把几乎所有的汉族群体都集合成一个紧密的聚类;北方汉族对南方汉族的遗传贡献无论父系方面还是母系方面都是可观的。
该研究观察到壮族、黎族、侗族、仫佬族、毛南族、仡佬族等少数民族遗传距离较近,实际上这些民族也同属于同一语族——南方壮侗语族。作者观察到达斡尔族和壮族之间遗传距离最远,解释如下:从地理分布上讲,两者之间地理距离遥远,达斡尔族主要分布在内蒙古自治区、黑龙江,少数居住在新疆、辽宁,而壮族主要位于广西;从语言上讲,两者分属不同语族,达斡尔族属于阿尔泰语系中的蒙古语族,而壮族属于汉藏语系中的壮侗语族。该研究现发现该两者之间的遗传距离在40个人群间最大,此结果以前少有报道。该研究还发现,福建汉族和湖南土家族之间遗传距离最小,可能是因为缺乏湖南汉族数据的缘故,因为存在着湖南汉族和湖南土家族之间遗传距离更小的可能性。
作者选择采用Neighbor-Joining法构建系统发生树,因为如果群体间存在着基因交流,用Neighbor-Joining法构建系统发生树比用UPGMA法所受的影响要小[60]。该树状图结果有以下特点:一是汉族呈现愈来愈多样化的趋势。在各民族通婚的背景下,某些区域的汉族与当地少数民族已经有融合的趋势,如内蒙汉族与新疆哈萨克族遗传距离较近,再与内蒙蒙古族、天津汉族聚合在一起;广西汉族与广西京族、壮族遗传距离较近,再与重庆土家族、江苏汉族等聚合。二是一些距离稍远的少数民族之间存在一定的聚合趋势,如浙江畲族与西藏珞巴族聚合在一起,湖南土家和广西瑶族聚合在一起,广西毛南族与广西苗族聚合在一起,广西仫佬族与云南白族聚合在一起。从该发生树结果来看,汉族基本上分为南北两大群,北群包括在北方居住的汉族,南群包括在南方居住的汉族;但是河南汉族与安徽汉族出现于南方群,有待纳入同一地域的更多分型结果进行校正。同样,该研究观察到汉族与当地的少数民族的融合趋势,既可能是汉族人与当地少数民族基因交流的结果,也可能是取样过程中少数民族样品中混有汉族人的血液。
综上所述,根据40个人群的9个常色体STR遗传标记计算的遗传距离结果与它们的地理分布和民族历史记载基本一致,能够比较全面地反映我国多民族间的遗传关系和近期以来的基因漂变。当然,要阐明这些群体间的遗传结构,需要增加研究位点数,特别是与Y-SNPs分型、线粒体DNA分型进行互补[60],才能提高分辨率,更加全面地揭示出各个人群的进化历史以及群体间的亲缘关系。
[1]GILL P,HANED H,BLEKA O,et al.Genotyping and interpretation of STR-DNA:low-template, mixtures and database matches-twenty years of research and development[J].Forensic Sci Int Genet,2015,18:100
[2]石美森,百茹峰,张金生,等.辽宁满族11个Y-STR基因座多态性及遗传关系的分析[J].遗传,2008,30(5):583
[3]王文菲.法医学常用15个STRs的检测及其应用于人群遗传关系推断的研究[D].郑州:郑州大学,2012.
[4]梁晓华,于卫建,胡荣花.辽宁汉族人群9个短串联重复序列基因座的遗传多态性[J].临床输血与检验,2005,7(3):192
[5]刘孟男,匡金枝,聂同钢,等.天津地区朝鲜族9个STR基因座遗传多态性[J].刑事技术,2004(4):23
[6]匡金枝,朱巍,聂同钢,等.天津地区汉族群体9个STR基因座的频率调查[J].中国法医学杂志,2002,17(3):151
[7]顾明波,李晓平,杜庆新,等.中国内蒙古达斡尔族15个STR基因座的多态性[J].中国法医学杂志,2004,19(3):166
[8]张贵芹,裴黎,马原,等.内蒙古汉族群体15个STR基因座遗传多态性[J].中国法医学杂志,2008,23(4):265
[9]顾明波,李晓平,杜庆新,等.中国布里亚特蒙古族人群15个STR基因座的遗传多态性[J].中国法医学杂志,2004,19(4):229
[10]张艳霞,李爱强,赵丽,等.河北地区汉族群体19个STR基因座的遗传多态性[J].刑事技术,2011(2):48
[11]裴黎,张贵芹,丛斌,等.河北汉族人群15个STR基因座的遗传多态性[J].河北医科大学学报,2008,29(3):455
[12]迟晓云,焦淑贤,胡彬,等.青岛地区汉族群体15个STR基因座遗传多态性[J].中国法医学杂志,2008,23(5):339
[13]黄磊,吕桂平,姚桂法,等.山东地区人群9个STR基因座遗传多态性及频率调查[J].刑事技术,2002(4):23
[14]吴元明,张晓楠,邹游,等.陕南地区汉族人群9个STR基因座遗传多态性研究[J].第四军医大学学报,2008,29(5):388
[15]王占海,郭燕霞,刘开会,等.青海地区藏族人群9个STR基因座的遗传多态性[J].中国法医学杂志,2004,19(2):100
[16]郭燕霞,王占海,刘开会,等.青海地区回族人群9个STR基因座的遗传多态性[J].中国法医学杂志,2003,18(4):244
[17]宋兴勃,陈鹏宇,应斌武,等.中国新疆维吾尔族群体15个STR基因座的遗传多态性[J].法医学杂志,2008,24(5):349
[18] 匡金枝, 张颖, 阿不都艾尼. 新疆维吾尔族15个STR基因座的遗传多态性[J].刑事技术,2004(3):14
[19]李宁,苏玉虹,席焕久,等.拉萨市藏族人群15个STR基因座多态性的研究[J].遗传,2006,28(11):1361
[20]李宁,苏玉虹,席焕久,等.西藏藏族人群15个短串联重复序列基因座的遗传多态性[J].遗传,2008,30(7):851
[21]李宁,苏玉虹,席焕久,等.西藏那曲地区藏族人群15个短串联重复序列位点多态性的研究[J].人类学学报,2007,26(1):70
[22]袁东亚,康龙丽,赵健民,等.西藏珞巴族15个STR位点遗传多态性研究[J].西安交通大学学报(医学版),2006,27(4):319
[23]ZENG ZS,ZHENG XD,ZHU YL,et al.Population genetic data of 15 STR loci in Han population of Henan province (central China)[J].Leg Med (Tokyo),2007,9(1):30
[24]杨玉玲,王冬花,陈玲,等.安徽汉族人群15个STR基因座遗传多态性[J].法医学杂志,2007,23(1):36
[25]李霞,周新,李杰,等.湖北汉族群体9个STR位点遗传多态性分析[J].武汉大学学报(医学版),2003,24(4):307
[26]易少华,杨荣芝,梅焜,等.湖北汉族人群15个STR基因座的遗传多态性[J].华中科技大学学报(医学版),2010,39(1):91
[27]王翔,蔡继峰,钟鸣,等.湖南石门土家族人群15个STR基因座遗传多态性[J].法医学杂志,2010,26(3):214
[28]徐庆,高金林,裴军昌,等.江苏泰州地区汉族人群9个STR基因座的遗传多态性[J].刑事技术,2008(2):59
[29]王军,邢锦山.连云港地区汉族人群15个STR基因座的遗传多态性[J].苏州大学学报(医学版),2010,30(2):333
[30]孙耀东,李健,陈月勇,等.盐城地区汉族人群15个STR基因座的遗传多态性[J].中国法医学杂志,2006,21(4):235
[31]周文斌,裴君.江西汉族人群15个STR基因座的多态性调查[J].法律与医学杂志,2006,13(1):53
[32]陈雁,朱宇宁,吕时铭,等.浙江省汉族人群18-STR基因座的分型资料及其应用[J].中山大学学报(医学科学版),2010,31(1):122
[33]张幼芳,李佑英,兰樟彩,等.浙江畲族人群9个STR基因座的遗传多态性[J].刑事技术,2003(4):22
[34]黄霞,王芳,王蓉感,等.重庆地区汉族人群15个STR基因座遗传多态性研究[J].重庆医学,2008,37(1):58
[35]邓世雄,陈伟.重庆土家族群体15个STR基因座的遗传多态性[J].重庆医科大学学报,2009,34(11):1499
[36]宋兴勃,范红,李永生,等.四川汉族人群15个短串联重复序列基因座遗传多态性分析[J].中国输血杂志,2009,22(12):977
[37]程祖建,杨滨,林寿榕,等.福建汉族人群STR位点遗传多态性分析及其在亲子鉴定中的应用[J].福建医药杂志,2005,27(1):117
[38]吴德清,胡盛平,刘静文,等.闽南地区汉族人群15个STR基因座遗传多态性调查[J].中国法医学杂志,2005,20(2):101
[39]赖江华,张保华,朱波峰,等.云南白族STR遗传多态性研究[J].西安医科大学学报,2002,23(3):242
[40]刘超,朱少建,汪萍,等.广东广西地区5个群体9个STR基因座的频率调查[J].中国法医学杂志,2002,17(5):263
[41]吴小洁,孙华明,刘超,等.广东省瑶族人群15个STR基因座的多态性调查[J].刑事技术,2005(2):6
[42]杨电,刘超,朱少建,等.广西地区京族人群9个STR基因座遗传多态性[J].中国法医学杂志,2002,17(3):153
[43]周丽宁,李松峰,徐林,等.广西环江县毛南族九个短串联重复序列基因座的遗传学分析[J].人类学学报,2007,26(3):264
[44]徐林,李松峰,邓琼英,等.广西毛南族群体15个短串联重复序列的遗传学研究[J].广西医科大学学报,2007,24(2):180
[45]周丽宁,覃耀春,徐林,等.FGA等15个短串联重复序列在融水县苗族群体中的遗传学分析[J].解剖学报,2008,39(5):683
[46]刘超,杨电,刘长晖,等.广西苗族人群15个STR基因座的多态性调查[J].法医学杂志,2003,19(4):204
[47]杨电,刘长晖,朱少建.广西地区3个群体15个STR基因座频率多态性[J].中国法医学杂志,2002,17(S1):24
[48]高放,毕世华,赖江华,等.中国瑶族人群(广西)9个STR基因多态性研究[J].遗传,2002,24(5):537
[49]唐秋萍,杨向萍,章雅清.海南地区汉族15个STR基因座群体遗传学研究[J].中国输血杂志,2010,23(6):445
[50]金鑫,姜成涛,涂政,等.海南黎族群体14个STR基因座遗传多态性[J].中国法医学杂志,2007,22(6):403
[51]YANG C,BA H,YU H,et al.Association of 15 short tandem repeats loci with aggressive behavior[J].Zhonghua Yi Xue Yi Chuan Xue Za Zhi,2014,31(1):101
[52]KUANG WJ,HUANG P,TUO Y,et al.Genetic differentiation and patterns of gene flow of ten minorities in Yunnan province[J].Fa Yi Xue Za Zhi,2011,27(2):112
[53]SAITOU N,NEI M.The neighbor-joining method:a new method for reconstructing phylogenetic trees[J].Mol Biol Evol,1987,4(4):406
[54]谢兆辉.人类STR标记的研究进展[J].德州学院学报,2005,21(6):40
[55]俞建昆,褚嘉档,钱亚屏,等.应用30个常染色体STR位点研究中国6个民族群体的遗传关系[J].遗传学报,2001,28(8):699
[56]范浩.应用STR位点研究中国西、南部28个民族群体族源关系[D].昆明:昆明医学院,2006:43
[57]龙友国,邱祥智,龙思方,等.中国17个汉族人群3个STR基因座的遗传关系研究[J].右江民族医学院学报,2007,29(3):336
[58]HUANG H,SU M,LI X,et al.Y-chromosome evidence for common ancestry of three Chinese populations with a high risk of esophageal cancer[J].PLoS One,2010,5(6):e11118
[59]WEN B,LI H,LU D,et al.Genetic evidence supports demic diffusion of Han culture[J].Nature,2004,431(76):302
[60]WANG CC,LI H.Inferring human history in East Asia from Y chromosomes[J].Investig Genet,2013,4(1):11
(2016-07-02收稿 责任编辑李沛寰)
Analysis of 15 autosomal STR among Xinjiang Kazaks and the inference of genetic relationship among 40 Chinese populations based on the 15 STRs
WANGWenfei1,2),LYUZili3,4),ZHANGYong4),ZHONGMingxia2,5),JIAXingyi6),ZENGZhaoshu2)
1)InstituteofCriminalSciences,FuyangBureauofPublicSafety,Fuyang,Anhui236000 2)DepartmentofForensicMedicine,CollegeofBasicMedicalSciences,ZhengzhouUniversity,Zhengzhou450001 3)TheSecondAffiliatedHospital,ChengduUniversityofTraditionalChineseMedicine,Chengdu610075 4)TheMaternalandChildHealthHospitalofUrumqiCity,Urumqi830001 5)YunchengNursingVocationalCollege,Yuncheng,Shanxi044000 6)LawSchool,ZhengzhouUniversity,Zhengzhou450001
forensic medicine;autosome;STR;genetic distance;Chinese population
Aim: To investigate the population genetic data of Xinjiang Kazaks and to analyze the genetic distance and genetic relationship among 40 Chinese populations including Kazaks. Methods: The 15 autosomal STR loci in Xinjiang Kazaks samples were analyzed; corresponding frequency data of 15 STR loci of other Chinese populations was collected from published literatures. The genetic distance(Rst) among the 40 populations was calculated by Phylip 3.69 and Arlequin 3.5, and the phylogenetic tree was constructed with Neighbor-Joining method. Results: All the other 14 STRs except TPOX had high He and PIC values more than 0.7 in Xinjiang Kazaks. The mean Rst among the 40 populations was 0.055 1, the mean Rst was 0.044 9 among the 18 Han populations, and the mean Rst was 0.057 6 among the other 22 minorities. In the tree graph, the Han nationality displayed a lot of branches, showing a trend of more and more diversity; some of the minorities gathered together although they were geographically distant. Conclusion: The 15 autosomal STR are highly polymorphic in Xinjiang Kazaks and the 15 STR frequency data published by the forensic science community can be conveniently used for the study of human evolution, ethnic origin and ethnic group relationships.
10.13705/j.issn.1671-6825.2017.03.009
*国家自然科学基金资助项目 31071100;河南省教育厅高等学校重点科研项目 15A180021
D919.4
