一株牛源肺炎克雷伯氏菌的分离鉴定与耐药基因型检测
2017-04-26蒙正群冷依伊任梅渗刘亚东姚学萍杨泽晓
蒙正群,冷依伊,任梅渗,刘亚东,王 印,2,姚学萍,杨泽晓,*
(1.四川农业大学 动物医学院,四川 温江 611130; 2.动物疫病与人类健康四川省重点实验室,四川 温江 611130)
一株牛源肺炎克雷伯氏菌的分离鉴定与耐药基因型检测
蒙正群1,冷依伊1,任梅渗1,刘亚东1,王 印1,2,姚学萍1,杨泽晓1,*
(1.四川农业大学 动物医学院,四川 温江 611130; 2.动物疫病与人类健康四川省重点实验室,四川 温江 611130)
从病死犊牛肺脏组织中分离到一株革兰阴性短杆菌,进行了细菌分离纯化、生化试验、16SrDNA测序鉴定、小鼠致病性试验、药敏试验,及碳青霉烯类酶与产超广谱β-内酰胺酶(ESBLs)等7种耐药基因的PCR检测。结果鉴定该菌为肺炎克雷伯氏菌,其16S rDNA扩增序列与GenBank中肺炎克雷伯氏菌同源性达99%。对小鼠致病性强。该菌分离株对氨苄西林、头孢呋辛、复方新诺明、红霉素耐药,而对碳青霉烯类、第四代头孢等受试药物均为敏感。耐药基因PCR检测仅扩增出约862 bp的SHV基因片段,与公布的SHV-1基因序列同源性达99.3%。该牛源克雷伯氏菌分离株耐药表型与耐药基因检测结果一致。
肺炎克雷伯氏菌;分离鉴定;耐药基因
肺炎克雷伯氏菌为肠杆菌科克雷伯氏菌属的条件致病性革兰阴性杆菌。该菌在人和动物肠道、呼吸道,以及外界环境中广泛存在,当人和动物机体免疫力下降时,可引起肺炎、呼吸道感染、泌尿系统感染、腹膜炎,甚至菌血症、败血症等[1]。近年来,该病在全世界范围内广泛流行,耐药性严重,且多为多重耐药,在大多数研究报告中该菌对碳青霉烯类,β-内酰胺类抗生素具有耐药性,对碳青霉烯类的耐药性主要是产生碳青霉烯酶(Klebsiellapneumoniaecarbapenemases, KPC),同时产超广谱β-内酰胺酶,导致其对β-内酰胺类抗生素产生了广泛的耐药性,从而为该病的治疗造成了很大的困难,在临床上引起了高度的关注[2-5]。
2016年3月,四川温江区某牛场送检一头病死30日龄犊牛,主述出现发热、食欲减退症状,第2天呼吸道症状加重,经磺胺类药物治疗未见好转,次日死亡。临床检查无明显症状,解剖可见肺脏出现水肿和肉变区,其他组织器官未见明显病变,疑似为克雷伯氏菌肺炎。为此,本试验采用常规微生物学实验技术,通过纯化分离、生化试验、16S rDNA扩增测序、动物试验、药敏试验及7种耐药基因的PCR扩增分析,对临床送检疑似克雷伯氏菌感染的病死犊牛肺脏组织中细菌进行了检测,以期为临床上肺炎克雷伯氏菌病的防治与耐药性相关研究提供参考。
1 材料与方法
1.1 材料
1.1.1 主要试剂和仪器
营养琼脂培养基、麦康凯琼脂培养基、微量生化反应管、碳青霉烯类抗生素药敏纸片、头孢吡肟、头孢匹罗、头孢噻吩、四环素、萘啶酸、呋喃妥因、氨曲南购自温州市康泰生物科技有限公司,其他药敏纸片购自杭州微生物试剂有限公司。2×Taq PCR MasterMix、凝胶回收试剂盒、大肠埃希菌感受态DH5α等均购自天根生化科技(北京)有限公司。头孢菌素β-内酰胺酶(AmpC酶)、DHA-1阳性猪肺炎克雷伯氏菌系本实验室保存,pMD19-T载体购自宝生物(大连)有限公司。
1.1.2 病料和试验动物
病死犊牛肺脏组织来自四川温江某牛场;健康小鼠购自四川大学华西实验动物中心。
1.1.3 引物
参照文献[6-8],设计细菌16S rDNA 基因通用引物P1/P2,常见耐受碳青霉烯类药物的耐药基因KPC、OXA-48、NDM-1和引起耐受青霉素、单酰胺类(氨曲南)、β-内酰胺类抗生素相关酶的耐药基因CTX-M1、TEM、SHV、DHA-1等7种耐药基因的PCR扩增引物P3~P16(表1),由上海生工生物工程技术服务有限公司合成,按照说明书稀释至10 μmol·L-1,-20 ℃保存备用。
表1 试验用引物序列
Table 1 Primer sequences used in this experiment

基因名称Genename引物序列Primersequence(5’-3’)片段长度Productslength/bp扩增基因名称Amplifiedgenename16SrDNAP1:AGTTTGATCCTGGCTCAG1500细菌鉴定P2:TTACCTTGTTACGACTBacterialidentificationKPCP3:ATCGCCGTCTAGTTCTGC638碳青霉烯酶P4:GTGTTTCCCTTTAGCCCarbapenemaseOXA-48P5:GGTGGCATCGATTATCGG743碳青霉烯酶P6:GCACTTCTTTTGTGATGGCCarbapenemaseDHA-1P7:CTTTCACAGGTGTGCTGGGT405产头孢菌素β-内酰胺类酶P8:GTACGCATACTGGCTTTGCAmpCbeta-lactamaseCTX-M1P9:GCGCTTTTGCCGTCGTCTAAG944产超广谱β-内酰胺类酶P10:CCATGGTTAAAAAATCACTGCExtendedSpectrumbeta-lactamasesNDM-1P11:TTGGCGATCTGGTTTTC621碳青霉烯类酶P12:AATGGCTCATCACGATCCarbapenemaseTEMP13:AAATTCTTGAAGAC1075产超广谱β-内酰胺类酶P14:CCAATGCTTAATCAExtendedSpectrumbeta-lactamasesSHVP15:CGTTATATTCGCCTGTG862产超广谱β-内酰胺类酶P16:GCGTTGCCAGTGCTCGExtendedSpectrumbeta-lactamases
1.2 方法
1.2.1 培养基制备
5%血平板、麦康凯培养基、普通平板等均按照文献[9]中方法配制。凝固后用保鲜膜包好放于4 ℃备用。
1.2.2 细菌的分离纯化
采集病理剖检有典型病理变化的组织,无菌操作,接种于血平板进行分离培养,37 ℃恒温培养18~24 h,同时采集病料进行厌氧培养。对各培养条件下细菌菌落进行观察,将分离株纯培养细菌经革兰氏染色镜检,观察细菌的个体形态及染色特征,然后接种于麦康凯培养基观察其培养特征。
1.2.3 生化试验
将以上纯培养得到的细菌接种于普通营养琼脂中培养24 h,按常规方法将菌液接种微量生化管进行生化试验。
1.2.4 16S rDNA 基因鉴定
以纯培养细菌染色体DNA为模板,扩增16S rDNA基因片段。反应体系25 μL:2×Taq PCR MasterMix 12.5 μL,ddH2O 8.5 μL,P1、P2各1.0 μL,模板2.0 μL,反应条件:94 ℃ 5 min;94 ℃ 45 s、55 ℃ 30 s、72 ℃ 30 s,30个循环;72 ℃ 10 min。PCR产物经10 g·L-1的琼脂糖凝胶电泳检测,并纯化回收目的片段,由擎科生物技术有限公司测序。
1.2.5 小鼠致病性实验
按照参考文献[10]的方法进行小鼠致病性试验,将挑纯菌株接种普通培养基培养24 h后,以无菌生理盐水冲洗菌苔,采用麦氏比浊法测定菌液浓度后稀释至1×108cfu·mL-1。将8只小鼠随机分为2组,每组4只,每只腹腔注射菌液0.2 mL测定其致病力,对照组注射0.2 mL生理盐水。
1.2.6 药敏试验
采用KB纸片扩散法进行药敏试验,参照《全国临床检验操作规程》及CLSI《抗菌药物敏感性试验标准》2013版,按抑菌圈直径大小和各抗菌药的具体标准评定为敏感、中介和耐药。
1.2.7 耐药基因PCR检测
以分离菌株的纯培养物为模板, 用各种耐药基因引物(表1)进行7种耐药基因的PCR检测,同时以实验室保存DHA-1阳性猪肺炎克雷伯氏菌菌株为检测对照。反应体系25 μL:2×Taq PCR MasterMix 12.5 μL,ddH2O 8.5 μL,上下游引物各1.0 μL,模板2.0 μL,PCR 反应条件:94 ℃ 5 min;94 ℃ 45 s,55 ℃ 30 s,72 ℃ 30 s,30个循环;72 ℃ 10 min。PCR产物经10 g·L-1的琼脂糖凝胶电泳检测。将扩增出的特异性DNA条带进行胶回收,连接转化,将产物送至生物工程公司进行测序。将测序结果进行BLAST分析。
2 结果与分析
2.1 细菌的分离纯化
细菌在血平板和普通琼脂培养基上为中等大小的菌落,半透明,灰白色,圆凸,闪光,丰富而黏稠,有一部相互交融、表面湿润的光滑型菌落,拉动成丝线状。在麦康凯平板上生长形成隆起大而黏液样粉红色菌落,经革兰染色镜检显示为革兰阴性短球杆状,如图1所示。
2.2 生化鉴定
以无菌生理盐水冲洗菌苔,将菌液接种于生化管中进行生化鉴定,培养特性结果显示,该菌可以发酵多种糖类,产酸或同时产气,不产生H2S,可分解尿素,利用枸橼酸盐等,为兼性厌氧发酵型无运动能力的革兰阴性杆菌(表2),与克雷伯氏菌的生化特征相一致[11],初步鉴定为牛源肺炎克雷伯氏菌。
2.3 16S rDNA鉴定
挑取纯培养的菌落进行PCR扩增测序,分离株的16S rDNA 扩增结果经琼脂糖凝胶电泳,片段约1 500 bp,与预期目的片段大小一致(图2)。将纯化回收的目的片段测序结果(已提交GenBank,登录号KX237750)经BLAST分析比对,显示该菌株16S rDNA 基因部分序列与肺炎克雷伯氏菌同源性达99%,结合培养特性和生化实验结果,鉴定该菌为牛源肺炎克雷伯氏菌,并命名为CK2016。
图1 分离菌株的革兰染色Fig.1 Gram staining of isolated strain
表2 生化鉴定结果
Table 2 Result of biochemical tests

底物Substrate结果Result底物Substrate结果Result葡萄糖Glucose精氨酸Arginine-麦芽糖Maltose甘露醇Mannitol蔗糖Sucrose丙二酸盐Malonate+乳糖Lactose苯丙氨酸Phenylalanine-硫化氢Hydrogensulfide-水杨酸Salicylicacid+黏液酸Mucusacid-卫矛醇Dulcite-山梨醇Sorbitol鼠李糖Rhamnose+阿拉伯糖Arabinose纤维二糖Cellobiose枸橼酸钠Sodiumcitrate+蕈糖Mushroomsugar尿素Urea+鸟氨酸Ornithin+蜜二糖Melibiose半乳糖Galactose+七叶苷Esculoside氰化钾Editpotassiumcyanide+
“⊕”表示产酸产气;“+”表示产酸;“-”表示阴性。
“⊕” represented producing acid and gas; “+” represented producing acid; “-” represented negative.

M,DL2000 marker;1,分离株16SrDNA ;2,阴性对照M, DL2000 marker; 1, 16S rDNA of isolated strain; 2, Negative control图2 分离株16S rDNA 基因扩增电泳图谱Fig.2 Electrophoresis diagram of 16S rDNA gene amplification products of isolated strain
2.4 小鼠致病性试验
感染组小鼠于接种4 h后,开始表现出精神沉郁,在接种后24 h 内全部死亡。剖检小鼠组织病变表现为肝脏肿胀,边缘瘀血,脾脏肿大充血,剖检后取内脏器官进行细菌的分离,通过革兰染色镜检、生化试验和16S rDNA PCR 法鉴定, 结果与犊牛病变组织中分离到的细菌一致,对照组无异常反应。
2.5 药敏试验结果
按照1.2.6节操作进行分离菌株的药物敏感性检测,结果如表3所示,该菌对亚胺培南、美培南、头孢匹罗等大部分的抗生素敏感,对氨苄西林、头孢呋辛、复方新诺明以及红霉素耐药。对萘定酸、头孢噻吩、氧氟沙星表现为中敏。
表3 药敏试验结果
Table 3 Result of antimicrobial susceptibility
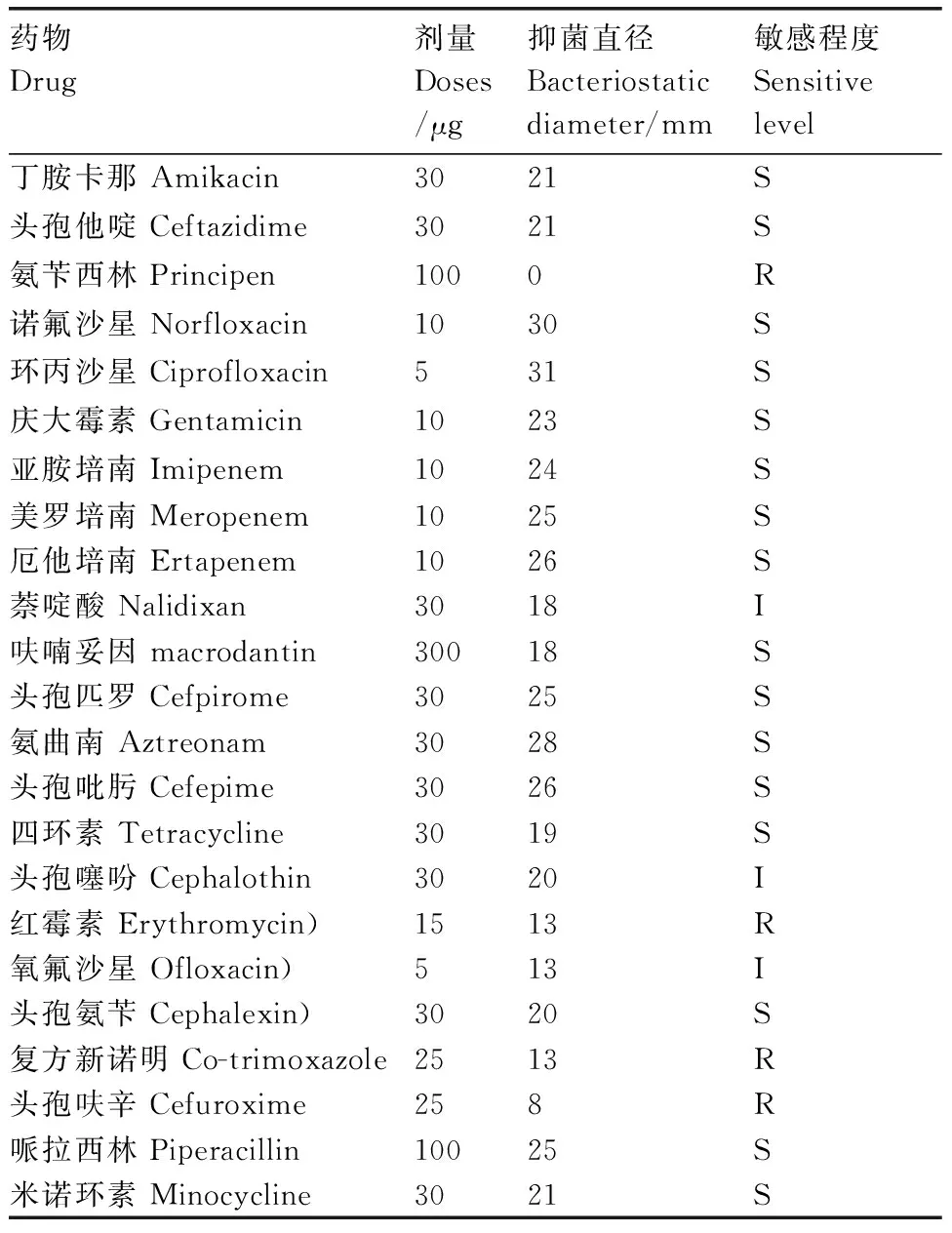
药物Drug剂量Doses/μg抑菌直径Bacteriostaticdiameter/mm敏感程度Sensitivelevel丁胺卡那Amikacin3021S头孢他啶Ceftazidime3021S氨苄西林Principen1000R诺氟沙星Norfloxacin1030S环丙沙星Ciprofloxacin531S庆大霉素Gentamicin1023S亚胺培南Imipenem1024S美罗培南Meropenem1025S厄他培南Ertapenem1026S萘啶酸Nalidixan3018I呋喃妥因macrodantin30018S头孢匹罗Cefpirome3025S氨曲南Aztreonam3028S头孢吡肟Cefepime3026S四环素Tetracycline3019S头孢噻吩Cephalothin3020I红霉素Erythromycin)1513R氧氟沙星Ofloxacin)513I头孢氨苄Cephalexin)3020S复方新诺明Co-trimoxazole2513R头孢呋辛Cefuroxime258R哌拉西林Piperacillin10025S米诺环素Minocycline3021S
S表示敏感;I表示中介;R表示耐药。
S represented sensitive; I represented intermediary; R represented resistance.
2.6 耐药基因检测
按照1.2.7节操作进行分离菌株7种耐药基因的PCR检测,结果如图3所示,仅SHV基因扩增阳性,在862 bp左右出现预期大小DNA条带(泳道7),其DNA片段的测序结果BLAST分析显示与NCBI中公布的SHV-1基因序列(登录号:X98098)同源性达到99.3%;DHA-1基因型的检测产物在245 bp左右虽出现一特异性DNA条带(图3泳道5和图4泳道2),而DHA-1阳性猪肺炎克雷伯氏菌菌株扩增出405 bp大小DNA条带,二者大小不同。将其DNA扩增条带的序列测定进行BLSAT分析,DHA-1阳性对照菌株扩增的DNA序列与已公布DHA-1基因序列(登录号:AY635140)同源性比对达97.2%以上,而菌株CK2016扩增的DNA片段与克雷伯氏菌染色体上基因序列同源性达99%以上。为比较二者序列差异,分别登录NCBI调取3条DHA-1靶基因序列KJ135976、KJ135977 和KJ135990,以及与菌株CK2016扩增的DNA片段同源性99%的克雷伯氏菌基因序列CP003200、CP003999、CP006648进行比对,结果如图5所示,该克雷伯氏菌扩增基因片段与耐药基因DHA-1靶基因片段出现大量缺失,同源性仅有30%左右,提示菌株CK2016的耐药基因DHA-1阴性。加之碳青霉烯类酶基因、TEM-1和CTX-M1等其他5种耐药基因的PCR扩增均为阴性(图3泳道1-6),表明该牛源克雷伯氏菌菌株CK2016只有SHV-1耐药基因,与药敏试验的结果基本一致。

M, DL2000 marker;1、2、3、4、6分别是KPC,CTX-M1,OXA-48,NDM-1,TEM-1;5和7分别是DHA-1(245 bp),SHV-1(860 bp)M : DL2000 marker; 1, 2, 3, 4, 6 were KPC, CTX-M1, OXA-48, NDM-1, TEM-1, respectively; 5 and 7 were DHA-1(245 bp), SHV-1(860 bp)图3 基因PCR扩增电泳Fig.3 Electrophoresis diagram of resistance genes amplification

M,DL2000 marker;1,犊牛分离株(245 bp);2,猪分离株(405 bp);3,阴性对照M, DL2000 marker; 1, Strain from calf (245 bp); 2, Strain from swine (405 bp); 3, Negative control图4 两株克雷伯氏菌DHA-1 PCR产物电泳图Fig.4 Electrophoresis diagram of amplification results of two Klebsiella pneumonia strains
3 讨论
近年来克雷伯氏菌在人医临床上是仅次于大肠埃希菌的条件性致病菌,而且随着抗生素的大量使用,使该菌变异产生各种抗生素水解酶,对于其治疗的首选药物碳青霉烯类抗生素产生广泛的耐药性,使得该病的治疗非常棘手[12]。在很多感染克雷伯氏菌的病例报道中,主要是引起肺炎、呼吸道感染、腹膜炎等,奶牛则主要引起乳房炎[13]。本研究根据纯化培养结果、镜检观察,以及生化鉴定结果,可以初步认定其为克雷伯氏菌,再通过16S rDNA扩增测序从而确证该病原菌为肺炎克雷伯氏菌,小鼠致病性实验显示,该菌的致病性强,提示犊牛死亡可能与该菌密切相关,加之药敏试验显示该菌株对复方磺胺等药物耐受,这与用药后病情最终未得到控制相符合。

*为扩增245 bp基因片段测序* indicated 245 bp fragment of amplified gene sequence图5 DHA-1基因序列及克雷伯氏菌扩增序列Align分析结果Fig.5 Align analysis of DHA-1 gene sequences and amplified Klebsiella pneumoniae gene sequences
SHV为细菌的质粒介导,是巯基变量(sulphydryl variable)的缩写,为ESBLs的一种基因型,可通过传导、转化、转座、结合转移和整合等方式将耐药性在同种或不同种细菌间传播和扩散,而且其耐药基因编码位点往往与喹诺酮类、氨基糖苷类和磺胺类药物耐药基因相近而导致多药耐性。该酶类主要水解β-内酰胺类抗生素,如青霉素、头孢、单环酰胺类等[14-15]。同时在肺炎克雷伯氏菌中也有染色体介导的SHV-1型基因,该基因所编码的酶不仅可以水解广谱β-内酰胺类抗生素,还可以水解亚胺培南[16]。本研究经PCR检测扩增出了SHV-1耐药基因,这与该菌耐受氨苄西林、头孢呋辛等药物相一致。AmpC酶编码基因存在于大多数细菌的染色体上,但肺炎克雷伯菌出现质粒介导的AmpC酶,国内以DHA-1基因型为主[17],该类基因介导的水解酶主要对一代到三代头孢、氨曲南等药物水解作用强,该耐药基因可以通过质粒的复制、转化以及一些转座元件的转移,使得耐药基因可以在种属内(间)类扩散[18]。本研究进行DHA-1 的PCR检测时直接取培养菌液为模板,避免了提取DNA和抽提质粒,结果未扩增出预期大小基因条带,表明该菌不存在DHA-1耐药基因,这与该菌株对头孢匹罗、头孢他啶、亚胺培南等药物敏感的表型也是符合的。值得说明的是,将扩增出克雷伯氏菌的非预期大小DNA条带与DHA-1耐药基因靶片段序列进行分析显示,二者存在约30%的同源性,DHA-1耐药基因靶片段序列更长,从敏感菌株到DHA-1耐药基因形成是否通过插入突变和点突变形成,抑或通过缺失突变能否将耐药菌株耐药性去除将是非常让人感兴趣的问题。
肺炎克雷伯氏菌作为医院细菌感染的重要成员,且随着耐药基因的变体增加,多重耐药严重,同时质粒介导类的耐药基因在种类间的转移,使得耐药基因很容易扩散,已成为临床工作中不容忽视的问题。人医临床上,抗生素使用种类多,碳青霉烯类常作为治疗该菌感染的首选药物,所以人医临床上有大量的耐药性研究报道。而本研究分离的克雷伯氏菌对大部分碳青霉烯类、第四代头孢等敏感抗生素敏感,宏观上讲很可能与兽医临床上相关抗生素尤其是碳青霉烯类抗生素使用较少相关。这从一个侧面说明在兽药管理和使用方面,应严防人药用于畜禽养殖。总之,本研究通过细菌分离鉴定、药敏试验和耐药基因的检测对该例犊牛死亡的原因进行了细菌学检测与耐药表型和基因分析,为克雷伯氏菌感染的临床用药提供了科学参考,也为克雷伯氏菌耐药机理的相关研究提供了科学材料。
[1] 和晋渝.肺炎克雷伯菌的血清分型及毒力基因分布的研究[D].重庆:重庆医科大学,2012. HE J Y. Study on serotypes and distribution characteristic of virulence gene ofKlebsiellapneumoniae[D].Chongqing: Chongqing Medical University, 2012. (in Chinese with English abstract)
[2] 刘保光,吴华,张萍英. 肺炎克雷伯菌耐药研究进展[J]. 畜牧与兽医,2014,46(2):108-111. LIU B G, WU H, ZHANG P Y. Research progress of resistance ofKlebsiellapneumoniae[J].AnimalHusbandryandVeterinaryMedicine, 2014, 46(2):108-111. (in Chinese)
[3] 管希周,罗燕萍,刘又宁,等. 高产AmpC酶肺炎克雷伯菌的临床报道[J]. 中华医院感染学杂志,2004,14(4):461-464. GUAN X Z, LUO Y P, LIU Y N, et al. High AmpC-producing clinical isolates ofKlebsiellapneumoniae: A clinical report [J].ChineseJournalofNosocomiology, 2004, 14(4):461-464. (in Chinese with English abstract)
[4] KOLA A, PIENING B, PAPE U F, et al. An outbreak of carbapenem-resistant OXA-48-producingKlebsiellapneumoniaeassociated to duodenoscopy [J].AntimicrobialResistanceandInfectionControl, 2015, 4(1):8.
[5] 陈燕敏,廖于峰,芮球红. 产ESBLs肺炎克雷伯菌耐药表型分析与整合子基因检测的研究[J]. 中华医院感染学杂志,2015,25(23):5301-5304. CHEN Y M, LIAO Y F, RUI Q H. Detection of resistance phenotype and integrons among ESBLs-producingKlebsiellapneumoniaeisolated from intensive care unit [J].ChineseJournalofNosocomiology, 2015, 25(23):5301-5304. (in Chinese with English abstract)
[6] GREISEN K, LOEFFELHOLZ M, PUROHIT A, et al. PCR primers and probes for the 16S rRNA gene of most species of pathogenic bacteria, including bacteria found in cerebrospinal fluid [J].JournalofClinicalMicrobiology, 1994, 32(2): 335-351.
[7] 王建友,周阿旺,陈丹. 碳青霉烯酶在碳青霉烯类耐药肺炎克雷伯杆菌的分布及分子流行病学[J]. 中国微生态学杂志,2015,27(5):517-526. WANG J Y, ZHOU A W, CHEN D.The distribution and molecular epidemiology of carbapenemase in carbapenem-resistantKlebsiellapneumoniae[J].ChineseJournalofMicroecology, 2015, 27(5):517-526. (in Chinese with English abstract)
[8] 唐愈菲,杨成,王铠佳. 耐头孢西丁肺炎克雷伯菌AmpC酶的检测及耐药性基因分析[J]. 实用预防医学,2010,17(8):1516-1519. TANG Y F, YANG C, WANG K J. Detection of AmpC β-lactamases in cefoxitin-resistantKlebsiellapneumoniaeand analysis on its antimicrobial resistance genes[J].PracticalPreventiveMedicine, 2010, 17(8):1516-1519. (in Chinese with English abstract)
[9] 杨泽晓,刘常钰,王印. 一株鹅源多杀性巴氏杆菌强毒株的分离鉴定及其血清型鉴定相关基因的克隆[J]. 浙江农业学报,2016,28(6):935-943. YANG Z X, LIU C Y, WANG Y. Isolation, identification of a virulent strain goosePasteurellamultocidaand gene cloning of its serotype associated genes [J].ActaAgricultureZhejiangensis, 2016, 28(6):935-943. (in Chinese with English abstract)
[10] 林星宇,王印,杨泽晓. 猪源肺炎克雷伯菌的分离鉴定[J]. 中国预防兽医学报,2015,37(5):375-378. LIN X Y, WANG Y, YANG Z X. Isolation and identification ofKlebsiellapneumoniaefrom swine [J].ChineseJournalofPreventiveVeterinaryMedicine, 2015, 37(5):375-378. (in Chinese with English abstract)
[11] 陆承平. 兽医微生物学 [M]. 5版. 北京:中国农业大学出版社,2012:112.
[12] 刘保光,贺志沛,吴华. 兽医临床肺炎克雷伯菌的流行现状及防治措施研究[J]. 农业灾害研究,2013,3(8):32-33, 41. LIU B G, HE Z P, WU H. Research on situation and control measures ofKlebsiellapneumoniaeepidemic in veterinary clinical [J].JournalofAgriculturalCatastrophology, 2013, 3(8):32-33, 41. (in Chinese with English abstract)
[13] 海岩. 内蒙地区奶牛环境性乳房炎主要病原菌的分离鉴定及克雷伯氏菌的基因分型[D].呼和浩特:内蒙古农业大学,2007. HAI Y. Isolation and identification of environmental mastitis pathogens and genetyping ofKlebsiellain Inner Mongolia [D].Huhehaote: Inner Mongolia Agricultural University, 2007. (in Chinese with English abstract)
[14] MEDEIROS A A. Evolution and dissemination of β-lactamases accelerated by generation of β-lactam antibiotics [J].ClinicalInfectiousDiseases, 1997, 24(Suppl. 1):S19-S45.
[15] 张玲,黄永茂. SHV型超广谱β-内酰胺酶的研究进展[J]. 国际检验医学杂志,2016, 37(7):944-946. ZHANG L, HUANG Y M. Advances in research of SHV extended broad spectrum β-lactamases [J].InternationalJournalofLaboratoryMedicine, 2016, 37(7):944-946. (in Chinese)
[16] LAURENT P, CLAIRE H, ISABELLE P, et al. Emergence inKlebsiellapneumoniaeof a chromosome-encoded SHV β-lactamase that compromises the efficacy of imipenem [J].AntimicrobialAgentsandChemotherapy, 2003, 47(2):755-758.
[17] DING H, YANG Y, LU Q, et al. The prevalence of plasmid-mediated AmpC beta-lactamases among clinical isolates ofEscherichiacoliandKlebsiellapneumoniaefrom five children’s hospitals in China [J].EuropeanJournalofClinicalMicrobiologyandInfectiousDiseases, 2008, 27(10):915-921.
[18] 张运丽. 肺炎克雷伯菌质粒介导AmpC β-内酰胺酶耐药基因研究[D]. 长沙:中南大学,2009. ZHANG Y L. Study on drug resistance genes of plasmid-mediated AmpC β-lactamases produced byKlebsiellapneumoniae[D]. Changsha: Central South University, 2009. (in Chinese with English abstract)
(责任编辑 卢福庄)
Isolation, identification and drug resistance detection of a calfKlebsiellapneumoniaestrain
MENG Zhengqun1, LENG Yiyi1, REN Meishen1, LIU Yadong1, WANG Yin1,2, YAO Xueping1,YANG Zexiao1,*
(1.CollegeofVeterinaryMedicine,SichuanAgriculturalUniversity,Wenjiang611130,China; 2.KeyLaboratoryofAnimalDiseaseandHumanHealthofSichuanProvince,Wenjiang611130,China)
A gram-negative bacillus was isolated and purified from the lung of a died calf, and was identified by morphological and cultured characteristics, biochemical tests, 16S rDNA gene sequencing, pathogenicity tests, and drug sensitivity tests. Then its drug-resistance genes were detected and analyzed by PCR methods using 7 pairs of primers for drug-resistance genes that produce carbapenemase and extended-spectrum β-lactamase (ESBLs) etc. The results showed that this isolate was identified as cow-derivedKlebsiellapneumoniae, which was pathogenic to mice. Its 16S rDNA gene sequence shared 99% homology withKlebsiellapneumoniaegene sequences published in GenBank. The isolate was highly resistant with penbritin, cefuroxime, co-trimoxazole and erythromycin, while sensitive to the gross antibiotics such as carbapenems and the fourth generation cephalosporin. A resistance gene fragment about 862 bp in the length ofSHVgene was amplified by PCR, which had a 99.3% homology with publishedSHV-1 gene sequences in GenBank. The cow-derivedKlebsiellapneumoniaestrain had strong virulence and drug-resistance, and its drug-resistant phenotype was consistent with the drug-resistant testing results.
Klebsiellapneumoniae; isolation and identification; drug resistance genes
http://www.zjnyxb.cn
10.3969/j.issn.1004-1524.2017.04.03
2016-10-26
“十二五”国家科技支撑计划(2013BAD12B04);四川农业大学学科建设双支计划(03571806)
蒙正群(1992—),女,贵州瓮安人,硕士研究生,研究方向为预防兽医学。E-mail: m15983544718_1@163.com
* 通信作者,杨泽晓,E-mail: yzxyang2003@126.com
S858.23;S855.1+2
A
1004-1524(2017)04-0534-08
浙江农业学报ActaAgriculturaeZhejiangensis, 2017,29(4): 534-541
蒙正群,冷依伊,任梅渗,等. 一株牛源肺炎克雷伯氏菌的分离鉴定与耐药基因型检测[J]. 浙江农业学报,2017,29(4): 534-541.
