耐氟喹诺酮类药物禽源大肠埃希菌gyrA基因的检测分析
2017-04-13罗青平张腾飞汪宏才王红琳温国元张蓉蓉邵华斌
卢 琴 ,罗青平,张腾飞,汪宏才,罗 玲,王红琳,温国元,张蓉蓉,邵华斌
(湖北省农业科学院畜牧兽医研究所/农业部畜禽细菌病防治制剂创制重点实验室/动物胚胎工程及分子育种湖北省重点实验室,湖北武汉 430064)
耐氟喹诺酮类药物禽源大肠埃希菌gyrA基因的检测分析
卢 琴 ,罗青平,张腾飞,汪宏才,罗 玲,王红琳,温国元,张蓉蓉,邵华斌*
(湖北省农业科学院畜牧兽医研究所/农业部畜禽细菌病防治制剂创制重点实验室/动物胚胎工程及分子育种湖北省重点实验室,湖北武汉 430064)
探讨不同禽源大肠埃希菌中喹诺酮类药物的耐药情况及耐药基因gyrA的分布和突变特征。采用K-B药敏纸片法、gyrA基因的PCR扩增,对9株大肠埃希菌进行喹诺酮类药物试验,并将gyrA基因的PCR产物测序,对测序结果采用DNA MAN、DNA Star、MEGA6等软件分析。药敏试验结果表明,C1、C2、C3菌株对左氧沙星、氧氟沙星、环丙沙星、诺氟沙星敏感,D1、D2、D3、B1、B2和B3菌株对左氧沙星、氧氟沙星、环丙沙星、诺氟沙星均表现为耐药和中介;gyrA基因的测序结果表明,除B1菌株有1处核苷酸突变位点和B2菌株有14处核苷酸突变位点;B2菌株gyrA基因的氨基酸突变发生在87位Ile→Val替代、101位Leu→Met替代、102位Ala→Ser替代、129位Lys→Gln替代。9株禽源大肠埃希菌的同源性和进化树分析表明,不同禽源耐氟喹诺酮类药物的大肠埃希菌菌株中B2菌株gyrA基因与其他9株菌株相比,同源性在90%左右,进化树不在一个分支上,研究中的B2菌株将为大肠埃希菌的氟喹诺酮类耐药机制的研究提供候选菌株。
禽源大肠埃希菌;氟喹诺酮;耐药菌株;gyrA基因
氟喹诺酮类药物(fluoroquinolones,FQs)为人工合成化学抗菌药,在人和动物治疗细菌感染中使用最为广泛。氟喹诺酮类药物与DNA结合和拓扑异构酶形成喹诺酮DNA拓扑异构酶络合物,阻断DNA的转录或复制,而对喹诺酮类药物耐药的主要机制是DNA旋转酶和Ⅳ型拓扑异构酶2种酶的突变[1-2],细菌膜通透性的降低以及膜的主动外排泵激活;编码靶酶DNA促旋酶的gyrA基因突变范围主要集中于第67-106位氨基酸的区域,称为喹诺酮耐药性决定区(QRDR),抑制菌体DNA的合成而起到杀菌作用[3]。质粒介导的喹诺酮类耐药基因有3种,即通过导致质粒介导的水平转移基因qnr产生喹诺酮的耐药,其他2种质粒介导喹诺酮类耐药基因为AAC(6′)-Ib-cr基因和外排泵qepA基因[4-6]。经过20多年的临床应用,使细菌对该类药物普遍具有耐药性,耐药谱也日益增大,严重影响药物治疗的效果及药物的合理使用。本试验选择鸡、鸭、野鸟分离到的致病性和非致病性大肠埃希菌作为研究对象,了解禽类不同种属间大肠埃希菌FQs的耐药情况及靶位基因gyrA突变,从而正确地使用药物进行治疗,为阐述耐药机制的研究提供实验数据基础。
1 材料与方法
1.1 材料
1.1.1 菌株 9株大肠埃希菌分离自湖北大冶、天门、汉口等地区鸡、鸭、野鸟的肝、脾、脑等组织及粪便,划线接种于培养基上,其编号和来源如下:菌株C1~C3(鸡脾脏、脑、肝等组织),菌株D1~D3(鸭心、肝、脾脏等组织),菌株B1~B3(野鸟粪便)。ATCC25922为药物敏感性试验质控菌,中国兽医药品监察所产品。
1.1.2 检测药物与试剂 左氧沙星(5 μg/片)、氧氟沙星(5 μg/片)、环丙沙星(5 μg/片)、诺氟沙星(10 μg/片),杭州天和微生物试剂有限公司产品;DNA Marker DL 2 000、MIX,宝生物工程(大连)有限公司产品。
1.2 方法1.2.1 药敏试验 药敏试验按照CLSI(Clinical and Laboratory Standards Institute)推荐的K-B法进行。
1.2.2 引物设计 根据GenBank(登录号:KC493121)中大肠埃希菌的引物序列,采用Primer 5.0软件设计gyrA基因引物gyrA(F:5′-CGATGTCGGTCATTGTTG-3′,R:5′-ACTTCCGTCAGGTTGTGC-3′),扩增产物大小为496 bp。
1.2.3 模板DNA提取和PCR扩增 采用水煮沸法,将过夜培养的大肠埃希菌菌液于沸水中煮10 min后置于冰上20 min~30 min,12 000 r/min离心5 min取上清,即为模板DNA。PCR反应条件:95℃ 5 min;95℃ 30 s,55℃ 30 s,72℃ 40 s,30个循环;72℃ 10 min,4℃保存。反应体系为25 μL体系,即模板DNA 2 μL,上、下游引物各1 μL,MIX12.5 μL,加ddH2O至25 μL。PCR扩增完成后将PCR产物点样于10 g/L琼脂糖凝胶电泳,恒压为120 V,电流18 mA,15 min,于凝胶成像系统将凝胶结果进行拍照保存。
1.2.4 测序及分析 将gyrA基因PCR扩增产物送到上海生工生物工程技术服务有限公司测序。测序结果用DNA Man软件与GenBank中引物的gyrA基因参考序列进行核苷酸和氨基酸序列比对分析,采用DNA Star和MEGA6进行不同禽源gyrA基因的同源性和进化树分析。
2 结果
2.1 药敏试验
2.1.1 K-B纸片法的质控菌 大肠埃希菌(ATCC25922)的药敏结果质控范围见表1,符合大肠埃希菌药敏试验的药敏结果质控范围。
2.1.2 9株大肠埃希菌的药物敏感性检测结果 结果见表2。
2.2 大肠埃希菌gyrA基因的PCR扩增
琼脂糖凝胶电泳显示,9株禽源大肠埃希菌和质控菌株ATCC2599均扩增出分子质量大小为496 bp的目的条带,这与gyrA基因的预期片段大小一致(图1)。

表1 质控菌大肠埃希菌(ATCC25922)的药敏结果质控范围
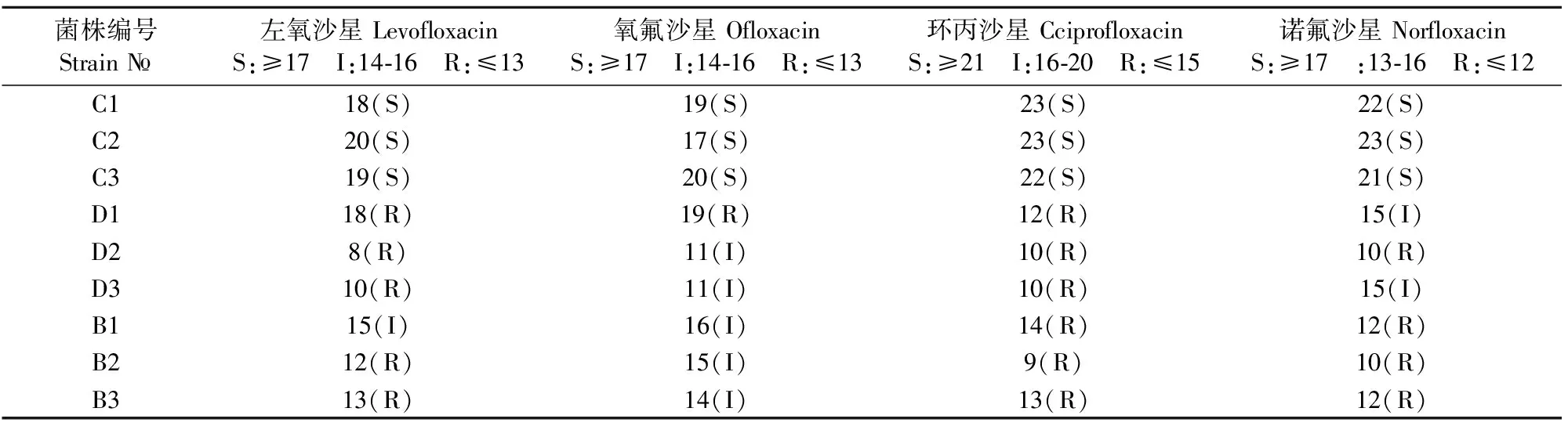
表2 K-B法药敏试验结果

M.DNA 标准DL 2 000;1~3.鸡病料;4~6.鸭病料;7~9.鸟病料;10.ATCC25922
2.3 大肠埃希菌gyrA基因突变
9株不同来源大肠埃希菌分离株的gryA基因氟喹诺酮类药物耐药决定区突变引起的不同碱基和氨基酸替代类型及其发生的菌株数见表3、表4、图2和图3。
2.4 不同禽源大肠埃希菌gyrA基因的同源性和进化树分析
B2菌株与参考菌株及其他8个菌株的同源在90%左右,而另外8株菌株的同源性与参考菌株的同源均在98%左右,说明了B2菌株的差异明显高于另外8株试验菌株。进化树分析表明B2菌株与其他8株禽源大肠埃希菌菌株不在一个分支上,分析结果结果见图4和图5。

表3 大肠埃希菌gyrA基因核苷酸序列比较分析

表4 大肠埃希菌gyrA基因氨基酸序列比较分析
3 讨论
目前对于喹诺酮类药物的敏感性降低导致临床上使用此类药物进行预防和治疗没有效果。而国内外对大肠埃希菌耐氟喹诺酮类药物耐药机制的研究多集中于人源或动物源致病性大肠埃希菌的研究层面。至于用K-B药敏纸片法检测,对喹诺酮的耐药表型差异不显著,故现多采用PCR结合测序对突变位点进行分析,从而更好地了解细菌及病毒样品中gyrA基因的分布情况。本研究采用PCR扩增均能扩增出gyrA基因,但其药敏试验结果与PCR结果不完全一致,故用测序方法找出菌株的gyrA QRDR区的点突变。Farrera G P等[7]表明猪沙门菌gyrA QRDR区第83位和87位氨基酸突变,导致了发病率和病死率增加,降低了抗菌药物的治疗效果。Poramathikul K等[8]对2014年—2015年柬埔寨感染患者的腹泻样品中检测出对gyrA、 parC、 β-内酰胺酶和mphA等多重耐药,多重耐药问题引起了人们对志贺菌属的关注。表明gyrA QRDR区的有义突变常发生在第81、82、83、84、87位和106位氨基酸的编码基因,单位点突变以83位ser→leu最常见,双位点突变有第83、87位氨基酸取代[9-11]。本试验中B2菌株的突变发生在87位Ile→Val替代,而B2菌株的突变发生在87位Ile→Val替代、101位Leu→Met替代、102位Ala→Ser替代、129位Lys→Gln替代。这与曹丽婷等[12]报道的耐药菌株的突变发生在83位ser→leu替代和87位Asp→Asn替代,闫东辉等[13]表明屎肠球菌的gyrA亚单位的83位氨基酸由ser→pro,粪肠球菌gyrA亚单位的87位氨基酸由Glu变为Gly,粪肠球菌和屎肠球菌gyrA基因的氨基酸突变不同。对加拿大野鸟大肠埃希菌gyrA、gyrB、parC和parE基因的QRDR区进行测序分析,在所有分离株中gyrA仅在83位ser→leu,而gyrB、 parC、parE在分离株中未见突变位点[14]。Anna J等[15]表明gyrA的氨基酸突变在83位ser→leu和87位asp→asn,检出率分别为86%、32%, 有31%菌株存在这2种氨基酸突变。因此,说明了各类别大肠埃希菌中gyrA基因的分布不同,导致对喹诺酮的耐药谱不一样。不同位点的氨基酸替代,同一位点的不同氨基酸替代、单个氨基酸替代或氨基酸双替代都可导致不同氟喹诺酮的药物敏感度不同。药敏结果表明C1、C2、C3菌株对左氧沙星、氧氟沙星、环丙沙星、诺氟沙星敏感,D1、D2、D3、B1、B2和B3菌株对左氧沙星、氧氟沙星、环丙沙星、诺氟沙星均表现为耐药和中介,说明了在临床用药时,应了解药物的使用情况及发病情况,用药间隔时间及日龄大小,以避免多重用药导致耐药,从源头上控制抗菌药物的使用。gyrA基因的突变与大肠埃希菌的耐药性密切相关,同源性和进化树分析表明,B2菌株的点突变多,B2菌株与参考菌株及其他8个菌株的同源在90%左右,而另外8株菌株的同源性与参考菌株的同源均在98%左右,说明了B2菌株的差异明显高于另外8株试验菌株。致肾盂肾炎大肠埃希菌gyrA基因突变能降低细菌的毒力,可能与几种毒力因子(fimA、papB、ompA 基因)和蛋白表达的DNA超螺旋结构作用有关,由此降低感染膀胱炎和肾盂肾炎的能力[16-17]。刘果等[18]和阮周曦等[19]分别对长春地区宠物源和进境冻畜禽产品中大肠埃希菌对喹诺酮中的环丙沙星和左氧氟沙星的耐药率均在50%左右,说明临床生产中对喹诺酮类药物的滥用,使得不同动物源的大肠埃希菌均能产生较高的耐药率。进化树分析表明B2菌株与其他8株禽源大肠埃希菌菌株不在一个分支上,B2菌株的点突变结果可能与毒力基因、进化树、耐药基因的分布特点有关[20-23],这为后续的耐药机理研究提供背景材料,另外针对临床分离病料,了解药物使用情况,以减少交叉耐药,并从源头上合理选用抗菌药物及其替代品,最终根据检测结果指导临床用药提供参考。

图2 大肠埃希菌gyrA基因核苷酸序列比较分析

图3 大肠埃希菌gyrA基因氨基酸序列比较分析

图4 9株禽源大肠埃希菌的同源性分析

图5 9株禽源大肠埃希菌的进化树分析
[1] Calvo J,Martínez-Martínez L.Antimicrobial mechanisms of action[J].Enferm Infecc Microbiol Clin,2009,27(1):44-52.
[2] Hooper D C.Mechanisms of action and resistance of older and newer fluoroquinolones[J].Clin Infect Dis,2000,31:24-28.
[3] Piddock L J V. Mechanisms of fluoroquinolone resistance:An update 1994-1998[J].Drugs,1999,58(2):11-18.
[4] Martinez-Martinez L,Pascual A,Jacoby G A,et al.Quinolone resistance from a transferable plasmid[J].Lancet Infect Dis,1998,351:797-799.
[5] Robicsek A,Jacoby G A,Hooper D C,et al.The worldwide emergence of plasmid-mediated quinolone resistance[J].Lancet Infect Dis,2006,6:629-640.
[6] Yamane K,Wachino J,Suzuki S,et al.New plasmid-mediated fluoroquinolone efflux pump,QepA,found in anEscherichiacoliclinical isolate[J].Antimicrob Agents Chemother,2007,51:3354-3360.
[7] Farrera G P,Borroto E T,Ramírez F R,et al.Detection of quinolone resistance inSalmonellatyphimuriumpig isolates determined by gyrA gene mutation using PCR and sequence-based techniques within the gyrA gene[J].Curr Pharm Des,2016,8.PMID: 27494067.
[8] Poramathikul K,Bodhidatta L,Chiek S,et al.Multidrug-resistant shigella infections in patients with diarrhea,cambodia,2014-2015[J].Emerg Infect Dis,2016,22(9):1640-1643.
[9] Yoshia H,Bogaki M,Nakamura M,et al.Quiolone resistance-determing region in the DNA gyrase gyrB gene ofEscherichiacoli[J].Antimicrob Agents Chemother,1991,35(8):1647-1650.
[10] Tamang M D,Nam H M,Chae M H,et al.Prevalence of plasmid-mediated quinolone resistance determinants amongEscherichiacoliIsolated from food animals in Korea[J].Foodb Pathog Dis,2012,9(12):1057-1063.
[11] De la Fuente C M,Dauros S P,Bello T H,et al.Mutations in gyrA and gyrB genes among strains of Gram-negative bacilli isolated from Chilean hospitals and their relation with resistance to fluoroquinolones[J].Rev Med Chil,2007,135(9):1103-1110.
[12] 曹立亭,周晏丞,仲崇华,等.养殖环境中耐氟喹诺酮类药物大肠杆菌gyrA基因的检测与序列分析[J].西南大学学报,2014,36(9):9-14.
[13] 闫东辉,许淑珍,苏建荣,等.肠球菌gyrA基因突变和多重耐药泵与氟喹诺酮耐药性关系研究[J].首都医科大学学报,2012,33(2):161-164.
[14] Jiménez P A,Gómez J E,Garcíade los Ríos A,et al.Molecular basis of quinolone resistance inEscherichiacolifrom wild birds[J].Can J Vet Res,2004,68:229-231.
[15] Johnning A,Kristiansson E,Fick J,et al.Resistance mutations in gyrA and parC are common in escherichia communities of both fluoroquinolone-polluted and uncontaminated aquatic environments[J].Front Microbiol,2015,12(6):1-9.
[16] Basu S,Mukherjee S K,Hazra A,et al.Molecular characterization of uropathogenicEscherichiacoli:nalidixic acid and ciprofloxacin resistance,virulent factors and phylogenetic background[J].J Clin Diagn Res,2013,7(12):2727-2731.
[17] Sheehan J.Effects of a Mutation in the gyrA gene on the virulence of uropathogenicEscherichiacoli[J].Antimicrob Agents Chemother,2015,59(8):1235-1239.
[18] 刘 果,纪 雪,孙 洋,等.宠物源大肠埃希菌的分离鉴定和耐药性研究[J].动物医学进展,2016,37(2):120-124.
[19] 阮周曦,唐金明,徐 鹏,等.进境冻畜禽产品中产超广谱β-内酰胺酶大肠埃希菌耐药性的监测与分析[J].动物医学进展,2013,34(9):6-11.
[20] Korona-Glowniak I,Skrzypek K,Siwiec R,et al.Fluoroquinolone-resistance mechanisms and phylogenetic background of clinicalEscherichiacolistrains isolated in south-east Poland[J].New Microbiol,2016,39(3):1-17.
[21] Mosquito S,Pons M J,Riveros M,et al.DiarrheagenicEscherichiacoliphylogroups are associated with antibiotic resistance and duration of diarrheal episode[J].Sci World J,2015,2:1-6.
[22] Piatti G,Mannini A,Balistreri M,et al.Virulence factors in urinaryEscherichiacolistrains:phylogenetic background and quinolone and fluoroquinolone resistance[J].J Clin Microbiol,2008,46(2):480-487.
[23] Aldred K J,Kerns R J,Osheroff N,et al.Mechanism of quinolone action and resistance[J].Biochemistry,2014,53(10):1565-1574.
Detection and Analysis of gyrA Gene in Fluoroquinolone-resistant AvianEscherichiacoli
LU Qin,LUO Qing-ping,ZHANG Teng-fei,WANG Hong-cai,LUO Ling,WANG Hong-lin,WEN Guo-yuan,ZHANG Rong-rong,SHAO Hua-bin
(InstituteofAnimalHusbandryandVeterinary,HubeiAcademyofAgriculturalSciences,KeyLaboratoryofPreventionandControlAgentsforAnimalBacteriosis,HubeiKeyLaboratoryofAnimalEmbryoEngineeringandMolecularBreeding,Wuhan,Hubei,430064,China)
This paper discussed the distribution and mutation characteristics of gyrA gene and drug resistance of quinolones resistant gene in different avianEscherichiacoli.The quinolone resistance test of 9E.coliwere determinded by the K-B method.After PCR amplification of gyrA gene,sequencing,the softwares as DNA MAN,DNA STAR,MEGA6 were used to analyze the results.The result of drug sensitivity test showed that strains of C1,C2 and C3 were sensitive to levofloxacin,ofloxacin,ciprofloxacin and norfloxacin,strains of D1,D2,D3, B1,B2 and B3 were resistant and intermediate to levofloxacin,ofloxacin,ciprofloxacin and norfloxacin.Sequence analysis of gyrA gene showed 1 mutation in the nucleotide in strain B1 and 14 mutations in stain B2;gyrA gene showed 4 mutations in the amino acids in strain B2,which resulted in 87 Ile →Val,101 Leu→Met,102 Ala→Ser and 129 Lys→ Gln.Homology and phylogenetic tree analysis of 9 strains of avianEscherichiacolishowed fluoroquinolone-resistant strains from different avianE.coli,compared with other 9 strains,the homology of gyrA gene of B2 strain was at 90%,phylogenetic tree was not in a same branch.B2 strain in this resarch provided candidate strain to study the fluoroquinolone-resistance mechanism ofE.coli.
avianEscherichiacoli;fluoroquinolone;drug-resistance strain;gyrA gene
2016-08-26
国家重点研发计划项目 (2016YFD0501305,2016YFD0500800);公益性行业(农业)科研专项项目(201303044); 现代农业产业技术体系建设专项资金项目(CARS-42-G11); 湖北省农业科学院青年科学基金项目(2016NKYJJ16)
卢 琴(1989-),女,湖南常德人,研究实习员,硕士,主要从事禽病防控研究工作。*通讯作者
S852.612
A
1007-5038(2017)04-0052-06
