水城县米箩乡中华蟾蜍的分子鉴定
2017-04-04熊荣川喻廷君李正富任梅梅詹秀秀
熊荣川 喻廷君 李正富 曹 东 任梅梅 詹秀秀
(1六盘水师范学院生物科学与技术学院,贵州六盘水553001;2六盘水市生物研究所,贵州六盘水553001;3贵州大学昆虫研究所,贵阳550025;4水城县石丫口小学,贵州六盘水553000)
中华蟾蜍(Bufo gargarizans)隶属蟾蜍科(Bufonidae)蟾蜍属(Bufo),包含三个亚种,即指名亚种(Bufo gararizans gararizans)、华西亚种(Bufo gararizans andrewsi)以及岷山亚种(Bufo gararizans minshanicus)。中华蟾蜍在贵州省记载有两个亚种,即指名亚种(Bufo gararizans gararizans)及华西亚种(Bufo gararizans andrewsi)。指名亚种分布于遵义、绥阳、正安、仁怀、赤水、江口、印江、德江、松桃、水城、毕节、大方、金沙、雷山、贵定、贵阳、务川等地;而华西亚种分布于威宁、兴义、荔波、绥阳、江口等(费梁等,2009)。已有的分子遗传研究报道了绥阳、江口和威宁种群的分子数据,3个种群分别属于不同的支系(Fu J Z等,2005;Zhan A B,Fu J Z,2011;熊荣川 等,2014)。本研究针对采自贵州省水城县米箩乡的蟾蜍标本进行DNA提取,并克隆其线粒体16S rRNA基因,对其物种进行准确而有效的鉴定。
1 材料与方法
1.1 标本信息
研究材料于2014年6月1日采自贵州省水城县米箩乡石丫口民裕小学附近(标本号:LPSSC130563、LPSSC130564),标本保存于六盘水师范学院生物科学与技术学院标本馆。
1.2 总DNA提取及目的基因片段的扩增
取标本腿部新鲜肌肉组织适量,经90%浓度酒精固定后,使用动物组织DNA提取试剂盒(FOREGENE,DE-05011:250 Preps)提 取 总DNA,-20℃保存备用。
扩增引物为1对无尾两栖动物通用的线粒体16S rRNA片段扩增引物(Simons C等,1994)。所扩增目的序列对应中华蟾蜍线粒体基因组(Genbank索取号KM587710)2135-2675bp区间位置,对应中华蟾蜍16S rRNA全基因(Genbank索取号KM587710)856-1396bp区间位置。
PCR反应条件:反应体积为50 ul,其中北京全式金公司配备反应缓冲液2×EasyTaq PCR Super-Mix 25 ul,总DNA模板2 ul(含10~100 ng),上、下游引物各2ul(10 uM),用ddH2O补足50 ul。扩增PCR反应程序为94℃预变性4 min;94℃变性40 s,52℃退火40 s,72℃延伸40 s,循环次数为35次;72℃再延伸10 min。PCR产物经1%琼脂糖凝胶电泳检测后送测序公司测序。
1.3 参考基因序列下载及系统发育分析
所测得的中华蟾蜍16S基因序列经过上传GenBank进行搜索比对(Megablast)获得100条初步的参考序列,与待定基因序列一起构成数据集。进行系统发育分析时不预先设定外群,构建一棵无根系统发育树。用MUSCLE(Edgar R C,2004)程序对序列进行比对,辅以人工校对。使用raxmlGUΙ1.3(Silvestro D,Michalak Ι,2012)软件构建最大似然树,使用Mrbayes3.2(Ronquist F等,2012)构建贝叶斯树。在jModelTest 2(Darriba D等,2012)中筛选最适合该数据集序列演化模型以供最大似然法(Maximum likelihood,ML)分析和贝叶斯分析(Bayesian inference methods)。根据AΙC标准,适合本数据集的模型TΙM2+Ι+G。使用构建最大似然树(maximum likelihood tree),由于该软件不支持TΙM2+Ι+G模型,使用次优模型GTR+Ι+G,其中Ι=0.462,G=0.567。最大似然树支持率大于75%表明该支系关系得到充分解决,在50%~70%之间为中度支持,否则视为未解决。贝叶斯树支持率大于95%表明该支系关系得到充分解决,在75%~95%之间为中度支持,否则视为未解决。
1.4 遗传距离的计算
选用MEGA6(Tamura K等,2013)分析各序列间差异,依据Kimura2-parameter模型计算两两序列间的遗传距离以及主要支系间的平均遗传距离。
2 研究结果及分析
2.1 PCR扩增及测序结果
经1%琼脂糖凝胶电泳检测,引物P7/P8扩增到560bp左右的基因片段(GenBank索取号:KX870122,KX870123)。基因序列(单链)碱基组成存在明显的偏向性:AT含量较高(A:30.6%,T:25.5%);GC含量较低(G:19.7%,C:24.2%)。
2.2 支系分化
将所测得蟾蜍16S基因序列提交Genbank进行搜索比对,其与Genbank中的已有中华蟾蜍序列相似度最高,下载搜索得到的100条同源序列与待鉴定目标序列构成16S序列数据集。基于该数据集构建的系统发育树中(图1),56条来自Bufo bufo和Bufo eichwaldi参考序列聚为一个单系,将之设置为外群;另外包含目标序列在内的46条序列构成一个支持率较高的单系,该单系在系统发育分析中作为内群(各序列的Genbank索取号及采集地点已标注于图1)。
基于最大似然法和贝叶斯法构建的系统发育树拓扑结构基本一致(图1),内群内部均聚为7个主要支系:支系L.A包含了中华大蟾蜍水城种群、中国东部、北部地区种群以及四川康定和丹巴两种群(支系内遗传距离均值0.008);支系L.B主要包含了中华大蟾蜍俄罗斯远东种群、韩国种群以及中国东北部地区种群(支系内遗传距离均值0.003);支系L.C主要包含了中华大蟾蜍云南昆明种群;支系L.D主要包含了中华大蟾蜍云南怒江、中甸及四川石棉种群(支系内遗传距离均值0.005);支系L.S主要包含了分布于中国辽宁和韩国江原道地区的史氏蟾蜍(Bufo stejnegeri)种群(支系内遗传距离均值0);L1和L2为两个日本蟾蜍支系(支系内遗传距离均值分别为0.018和0.014)。
基于16S序列的遗传距离(表1)表明,内外群间平均遗传距离为0.059;内群各支系间平均遗传距离为0.028~0.051.

图1 基于16SrRNA的蟾蜍类物种的最大似然树。支系附近数字表示各主要支系的支持率(最大似然率/贝叶斯后验概率),-表示没有数据。Fig.1 The maximum likelihood(ML)tree based on the 16SrRNAfromBofospecies.Numbers around the branches are statistical supports(ML bootstrap values/Bayesian posterior probability),-means no available data.
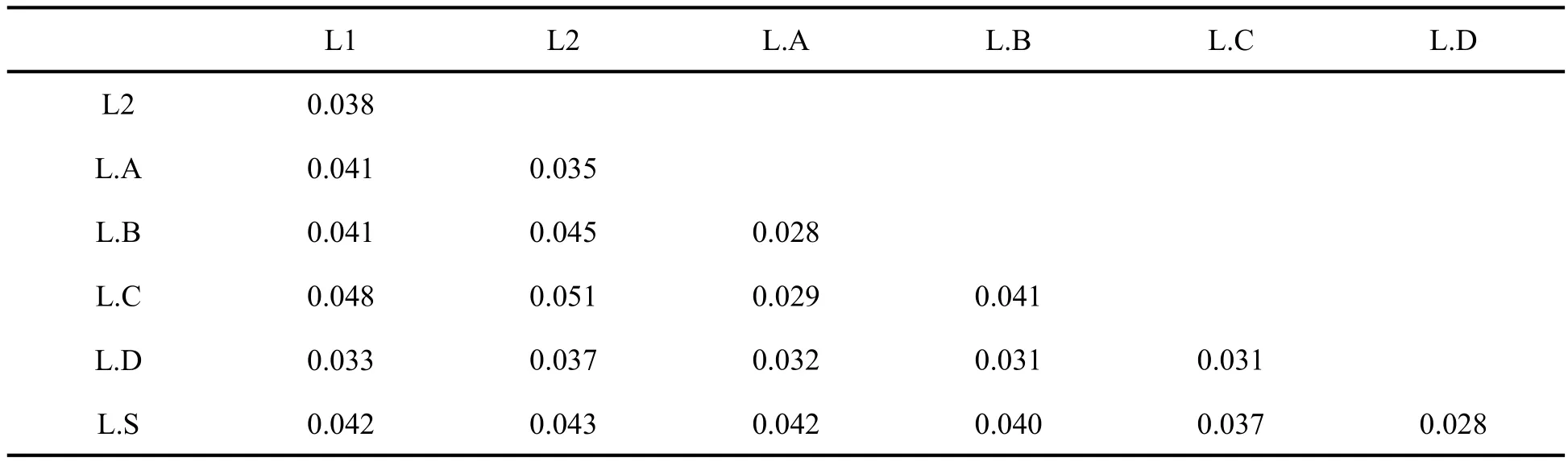
表1 本研究中内群各支系间平均遗传距离Table 1 The average genetic distances among the lineages of the ingroup in this study.
3 结论及讨论
本研究成功扩增了水城县米箩乡分布蟾蜍的线粒体16S rRNA基因,经过系统发育分析,其与44条蟾蜍序列构成一个支持率较高的单系群,该单系群进一步分化为7个主要支系,水城种群所在的L.A支系最大似然法支持率较低(41);但得到贝叶斯推断法的中度支持(0.83)。同时,该支系内部序列间平均遗传距离为0.008,远小于该支系与其他支系间的平均遗传距离(0.29~0.42),因此可以初步判断水城种群与该支系内其他种群为同一个物种。
支系L.A包含的序列分别被原始研究鉴定为中华蟾蜍(Bufo gargarizans)(Liu W Z等,2000;Jiang J P,Zhou K Y,2005;Cao S等,2006;Ιgawa T等,2006;Yang J等,2015)、盘古蟾蜍(Bufo bankorensis)(Liu W Z等,2000;Ιgawa T等,2006;Recuero E等,2012)、西藏蟾蜍(Bufo tibetanus)(Wang X等,2013)。然而有研究认为盘古蟾蜍和西藏蟾蜍是中华蟾蜍的同物异名(Matsui M,1986;Liu W Z等,2000;Zhan A B,Fu J Z,2011),因此支系L.A的物种应为中华蟾蜍,由此得出采自水城米箩乡的蟾蜍标本为中华蟾蜍(Bufo gargarizans)。
参考文献:
费梁,胡淑琴,叶昌媛.2009.中国动物志(两栖纲中卷无尾目)[M].北京:科学出版社,1-957.
熊荣川,田应洲,李松,等.2014.威宁中华蟾蜍的分子鉴定[J].贵州农业科学,42(12):32-36.
Cao S,Wu X,Yan P,et al.2006.Complete nucleotide sequences and gene organization of mitochondrial genome ofBufo gargarizans[J].Mitochondrion,6(4):186-193.
Darriba D,Taboada G L,Doallo R,et al.2012.jModelTest 2:more models,new heuristics and parallel computing[J].Nature Methods,9(8):772-772.
Edgar R C.2004.MUSCLE:multiple sequence alignment with high accuracy and high throughput[J].Nucleic acids research,32(5):1792-1797.
Fu J Z,Weadick C J,Zeng X M,et al.2005.Phylogeographic analysis of theBufo gargarizansspecies complex:Arevisit[J].Molecular Phylogenetics and Evolution,37(1):202-213.
Ιgawa T,Kurabayashi A,Nishioka M,et al.2006.Molecular phylogenetic relationship of toads distributed in the Far East and Europe inferred from the nucleotide sequences of mitochondrial DNA genes[J].Molecular phylogenetics and evolution,38(1):250-260.
Jiang J P,Zhou K Y.2005.Phylogenetic relationships among Chinese ranids inferred from sequence data set of 12S and 16S rDNA[J].The Herpetological Journal,15(1):1-8.
Liu W Z,Lathrop A,Fu J Z,et al.2000.Phylogeny of East Asian Bufonids Ιnferred from Mitochondrial DNA Sequences(Anura:Amphibia)[J].Molecular Phylogenetics and Evolution,14,(3):423-435.
Matsui M.1986.Geographic variation in toads of the Bufo bufo complex from the Far East,with a description of a new subspecies[J].Copeia,(3):561-579.
Recuero E,Canestrelli D,Voros J,et al.2012.Multilocus species tree analyses resolve the radiation of the widespread Bufo bufo species group(Anura,Bufonidae)[J].Molecular Phylogenetics and Evolution,62(1):71-86.
Ronquist F,Teslenko M,van der Mark P,et al.2012.MrBayes 3.2:efficient Bayesian phylogenetic inference and model choice across a large model space[J].Systematic Biology,61(3):539-542.
Silvestro D,Michalak Ι.2012.raxmlGUΙ:a graphical front-end for RAxML[J].Organisms Diversity&Evolution,12(4):335-337.
Simons C,Frati F,Beckenbach A,et al.1994.Evolution,weighting,and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers[J].Annals of the entomological Society of America,87(6):651-701.
Tamura K,Stecher G,Peterson D,et al.2013.MEGA6:Molecular Evolutionary Genetics Analysis Version 6.0[J].Molecular biology and evolution,30(12):2725-2729.
Wang X,Wang Y,Yue B,et al.2013.The complete mitochondrial genome of the Bufo tibetanus(Anura:Bufonidae)[J].Mitochondrial DNA,24(3):186-188.
Yang J,Liu J,Xue R,et al.2015.Characterization of the mitochondrial genome ofBufo gargarizansminshanicus(Anura:Bufonidae)[J].Mitochondrial DNA,27(5):3327-3328.
Zhan A B,Fu J Z.2011.Past and present:Phylogeography of theBufo gargarizansspecies complex inferred from multi-loci allele sequence and frequency data[J].Molecular phylogenetics and evolution,61(1):136-148.
