HIV⁃1耐药基因检测的研究进展
2017-03-30陈瑶吴英松
陈瑶 吴英松
HIV⁃1耐药基因检测的研究进展
陈瑶 吴英松★
HIV⁃1耐药是影响艾滋病抗病毒治疗效果的主要因素。随着抗病毒疗法的普及和接受治疗者的增多,HIV⁃1耐药的问题日益凸显,使艾滋病治疗面临巨大的挑战。因此,了解各抗病毒药物治疗引起的HIV⁃1耐药突变位点并及时采用有效方法进行耐药基因检测对临床治疗具有重要指导意义。HIV⁃1耐药位点基因型检测方法因其快速准确的特点,目前是HIV⁃1耐药检测最常用也是最有前景的方法。本文对HIV⁃1耐药位点和耐药基因检测的研究进展做简单综述。
HIV⁃1;耐药位点;基因型;耐药检测
艾滋病(acquired immune deficiency syndrome,AIDS)是由人免疫缺陷病毒(human immunodefi⁃ciency virus,HIV)引起的传染病,目前世界范围内主要流行HIV⁃1。HIV耐药是由于病毒基因发生突变,使得药物作用靶点的生化特点或者结构特征发生改变,导致病毒对抗病毒药物敏感性降低,或者失去敏感性[1⁃3]。随着高效联合抗逆转录病毒(highly active antiretroviral therapy,HAART)药物的广泛应用,导致HIV⁃1病毒发生突变。耐药突变株在药物选择压力下逐渐成为优势病毒株,导致耐药菌株感染的病例不断出现。因此,作为帮助临床医生选择联合用药方案的重要工具,耐药检测就显得十分重要。目前HIV⁃1耐药检测方法主要有表型检测和基因型检测。其中,基因型检测因其快速准确的特点,成为调整治疗方案的首选[3]。
1 耐药基因的分类
随着抗病毒治疗的发展及新型靶点抗病毒药物的临床应用,已确定了200多个各种类型HIV⁃1耐药相关的基因突变,它们对于耐药及病毒的生物学特性产生不同程度的影响[4⁃5]。其中有不少受到严重的关切,并且已经应用到临床的耐药位点筛查,下表(表1)列出了临床检测常用的HIV⁃1的耐药基因突变及其针对的药物[6⁃13]。临床医生可通过不同方法检测相应的位点,推测基因突变对临床耐药的影响,从而更好地指导临床药物的选择和调换。
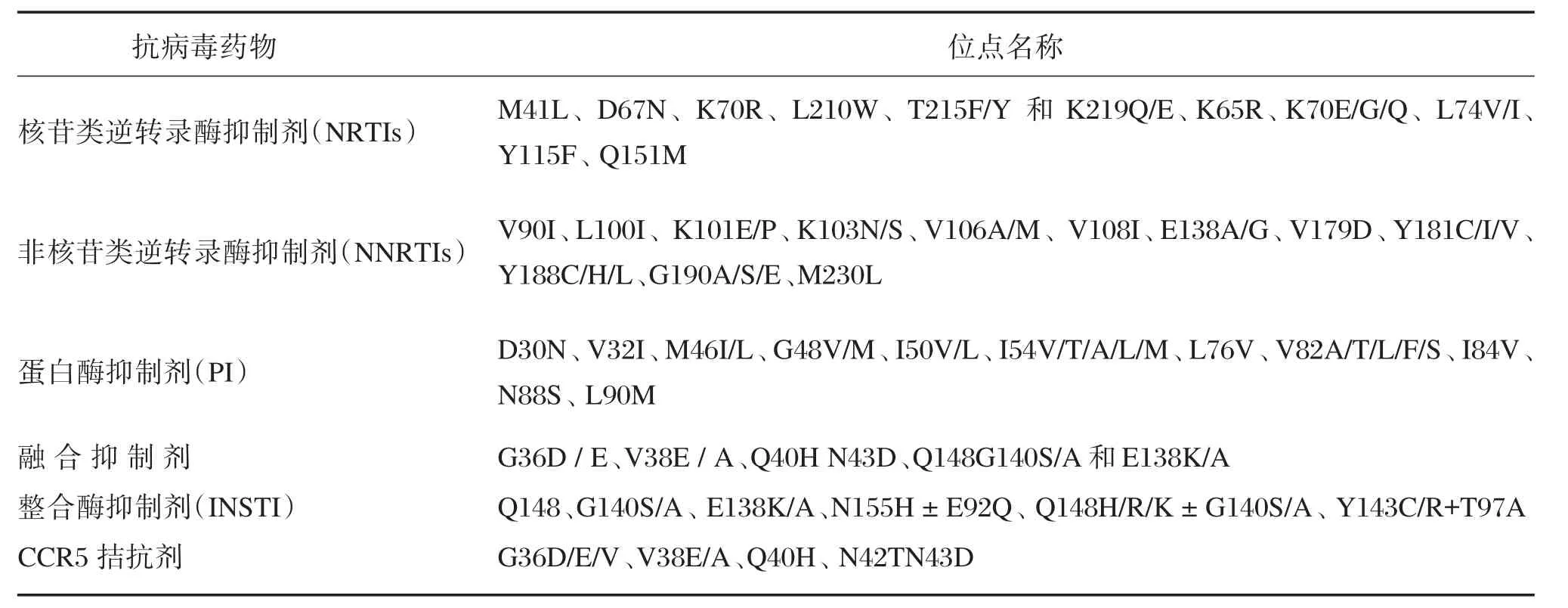
表1 HIV⁃1耐药基因检测靶点Table 1 Targets of HIV⁃1 drug⁃resistant gene detection
2 耐药基因的检测对象和检测时机
对HIV⁃1耐药及其临床意义的认识随着抗HIV药物的研发和应用逐步积累和深入。随着耐药菌株的不断出现,不管是新发的患者选择用药,还是治疗中的患者监控用药效果,都需要进行耐药性检测。
初始抗病毒治疗的患者应进行耐药检测,以指导并制定抗病毒治疗方案。已经证实耐药HIV⁃1毒株能够传播,因此现如今的观点是,从未治疗过的患者也可能携带耐药毒株,这将导致对初始治疗方案的无应答或应答差。对这类患者进行耐药检测有助于选择能够取得最大病毒抑制效果的治疗方案[14]。在治疗过程中,对于接受抗病毒治疗的患者在改变抗病毒治疗方案时,病毒学应答失败(HIV⁃1 RNA>1 000 copies/mL)或病毒抑制不理想时,耐药监测是判断是否发生耐药以及耐药类型,从而指导药物选择和调换进行后续治疗的必须手段[15⁃16]。
目前耐药性检测有两种方法,一种是表型耐药分析,需要对病毒进行体外培养,时间较长,操作繁复,价格昂贵。因此基因检测的快速准确的特点让其越来越成为临床指导药物选择的重要手段。尤其是在对抗病毒联合治疗效果不好,病毒学应答失败的患者的药物调整的过程中,快速准确的基因治疗有利于快速指导药物选择和调换[15⁃16]。
3 HIV⁃1耐药基因检测的常用方法
3.1 等位基因特异扩增PCR(Allele⁃specific PCR,AS⁃PCR)方法
AS⁃PCR是一种基于单核苷酸多态(single nu⁃cleotide polymorphism,SNP)的PCR标记方法。上世纪90年代首次报道使用AS⁃PCR方法检测HIV⁃1耐药突变,后经技术改良,该方法已越来越成熟,具有高灵敏度和准确度,重复性好,较为省时省力,快速且经济实惠,也可检测变异率低于1%的病毒准种等特点。Hunt等[17]采用AS⁃PCR和深度测序法同时检测了105例使用奈韦拉平预防失败的HIV⁃1感染患儿,两种方法均可检测到低水平的变异株,本研究发现两种方法检测突变位点Y181C和K103N的相关系数分别为0.94和0.89。但该方法存在一些不足,即需知道变异位点在核酸的具体位置;若引物或探针结合位点处出现多态性可显著降低该方法的准确性。另外,为了确保扩增效率相同,AS⁃PCR要求引物退火位置相同。这局限了这种方法应对不断出现的新的耐药位点,不利于临床综合判断解释耐药检测的结果。
3.2 限制性片段质谱多态性技术(restriction frag⁃ment mass polymorphism,RFMP)
限制性片段长度多态性技术的原理是检测DNA在限制性内切酶酶切后形成的特定DNA片段的大小,该技术将基质辅助激光解吸电离飞行时间质谱(matrix assisted laser desorption/ionization time⁃of⁃flight mass spectrometry,MALDI⁃TOF MS)技术结合,根据被测物质的相对分子质量进行定量检测。该方法敏感,准确,重复性好,能够发现数量不足1%的变异株。Lee等采用该技术检测100份血浆标本,并与Sanger测序法进行比较,发现RFMP方法要求的病毒载量水平更低,两种方法在检测逆转录酶和蛋白酶编码区的K65R、T69Ins/D、L74V/I、V106A/M、Q151M、Y181C、M184V/I、T215Y/F、D30N、M46I、G48V、I50V、I54L/S、V82A、I84V、L90M等位点时的一致性均≥94%[18]。但该方法仅能检测已知位点变异,且价格昂贵,很难在临床推广应用。
3.3 基因芯片技术
基因芯片技术是将大量特定的基因片段或寡核苷酸片段作为探针有序和高密度地排列,固定在玻璃或膜等载体上,然后与待测有标记的样品核酸按碱基配对的原则杂交然后检测结果。它借助荧光标记进行扫描观察,通量高,可实现大量SNP的检测。据报道,学者采用本方法共检测了来自瑞士和坦桑尼亚人群的HIV⁃1基因组片段序列,共获得2 550个SNP,与Sanger测序的一致性为92.7%[19]。本方法同样主要针对已知变异位点,现随着基因芯片高通量测序技术的发展,也可用于检测未知变异位点。但本方法的成本高,受实验条件所限,尚不能在实验室普及开展。
3.4 Sanger测序法
随着测序技术不断深入各级实验室,许多分子生物实验室具备Sanger测序的条件,其价格合理,且可用于自建方法分析,该方法已经作为HIV⁃1耐药检测的主要技术平台和金标准,在临床和科研中均已被采用。目前基于该技术原理已产生了两种成熟的用于HIV⁃1耐药检测的商业化系统TRUGENETM和ViroSeqTM以及实验室自建的In⁃house检测方法。耐药性的分析需要通过在线数据库完成,即用户将测序序列提交到数据库便可直接得到耐药突变分析结果。该方法便捷,但在碱基判读时设定的阈值较高[20],且灵敏度低,只有当HIV⁃1变异株超过准种池的20%时才可被检测到[21],即低频率的突变易发生漏检,从而低估耐药突变情况,影响耐药监测的结果。
3.5 深度测序
随着测序技术的快速发展,深度测序通量高,已在发达国家广泛应用于HIV⁃1耐药检测。该方法可精确检测到只发生在1%的HIV⁃1毒株中的耐药性突变[22],极大程度提高了检测的灵敏度,因低频突变对高效联合抗反转录病毒治疗(highly active antiroviral therapy,HAART)治疗的影响存在争议,比利时学者采用深度测序方法检测了70例接受NNRTI治疗患者的突变位点,发现该方法并不能确证逆转录区出现低频突变与治疗失败的关联性[23]。有比较发现相同样本采用深度测序可检测到的HIV⁃1基因型耐药位点数目是Sanger测序法的1.5~3倍[22]。但深度测序的检测质量受文库构建偏倚的影响,如引物结合位点耐药突变或多样性的出现或PCR扩增效应等。近年来深度测序技术已出现多种平台,其成本也已逐渐降低,不过对于低收入国家来说仍难以负担。此外,该技术前期手工操作步骤多,上机收集的数据量大,目前仍缺乏简单便捷的生物信息学分析流程。因此,深度测序尚不能在各实验室广泛开展应用于HIV⁃1耐药检测分析。
4 展望
患者服药依从性、药物间的相互作用等均可影响HIV⁃1耐药的产生。随着抗病毒药物的广泛应用,耐药问题凸显,出现的耐药突变位点极为多样,且突变位点间的相互作用也可引起耐药的发生,从而导致治疗失败。中低收入国家由于可使用的抗病毒治疗药物种类有限,主要存在NRTI、NNRTI耐药现象,约有10%~30%在接受NRTI或NNRTI治疗过程中出现病毒抑制失败,大多因出现了耐药病毒株[1⁃2]。虽然PI、INSTI等药物在临床应用尚少,耐药率明显低于欧美发达国家,但随着条件许可,可使用的抗病毒药物逐渐增加。因此,有必要加强了解突变位点与各类耐药的相关性,以助于临床提供准确的治疗方案。此外,HIV⁃1耐药检测已被发达国家纳入监测指标,它既可为初始抗病毒治疗也可为已出现病毒学抑制失败的后续治疗选择或更换药物提供指导[3⁃4]。多数情况下推荐使用基因型检测,因其相对操作简便、省时、花费少,在野生毒株与耐药毒株混合存在的情况下具有较高的灵敏性。在常见的基因型检测方法中,质谱和基因芯片的方法因为成本较高较少应用于临床。随着成本降低及条件优化,未来传统的标准检测方法有望被深度测序所取代[21⁃22]。
总而言之,与其他病毒性疾病的临床检验方法相比,HIV⁃1基因型耐药检测的程序复杂,影响因素多,受HIV⁃1固有的高度变异和准种的影响,结果的解释比较困难。检测出耐药基因是一方面,后续复杂和繁琐的生物信息学处理以获得与结果的解释也存在极大的复杂性和难度。存在解释的结果与临床状况不能一致的现象[19⁃20]。近年来随着深度测序技术的不断发展关于基因型变异的数据大量积累,前瞻性检验设计和回顾性临床研究的不断发展,将有助于改进分析方法,更好的理解突变、耐药和临床治疗应答3方面的关系,为耐药基因检测更好地应用于临床提供更有力的支持。
[1]Sluis⁃Cremer N,Wainberg MA,Schinazi RF.Resis⁃tance to reverse transcriptase inhibitors used in the treatment and prevention of HIV⁃1 infection[J].Fu⁃ture Microbiology,2015,10(11):1773⁃1782.
[2]Rhee SY,Liu TF,Holmes SP,et al.HIV⁃1 Subtype B Protease and Reverse Transcriptase Amino Acid Co⁃variation[J].PLoS Comput Biol,2007,3(5):e87.
[3]Tang MW,Shafer RW.HIV⁃1 Antiretroviral Resis⁃tance[J].Drugs,2012,72(9):e1⁃e25.
[4]Wensing AM,Calvez V,Gunthard HF,et al.2015 Update of the Drug Resistance Mutations in HIV⁃1[J]. Topics in Antivir Med,2015,23(4):132⁃141.
[5]TenoRes Study Group.Global epidemiology of drug re⁃sistance after failure of WHO recommended fi rst⁃line regimens for adult HIV⁃1 infection:a multicentre retro⁃spective cohort study[J].Lancet Infect Dis,2016,16(5):565⁃575.
[6]Melikian GL,Rhee SY,Varghese V,et al.Non⁃nu⁃cleoside reverse transcriptase inhibitor(NNRTI)cross⁃resistance:implications for preclinical evaluation of novel NNRTIs and clinical genotypic resistance testing[J].J Antimicrob Chemother,2014,69(1):12⁃20.
[7]Codoñer FM,Pou C,Thielen A,et al.Added Value of Deep Sequencing Relative to Population Sequencing in Heavily Pre⁃Treated HIV⁃1⁃Infected Subjects[J]. PloS One,2011,6(5):e19461.
[8]Shadrina OA,Zatsepin TS,Agapkina YY,et al.Influ⁃ ence of drug resistance mutations on the activity of HIV⁃1 subtypes A and B integrases:a comparative study[J].Acta Naturae,2015,7(1):78⁃86.
[9]Blanco JL,Varghese V,Rhee SY,et al.HIV⁃1 inte⁃grase inhibitor resistance and its clinical implications[J].J Infect Dis,2011,203(9):1204⁃1214.
[10]Geretti AM,Armenia D,Ceccherini⁃Silberstein F.Ce⁃ccherini⁃Silberstein,Emerging patterns and implica⁃tions of HIV⁃1 integrase inhibitor resistance[J].Curr Opin Infect Dis,2012,25(6):677⁃686.
[11]Menzo S,Castagna A,Monachetti A,et al.Genotype and phenotype patterns of human immunodeficiency vi⁃rus type 1 resistance to Enfuvirtide during long⁃term treatment[J].Antimicrob Agents Chemother,2004,48(9):3253⁃3259.
[12]Skhosana L,Steegen K,Bronze M,et al.High preva⁃lence of the K65R mutation in HIV⁃1 subtype C infect⁃ed patients failing tenofovir⁃based first⁃line regimens in South Africa[J].Plos One,2015,10(2):e0118145.
[13]Skhosana L,Steegen K,Bronze M,et al.High preva⁃lence of the K65R mutation in HIV⁃1 subtype C infect⁃ed patients failing tenofovir⁃based first⁃line regimens in South Africa[J].Plos One,2015,10(2):e0118145.
[14]Rhee SY,Jordan MR,Raizes E,et al.HIV⁃1 Drug Resistance Mutations:Potential Applications for Point⁃of⁃Care Genotypic Resistance Testing[J].Plos One,2015,10(12):e0145772.
[15]Rimsky L,Vingerhoets J,Eygen VV,et al.Genotyp⁃ic and phenotypic characterization of HIV⁃1 isolates ob⁃tained from patients on rilpivirine therapy experiencing virologic failure in the phase 3 ECHO and THRIVE studies:48⁃week analysis[J].J Acquir Immune Defic Syndr,2012,59:39⁃46.
[16]Lathouwers E,Meyer SD,Dierynck I,et al.Virologi⁃cal characterization of patients failing darunavir/ritona⁃vir or lopinavir/ritonavir treatment in the ARTEMIS study:96⁃week analysis.Antiviral Therapy,2011,16(1):99⁃108.
[17]Hunt GM,Morris L,Moorthy A,et al.Concordance between allele⁃specific PCR and ultra⁃deep pyrose⁃quencing for the detection of HIV⁃1 non⁃nucleoside re⁃verse transcriptase inhibitor resistance mutations[J].J Virol Methods,2014,207:182⁃187.
[18]Lee JH,Hachiya A,Shin SK,et al.Restriction frag⁃ment mass polymorphism(RFMP)analysis based on MALDI⁃TOF mass spectrometry for detecting antiretro⁃viral resistance in HIV⁃1 infected patients[J].Clin Mi⁃crobiol Infect,2013,19(6):E263⁃270.
[19]Masimba P,Gare J,Klimkait T,et al.Development of a simple microarray for genotyping HIV⁃1 drug re⁃sistance mutations in the reverse transcriptase gene in rural Tanzania[J].Trop Med Int Health,2014,19(6):664⁃671.
[20]Ji H,Liang B,Li Y,et al.Low abundance drug resis⁃tance variants in transmitted HIV drug resistance sur⁃veillance specimens identified using tagged pooled py⁃rosequencing[J].J Virol Methods,2013.187(2):314⁃320.
[21]Garcia⁃Diaz A,Guerrero⁃Ramos A,McCormick AL,et al.Evaluation of the Roche prototype 454 HIV⁃1 ul⁃ tradeep sequencing drug resistance assay in a routine diagnostic laboratory[J].J Clin Virol,2013,58(2):468⁃473.
[22]Chang MW,Oliveira G,Yuan J,et al.Rapid deep se⁃quencing of patient⁃derived HIV with ion semiconduc⁃tor technology[J].J Virol Methods,2013,189(1):232⁃234.
[23]Messiaen P,Verhofstede C,Vandenbroucke I,et al. Ultra⁃deep sequencing of HIV⁃1 reverse transcriptase before start of an NNRTI⁃based regimen in treatment⁃naive patients[J].Virology,2012,426(1):7⁃11.
Advances in the drug resistance genotype detection of HIV⁃1
CHEN Yao,WU Yingsong★
(School of Laboratory Medicine and Biotechnology,Southern Medical University,Guangzhou,Guangdong, China,510515)
Anti⁃retroviral therapy(ART)can help reduce viral loads and infection⁃associated morbid⁃ity.It can also prolong survival,prevent transmission,and improve clinical outcomes.However,with the spread of ART and increasing numbers of patients receiving treatment,more and more HIV⁃1 drug resistant strains have appeared,which brings about a great challenge to the therapy of acquired immune deficiency syndrome (AIDS).Therefore,to be familiar with and to detect HIV⁃1 resistance mutations caused by various anti⁃virus drugs in a timely manner will be of great significance for clinical treatment.In this review,we will summarize the advances in the detection of HIV⁃1 resistance mutations and genotypes.
HIV⁃1;Drug resistance mutation;Genotype;Drug⁃resistance detection
重大传染病创新检测试剂的研制和应用研究(广州市协同创新重大专项,201400000004⁃1)作者单位:南方医科大学检验与生物技术学院,广东,广州510515
★通讯作者:吴英松,E⁃mail:wg@smu.edu.cn
