内蒙古自然发酵绵羊奶油中乳酸菌的分离鉴定及优势菌群q-PCR定量分析
2017-01-04
(内蒙古农业大学乳品生物技术与工程教育部重点实验室,呼和浩特010018)
内蒙古自然发酵绵羊奶油中乳酸菌的分离鉴定及优势菌群q-PCR定量分析
刘红新,德亮亮,陈红霞,刘文俊,孙天松
(内蒙古农业大学乳品生物技术与工程教育部重点实验室,呼和浩特010018)
采用传统纯培养方法对内蒙古不同地区的12份绵羊奶油样品中的乳酸菌进行分离纯化,运用16S rDNA序列分析方法进行属种鉴定,同时采用实时荧光定量聚合酶链反应(q-PCR)技术对包头地区样品中优势菌群数量进行了定量研究。分离鉴定结果表明三个地区绵羊发酵奶油中分离鉴定的44株乳酸菌,其中Lactococcus lactis subsp.Lactis为优势菌群。q-PCR定量结果表明3种优势菌属的数量关系为Lac.Lactis.subsp.lactis>L.plantarum>Leu.Mesenteroides。
乳酸菌;分离鉴定;实时荧光定量聚合酶链反应(q-PCR);天然发酵奶油
0 引言
自然发酵奶油(Naturally fermented cream),蒙古语又称Zhouhei,是从牛奶、羊奶中提取的黄色或白色脂肪性半固体食品[1],富含人体所需多种脂肪酸及有益脂类,赋予乳制品良好的风味,改善乳制品的口感[2]。然而,关于发酵奶油的系统性研究还较少,特别是对自然发酵羊奶油中微生物的研究更少。
实时荧光定量聚合酶链式反应(real-time quanti⁃tative polymerase chain reaction,q-PCR)是将普通PCR和光谱分析、实时检测等手段巧妙结合到一起的一项技术[3]。在乳酸菌定量分析中较传统活菌计数具有快速、准确、高效等[4]优势。
本研究运用16S rDNA序列分析方法对内蒙古不同地区绵羊奶油样品中的乳酸菌进行种属鉴定,并结合实时荧光定量PCR技术进行分析与研究,不仅丰富中国的乳制品市场,也为乳酸菌资源的开发与利用提供有力依据。
1 实验
1.1材料与试剂
本研究中12份奶油样品由实验室的研究人员于2014年6月14日-2014年6月17日在内蒙古鄂尔多斯、包头、巴彦淖尔三个地区采集(采集条件:温度为18~23℃,pH值在4.5以下,2~6 d发酵)。样品采集后低温保存,于-80℃超低温保藏,进行后续实验。
菌株分离、传代培养基分别为MRS固体和MRS液体培养基分离、纯化乳杆菌;M17固体和M17液体培养基用于分离、纯化、培养乳球菌。
实验所用试剂:PBS保护液;PBS缓冲液,所用试剂级缓冲液均参照文献[5]要求配制;DNA提取试剂,DNA片段胶回收纯化试剂,PCR扩增及凝胶电泳所用试剂和酶类和Marker。
16S rRNA基因的PCR扩增引物序列参考细菌通用引物序列[6]并进行了完善[7]。用于菌株q-PCR特异性引物采用Primer5软件合成,引物序列如表1所示。
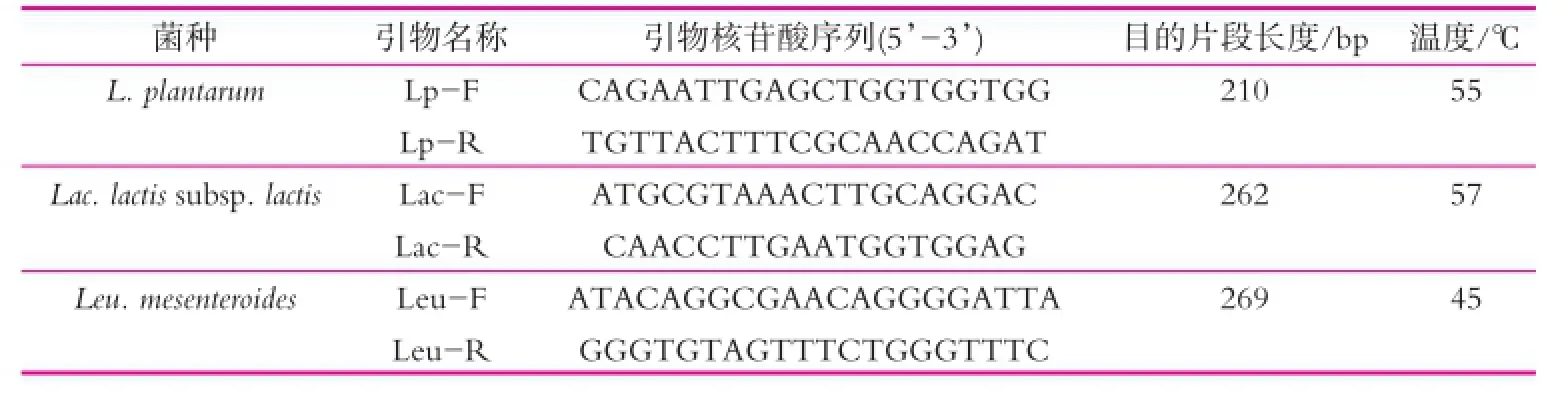
表1 q-PCR特异性引物信息
1.2仪器与设备
便携式冰箱、温度计、便携式PH计、采样箱,ZHJH-C1214C型超净工作台;TGL-168高速台式离心机;HWS28型电热恒温水浴锅;AR2202CN型电子天平;KDC-1044型低速离心机;LRH-250生化培养箱;BX50型光学显微镜;HA-300M型全自动高压蒸汽灭菌器;SP-650型全自动高压干热灭菌器;WD-9405B型水平摇床;LL-6SFPY型真空冷冻干燥机;PTC-200梯度基因扩增仪;DYY-12型电泳仪;GDS-8000型凝胶成像仪;ND-1000型微量紫外分光光度计等。
1.3样品采集及乳酸菌分离、鉴定
1.3.1 样品采集
本研究在采集奶油样品时采用直接取样法。将样品充分混匀,吸取样品至装有灭菌中和剂(内含0.5 g,CaCO3/淀粉=1/50,质量比)的无菌螺口冻存管中,混匀后标记样品号并作记录,封口保存。最后将采集的样品置于4℃便携式冰箱中以维持其低温状态,进行乳酸菌的分离纯化等试验。
1.3.2 样品中乳酸菌的计数和分离
乳酸菌的活菌计数,采用倾注培养法[8]。将奶油样品梯度稀释(10-4,10-5,10-6),后涂于MRS和M17固体培养基上,随机选择不同菌落形态的单菌落进行乳酸菌的初步分析,选取过氧化氢阴性、革兰氏阳性的纯培养物,将此培养物暂定为乳酸菌,然后进一步运用16S rDNA序列分析方法进行属种鉴定[9]。
1.3.3 16S rDNA序列测序鉴定
采用液氮反复冻融-CTAB法提取乳酸菌基因组DNA[10],再用ND-1000型微量紫外分光光度计测其浓度以及OD260/280值。
PCR扩增体系:5 μL 10×EasyTaq Buffer(Mg2+);4 μL High Pure dNTPs(2.5 mMol/L);1.5 μL引物FA-27F(10 mmol/L);1.5 μL引物RA-1495R(10 mmol/L);0.5 μL EasyTaq DNA polymerase(5 U/μL);2 μL DNA模板(100 ng/μL);35.5 μL ddH2O。
PCR扩增循环参数为:94℃预变性5 min;94℃变性1 min,58℃退火1 min,72℃延伸2 min,30个循环;72℃末端延伸10 min。
扩增完毕后,取约2 μL的PCR扩增产物用质量分数为1.0%的琼脂糖凝胶电泳检测,若在1 500 bp处有清晰的扩增条带且无拖尾、弥散现象,则PCR扩增成功。将PCR扩增产物送往上海美吉生物医药有限公司进行纯化和DNA测序。将测序得到的DNA序列运用SeqMan(DNAStar 5.01)软件进行序列拼接后,再利用NCBI中的BLAST将拼接好的序列与数据库中已鉴定的乳酸菌16S rDNA序列进行同源性分析[11],将待鉴定菌株序列与同源性最高的模式菌株序列应用MEGA 5.0(http://www.megasoftware.net)软件构建系统发育树,进行遗传进化研究。
1.4样品中优势菌群的q-PCR定量分析
1.4.1 绵羊奶油样品及标准菌株宏基因组DNA的提取
绵羊奶油样品宏基因组DNA的提取采用玻璃珠吸附法,具体方法根据参考文献[12]。将提取的DNA原液稀释至100 ng/μ L左右,作为备用。标准菌株L. plantarum(ATCC14931)、Leu.mesenteroides(AS1.544)、Lac.lactis subsp.lactis(ATCC19435)用液体MRS培养基活化培养后,收集菌体,采用SDS-CTAB液氮反复冻融法提取标准菌株的基因组DNA[13],并检测其纯度和浓度。采用ND-1000型微量紫外分光光度计检测标品DNA原液的OD值和浓度,并对其进行十倍稀释。
以模式菌株DNA为阳性对照,以标准菌株的10-1~10-8梯度稀释液为模板构建q-PCR定量标准曲线。,以绵羊奶油样品的宏基因组DNA稀释液为样品模板,进行样品中优势菌群-q-PCR定量分析,设置两个重复,并以ddH2O为阴性对照模板,采用特异性引物按照表2中的定量PCR反应体系及扩增方法在实时荧光定量仪上进行PCR扩增。反应结束后,利用StepOne v2.3软件制作标准曲线。以标准曲线为依据,对样品中的优势乳杆菌属菌种进行定量分析。

表2 q-PCR扩增体系
实时定量PCR采用两步法,具体扩增条件如下:预热阶段:95℃20 s;循环阶段:95℃、20 s预变性,然后95℃变性5 s,退火40 s,72℃延伸50 s,共40个循环;扩增阶段:95℃(15 s),75℃(1 min)。
2 结果与分析
2.1地区绵羊奶油样品中乳酸菌计数结果
内蒙古不同地区绵羊奶油样品中乳酸菌计数结果如表3所示。
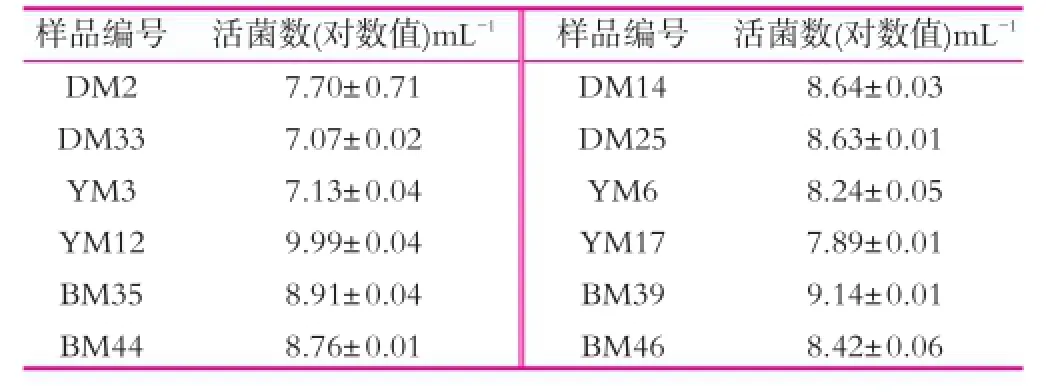
表3 内蒙古不同地区绵羊奶油中乳酸菌计数结果
内蒙古不同地区(包头、巴彦淖尔、鄂尔多斯)奶油中乳酸菌的活菌数见表3,从中可以看出,本研究奶油样品活菌数均大于107mL-1,在7.07~9.99 mL-1(对数值)之间,平均值为(8.38±0.09)mL-1(对数值)。可见内蒙古三个地区的绵羊奶油制品新鲜度较高,菌株存活能力较强,有利于下一步的分离鉴定及研究。张哲等[14]人对俄罗斯卡尔梅克地区酸奶油样品进行研究,其中乳酸菌活菌数在7.30~8.62 mL-1(对数值)之间。张文羿等[15]对11份青海海西州酸山羊奶中的乳酸菌进行研究,活菌数在8.40~9.48 mL-1(对数值)之间。张冬蕾[16]等人对内蒙古鄂尔多斯地区酸羊奶进行分析,活菌数在7.78~9.84 mL-1(对数值)之间。包秋华等[17]对13份云南酸山羊奶的研究表明其乳酸菌活菌数为8.32~10.34 mL-1(对数值)之间。由此可见,采样地点不同,其中乳酸菌活菌数量存在差异。即使同一地区采样,随着发酵和贮藏时间的延长,自然发酵乳制品中微生物乳酸菌种属和数量也会有变化。
2.2乳酸菌分离鉴定结果
根据乳酸菌的形态学特征及理化性质[18]初步将44株菌定为乳酸菌。
2.2.1 DNA提取和PCR扩增结果
DNA浓度和OD260/280值的测定:提取44株乳酸菌的DNA并测其浓度以及OD260/280值,OD260/280值在1.8~2.0之间即为纯DNA样品。将DNA原液稀释至浓度为100 ng/μL后进行16S rRNA基因的PCR扩增。扩增结果经1.0%琼脂糖凝胶电泳检测后,如图1可以观察到在大约1 500 bp的位置处有一条清晰明亮的条带,无拖尾、弥散现象,未发现明显非特异扩增现象,说明分析菌株的16S rRNA基因扩增成功。将PCR扩增产物送于上海美吉生物科技有限公司进行测序。
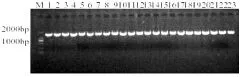
图1 部分分离株16S rRNA基因的PCR扩增产物琼脂糖凝胶电泳
图1中,1~23为部分分离株16S rRNA基因的PCR产物;M为DL 2000 DNA Markers。
2.2.2 16S rRNA基因同源性比对及系统发育树构建
将测序获得的16S rRNA基因序列通过NCBI数据库中BLAST进行同源序列比对分析,若同源性高于99%则可直接将其归为此种模式株菌种,选取部分具有代表性的菌株用MEGA 5.0(http://www.megas⁃oftware.net)软件构建系统发育树。分离株与模式株系统发育树如图2所示(选取部分作图)。
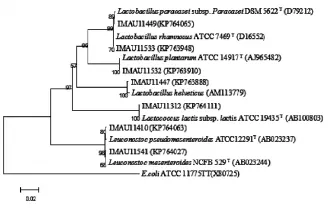
图2 内蒙古不同地区代表性分离株的16S rRNA基因序列的系统发育树
由图2可以看出,所有分离株集中为杆菌,IMAU11449(KP764065)与模式菌Lactobacillus paracasei subsp.Paracasei DSM 5622T(D79212)聚为一类,同源性达99%以上,将其鉴定为Lactobacillus paracasei subsp.Pa⁃racasei。IMAU11533(KP763948)与模式菌Lactobacillus rhamnosus ATCC 7469T(D16552)聚为一类,同源性达99%以上,因此将归其为 Lactobacillus rhamnosus。IMAU11532(KP763910)与模式菌Lactobacillus planta⁃rum ATCC 14917T(AJ965482)聚为一类,同源性为99%,故将其鉴定为Lactobacillus plantarum。IMAU11447 (KP763888)与模式菌Lactobacillus helveticus(AM113779)聚为一类,同源性达99%以上,故鉴定为Lactobacillus helveticus。IMAU11312(KP764111)与模式菌Lactococ⁃cus lactis subsp.lactis ATCC 19435T(AB100803)聚为一类,同源性达99%以上,故将其鉴定为Lactococcus lactis subsp.lactis。IMAU11410(KP764063)与模式菌Leuco⁃nostoc pseudomesenteroides ATCC12291T(AB023237)聚为一类,同源性为99%以上,故鉴定为Leuconostoc pseudo⁃mesenteroides。IMAU11541(KP764027)与模式菌Leuco⁃nostoc mesenteroides NCFB 529T(AB023244)聚为一类,同源性达99%以上,故将其归为Leuconostoc mesenteroi⁃des。根据16S rDNA序列分析,分离的44株菌均与模式菌株的同源性达到99%以上。
2.2.3 内蒙古不同地区绵羊奶油中乳酸菌的优势菌种分析
研究表明,12份绵羊奶油中共分离出44株乳酸菌,分别为3个属,7个种或亚种。内蒙古不同地区绵羊奶油中乳酸菌的分离结果如表4所示。
由表4可以看出,分离的44株乳酸菌中Lactococcus lactis subsp.lactis数量最多,12份样品中8份样品均有乳酸亚种,共分离15株,占总分离株的34%,为三个地区绵羊奶油中的优势菌种。12份样品中4份样品分离到了Lactobacillus helveticus,共分离13株,占总分离株的29%,为三个地区次优势菌种。其中鄂尔多斯地区优势菌种为Lactobacillus helveticus,包头、巴彦淖尔地区优势菌种为Lactococcus lactis subsp.lactis。许多研究表明,不同地区和种类传统乳制品中的乳酸菌组成存在一定的差异。Mourad等[19]人对采集自阿尔及利亚撒哈拉地区的20份传统奶油中乳酸菌进行研究,结果显示,Lactobacillus plantarum为优势菌种。杜晓华等[20]人对采集于蒙古国牧民家庭的17份发酵乳样品中乳杆菌进行分离鉴定,发现Lactobacillus ermentum为样品中优势菌群。由此可见不同地区、不同地理环境优势菌种不同,即使同一地区采样时间、贮藏时间等不同,结果也会有差异。

表4 内蒙古不同地区绵羊奶油中乳酸菌的分离结果
2.3样品中优势菌群定量分析
提取绵羊奶油样品中的宏基因组DNA,并应用紫外分光光度计检测样品中宏基因组DNA的浓度和纯度,结果显示,所有分离株的基因组DNA的浓度均在于100~2000 ng/μL之间,纯度值(A260/A280比值)在1.8~2.0范围内。分离株基因组DNA的琼脂糖凝胶电泳结果显示,大约在23 Kb处有清晰、明亮的条带,且无拖尾和弥散现象,由此可知样品中宏基因组DNA满足后续试验要求。将胶回收后的标准品进行稀释,作为模板对相对应的标菌进行qPCR扩增,结果表明3种标准曲线的相关系数(R2)均达到0.99以上,满足后续实验要求。包头地区绵羊奶油中Lac.lac⁃tis.subsp.lactis、Leu.mesenteroides、L.plantarum的基因拷贝数,如图3所示。

图3 绵羊奶油样品中优势菌种群的定量结果比较
由图3可以看出,Lac.lactis subsp.lactis的基因拷贝数在包头市奶油中平均达到(8.31±0.10)×108copies/ mL,Leu.mesenteroides的基因拷贝数在包头市奶油中平均达到(3.94±0.25)×103copies/mL,L.plantarum的基因拷贝数在包头市奶油中平均达到(5.78±0.27)×105cop⁃ ies/mL,3种优势菌属的数量关系为Lac.Lactis.subsp. Lactis>L.plantarum>Leu.Mesenteroides,且3个样品均符合这一数量关系。
3 结论
本研究对内蒙古不同地区的12份自然发酵绵羊奶油中的乳酸菌进行分离鉴定和定量研究,样品中活菌数平均为(8.38±0.09)mL-1(对数值)。共分离出44株菌,通过16S rDNA基因同源性分析,将44株鉴定为3个属,7个种或亚种。其中Lactococcus lactis subsp. Lactis为三个地区绵羊奶油中的优势菌种,占总分离株的34%。同时采用实时荧光定量PCR(q-PCR)技术对包头地区样品中优势菌群数量进行了比较分析,结果表明包头地区绵羊奶油中,3种优势菌属的数量关系 为 Lac.Lactis.subsp.Lactis>L.plantarum>Leu.Mesen⁃teroides。
[1]ZHANG H P.Traditional Dairy Products for Chinese Minorities(Re⁃view).In:Asian Indigenous Dairy Products.Bulletin of The Interna⁃tional Dairy Federation,2007,415:21-26.
[2]史瑛,赵建新,田丰伟,陈卫.天然发酵稀奶油的乳酸菌菌种选育与风味图谱测定[A].中国食品科学技术学会.乳酸菌与营养健康:第九届乳酸菌与健康国际研讨会摘要汇编[C].中国食品科学技术学会, 2014:2.
[3]路虎,杜海,徐岩,杜小威,王栋.荧光定量PCR法定量白酒固态发酵过程中产土味素的链霉菌[J].微生物学通报,2014,04:786-792.
[4]邓文星,张映.实时荧光定量PCR技术综述[J].生物技术通报,2007, 05:93-95.
[5]杨彦荣,任艳,德亮亮,陈红霞,张冬蕾,刘文俊,张和平.传统奶酪样品中乳酸菌的分离鉴定[J].中国乳品工业,2015,09:19-22.
[6]MAURO SCARPELLINI(A1);Diego Mora(A2);Silvia Colombo (A1);Laura Franzetti(A1).Developmentofgenus/species-specific PCR analysis for identification of Carnobacterium strains.[J].Current Microbiology.2002(NO.1).
[7]张和平,孙志宏,张家超,等.一种适合乳酸菌16SrRNA序列测定的引物设计[P].内蒙古:CN101705305A,2010-05-12.
[8]PANG H L,ZHANG M,QIN G Y,TAN Z F,LI Z W,WANG Y P,CAI Y M.Identification of lactic acid bacteria isolated from corn stovers[J].Animal Science Journal.2011(No.5).
[9]樊哲新,李宝坤,李开雄,等.传统分离培养结合DGGE技术研究新疆传统发酵酸驼乳中乳酸菌的多样性[J].中国食品学报,2015,04: 208-217.
[10]YU J,SUN Z H,LIU W J,et al.Phylogenetic study of Lactobacillus acidophilus group,L.casei group and L.plantarun group based on partial hsp60,pheS and tuf gene sequences[J].European Food Re⁃search and Technology,2012,234(6):927-934.
[11]ALTSCHUL S F,MADDEN T L,SCHÄFFER A A,et al.Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J].Nucleic acids research,1997,25(17):3389-3402.
[12]方光伟,洪雪梅,蔡丽希,彭锟,林毅.土壤宏基因组的提取及基于免培养技术分析细菌16S rDNA[J].江西农业大学学报,2005,04: 505-507.
[13]孙志宏,刘文俊,张和平.基于宏基因组方法对西藏传统发酵牦牛奶中微生物多样性的研究[J].北京工商大学学报(自然科学版),2012, 04:19-24.
[14]张哲,徐海燕,于洁.俄罗斯卡尔梅克共和国酸奶油中乳酸菌的多样性分析[J].中国乳品工业,2015,02:37-39+43.
[15]ZHANG W Y,YUN Y Y,SUN T S,et al.Isolation and identifica⁃tion of dominant microorganisms involved in naturally fermented goat milk in Haixi region of Qinghai,China[J].Annals of microbiolo⁃gy,2008,58(2):213-217.
[16]张冬蕾,任艳,德亮亮,陈红霞,杨彦荣,刘文俊,孙天松.内蒙古鄂尔多斯地区羊鲜乳与酸乳中乳酸菌的分离鉴定[J].中国乳品工业, 2015,07:30-34.
[17]B Q H,C X,L H X,et al.Isolation and identification of cultivable lactic acid bacteria from traditional goat milk cake in Yunnan prov⁃ince of China[J].African Journal of Microbiology Research,2011,5 (29):5284-5291.
[18]东秀珠,蔡妙英.常见细菌系统鉴定手册[M].北京:科学出版社, 2001:289-294.
[19]MOURAD K,NOUR-EDDINE K.Physicochemical and microbi⁃ological study of“shmen”,a traditional butter made from camel milk in the Sahara(Algeria):isolation and identification of lactic acid bacte⁃ria and yeasts[J].Grasas y Aceites,2006,57(2):198-204.
[20]杜晓华,艾日登才次克,李莉,等.蒙古国地区酸乳中乳酸菌的鉴定及耐酸菌株筛选[J].微生物学通报,2009,36(7):994-1000.
Isolation and identification of Lactic acid bacteria from naturally fermented sheep cream in Inner Mongolia as well as quantification of predominant species by q-PCR
LIU Hong-xin,DE Liang-liang,CHEN Hong-xia,LIU Wen-jun,SUN Tian-song
(Key Laboratory of Dairy Biotechnology and Engineering,Ministry of Education,Inner Mongolia Agricultural Universi⁃ty,Hohhot 010018,China)
In this study,Lactic acid bacteria(LAB)were isolated and purified by traditional pure culture method from 12 samples of naturally fermented sheep cream collected in Inner Mongolia,and then identified by 16S rDNA sequencing and phylogenetic analysis method.Mean⁃while,quantitive real-time quantity PCR(q-PCR)was applied to compare and quantitatively analyze the predominant species in traditional dairy products in Baotou,The results showed that the content of three predominant species was in the order of Lac.Lactis.subsp.lactis>L. plantarum>Leu.mesenteroides.in these sheep cream samples.
Lactic acid bacteria;isolation and identification;q-PCR;naturally fermented cream
Q939.11+7
A
1001-2230(2016)09-0016-04
2016-04-30
刘红新(1991-),女,硕士研究生,研究方向为微生物生理与发酵工艺工程。
孙天松
