水牛全血基因组抽提方法优化
2016-12-24陆杏蓉庞春英邓廷贤段安琴梁贤威
陆杏蓉,庞春英,邓廷贤,朱 鹏,段安琴,梁贤威
(中国农业科学院广西水牛研究所 广西水牛遗传繁育重点实验室,南宁 530001)
水牛全血基因组抽提方法优化
陆杏蓉,庞春英,邓廷贤,朱 鹏,段安琴,梁贤威*
(中国农业科学院广西水牛研究所 广西水牛遗传繁育重点实验室,南宁 530001)
[目的]通过比较三种提取水牛全血基因组DNA的方法,获得一种安全、高效、快速的DNA提取方法,为水牛基因功能的研究奠定基础。[方法]收集43头水牛抗凝血样,每份血样随机分为等量3份,每份血样2 mL,分别使用试剂盒改良法、试剂盒说明书法及酚仿抽提法提取全血基因组 DNA。比较分析三种方法提取的同一头牛三份血样DNA的含量、OD260/280值、琼脂糖凝胶电泳和基因扩增结果。[结果]三种方法提取DNA的含量由高到低依次为试剂盒改良法、酚仿抽提法、试剂盒说明书法,各组间差异显著(1376.72±127.54 VS 1121.32±81.64 VS 326.18±21.17,\%P\%<0.05)。三种方法提取DNA的OD-260/280值依次增加,试剂盒改良法与试剂盒说明书法OD260/280值差异不显著(1.85±0.0017 VS 1.86±0.0021,\%P\%>0.05),与酚仿抽提法OD260/280值有显著差异(1.85±0.0017 VS 1.88±0.0029,1.86±0.0021 VS 1.88±0.0029,\%P\%<0.05)。三种方法提取 DNA 琼脂糖凝胶电泳结果显示,试剂盒改良法和试剂盒说明书法DNA条带清晰、光密度高,酚仿抽提法条带模糊有拖带现象。三种方法所提取的基因组PCR扩增stat5基因获得较清晰准确的条带,扩增效果良好。[结论]试剂盒改良法用于水牛全血基因组的提取比试剂盒说明书法和酚氯仿法更安全、高效、便捷。
水牛血液;基因组抽提;试剂盒改良法;试剂盒说明书法;酚仿抽提法
随着人类基因组全序列的公布,分子生物学研究取得了突飞猛进的发展,人们对基因的研究也越来越深入,而模板 DNA 提取的质量是进行基因组研究的基本前提[1]。在分子遗传学实验中,用于提取 DNA的样本多种多样,包括血液、唾液、尿液、粪便、脑脊液以及组织等[2]。其中,血液可以作为基因组 DNA 的便捷来源,从血液中提取全基因组 DNA 是进行各种分子遗传学实验的基础,聚合酶链反应、限制性内切酶处理、突变检测、基因型分型以及连锁分析等遗传学实验对于DNA的质和量都有较高的要求[3]。因此使用一种操作简单、快捷高效的提取方法对于进一步的基因分型及分子育种具有重要意义。目前,许多实验室都开展了基因组方面的研究工作,对于基因组DNA的提取各实验室也采用不同的提取方法,本研究将对3种常规的 DNA提取方法[酚氯仿法,硅胶膜吸附法(天根血液基因组DNA提取试剂盒-离心柱型),改良的硅胶膜吸附法]进行比较分析,从而获得一种安全、高效、快速的DNA提取方法。
1 材料与方法
1.1 试验材料、试剂与仪器
无水乙醇、Tris-酚、氯仿、蛋白酶 K、EDTA、ddH2O、DNA 提取试剂盒离心柱型 (北京天根生化科技公司)、超微量核酸分析仪(Nanodrop 2000 美国)、离心机(Thermo Sorvall ST16R 美国)、移液器(Eppendorf 德国)、恒温水浴锅、凝胶成像系统(UVI FireReader 英国)。
1.2 试验方法
1.2.1 样本处理方法 新鲜水牛血样2 mL (肝素钠抗凝)加2 mL红细胞裂解液,混匀,室温放置至上清为透明红色,离心去上清,重复 3~4次至沉淀为白色。
1.2.2 试剂盒改良法 血液样本经细胞裂解液Buffer CL(NH4Cl)处理至白色沉淀后置于4℃过夜,除了裂解过程,其它步骤均按照天根血液基因组 DNA 提取试剂盒说明书操作,所得DNA 溶于70 μL ddH2O中。
1.2.3 离心柱型试剂盒抽提法 全程完全按照天根血液基因组 DNA 提取试剂盒说明书操作,所得DNA溶于70 μL ddH2O中。
1.2.4 酚仿抽提法 裂解过夜后,离心,收集白色沉淀,加入600 μL DNA抽提缓冲液,20 μL 蛋白酶K,55℃ 过夜,期间颠倒混匀数次。次日加入600 μL饱和酚,垂直混匀10 min,12 000 r/min 离心10 min,取上清。300 μL饱和酚与300 μL氯仿混匀,加入上步所得上清,垂直混匀10 min,12 000 r/min离心10 min,取上清。加入600 μL氯仿,垂直混匀10 min,12 000 r/min 离心10 min,取上清。加入1 200 μL 无水乙醇,离心,取絮状沉淀,用70%酒精洗涤,风干后溶于70 μL ddH2O中。
1.2.5 统计学分析 三种方法所抽提DNA的浓度及纯度用超微量核酸分析仪(Nanodrop 2000 )检测,结果用SPSS软件进行显著性分析,\%P\%<0.05认为差异显著,\%P\%>0.05为差异不显著。DNA含量(ng)=浓度(ng/μL)×体积(μL);DNA纯度= OD260/280
2 结果与分析
2.1 不同方法抽提水牛全血基因组含量
运用超微量核酸分析仪检测比较水牛DNA提取液浓度, 计算出2 mL所提取的水牛血液DNA含量,试剂盒改良法所提取DNA含量显著高于其它两种方法,如表1。
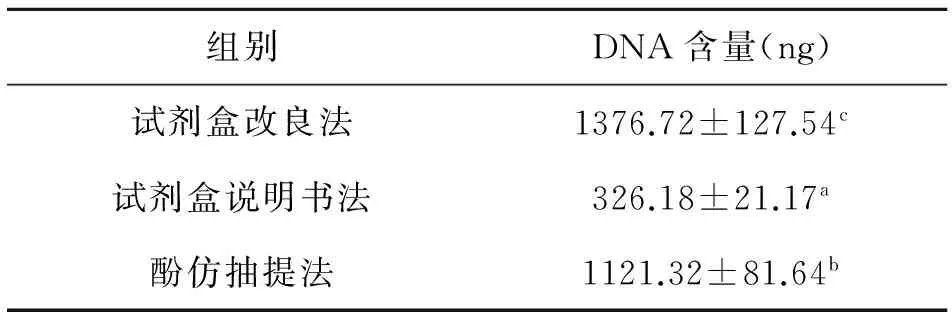
表1 不同方法抽提水牛全血基因组含量比较
2.2 不同方法抽提水牛全血基因组纯度比较
运用超微量核酸分析仪检测所提取的水牛血液DNA OD260/280,三组纯度OD260/280都在1.8附近,而试剂盒法更趋近1.8显著优于酚仿抽提法,如表2。
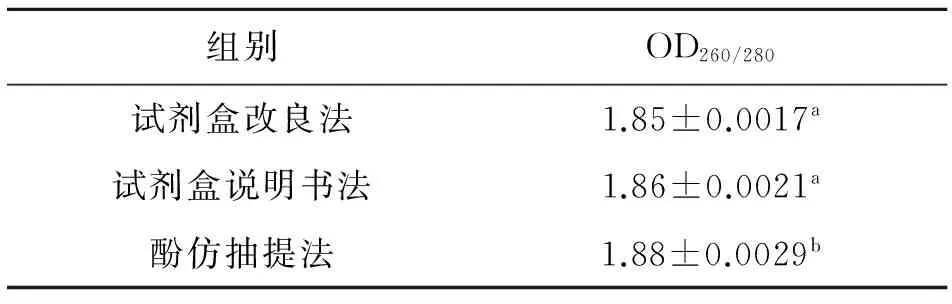
表2 不同方法抽提水牛全血基因组OD260/280比较
2.3 琼脂糖凝胶电泳检测
使用 1%的琼脂糖凝胶进行电泳,将DNA样品与加样缓冲液按1:1的比例混匀加入加样孔中,当溴酚蓝移到距凝胶前沿1~2 cm 时,停止电泳,取出凝胶,在 245 nm 紫外灯下照相记录电泳图谱,图1.A显示琼脂糖电泳DNA带型整齐、集中、无拖尾现象,说明所提取基因组DNA完整无降解;图1.B琼脂糖电泳DNA带型整齐、集中、个别样本有拖尾现象;图1.C琼脂糖电泳DNA带型略有拖尾现象。
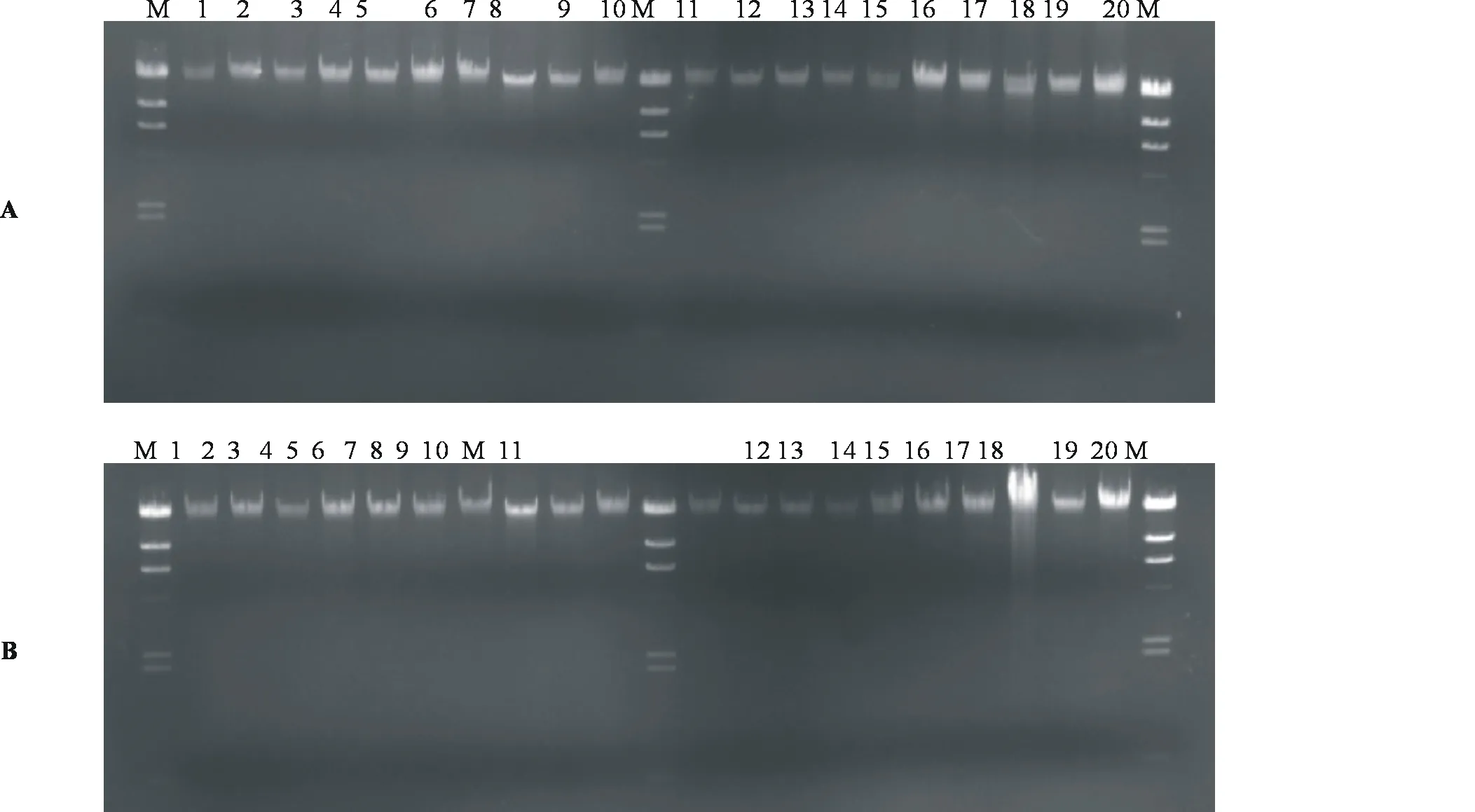
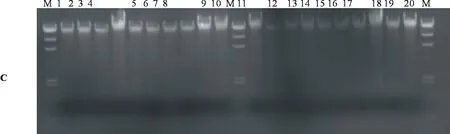
图1 不同抽提方法所得DNA琼脂糖凝胶电泳结果
2.4 PCR扩增检测
以三组提取的基因组为模板进行stat5基因PCR扩增,利用1%琼脂糖凝胶电泳检测结果如图2所示。
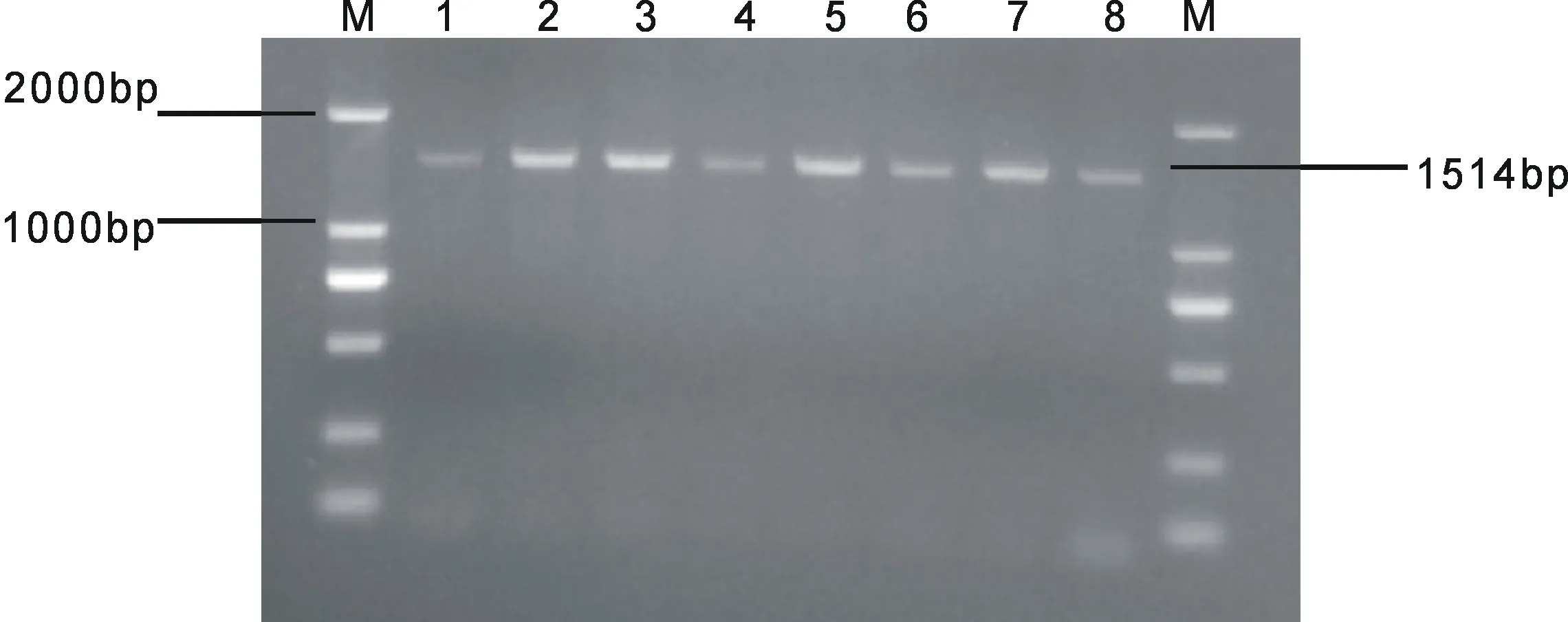
图2 三种方法提取基因组的stat5基因PCR扩增
3 讨论
3.1 试剂盒改良法在提取效果上的优势
核酸提取是下游操作和分析的前提,高质量核酸的获取是下游基因组实验成功进行的关键因素之一。哺乳动物血液由血浆和血细胞构成,而血细胞中又包括红细胞、白细胞和血小板三类细胞,这三类细胞中只有白细胞含有细胞核,核内含有遗传物质基因组DNA[4]。本实验中,试剂盒改良法主要通过充分裂解红细胞,从而达到了提高基因组提取浓度和纯度的效果(表1)。王大明等[5]报道了血液样本反复冻融虽对提取基因组的纯度无影响但却降低了基因组数量(浓度)。本试验中改良法采用了裂解过夜的方法,如若不能及时做后续处理,则可在4℃较长时间保存,既可使裂解充分,又不影响后面的操作,避免了血液样本未能及时处理反复冻融造成提取基因组的损失。值得注意的是,该法裂解过夜并非简单添加裂解液放置过夜,而是将首轮裂解离心沉淀后的红-白细胞混合物再次添加裂解重悬放置过夜,目的在于使白细胞核基因组的充分释放,如若多次离心沉淀后再裂解过夜则有可能在这一过程中损失了大量的DNA。从产出量方面考虑,酚仿抽提法和试剂盒改良法提取的DNA产量最高,而采用酚仿抽提法提取的DNA纯度却不及试剂盒改良法。经琼脂糖凝胶电泳检测(图1)后发现试剂盒改良后并不影响DNA的质量,对后续的基因组研究无不良影响,电泳图中有些条带略有抹带现在可能是由于DNA浓度过高或者基因组发生降解的缘故,本试验的不足之处在于没有进一步探讨其中的原因。从PCR扩增结果看,图2为stat5基因PCR扩增结果(1514 bp),三种方法提取的基因组DNA均可作为PCR扩增模版,且扩增效果良好,条带清晰,片段大小准确。由此可见试剂盒改良法可以确保获得稳定的DNA质量和数量,保证实验室DNA模板的正常使用。
3.2 试剂盒改良法在在时效性和安全性上的优势
从时效性看,完全按照试剂盒说明书提取便捷省时,单个样品可在30min内操作完毕[6],但是提取的DNA 浓度较低。酚仿抽提法所提取的DNA基本没有蛋白质残余,适合做各种分析,但操作繁琐,抽提步骤多,耗时长,这一点与陈丽霞等报道相符[7]。试剂盒改良法除了裂解部分需过夜外,其它操作与试剂盒说明书操作方法无异。从安全环保方面看,氯仿对人体中枢神经系统、心、肝、肾、皮肤具有损害,对环境、水体可造成污染,易爆,疑可致癌。低剂量的酚对皮肤、粘膜有强烈腐蚀作用,抑制中枢神经系统,损害肝、肾功能,高剂量可致死。酚仿抽提法使用酚试剂对人体有伤害,试剂盒说明书提取和改良法避免了使用有毒的苯酚、氯仿等有毒试剂,减少了操作过程中对操作人员身体健康的损害且有助于环保。
4 结论
酚仿抽提法提取DNA效率高、成本低,但对人体危害大,不安全;试剂盒法操作简便、省时,但成本高,效率较低;而经试剂盒法改良的基因组抽提方法能够大幅度提高水牛血液基因组的提取效率,是一种安全、高效、便捷、快速的 DNA 提取方法,具有推广价值。
[1] 王小飞, 陈玉红和王惠民. 人类基因组 DNA 四种不同抽提方法比较[J]. 陕西医学检验, 2001,16(4):9-10.
[2] TAN S C and YIAP B C. DNA, RNA, and protein extraction: the past and the present[J]. BioMed Research International, 2009.
[3] SHAMS S S, VAHED S Z, SOLTANZAD F, et al. Highly effective DNA extraction method from fresh, frozen, dried and clotted blood samples[J]. BioImpacts: BI, 2011,1(3):183.
[4] 成令忠, 组织与胚胎学[M]. 1990, 北京:人民卫生出版社.
[5] 王大明, 李桢, 邹红岩等. 全血抽提基因组 DNA 数量与纯度影响因素的探讨[J]. 江西医学检验, 2005,23(4):297-298.
[6] 王春艳, 郑旭, 高月等. 两种动物组织 DNA 提取方法的比较[J]. 现代畜牧兽医, 2012,41(10):57-59.
[7] 陈丽霞, 张振昶,谢小冬,等. 3 种提取全血基因组 DNA 的方法比较[J]. 基因组学与应用生物学, 2014. 33(5):1110-1113
Optimization of Methods for Genomic Extraction of Buffalo Blood
LU Xing-rong, PANG Chun-ying, DENG Ting-xian, ZHU Peng, DUAN An-qin, LIANG Xian-wei*
(GuangxiBuffaloResearchInstituteofChineseAcademyofAgriculturalScience,TheKeyLaboratoryofBuffaloGeneticsBreedingandReproductioninGuangxi,Nanning,Guangxi, 530001,China)
【Objective】The aim of this study was to obtain an security, efficient and rapid DNA extraction method by comparing three genomic DNA extraction methods of buffalo, which would lay the foundation for gene function in buffalo.【Method】 In this study, a total of 43 anticoagulant blood samples of buffalo were collected. Eachblood samples were randomly divided into three equal parts, and the volume of every part is 2 mL. Modified blood DNA Kit method, Blood DNA Kit instruction method and phenol-chloroform extraction method were used to extract the genomic DNA from blood sample of same individual, respectively. Then, several contents, including the content of DNA, OD260/280, agar sugar gel rubber electrophoresis and gene amplification, were compared by using these three extraction methods. 【Result】 A significantly difference was found in the content of DNA by using these three extraction methods, and the order from high to low was the modified Blood DNA Kit method, phenol extraction and Blood DNA Kit instruction method (1376.72 ± 127.54 VS 1121.32 ± 81.64 VS 326.18 ± 21.17, \%P\%<0.05). Moreover, comparing the value of OD260/280, it was added by order, and there was not significant difference between modified Blood DNA Kit method and Blood DNA Kit instruction method (1.85±0.0017 VS 1.86 ± 0.0021,\%P\%>0.05), but it was significant differences among the phenol-chloroform extraction and other two groups (1.85±0.0017 VS 1.88±0.0029, 1.86±0.0021 VS 1.88±0.0029,\% P\%< 0.05). Interestingly, the results of agarose gel electrophoresis by using these three DNA extraction groups showed that the DNA strap of modified Blood DNA Kit method and Blood DNA Kit instruction method was clear and high opticaldensity, but the DNA strap of phenol extraction method was fuzzy and towing. By amplifing stat5 gene, the results of three methods showed clear and accurate straps, and had good amplification effect consistently.【Conclusion】Compared with the Blood DNA Kit instruction method and phenol-chloroform extraction method, modified Blood DNA Kit method is a safer , more efficient and more convenient method for genomic extraction of buffalo blood.
buffalo blood;genomic extraction;modified Blood DNA Kit method;Blood DNA Kit instruction method;phenol-chloroform extraction method
2015-12-10 修改日期:2015-12-30
农业部转基因重点项目(2014ZX08010-012B);水牛基金(1504004)
陆杏蓉(1987-),广西百色人,女,壮族,实习研究员,硕士研究生,研究方向:动物繁殖生物技术,luxingrong074@163.com.
梁贤威(1962-),研究员,主要从事动物繁殖生物技术研究,liangbri@126.com。
S813
A
1001-9111(2016)02-0028-04
