广西PRRSV分离株GXNN1396全基因序列分析
2016-12-16黄加滨洪绍锋林思远何荣祥黄伟坚韦祖樟
黄加滨,韦 莹,洪绍锋,林思远,何荣祥,陈 樱,黄伟坚,韦祖樟*
(1.广西大学动物科学技术学院动物传染病研究室,广西南宁 530005;2.广西陆川县陆透水库管理所,广西陆川 537700)
研究论文
广西PRRSV分离株GXNN1396全基因序列分析
黄加滨1,韦 莹1,洪绍锋1,林思远1,何荣祥2,陈 樱1,黄伟坚1,韦祖樟1*
(1.广西大学动物科学技术学院动物传染病研究室,广西南宁 530005;2.广西陆川县陆透水库管理所,广西陆川 537700)
前期我们实验室分离鉴定了一株nsP2蛋白缺失49个氨基酸(aa)的猪繁殖与呼吸综合征病毒(PRRSV)GXNN1396株。为了进一步对该毒株全基因组序列特性进行分析,将GXNN1396株病毒在Marc-145细胞上传至第3代,收集细胞上清,抽提该病毒RNA。用RT-PCR检测方法对GXNN1396基因组进行分段扩增、克隆、序列比较和遗传进化分析。结果表明,GXNN1396全长15263核苷酸(nt),不包括5′ 帽子结构和3′Poly A 尾。其中,5′非翻译区(5′UTR)长为189nt,3′UTR长151nt,ORF1a、ORF1b、ORF2、ORF3、ORF4、ORF5、ORF6、ORF7分别为7422、4382、771、765、537、603、525nt和372nt。与国内外的PRRSV毒株核苷酸序列的同源性进行了比较显示,GXNN1396与欧洲经典毒株LV和VR2332株的同源性分别为61%和88.8%,与TJ、JXwn06、JXA1在全基因上的同源性高达97.2%~97.3%,说明GXNN1396株是一株典型的美洲型PRRSV毒株。遗传进化分析表明美洲型PRRSV主要分为3个亚群,GXNN1396与高致病性PRRSV毒株属于同一分支。
猪繁殖与呼吸综合征病毒;全基因序列分析;同源性分析;遗传进化分析
猪繁殖与呼吸综合征(Porcine reproductive and respiratory syndrome,PRRS)是猪繁殖与呼吸综合征病毒(Porcine reproductive and respiratory syndrome virus,PRRSV)引起的仔猪呼吸困难、体重下降、生长缓慢及母猪繁殖障碍为特征的一种接触性传染病,俗称“猪蓝耳病”。该病于1987年在北美首次暴发,现在已呈世界性分布。PRRSV为有囊膜、不分节段、单股正链RNA病毒,其基因组全长约有15kb,5′非翻译区(untranslated region,5′UTR)长约为190核苷酸(nucleotide,nt),3′UTR长约151nt。病毒编码区至少有9个开放阅读框(open reading frame,ORF)。左端(上游)占基因组总长3/4的开放阅读框1(open reading frame 1,ORF1)编码RNA依赖性RNA聚合酶(RdRp)等13个推测的非结构蛋白(nsP)。PRRSV基因组3′端结构蛋白基因区可编码至少8个ORF,其中,ORF2a、ORF3、ORF4和ORF5分别编码 GP2a、GP3、GP4和GP5等囊膜糖蛋白。ORF2b、ORF5a、ORF6和ORF7分别编码病毒非糖基化囊膜E蛋白、ORF5a蛋白、膜基质蛋白M和核衣壳蛋白N[1]。
在前期研究中,利用Marc-145细胞对从广西南宁市某猪场采集的RT-PCR检测PRRSV为阳性的肺脏组织进行病毒分离,分离到1株病毒,并命名为NN1396株[2]。同时,鉴定了GXNN1396为1株nsP2含有49aa缺失的PRRSV毒株;在与其他国内外的高致病性猪蓝耳病毒株在nsP2区除了1+29aa缺失外,上游还缺失了19个氨基酸(57nt)。由于RNA病毒易于发生抗原突变,近年来在世界各地出现了致病力增强的PRRSV变异株,表现为PRRSV感染怀孕母猪更加严重的流产和仔猪高病死率[3]。GXNN1396株在广西养猪业如此密集的区域是否为优势流行毒株的研究有重要意义,因此本研究对GXNN1396毒株的全基因组、核苷酸同源性和遗传进化了比较和分析。
1 材料与方法
1.1 材料
1.1.1 病毒、细胞和菌株 PRRSV毒株GNNN1396为广西大学传染病实验室分离、鉴定和保存;Marc-l45细胞、大肠埃希菌DH5α由本实验室保存;pMD18-T 载体为宝生物工程(大连)有限公司产品。
1.1.2 主要试剂 新生胎牛血清、MEM为GIBCO公司产品;Taq Mix、dNTP 、Mixture、RNase Inhibitor、M-MLV、LA Taq酶等为宝生物工程(大连)有限公司产品;DNA 凝胶回收试剂盒为Axyden Scientific,Inc产品。
1.1.3 引物设计 本研究使用的引物参考已发表文献引物[4],其中PF1/PR1203、PF1154/PR2379、PF2108/PR4238、PF4070/PR5373、PF5349/PR6663、PF6574/PR7976、PF7894/PR9232、PF9122/PR10481、PF10378/PR11569、PF11322/PR12186、PF11759/PR13401、PF13138/PR14371、PF14231/PR15300作为引物进行PCR扩增。
1.2 方法
1.2.1 病毒基因组的提取和RT-PCR扩增与克隆 第3代的病毒感染细胞后的上清液,并抽提病毒基因组RNA,以病毒RNA作为模板,用随机引物,分段进行反转录,合成与RNA模板互补的cDNA链。cDNA(2μL)用H2O(14.2μL)、buffer(2.5μL)、dNTP(4μL),在LATaq酶(0.3μL)的作用下,以各段P1/P2(1/1μL)为引物,配成25μL体系进行PCR反应,同时设阴性对照。PCR扩增条件:94℃ 1min;95℃ 0.5min,55℃ 0.5min,72℃ 1.5min,30个循环;72℃ 10min。PCR产物进行电泳,对所获得正确的PCR纯化产物与pMD18-T 载体4℃连接过夜、转化。孵育完成后,涂板到LA培养基上培养12h。挑取5个~7个单个菌落LB培养试管中,37℃摇床中培养12h~16h。最后用菌液PCR鉴定,条带正确的菌液抽取1mL送上海杰李生物技术有限公司测序。
1.2.2 生物信息分析 对测序结果用基因序列应用生物软件Lasergene进行序列的拼接和比较,用MEGA4.1对病毒进行遗传进化分析。
2 结果
2.1 病毒全基因组的克隆和序列分析
将GXNN1396感染Marc-145细胞,72h后收集上清,提取上清病毒RNA。使用引物(本文1.1.3引物设计)进行RT-PCR对GXNN1396的基因组进行分段扩增,电泳鉴定PCR产物条带大小(数据未显示)。接着,将PCR产物克隆到pMD18-T载体中,提取阳性克隆进行测序。用Lasergene软件包中的Seq Man对扩增的片段进行拼接,获得病毒的全长序列。
2.2 病毒基因组结构分析
根据表1在分离地区、年份和GenBank登陆号所获得9株PRRSV毒株核苷酸序列(1株经典型美洲株VR2332和8株国内的分离毒株CH-1a、BJ-4、HB-2(sh)2002、HN1、TJ、JXwn06、S1、JXA1)。如表2所示,对获得PRRSV分离株GXNN1396全长序列进行分析发现,GXNN1396全基因株序列全长为15263bp,比其他参考毒株的序列都短,说明该毒株的基因组存在核苷酸的缺失。在各参考PRRSV中,ORF1a的长度的变化是最大的,其中经典毒株VR2332、S1和CH-1a ORF1a为7512nt,BJ-4的ORF1a为7509nt,比VR2332少3nt。HB-2比VR2332少36nt。高致病性PRRSV毒株JXA1、JXwn06等在ORF1a比经典毒VR2332株少90nt,这是由于高致病性PRRSV的nsP2高变区不连续缺失了1+29aa所致,成为高致病性PRRSV独特的遗传标志。而本分离株的ORF1a为7365nt,说明了该毒株除了高致病性PRRSV 特征性的90nt缺失外,还有别的位点缺失。对各个毒株的nsP2蛋白进行比较,如图1所示,各个毒株ORF1a长度不一,主要体现在nsP2高变区存在不同程度的氨基酸缺失,与标准株VR2332相比,所有的高致病性PRRSV毒株如TJ、JXwn06和JXA1缺失了29+1aa。而HB-2(sh)2002毒株,也缺失了12aa。GXNN1396除了缺失在1+29aa外,在其上游,还缺失19aa。然而,GXNN1396株在ORF1b 和ORF 2~7的长度与其他毒株一致,分别为4383、771、765、537、603、525、372nt。
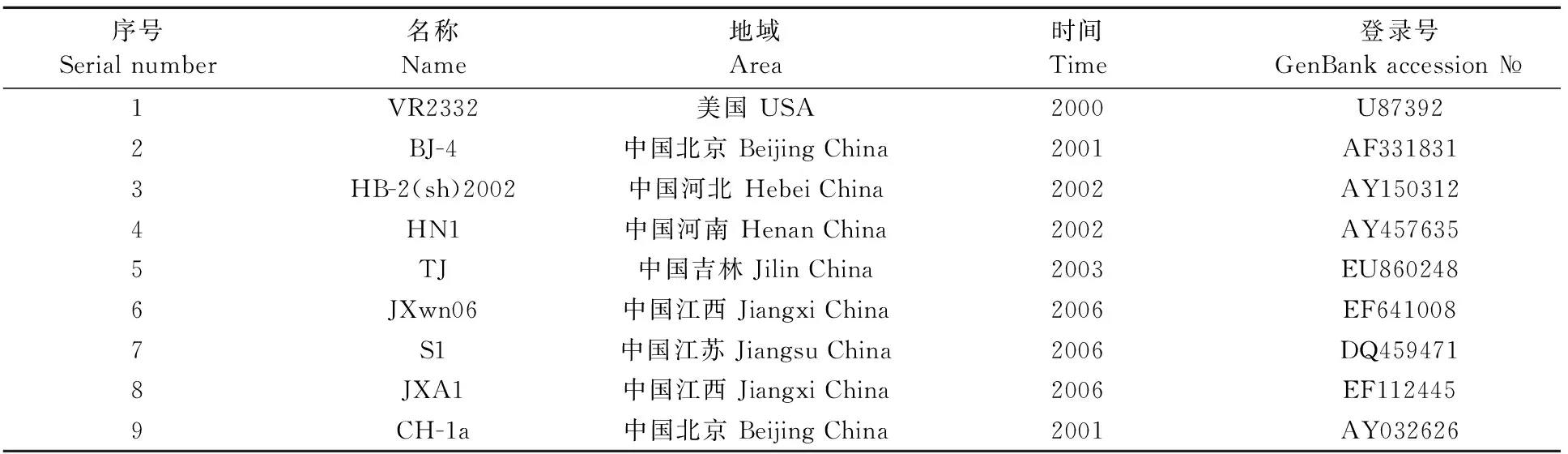
表1 本研究分析所用的PRRSV毒株Table 1 The strains of PRRSV used for analysis in this study
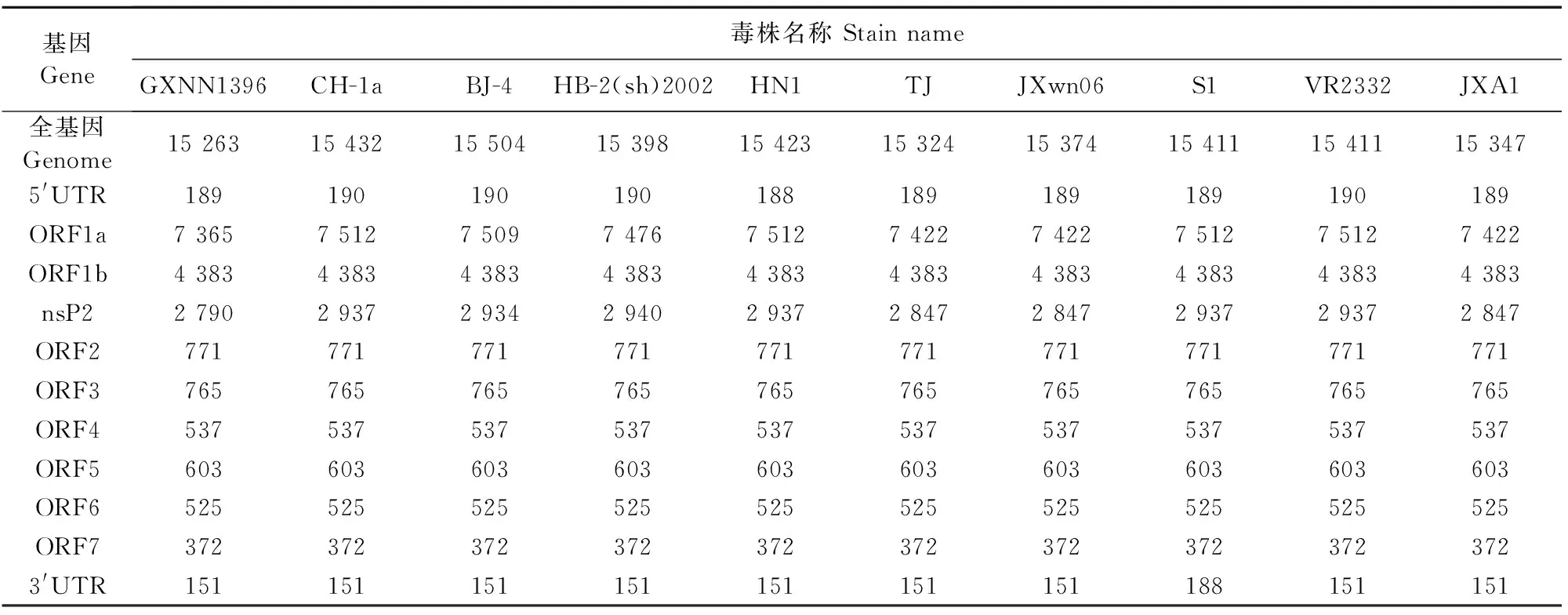
表2 GXNN-1396株与9株PRRSV毒株全基因组的长度比较Table 2 The full-length genome comparision between GXNN-1396stain and 9PRRSV strains bp

图1 GXNN-1396株与9株PRRSV毒株在nsP2高变区氨基酸序列比较:与VR2332氨基酸不同的氨基酸涂黑,缺失的氨基酸用“-”表示
Fig.1 Comparision of amino acid sequences of highly variable nsP2region between GXNN-1396and 9PRRSV strains:aa that differ with VR2332are shaded with black,aa deletion indicates as "-"
2.3 PRRSV-GXNN1396株和其他9株的全基因比较分析
对GXNN1396株全基因与国内外发表的部分毒株(JXA1、JXwn06、TJ、CH-1a、HB-2(sh)2002、HN1、S1、BJ-4、VR2332)序列进行比较,如表3所示,GXNN1396全基因组序列的相似性与JXwn06、TJ和JXA1等最高,为97.3%,与国内经典毒株BJ-4、S1和HN1毒株相似性为88.4%,与美洲型经典株VR2332相似性为88.8%。对非编码区和各个ORF进行比对发现,GXNN1396株在5′端UTR和3′端UTR的基因序列中,与高致病性PRRSV毒株JXwn06、JXA1相似性最高,为97.4%,与经典毒株VR2332和BJ-4只有91.5%,GXNN1396株与各株PRRSV之间在3′端UTR的相似性也有91.1%~97.3%。nsP2是PRRSV基因组中变异最大的基因,GXNN1396株与这些毒株最低相似性也有82%,ORF3、ORF4和ORF5的变异也较大,与HN1的相似性分别为88.8%、88.8%和87.1%;而ORF6和ORF7的保守性相对较高,分别高达93.9%和93.8%。
2.4 GXNN1396毒株全基因进化树分析
将GXNN1396株序列与9株参考毒株构建遗传进化树,如图2所示,PRRSV主要分成两大分支,以美洲型经典株VR2332为2型的PRRSV毒株和欧洲型经典株LV为代表1型的PRRSV毒株。而在2型PRRSV毒株中,可分为3个亚群,以VR2332、S1和HN1等经典株分为一群。而GXNN1396株则与高致病性PRRSV毒株JXA1、JXwn06和TJ分在同一亚群。经典株和高致病性亚群之间一个群则是Ch-1a和HB-2为代表。
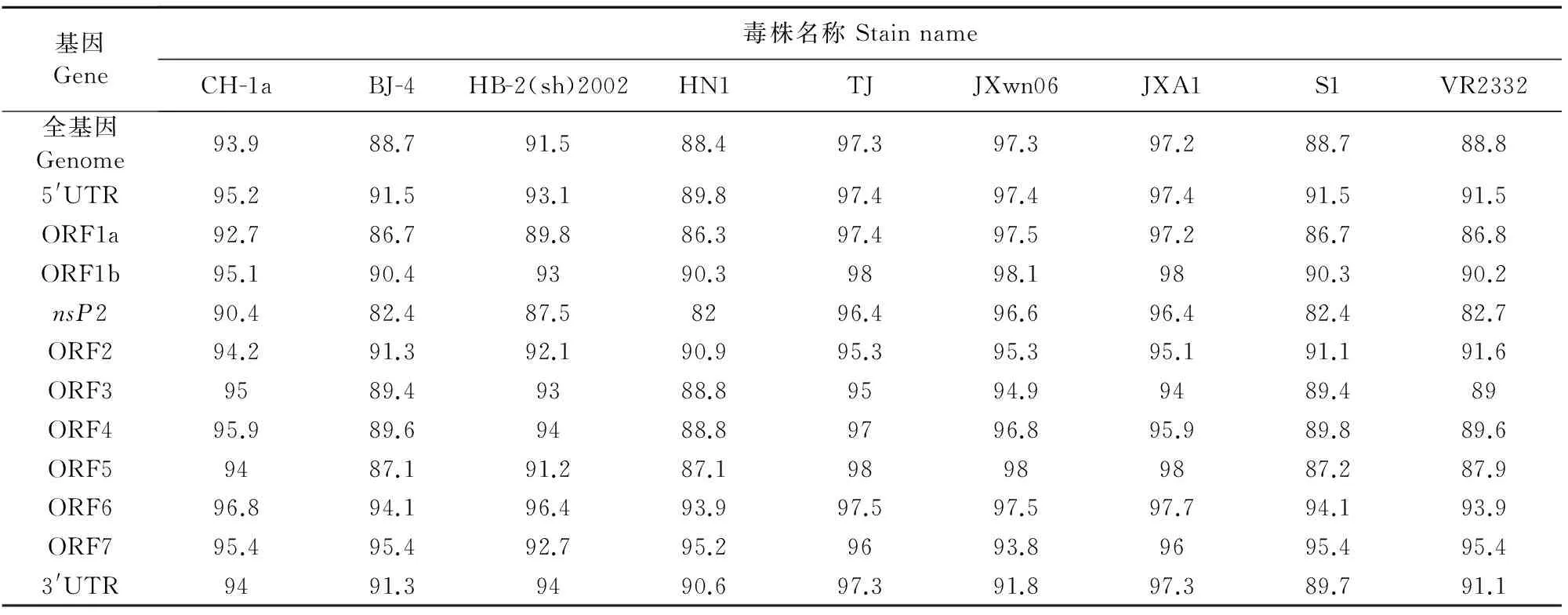
表3 GXNN1396株与其他9个毒株部分核苷酸同源性比较Table 3 Comparison of nucleotide identity between GXNN1396and other 9PRRSV strains %
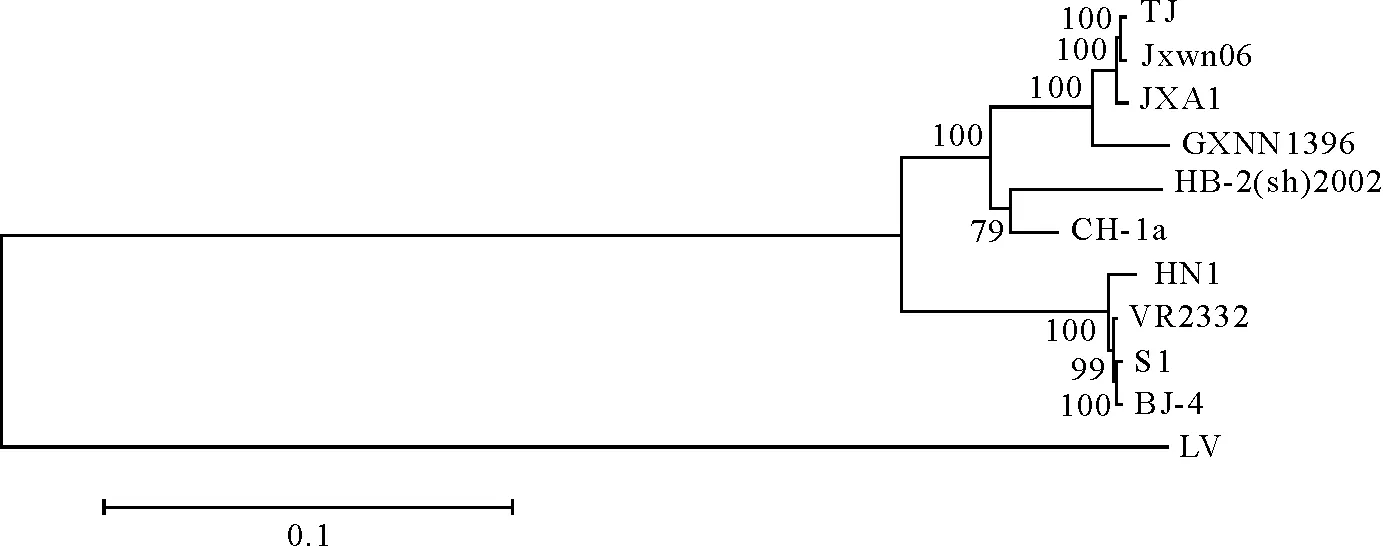
图2 GXNN1396株在部分PRRSV流行毒株的全基因遗传进化树
Fig.2 The phylogenetic tree of GXNN1396full-length genome in partial epidemic PRRSV strains
3 讨论
本研究中对PRRSV-GXNN1396全基因组进行扩增、同源性比较和进化树的分析,确定了GXNN1396毒株的核苷酸的长度、基因组结构特性和毒株进化情况。
本研究对GXNN1396毒株通过RT-PCR的方法进行全长序列分段扩增,通过克隆后进行测序,最后拼接得到病毒的全长序列。在与国内外高致病性PRRSV全基因序列比较中,与TJ、JXwn06、JXA1等核苷酸的同源性高达97.2%~97.3%。PRRSV有2个基因型,即VR2332株为代表的美洲型2型和以Lelystad virus(LV)株为代表欧洲1型,但这两者的基因序列的同源性仅为60%左右[7],而GXNN1396与欧洲经典毒株LV和VR2332株的核苷酸同源性分别为61%和88.8%。通过序列比对和遗传进化分析,确定了所分离的毒株是2型PRRSV毒株,并且与高致病性PRRSV毒株(JXA1、JXwn06和TJ)处于同一分支。目前为止,我国分离鉴定的毒株也多为2型的PRRSV毒株。有研究表明,以JXA1株为代表的美洲型HP-PRRSV株已成为我国PRRSV流行优势毒株[8-9]。显然,广西分离的毒株也是以高致病PRRSV 毒株为主,该毒株的致病性如何,还需进一步研究。我国自2006年以来,在nsP2有1+29aa的缺失已经证实是HP-PRRSV毒株的分子特性[10-11],但接着的研究表明,1+29aa的缺失与病毒毒力强弱没有关系[12],值得注意的是,国内著名蓝耳病专家杨汉春教授在寻找我国HP-PRRSV毒力决定区发现ORF1b在猪蓝耳病强毒与弱毒间替换,发现毒力发生反向变化[6]。尽管我们之前的研究确定了nsP2高变区存在着1+29aa的缺失,与其他高致病性PRRSV(如TJ、JXwn06、JXA1)缺失的部位一样,但不同是在上游还有19aa缺失。本研究对GXNN1396株的nsP2序列测通并比对发现,除了高变区缺失了49aa外,nsP2没有别的位点缺失。该毒株在nsP2中1+29aa的缺失有着何种病毒生物学特性还需进一步研究。对各个基因组序列比对结果显示,序列差异较大的基因有ORF1a、ORF3、ORF4和ORF5,而相对保守的基因为ORF6和ORF7,这与其他的研究结果相一致。值得注意的是,在本研究部分PRRSV流行毒株的全基因遗传进化树的分析中,GXNN1396株属于美洲型PRRSV中的一个亚型,虽然在与美洲型经典株VR2332不同属于一进化亚型。但在与当前我国流行优势毒株中,如JXA1、TJ、JXwn06等在同一个亚型进化分支中,这对研究GXNN1396株进化过程中是否存在由其他毒株基因的重组和变异,进而形成毒株来源提供了研究依据,也为日后候选为理想疫苗株奠定基础。
总之,在本研究中,我们实验室分离到的PRRSV毒株GXNN1396进行了全基因序列分析、同源性比较和遗传进化分析,确定了该毒株的来源与遗传变异情况,同时也丰富了PRRSV分离株的全基因组序列数据库,这对日后该病的流行病学调查提供了必要的数据,同时对研究PRRSV的致病力和遗传变异情况提供了理论依据。
[1] Johnson C R,Griggs T F,Gnanandarajah J,et al.Novel structural protein in porcine reproductive and respiratory syndrome virus encoded by an alternative ORF5present in all arteriviruses[J].J Gen Virol,2011,92(Pt 5):1107-1116.
[2] 洪绍锋,韦 莹,韦苗苗,等.一株nsp2蛋白部分氨基酸自然缺失的猪繁殖与呼吸综合征病毒的分离和鉴定[J].中国动物传染病学报,2014,22(6):6-12.
[3] Sutherland M A,Niekamp S R,Johnson R W,et al.Heat and social rank impact behavior and physiology of PRRS-virus-infected pigs [J].Physiol Behav,2007,90:73-81.
[4] Leng Xue,Li Zhenguang,Xia Mingqi,et al.Mutations in the genome of the highly pathogenic porcine reproductive and respiratory syndrome virus potentially related to attenuation[J].Vet Microbiol,2012,57(1-2): 50-60.
[5] 廖承球.广西PRRSV GXA株全基因序列测定、致病性实验及PRRSV分子流行病学调查研究[D].广西南宁:广西大学,2011:25-27.
[6] Li Yan,Zhou Lei,Zhong Jialong,et al.Nsp9and Nsp10contribute to the fatal virulence of highly pathogenic porcine reproductive and respiratory syndrome virus emerging in China[J].PLoS Pathog,2014,10(7):e1004216.
[7] Nelsen C J,Murtaugh M P,Faaberg K S.Porcine reproductive and respiratory syndrome virus compareison:divergent evolution on two continents[J].J Virol,1999,73(1):270-280.
[8] Zhou Lei,Chen Shuxian,Zhang Jialong,et al.Molecular variation analyses of porcine reproductive and respiratory syndrome virus in China [J].Virus Res,2011,155(2):473-486.
[9] Li Bin,Fang Liurong,Guo Xueliang,et al.Epidemiology and evolutionary characteristics of the porcine reproductive and respiratory syndrome virus in China between 2006and 2010[J].J Clin Microbial,2011,49(9):3175-3183.
[10] Wang Xiaomin,Sun Lichang,Lia Yanhua,et al.Development of a differentiable virus via a spontaneous deletion in the nsp2region associated with cell adaptation of porcine reproductive and respiratory syndrome virus[J].Virus Res,2013,171:150-160.
[11] Leng Chaoliang,Tian Zhijun,Zhang Wuchao,et al.Characterization of two newly emerged isolates of porcinereproductive and respiratory syndrome virus from Northeast China in 2013[J].Vet Microbiol,2014,171:41-52.
[12] Zhou Lei,Zhang Jialong,Zeng Jingwen,et al.The 30-aminoacid deletion in the nsp2of highly pathogenic porcine reproductive and respiratory syndrome virus emerging in China is not related to its virulence[J].J Virol,2009,83(10):5156-5167.
Full-length Genome Sequence Analysis of PRRSV Isolate GXNN1396in Guangxi
HUANG Jia-bin1,WEI Ying1,HONG Shao-feng1,LIN Si-yuan1,HE Rong-xiang,2CHEN Ying1,HUANG Wei-jian1,WEI Zu-zhang1
(1.AnimalInfectiousDiseaseLaboratory,CollegeofAnimalScienceandTechnology,GuangxiUniversity,Nanning,Guangxi,530005,China; 2.AdministrativeOfficeofLuchuanResevior,Luchuan,Guangxi,537700,China)
Previously,we isolated and identified a novel PRRSV strain (GXNN1396) with 49amino acids deletion in nsP2.In this study,to further characterize the full length genome of GXNN1396,Marc-145cells were inoculated with GXNN1396.The supernatant of infected cells was collected and used for virus RNA extraction.A set of primers were designed for RT-PCR to amplify the full-length genome of GXNN1396.The nucleotide sequence of GXNN1396was compared with the reference PRRSV strains retrieved from GenBank.The phylogenic tree of GXNN1396and the reference strains was constructed based on the full-length genome.The results showed that the complete genome of GXNN1396consists 15263nt,excluding the 5′cap and 3′poly A tail.The 5′UTR has 189nt and 3′UTR has 151nt.ORF1a,ORF1b,ORF2,ORF3,ORF4,ORF5,ORF6and ORF7contain 7422,4382,771,765,537,603,525and 372nucleotides,respectively.The complete genome of GXNN1396shares 88.8% identity with VR2332,and shares 97.2%-97.3% identity with highly pathogenic PRRSV strains TJ,JXwn06,JXA1,but only has 61% identity with type 1PRRSV stain LV,suggesting GXNN1396belongs to type 2PRRSV.The phylogenic analysis based on the full length genome revealed that PRRSV strains were classified into 3clades and GXNN1396was distributed with highly pathogenic PRRSV strains TJ,JXwn06,JXA1.
Porcine reproductive and respiratory syndrome virus; full-length sequence analysis;homology analysis; phylogenetic analysis
2015-07-11
国家自然科学基金项目(31372444);广西大学科研基金项目(XGZ130959)
黄加滨(1989-),男,浙江温州人,硕士研究生,主要从事预防兽医学研究。*通讯作者
S852.659.6
A
1007-5038(2016)01-0001-06
