基于下一代测序平台的结直肠癌KRAS基因检测
2016-12-02李丽娟罗婉珊张家彬王庆吴英松
李丽娟 罗婉珊 张家彬 王庆 吴英松
·论著·
基于下一代测序平台的结直肠癌KRAS基因检测
李丽娟1罗婉珊1张家彬1王庆1吴英松2★
目的基于下一代测序技术,检测和分析结直肠癌KRAS基因的突变情况,以及评估其应用于临床分子诊断中的可行性。方法应用Ion Proton半导体测序仪,检测108例结直肠癌患者的石蜡包埋组织样本的KRAS基因突变情况,并与作为金标准的一代测序检测结果进行对比。结果下一代测序检测结果与一代测序检测结果基本一致,符合率为94%,灵敏度高达100%。结论通过对108例临床样本的检测分析后,该下一代测序检测方法可用于检测结直肠癌KRAS基因突变情况。
下一代测序;结直肠癌;KRAS
RAS家族之一的KRAS基因位于12p12.1,编码分子量为21 kD、具有GTP酶活性的p21蛋白[1]。KRAS为EGFR信号传导通路RAS⁃RAF⁃MAPK中的重要因子。机体正常的KRAS基因,通过控制相关路径抑制癌细胞的生长。突变型KRAS基因,在无需活化EGFR的条件下,激活下游信号传导通路,导致细胞的生长、增殖和凋亡发生异常。细胞传导信号的紊乱以及增殖的失控会导致细胞癌变。KRAS基因主要在第12、13位密码子上发生突变,以点突变为主,在第61位密码子上可见少数突变[2]。
KRAS是人体肿瘤中常见的致癌基因,其中20%~50%的结直肠癌患者伴有KRAS基因突变。其突变出现在结直肠癌的早期阶段,不受治疗的影响而维持稳定状态[3]。KRAS是第一个对临床治疗结直肠癌的决策产生影响的生物标记物,是帮助了解相关基因和癌症的发展预后、疗效的关键指标。当KRAS基因为突变型时,则不建议采用
EGFR靶向药物。临床研究表明,当患者本身为KRAS基因野生型即不存在KRAS基因突变,服用EGFR有关靶向治疗药物,能获得明显的治疗效果。例如非KRAS基因突变的结直肠癌患者,可受益于西妥昔和帕尼单抗等EGFR抗体药物[4]。美国癌症综合网络(National Comprehensive Can⁃cer Network,NCCN)提出肿瘤患者必须先检测KRAS基因突变,再选择EGFR靶向药物。KRAS基因突变检测已被NCCN列为《结肠癌临床治疗指南》与《直肠癌临床治疗指南》临床用药必检项目。
目前,应用下一代测序技术检测KRAS基因突变的方法有多种。本研究应用Ion Torrent半导体测序技术[5],对108例结直肠癌病理样本进行KRAS基因的突变检测,并与一代测序检测结果进行对比。旨在探讨下一代测序检测方法在临床检测结直肠癌KRAS基因突变的可行性。
1 材料与方法
1.1 样本采集
随机选取2015年1月至2015年12月宁波市第二医院结直肠癌患者的石蜡包埋组织样本108例,其中男性64例,女性44例,年龄在32~90岁,平均年龄64岁。本研究经此医院医学伦理委员会批准,且患者均知情同意。
1.2 主要试剂和仪器
1.2.1 主要试剂
本研究主要使用的试剂见表1。
表1 试剂信息表Table 1 The information of reagents
表2 仪器信息表Table 2 The information of instruments
1.2.2 主要仪器
本研究主要使用的仪器见表2。
1.3 检测方法
1.3.1 DNA提取
采用环保脱蜡剂对石蜡包埋组织样本进行脱蜡处理,按照QIAamp DNA FFPE Tissue KIT(50)说明书提取样本的DNA,用Qubit®3.0荧光定量仪定量。
1.3.2 文库构建和定量
采用Ion AmpliSeq LibraryTMKit 2.0进行文库构建。关键步骤包括:目的片段的PCR扩增反应,消除部分引物序列,加P1接头和特异性接头以及文库的纯化。
采用Qubit®3.0荧光定量仪进行文库定量。根
据下列公式将文库稀释到200 pmol/L,稀释后采用荧光定量PCR仪定量,等质量混合后进行模板制备。一次测序反应,最多可以混合96个不同特异性接头的样本(包括阳性质控品和阴性质控品)。
1.3.3 模板制备与富集
按照Ion PITMHi⁃QTMOT2 Reagents200 kit说明书进行操作,适量的上样体积与ISP磁珠。关键步骤:通过反应过滤器形成油包水型,PCR扩增反应,离心回收制备好的模板颗粒,Ion One TouchTMES的磁珠技术分离得到阳性模板。
1.3.4 上机测序
采用Ion PITMHi⁃QTMSequencing 200 Kit,加入质控模板,退火,Loading测序芯片,加测序酶。108例样本分2张芯片进行上机测序。
1.3.5 结果分析
利用测序仪配套服务器和软件进行序列比对和分析。
运行VariantCaller和Coverage Analysis插件,得到样本的分析结果。点击“筛选”,在“Allele Call”选项中,筛选保留“Homozygous”和“Heterozygous”;在“Allele Source”选项中,筛选保留“Hotspot”;在“Frequency”选项中,筛选保留“≥5”的数值;在“Cov⁃erage”选项中,筛选保留“≥2500”的数值。
1.4 一代测序对照实验
将上述提取的108例DNA样本同时应用ABI 3500 DX基因分析仪及配套试剂进行检测,运行SeqScanner程序,导入测序结果。按Find快捷键,查找序列“AGTTGGAGCT”。此序列往后的第1~6个碱基,分别为KRAS第12、13密码子。12号密码子野生型为GGT,13号密码子野生型为GGC;否则为KRAS外显子2突变,详细见表3。

表3 突变信息表Table 3 The information ofmutations
1.5 统计学分析
采用SPSS 20.0统计分析软件,对结果进行一致性检验,以Kappa≥0.75为一致性较好。
2 结果
2.1 测序结果分析
通过对108例结直肠癌石蜡包埋组织样本测序结果分析,下一代测序结果检出65例为KRAS基因野生型并与一代测序结果完全一致,有43例为KRAS基因突变型,其中37例KRAS基因突变型与一代测序结果完全一致,具体信息见表4。
下一代测序检测结果中有6例KRAS基因突变型与一代测序结果不一致,见表5和图1。
样本NBE059、NBE071下一代测序结果显示6.7%、9.4%的突变频率;对应的一代测序峰图突变碱基荧光信号弱(灵敏度<20%),不足以判定其为阳性。样本NBE020、NBE074和NBE077下一代测序结果显示5.3%、7.4%、5.0%的突变频率;对应的一代测序峰图突变碱基峰与背景峰大体一致,判定其为野生型。样本NBE069下一代测序结果有7.4%的Gly12Cys突变,对应的一代测序峰图是野生型。
2.2 2种检测方法相关性分析
以金标准一代测序检测结果为基础,验证下一代测序检测方法的可行性。通过相应公式计算得到本实验灵敏度为100%,特异性为91.5%,阳性预测值为86%,阴性预测值为100%,总符合率为94%。以及采用SPSS 20.0统计分析软件,计算Kappa为0.881。2种测序结果统计具体见表6。
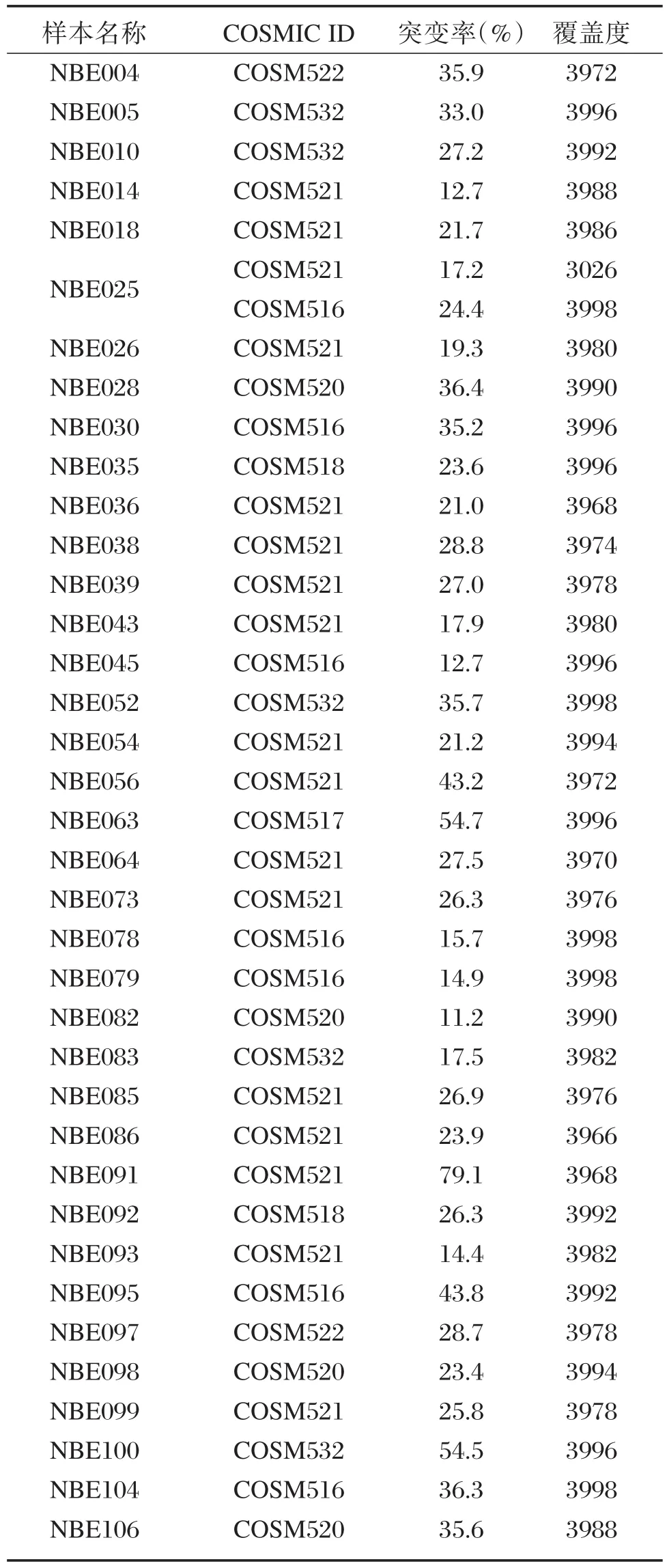
表4 下一代测序检测结果Table 4 The results of nextgeneration sequencing
3 讨论
作为金标准的一代测序法采用Sanger测序原理,4种荧光染料标记的ddNTP,经过单引物PCR反应,采用毛细管电泳技术,实现目的片段的所有碱基序列的检测。以准确、可靠、检测范围广等为特点,同时具有耗时长、成本高,操作复杂、易污染、灵敏度低等局限性[6]。主要针对已知突变基因的位点设计引物,所以要明确知道该基因位点的具体信息,对于没有明确的基因位点难以实现测序反应。一代测序的目的在于寻找与疾病相关的特定基因突变,若没有明确突变基因或筛查数量众多的基因则难以完成[7]。本研究采用下一代测序技术,经过Ion Proton半导体测序仪能够检测出目的片段的碱基序列。该系统采用天然核苷酸和聚合酶、无需激发光源、荧光标记、焦磷酸酶化学级联以及照相系统直接实时检测测序时产生的氢离子浓度,有效提高碱基判读准确性[8]。测序反应开始5m in内即开始得到测序数据,单次运行耗时2 h,测序芯片反应微孔大于1.6亿,产出数据量可达10G。相比一代测序具有检测快速,通量高,能同时检测出多种不同的突变型别等优势[9]。
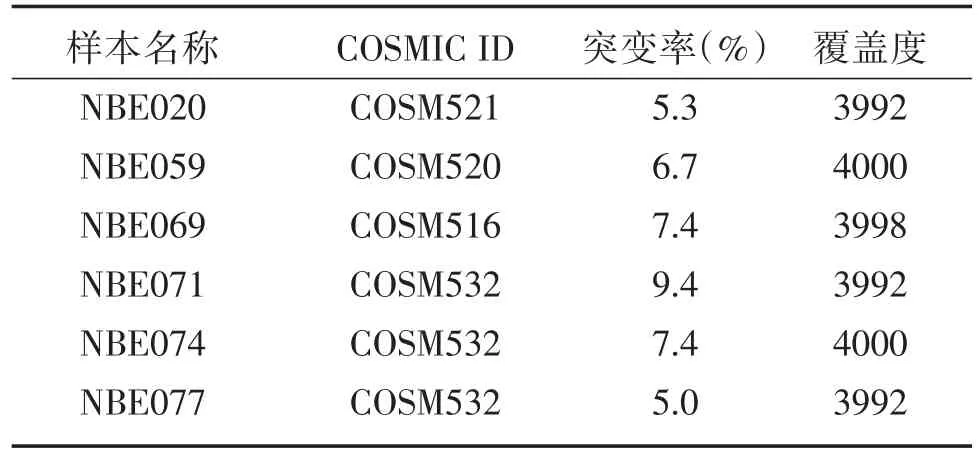
表5 下一代测序检测结果Table 5 The resultsof nextgeneration sequencing
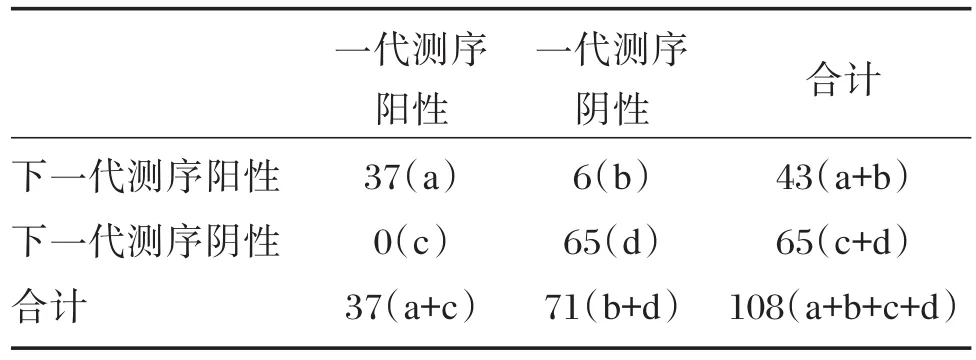
表6 测序结果比较Table 6 The comparision of sequencing results
本研究对108例结直肠癌临床样本KRAS基因检测结果显示,一代测序结果有37例阳性(突变率34.3%),下一代测序结果有43例阳性(突变率39.8%),2种方法符合率为94%。与国内外相关报告相符合。37例一代测序阳性样本在下一代测序结果中显示较高的突变频率,此外下一代测序还
检测出6例突变频率比较低的样本(突变频率>5%,结果可信)。对应的6例一代测序峰图由于突变碱基荧光信号弱(灵敏度<20%)或者突变碱基峰与背景峰大体一致,判定其为野生型。一代测序结果的准确性受测序仪器和设定条件的影响,背景峰的高低也会影响判定结果。肿瘤异质性较明显时,一代测序的峰图易受干扰,不能准确判断突变类型,因此其灵敏度低[10]。下一代测序灵敏度高达100%,特异性为91.5%,阳性预测值86%,阴性预测值100%以及Kappa值>0.75,验证下一代测序结果与一代测序有很好的一致性。但2种检测方法均未检测到第61密码子上的突变,可能是由于样本量少、癌组织处理以及这些突变较少见。因此需要扩大样本量进行深入研究加以验证。
目前下一代测序技术广泛应用于肿瘤基础科研中,鉴定肿瘤驱动基因突变,发现新的突变基因和位点,并应用于新的药物靶点和生物标志物的研发。在国外,下一代测序技术在临床诊断领域得到一定的发展。根据相关肿瘤基因组的信息预测药物的疗效,制订个体化诊疗方案,并为新药的临床试验筛选合适的患者[11]。本研究的下一代测序系统具有高通量特点,一张芯片可以同时检测1~96例样本;并能检测出一代测序无法检测的低频率突变位点,更好的满足结直肠癌临床分子诊断上的需求[12]。
[1]Cong T,Xiang D.KRASmutation testing inmetastat⁃ic colorectal cancer[J].World JGastroenterol,2012,18(37):5171-5180.
[2]Malapelle U,Carlomagno C,de Luca C,etal.KRAS testing in metastatic colorectal carcinoma:challenges,controversies,breakthroughs and beyond[J].J Clin Pathol,2014,67(1):1-9.
[3]Douillard JY,Oliner KS,Siena S,etal.Panitumumab⁃FOLFOX4 treatment and RASmutations in colorectal cancer[J].N Engl JMed,2013,369(11):1023-1034.
[4]Tougeron D,Lecom te T,Pagès JC,et al.Effect of low⁃frequency KRASmutationson the response to anti⁃EGFR therapy in metastatic colorectal cancer[J].An⁃nalsof Oncology,2013,24(5):1267-1273.
[5]Umberto M,Elena V,Roberta S,et al.Ion Torrent next⁃generation sequencing for routine identification of clinically relevant mutations in colorectal cancer pa⁃tients[J].JClin Pathol,2015,68(1):64-68.
[6]KothariN,Schell M J,Teer JK,et al.Comparison of KRAS mutation analysis of colorectal cancer samples by standard testing and next⁃generation sequencing[J]. JClin Pathol,2014,67(9):764-767.
[7]Normanno N,Rachiglio A,Roma C,et al.Comment on a comparison of threemethods for detecting KRAS mutations in formalin⁃fixed colorectal cancer specimens[J].Br JCancer,2012,107(10):1791-1792.
[8]Lin MT,Mosier SL,Thiess M,et al.Clinical valida⁃tion of KRAS,BRAF,and EGFR mutation detection using next⁃generation sequencing[J].Am J Clin Pathol,2014,141(6):856-866.
[9]Salto⁃Tellez M,de Castro DG.Next generation se⁃quencing:a change of paradigm inmolecular diagnos⁃tic validation[J].JPathol,2014,234(1):5-10.
[10]Angulo B,Garcia⁃Garcia E,Martinez R,et al.A com⁃mercial real⁃time PCR kit provides greater sensitivity than direct sequencing to detect KRAS mutations:a morphology⁃based approach in colorectal carcinoma[J].Molr Diagn,2010,12(3):292-299.
[11]Xuan J,Yu Y,Qing T,et al.Next⁃generation se⁃quencing in the clinic:prom ises and challenges[J]. Cancer Lett,2013,3409(2):284-295.
[12]van A llen EM,Wagle N,Stojanov P,et al.Whole⁃exome sequencing and clinical interpretation of forma⁃lin⁃fixed,paraffin⁃embedded tumor samples to guide precision cancer medicine[J].Nat Med,2014,20(6):682-688.
Based on the next generation sequencing platform for detection colorectal KRAS gene
LILijuan1,LUOWanshan1,ZHANG Jiabin1,WANGQing1,WU Yingsong2★
(1.Guangzhou Darui Biotechnology Co.,Ltd.,Guangzhou,Guangdong,China,510665;2.School of Laboratory Medicine Biotechnology,Southern MedicalUniversity,Guangzhou,Guangdong,China,510515)
Objective Based on next generation sequencing technology,to detect and analyze of KRAS genemutations in colorectal cancer,and to assess the feasibility of its application in clinicalmolecular diagnosis.Methods KRAS genemutation was detected in paraffin embedded tissue samples from 108 cases of colorectal cancer by Ion Proton sem iconductor sequencer.The resultswere compared w ith the results from Sanger sequencing which is the gold standard.Results The results of the next generation sequencing were consistent w ith the results of gold standard,w ith the accuracy rate of 94%and sensitivity up to 100%. Conclusion Through the detection and analysis of 108 cases of clinical samples,this next generation sequencingmethod isvalid fordetection of KRAS genemutation in colorectalcancer.
Nextgeneration sequencing;Colorectal cancer;KRAS
广东省科技计划项目应用型专项资金(2015B020233009)
1.广州市达瑞生物技术股份有限公司,广东,广州510665 2.南方医科大学检验与生物技术学院,广东,广州510515
★通讯作者:吴英松,E⁃mail:wg@smu.edu.cn
