应用下一代测序技术对α地中海贫血进行胚胎植入前遗传学检测
2016-12-02谢美娟邓权衡邓红辉卢绍月杨学习马强
谢美娟 邓权衡 邓红辉 卢绍月 杨学习 马强★
·论著·
应用下一代测序技术对α地中海贫血进行胚胎植入前遗传学检测
谢美娟1邓权衡1邓红辉1卢绍月1杨学习2马强2★
目的探讨下一代测序(nextgeneration sequencing,NGS)技术在α地中海贫血胚胎植入前遗传学检测中的应用。方法选取2对α地中海贫血⁃⁃SEA缺失型携带者夫妇体外受精胚胎活检后的6个胚胎样本应用全基因组扩增(whole genome amplification,WGA)技术、下一代测序技术进行胚胎植入前遗传学检测,同时采用跨越断裂点荧光PCR(gap⁃PCR)进行平行对照检测。结果2个家系6个胚胎样本的胚胎植入前遗传学诊断(preimplantation genetic diagnosis,PGD)结果分别为⁃⁃SEA/ αα母源携带、⁃⁃SEA/⁃⁃SEA、⁃⁃SEA/αα母源携带、⁃⁃SEA/⁃⁃SEA、⁃⁃SEA/αα母源携带和⁃⁃SEA/⁃⁃SEA;胚胎植入前遗传学筛查(preimplantation genetic screening,PGS)结果分别为45,XX,⁃5、46,XX、46,XY、47,XY,+1、46,XX和46,XY,+1,⁃2;Family 1 gap⁃PCR检测结果为父母均为SEA杂合子;E1为正常、E2为SEA纯合子、E3为正常;Family 2检测结果分别为:父母均为SEA杂合子;E4为SEA纯合子、E5为正常、E6为SEA纯合子。结论结果显示利用NGS不仅可以检测出23对染色体的核型,同时解决了单细胞扩增等位基因脱扣(allele drop⁃out,ADO)造成的假阳性和假阴性的风险,更具有市场潜力及应用前景。
下一代测序;α地中海贫血;胚胎植入前遗传学诊断;跨越断裂点荧光PCR
α⁃地中海贫血,是一组因α⁃珠蛋白链合成减少或不能合成、α⁃链/非α⁃链比例失衡为特征的遗传溶血性血红蛋白病。我国南方为α⁃地中海贫血的高发区,其携带率也高达8.53%[1];其中以东南亚(⁃⁃SEA)缺失型最为常见,其发生率为72.87%~82.87%[2]。α⁃地中海贫血与出生缺陷有密切关系,也给社会和家庭带来了沉重的负担,而目前,α⁃地中海贫血还没有有效的根治方法,主要以预防为主。因此,采用植入前期胚胎筛查来阻断患儿的出生极为重要。
胚胎植入前遗传学检测主要包括胚胎植入前遗传学诊断(preimplantation genetic diagnosis,PGD)和胚胎植入前遗传学筛查(preimplantation genetic screening,PGS)2方面。PGD是在胚胎着床之前即对配子或胚胎进行遗传物质分析,选择没有遗传物质异常的胚胎移植,可以有效地阻断患儿的出生;PGS是选择染色体整倍体的胚胎进行移植,进而提高体外受精⁃胚胎移植(in vitro fer⁃tilization⁃embryo transfer,IVF⁃ET)技术的成功率。
随着我国全面二孩政策的放开,高龄产妇数量猛增,再加上不孕不育率逐年上升,提高妊娠率和妊娠质量已经成为亟待解决的问题。目前,以PGD/PGS为代表的第三代试管婴儿正是解决这些问题的关键技术[3⁃4]。此外,NGS具有高通量、自动化、低成本的特征,自2008年以来,全基因组测序费用呈指数级下降,使得NGS在PGD/PGS临床上的应用得到迅速推广。本研究采用NGS对α⁃地中海贫血进行胚胎植入前遗传学检测,探讨NGS应用于PGD/PGS临床检测的可行性。
1 材料与方法
1.1 主要仪器和试剂
QIAamp®DNA M iniand QIAamp DNA Blood M ini Kits、The REPLI⁃g Single Cell Kit购自德国Qiagen公司;Ion AmpliseqTMlibrary kit 2.0、Ion PGM™Template OT2 200 Kit、Ion PGM™Sequenc⁃ing 200 Kit v2、Ion Personal Genome Machine®(PGM™)、Qubit®2.0均购自美国Life Technology公司;α⁃地中海贫血基因检测试剂盒(gap⁃PCR法)购自中山大学达安基因股份有限公司;Nuclease⁃FreeWater购自美国Life Technologies公司;无水乙醇(分析纯)购自广州化学试剂厂。
1.2 研究对象
2个家系双方均为α⁃地中海贫血⁃⁃SEA缺失型杂合子夫妇经体外受精胚胎活检后共6个胚胎囊胚期滋养外胚层细胞以及夫妻双方外周血。
1.3 实验方法
1.3.1 全基因组扩增、基因组DNA提取
使用The REPLI⁃g Single Cell Kit对6个胚胎囊胚期滋养外胚层细胞进行全基因组扩增;采用QIAamp®DNA M ini and QIAamp DNA Blood M ini Kits对2对夫妇的全血样本进行基因组DNA提取;具体操作步骤参见说明书。
1.3.2 下一代测序
根据测序需要,使用Ion AmpliseqTMlibrary kit 2.0将扩增好的DNA构建成文库DNA。PCR特异性扩增目的片段,在DNA片段两端加上通用测序接头,PCR扩增文库DNA,磁珠纯化去除杂质;建库后使用Qubit®2.0测定文库DNA的浓度;根据每个样本测序数据需求量和测序深度确定文库的混样个数为10个进行模板制备,使用Ion PGM™Template OT2 200 Kit制备测序模板;然后进行阳性模板的富集即去除杂质及扩增失败的测序模板;最后使用Ion PGM™Sequencing 200 Kit v2在PGM™上进行测序。
1.3.3 下一代测序数据分析
将上机测序得到的原始数据利用Tor⁃rent_Server_5.0_VM软件去除接头序列,通过Tor⁃rent Mapping A lignment Program(TMAP)软件比对到人类基因组参考序列hg19上,通过sam tools统计覆盖倍数,通过GATK calling SNP基因型分析,最后结合家系的SNP连锁分析,构建单体型。
1.3.4 gap⁃PCR
将2对夫妻双方外周血提取的基因组DNA以及6个胚胎全基因组扩增后的基因组DNA采用α⁃地中海贫血基因检测试剂盒进行gap⁃PCR检测,实验操作、结果判读参见说明书。
2 结果
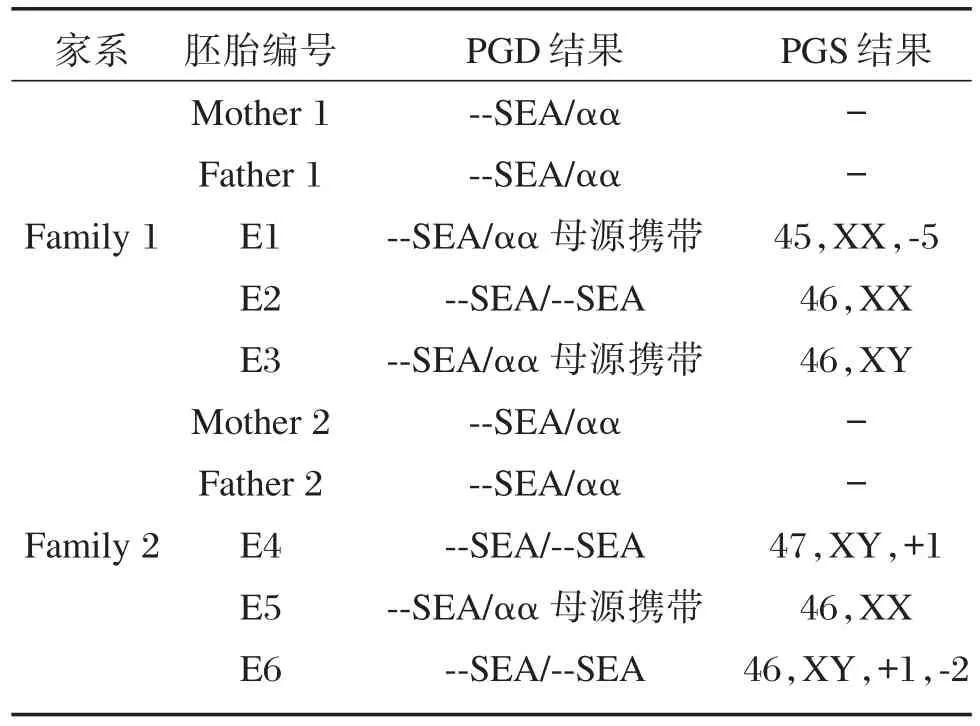
表1 6个胚胎下一代测序结果汇总表Table 1 Results of 6 embryos detected by NGS
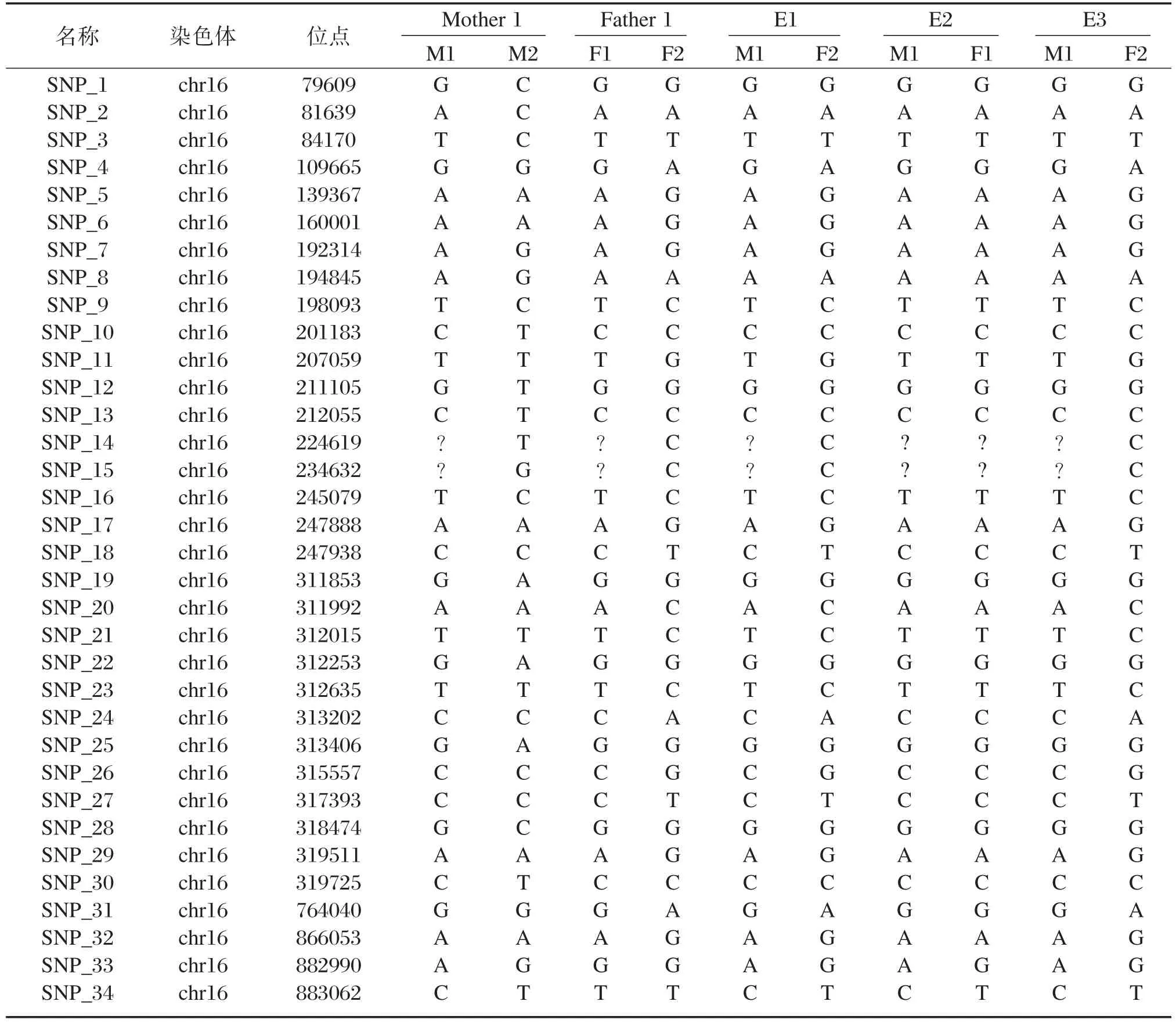
表2 Fam ily 1 PGD结果Table 2 PGD results of fam ily 1
2.1 下一代测序结果
本次研究收集2对地中海贫血携带者夫妇体外受精胚胎活检的6个囊胚期滋养外胚层细胞以及2对夫妻双方外周血基因组样本进行下一代测序。下一代测序检测结果汇总表见表1。
将夫妻双方外周血测序数据及其各自的胚胎测序数据进行家系的SNP连锁分析,构建单体型,进行结果的判断,6例胚胎样本中,PGD结果分别为E1:⁃⁃SEA/αα母源携带;E2:⁃⁃SEA/⁃⁃SEA;E3:⁃⁃SEA/αα母源携带;E4:⁃⁃SEA/⁃⁃SEA;E5:⁃⁃SEA/αα母源携带;E6:⁃⁃SEA/⁃⁃SEA,详见表2、表3。
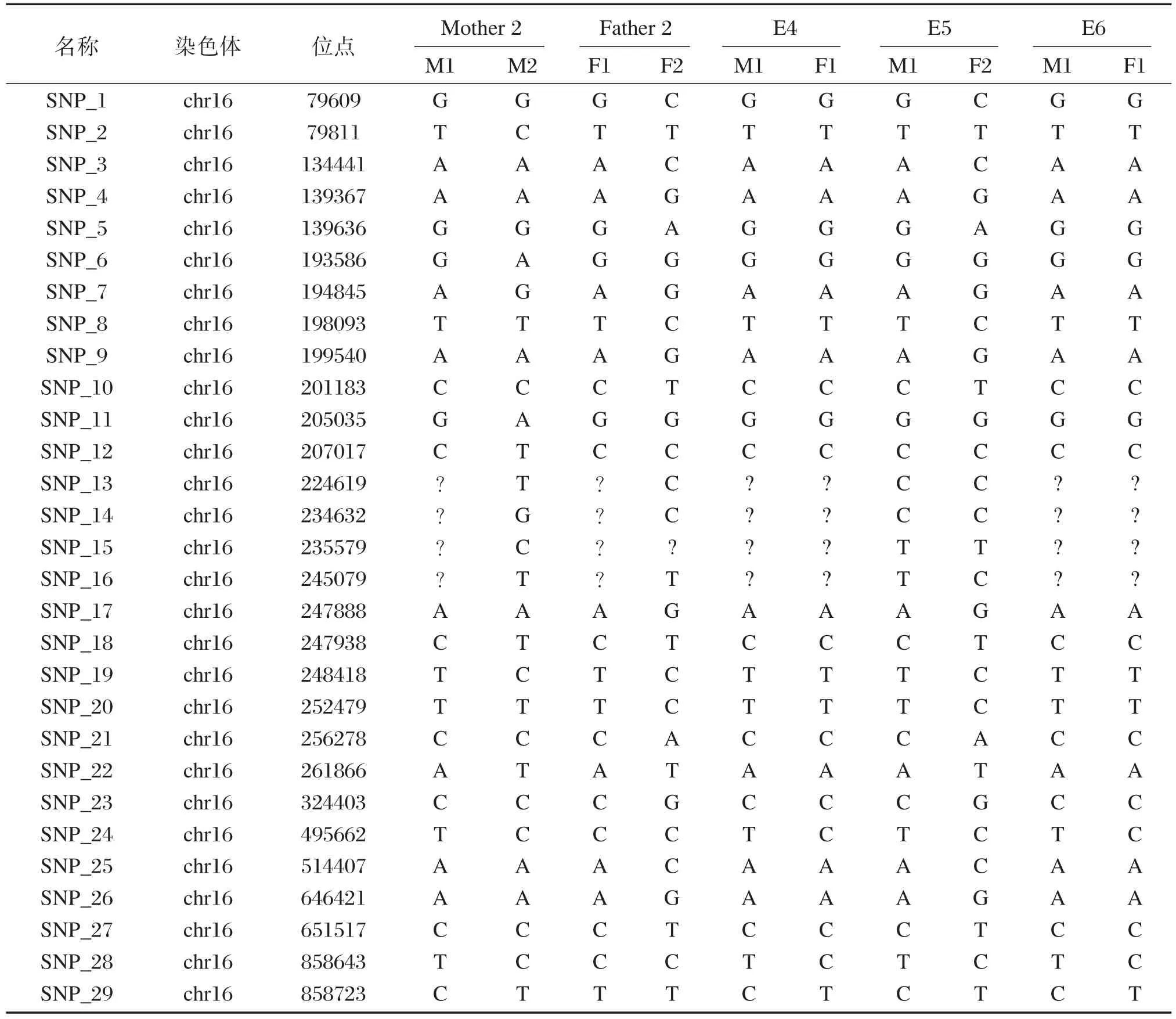
表3 Fam ily 2 PGD结果Table 3 PGD resultsof family 2

表4 2种检测方法结果比对Table 4 The comparison results of PGD and gap⁃PCR
为提高IVF⁃ET的成功率,再将6个胚胎进行PGS检测,PGS检测结果分别为E1:45,XX,⁃5;E2:46,XX;E3:46,XY;E4:47,XY,+1;E5:46,XX;E6:46,XY,+1,⁃2。详见图1、图2。
2.2 gap⁃PCR检测结果
为了验证本次检测结果的准确性,将2个家系样本同时进行gap⁃PCR的检测,Fam ily 1检测结果分别为:父母均为SEA杂合子;E1为正常、E2为 SEA纯合子、E3为正常;凝胶电泳图结果见图3;Fam ily 2检测结果分别为:父母均为SEA杂合子;E4为SEA纯合子、E5为正常、E6为SEA纯合子;凝胶电泳图结果见图4。
2.3 2种检测方法结果比对
将2种检测方法的检测结果进行对比,详见表4。结果显示对于SEA纯合缺失样本2种方法检测结果一致,而经过WGA的PCR产物对SEA携带者则检测结果为正常。
3 讨论
目前α⁃地中海贫血SEA缺失型的检测,文献报道的PCR技术已比较成熟[5⁃6]。但PCR技术在PGD中仍然面临着一些问题,如扩增偏倚和ADO等。ADO即一个细胞内来自双亲的2个等位基因只有一个扩增到可供检测的水平的现象,是影响PGD准确性的主要因素之一。ADO的发生率为5%~20%[7⁃8]。其危害在于可导致杂合子胚胎的误诊,是PCR技术进行显性遗传病PGD可靠诊断的最大障碍。
本次实验中共检测2个家系4个外周血样本及6例胚胎样本。PGD结果分别为E1:⁃⁃SEA/αα母源携带;E2:⁃⁃SEA/⁃⁃SEA;E3:⁃⁃SEA/αα母源携带;E4:⁃⁃SEA/⁃⁃SEA;E5:⁃⁃SEA/αα母源携带;E6:⁃⁃SEA/⁃⁃SEA;此外,在辅助生殖领域,染色体非整倍体也被认为是胚胎植入反复失败的重要原因之一[9⁃10]。据文献报道,约50%经IVF的胚胎存在染色体异常,约70%的IVF失败由染色体异常导致[11⁃12],因此为提高IVF⁃ET的成功率,将6个胚胎进行PGS检测,PGS检测结果分别为E1:45,XX,⁃5;E2:46,XX;E3:46,XY;E4:47,XY,+1;E5:46,XX;E6:46,XY,+1,⁃2;最后为了验证本次检测结果的准确性,将2个家系样本同时进行gap⁃PCR的检测。对比两者结果发现,对于SEA纯合缺失样本2种方法检测结果一致,而经过WGA的PCR产物对SEA携带者则检测结果为正常,与PGD检测结果不一致;这可能是由于在PCR过程中ADO所导致的[13],而本研究采用WGA结合NGS,通过单体型的构建可以推断出该胚胎2条DNA链的来源从而最终确定其是否携带致病基因;同时,通过低通量的全基因组测序来进行染色体核型的判断,从而选择核型正常且没有致病基因的胚胎进行植入,进而提高体外受精的妊娠率、阻断了地中海贫血患儿的出生。本研究结合WGA与NGS技术解决了样本微量的问题以及ADO造成的假阳性和假阴性的风险;而且还可以准确判断染色体非整倍体,更是解决了一些特殊基因不能直接诊断的局限性、样本污染等问题。
NGS当前已经广泛应用于无创产前诊断,同时也在PGD/PGS领域中应用。国内外均可见到NGS应用于PGD/PGS的报道。早期有报道称PGS不能显著提高植入率和活产率,这可能与早期的PGS技术主要是针对部分染色体异常进行检测有关。Kung等[14]采用活检的滋养外胚层细胞、卵裂球及已知的细胞系比较了NGS和微阵列比较基因组杂交(array⁃comparative genom ic hybridiza⁃tion,aCGH),结果显示,NGS的敏感度和特异度均为100%。而早期PGD主要应用荧光原位杂交(fluoresence in situ hybridization,FISH)和PCR等技术,存在一定的局限性,Moutou等[15]报道了PCR⁃PGD的误诊率为0.15%,而FISH⁃PGD误诊率为0.06%。目前尚未见NGS进行PGD的误诊率的相关报道。PGD/PGS还存在另外一个重要方面的争议是其子代的安全性问题,但目前尚缺乏大样本、前瞻性随机对照研究。无创胚胎染色体筛查(non⁃invasive chromosome screening,NICS)应运而生,该
新技术利用了游离在培养液中极微量的DNA,通过目前单细胞全基因组扩增技术实现对胚胎染色体的全面筛查,把从胚胎上提取细胞的活检形式改革成从培养液中提取DNA的检测形式,保护了胚胎样本的完整性和安全性,但该技术目前尚不成熟,还存在着WGA扩增效率低下、培养液污染等问题,要真正普遍应用于临床还有待进一步的探索。综合各方面因素本研究建立了基于NGS的PGD/ PGS技术,它可以获得基因组的全部信息,通过不同的检测和分析策略,完全有可能实现对植入前胚胎从染色体异常检测,到发现单基因突变甚至是新发突变等各个层面信息,同时该技术也可延伸到其他方面的临床应用,如无创单基因病产前诊断、HLA配型、染色体易位正常和携带者的区分以及亲子鉴定等,更具有市场潜力及应用前景。
[1]Xu XM,Zhou YQ,Luo GX,et al.The prevalence and spectrum of alpha and beta thalassaem ia in Guang⁃dong Province:implications for the future health bur⁃den and population screening[J].J Clin Pathol,2004,57(5):517-522.
[2]Xu YW,Zeng YH,Deng J,et al.Preimplantation ge⁃netic diagnosis for alpha⁃thalassaem ia in China[J].J AssistReprod Genet,2009,26(7):399-403.
[3]Xu Y,Chen S,Yin X,et al.Embryo genome profil⁃ing by single⁃cell sequencing for preimplantation genet⁃ic diagnosis in a beta⁃thalassemia family[J].Clin Chem,2015,61(4):617-626.
[4]Natesan SA,Bladon AJ,Coskun S,et al.Genome⁃w ide karyomapping accurately identifies the inheri⁃tance of single⁃gene defects in human preimplantation embryos in vitro[J].Genet Med,2014,16(11):838-845.
[5]Ko TM,Tseng LH,Hsieh FJ,et al.Carrier detection and prenatal diagnosis of alpha⁃thalassem ia of South⁃east Asian deletion by polymerase chain reaction[J]. Hum Genet,1992,88(3):245-248.
[6]Chang JG,Lee LS,Lin CP,et al.Rapid diagnosis of alpha⁃thalassem ia⁃1 of southeast Asia type and hydrops fetalis by polymerase chain reaction[J].Blood,1991,78(3):853-854.
[7]de Bourcy CF,de V lam inck I,Kanbar JN,et al.A quantitative comparison of single⁃cell whole genome amplification methods[J].Plos One,2014,9(8):e105585.
[8]Ning L,Li Z,Wang G,et al.Quantitative assessment of single⁃cellwhole genome amplificationmethods for detecting copy number variation using hippocampal neurons[J].Scientific Reports,2015,5(1):1.
[9]Handyside AH.24⁃chromosome copy number analy⁃sis:a comparison of available technologies[J].Fertil Steril,2013,100(3):595-602.
[10]Fiorentino F,Biricik A,Bono S,et al.Development and validation of a next⁃generation sequencing⁃based protocol for 24⁃chromosome aneuploidy screening of embryos[J].Fertil Steril,2014,101(5):1375-1382.
[11]Chung MK,Jeong HJ,Lee JH,et al.Comprehensive chromosome analysis of blastocysts before implanta⁃tion using array CGH[J].Mol Cytogenet,2013,6(1):22.
[12]SchoolcraftWB,Katz⁃Jaffe MG.Comprehensive chro⁃mosome screening of trophectoderm w ith vitrification facilitates elective single⁃embryo transfer for infertile women w ith advanced maternal age[J].Fertil Steril,2013,100(3):615-619.
[13]Yan L,Huang L,Xu L,et al.Live births after simul⁃taneous avoidance ofmonogenic diseases and chromo⁃some abnormality by next⁃generation sequencing w ith linkage analyses[J].Proc Natl Acad Sci USA,2015,112(52):15964-15969.
[14]Kung A,Wells D,Munne S,et al.Comparison of next generation sequencing(NGS)and aCGH for PGS[J].Human Reproduction,2014:17.
[15]Moutou C,Goossens V,Coonen E,et al.ESHRE PGD Consortium data collection XII:cycles from Jan⁃uary to December 2009 w ith pregnancy follow⁃up to October 2010[J].Hum Reprod,2014,29(5):880-903.
Successful preim p lantation genetic detection for alpha thalassaem ia by using next generation sequencing technology
XIEMeijuan1,DENG Quanheng1,DENG Honghui1,LU Shaoyue1,YANG Xuexi2,MA Qiang2★
(1.Guangzhou DaruiBiotechnology Co.,Ltd.,Guangzhou,Guangdong,China,510665;2.Schoolof Laboratory Medicine and Biotechnology,Southern Medical University,Guangzhou,Guangdong,China,510515)
Objective To explore the application of next generation sequencing(NGS)for alpha⁃thalassem ia in preimplantation genetic detection.Methods 2 couplesw ith alpha⁃thalassem ia Southeast Asia deletion(⁃⁃SEA deletion)and their embryos cells obtained after embryonic biopsy were selected to test by whole genome amplification(WGA)and NGS.Gap⁃PCR was done to all samples as a series of parallel tests. Results The PGD resultswere⁃⁃SEA/αα,⁃⁃SEA/⁃⁃SEA,⁃⁃SEA/αα,⁃⁃SEA/⁃⁃SEA,⁃⁃SEA/ααand⁃⁃SEA/⁃⁃SEA,respectively.And embryosw ith the abnormalαgenewere confirmed from thematernal side according to genealogicalanalysis.The PGS results of 6 embryos from 2 familieswere 45,XX,⁃5;46,XX;46,XY;47,XY, +1;46,XX and 46,XY+1⁃2.Both husbands and w ives of the 2 families gap⁃PCR resultswere heterozygotes w ith⁃⁃SEA deletion.The results of gap⁃PCR were normal in embryos 1,3 and 5 while⁃⁃SEA deletion were
Next generation sequencing;A lpha thalassaem ia;Preimplantation genetic diagnosis; Gap⁃PCR
广州市科技计划项目健康医疗协同创新重大专项(201400000004⁃4);广州市重大科技攻关项目子课题(2014Y2⁃00220);广州市科技计划产学研协同创新重大专项(201604020104);广东省科技计划项目公益研究与能力建设专项(2015A030401040)
1.广州市达瑞生物技术股份有限公司,广东,广州510665 2.南方医科大学检验与生物技术学院,广东,广州510515
★通讯作者:马强,E⁃mail:mqqqm2006@163.com
found in embryos 2,4 and 6 accordingly.Conclusion NGS not only can detect karyotype of 46 chromosomes but also avoid the risk of false positive and false negative caused by the allele drop⁃out(ADO) during the single cellamplification w ith a huge potentialmarketand w ide application prospect.
