微阵列比较基因组杂交技术与二代基因测序检测拷贝数变异的对比
2016-12-02陈新周王明珠何丹周慧林方欣李粉霞
陈新周 王明珠 何丹 周慧 林方欣 李粉霞★
·论著·
微阵列比较基因组杂交技术与二代基因测序检测拷贝数变异的对比
陈新周1王明珠2何丹1周慧1林方欣1李粉霞2★
目的对比分析微阵列比较基因组杂交技术(array⁃based comparative genomic hybridiza⁃tion,aCGH)和第二代基因测序技术(nextgeneration sequencing,NGS)在基因组拷贝数变异(copy number variation,CNV)上的一致性,为临床检测CNV提供新的检测方法。方法提取15例流产组织临床样本的基因组DNA,然后分别使用aCGH和NGS 2种方法对上述DNA进行检测,分析对比每个样本的CNV的数目。结果aCGH共检测到CNV数有109个,最小的片段有16 kb,最大的片段有34Mb,其中小于200 kb的有20个,大于200 kb小于1Mb的有34个,大于1Mb小于5Mb的有26个,大于5Mb小于34Mb的有29个。NGS共检出68个CNV,其中小于200 kb的有3个,检出率为15.0%;大于200 kb小于1Mb的有22个,检出率为64.7%;大于1Mb小于5 Mb的有20个,检出率为76.9%;大于5 Mb小于34Mb的有29个,检出率为100.0%。结论对于目前的数据量来说,NGS在检测大于5Mb的大片段CNV上检出率较高,与aCGH具有一定的一致性,可以应用于流产组织中CNV的检测。对于小片段的CNV的检测,需要增加相应的读取的数据量进行检测。
基因组拷贝数变异;第二代基因测序;微阵列比较基因组杂交技术
自然流产(spontaneous abortion,SA)在妊娠中的发生率为10%~15%[1]。孕12周之前的流产称为早期SA。与同一配偶发生连续2次或多次SA称为复发性流产。早期SA的病因复杂,50%以上的胚胎是由染色体异常造成的,染色体异常包括染色体数目异常,如单体、三体、多倍体;结构异常,如断裂、缺失、易位等。发生SA时的孕周越短,染色体异常的可能性越大。
拷贝数变异(copy number variation,CNV)指人类基因组中广泛存在的缺失、插入、重复和复杂位点的变异,长度为1 kb以上,突变率远高于单核苷酸多态性(single nucleotide polymorphism,SNP)。越来越多的研究表明[2⁃3],CNV与许多疾病有着密切的联系,在许多遗传疾病中扮演着重要的角色,特别是癌症、精神分裂等疾病,引起了研究者们的广泛兴趣。随着技术的发展,芯片技术[微阵列比较基因组杂交技术(array⁃based comparative genom ic hybridiza⁃tion,aCGH)或单核苷酸多态性微阵列技术(array single nucleotide polymorphism,SNP array)]也成为最近几年新兴的一种检测方法,能将检出率提高,不过受限于其技术原理,芯片探针无法覆盖的染色体区段将无法检测,这也限制发现新的染色体畸变,进而限制了发现新的染色体疾病的可能。然而高通量测序技术的迅猛发展,使得测序技术用于染色体拷贝数变异(CNV)检测的优越性越来越明显。本研究将通过aCGH和NGS 2种方法对流产组织CNV的检测,探讨NGS能否作为新的CNV的检测方法。
1 材料与方法
1.1 研究对象
研究对象为15例流产组织临床样本。
1.2 试剂
提取DNA所用的QIAamp®DNA M ini Kit试剂盒购自于德国QIAGEN公司,aCGH试剂盒购自于美国Agilent公司,NGS所用的建库试剂盒,模板制备试剂盒,测序试剂盒均购自于美国Life Technology公司。
1.3 仪器
aCGH芯片扫描仪SureScan M icroarray Scanner由美国Agilent公司研制。NGS使用的2100生物分析仪由美国Agilent公司研制,Ion One Touch 2和Ion One TouchTMES由美国Life公司研制,DA8600基因测序仪由中山大学达安基因股份有限公司提供。
1.4 方法
1.4.1 aCGH检测CNV
1.4.1.1 gDNA的提取
按照QIAamp®DNA M ini Kit说明书进行gDNA提取。
1.4.1.2 aCGH芯片上机前处理
将提取的DNA与试剂盒提供的reference进行片段化和随机扩增,然后使用Cy3和Cy5 2种染料标记DNA,并纯化标记后的DNA,使用NanoDrop 2000 UV⁃VIS分光光度计来检测并计算产量与比活度,只有产量和比活度符合要求后才能进行下一步的杂交。将符合要求的DNA加入到试剂盒提供的杂交室中,封闭杂交室并放入杂交箱中进行杂交。
1.4.1.3 上机检测
将芯片从杂交室中取出,并用纯水和试剂盒提供的Wash Buffer进行清洗,清洗后使用SureS⁃can M icroarray Scanner进行芯片的扫描,扫描后的图片结果可以使用Agilent公司提供的Agilent CytoGenom ics Edition软件进行分析。
1.4.2 NGS检测CNV
1.4.2.1 gDNA的提取
按照QIAamp®DNA M ini Kit说明书进行gDNA提取。
1.4.2.2 构建上机文库
将提取的gDNA按照文库构建试剂盒进行文库构建。首先DNA片段末端的补平,然后加上末端接头,最后PCR扩增DNA片段。扩增完后需要对所构建的文库进行检测,用Qubit检测DNA浓度,用生物分析仪检测片段长度。
1.4.2.3 DA8600上机测序
上机测序前,需要制备上机模板,并将模板进行富集。将DA8600进行清洗,先氯洗后再进行水洗,清洗完后将仪器初始化并设置上机Plan。芯
片上机前需要进行清洗和校准,校准完才能进行上机测序。上机测序结果可以在DA8600服务器上查看,利用染色体非整倍体检测数据分析系统进行数据分析,得到每个测序样本的Z值,根据参考范围进行结果判断。
2 结果
2.1 实验结果
2种实验的结果都可以通过检测的log2ratio值的大小来判断染色体的拷贝数变异,图1为随机选取实验中一个样本的2号染色体2种结果的对比图,图1a为aCGH结果,图1b为NGS的检测结果。2个结果中都显示出该样本的2号染色体在2q37.1q37.3(232,612,296⁃242,690,037)片段缺失,如图中蓝色箭头所示。
本次研究中,aCGH共检测到CNV数有109个,最小的片段有16 kb,最大的片段有34 Mb,其中小于200 kb的有20个,大于200 kb小于1 Mb的有34个,大于1Mb小于5Mb的有26个,大于5Mb的有29个。NGS的检测是共有74个CNV,其中小于200 kb的有3个,检出率为15.0%;大于200 kb小于1 Mb的有22个,检出率为64.7%;大于1Mb小于5Mb的有20个,检出率为76.9%;大于5Mb小于34Mb的有29个,检出率为100.0%,见表1所示。
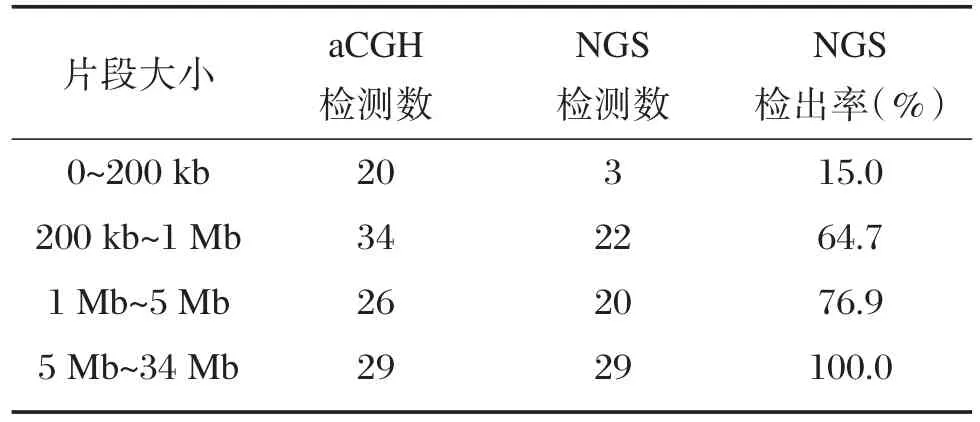
表1 aCGH与NGS的检测染色体拷贝数变异结果对比Table 1 Comparison of chromosomal copy number variants analysis by aCGH and NGS
3 讨论
遗传变异是生命进化演变的基本特征。从细胞遗传学水平看,DNA的变异主要包括染色体数目的变异和染色体结构的变异。数目变异包括染色体组成倍地增加或减少,以及单条染色体的增加或减少;结构变异主要指染色体片段的重复、缺失、倒位和易位等[4]。这些变异均会产生相应的遗传学效应,可能会导致变异个体表型发生严重改变,甚至死亡,也可能会发生现在未知的或许良性的改变。随着检测技术方法的不断发展,从染色体核型分析到原位荧光杂交,芯片到测序技术的使用,使得对染色体识别精确率逐步提高[5]。
染色体非整倍体改变仍然是流产胚胎的主要因素,集中在13、14、15、16、18、21、22号染色体,X单体也是早期流产常见的染色体核型[6]。双重三体较少见,约占流产胚胎所有核型的0.21%~2.8%[7]。
本研究中的15例流产物中,aCGH与NGS检出9例异常,异常率60%,与以往报道的异常率55%~66%[8]和51%[9]基本相符。15例流产物检测中,测序显示多态4例,还有2例检测阳性查询后显示致病性未知,而这些都是流产物中检出的,多态是否有致病效应存在一定争议,在公共数据库中查出致病性未知的这一部分是需要更多的经验积累,更进一步地研究这些多态和致病性间的关系。但从我们的结果可以看出,以目前的数据量来说,高通量测序技术相对于芯片方法来说,小片段的拷贝数变化并不能全部得到检测,但是大于5Mb的片段的检出率为100%。若相应的增加数据量,应该会达到更大的分辨率,这一点我们也会继续进行验证。
从对操作要求上来看,与已成相当规模的芯片杂交技术相比,NGS也有其独到的优势,在DNA质量的要求上,NGS对DNA的质量要求不
高,很低的浓度便可以满足实验的要求。而aCGH对于DNA的质量和纯度的要求都很高,如本次使用的安捷伦公司的芯片对于DNA浓度的要求需要在40 ng/μL,这就增加了对样本的要求。
本研究中用的测序方法是单端测序,则该测序方法与aCGH方法均不能解决平衡性的易位、倒位、整倍体等问题。而有研究表明随着测序读长的不断增加,NGS通过mate⁃pair文库或称跨步文库可能实现平衡易位的检测[10],不久的将来平衡异位可通过测序进行检测,这一个难点的突破将大大的提高测序的价值与意义,将会成为NGS最大的优势。NGS还有其他的优势,如其可以检测新的未知基因变异,同时因其可能更为低廉的高通量优势成本而利于临床推广;此外,与肿瘤发生相关基因的拷贝数变异和基因多态性分析等都有着足够应用的空间和优势[11⁃12]。
随着测序技术的不断发展和广泛运用,从产前诊断的游离DNA的测序[13⁃14],到流产物、羊水的测序CNV检测,到用母体外周血测序测胎儿CNV检测、单基因疾病检测,到单细胞植入前诊断测序检测、肿瘤游离DNA检测等广泛运用[15],测序技术用于流产物分析既可较为全面得出导致流产的遗传物质的改变原因,又可积累新染色体疾病的发现,还可用于生殖领域的促进,分析流产原因,测序技术用于胚胎植入前遗传学筛查(preimplantation ge⁃netic screening,PGS)可有效提高成功率[16⁃17]。
综上所述,对于较大片段的拷贝数变化的检测,NGS技术和aCGH技术的诊断成功率及染色体异常检出率均较高,在早期SA妊娠物核型分析中具有显著优越性,可作为遗传学检测的一种补充技术,也可为流产夫妇进行遗传学病因分析,从而为再次妊娠提供指导,有较高的临床应用价值。
[1]殷研.孕妇营养状况与枯萎卵妊娠关系的研究[D].北京:中国协和医科大学,2008,8-9.
[2]卢永娟,王瑞,倪华美,等.拷贝数变异(CNVs)导致遗传变异的研究进展[J].黑龙江畜牧兽医,2015,21:58-60,63.
[3]马天骏,邓祯祥,陈诗哲,等.DNA拷贝数变异及其研究进展[J].中华临床医师杂志:电子版,2013,7(14):6592-6595.
[4]夏家辉.医学遗传学[M].北京:人民卫生出版社,2004,166.
[5]眭维国,欧明林,戴勇,等.先天性疾病研究及产前诊断中Array CGH的应用[J].国际妇产科学杂志,2010,37(2):81-83.
[6]Stephenson MD,Awartani KA,Robinson WP.Cyto⁃genetic analysis of miscarriages from couples w ith re⁃currentmiscarriage:a case⁃control study[J].Hum Re⁃prod,2002,17(2):446-451.
[7]Diego⁃Alvarez D,Ramos⁃Corrales C,Garcia⁃Hoyos M,et al.Double trisomy in spontaneousmiscarriages:cytogenetic and molecular approach[J].Hum Reprod,2006,21(4):958-966.
[8]Lim JH,Kim MH,Han YJ,et al.Cell⁃free fetal DNA and cell⁃free total DNA levels in spontaneous abortion w ith fetal chromosomal aneuploidy[J].Plos One,2013,8(2):e56787.
[9]Sugiura⁃Ogasawara M,Ozaki Y,Katano K,et al.Abnonnal embryonic karyotype is the most frequent cause of recurrent m iscarriage[J].Hum Reprod,2012,27(8):2297-2303.
[10]TalkowskiME,Ordulu Z,Pillalamarri V,etal.Clini⁃cal diagnosis by whole genome sequencing of a prena⁃tal sample[J].N Engl JMed,2012,367(23):2226-2232.
[11]Pan Q,ShaiO,Lee LJ,etal.Deep surveying of alter⁃native splicing complexity in the human transcriptome by high⁃through put sequencing[J].NatGenet,2008,40(12):1413-1415.
[12]Pfifer GP,Besaratinia A.Mutational sqectra of human cancer[J].Hum Genet,2009,125(5/6):493-506.
[13]赖允丽,陈筠,易升,等.无创产前检测的临床应用及随访研究[J].重庆医学,2016,45(11):1471-1475.
[14]贾蓓,钟梅.高通量测序技术在无创产前检测中的应用[J].中国实用妇科与产科杂志,2015,31(9):807-810.
[15]张小珍,尤崇革.下一代基因测序技术新进展[J].兰州大学学报:医学版,2016,42(3):73-80.
[16]张静.人类早期胚胎植入前高通量测序遗传学筛查的研究[D].天津:天津医科大学,2015.
[17]谭跃球.下一代测序与PGS[A].2015年浙江省医学遗传学学术年会暨高通量基因测序产前筛查与诊断技术研讨会论文汇编,2015,10.
The com parison between array⁃CGH w ith next generation sequencing in the detection of copy number variations
CHEN Xinzhou1,WANGM ingzhu2,HEDan1,ZHOU Hui1,LIN Fangxin1,LIFenxia2★
(1.Guangzhou Darui Biotechnology Co.,Ltd.,Guangzhou,Guangdong,China,510665;2.Schoolof Labora⁃tory Medicine and Biotechnology,Southern Medical University,Guangzhou,Guangdong,China,510515)
Objective To provide a new method for clinical detection of copy number variants (CNV)by comparing the consistency between array⁃based comparative genomic hybridization(aCGH)and next generation sequencing(NGS)in the genome copy number variation.Methods DNA was extracted from 15 cases of clinical specimens and detected by both aCGH and NGSmethods.CNV numbers were analyzed in each sample.Results The results showed thataCGH identified 109 CNVs,in that the shortest CNV was 16 KB,and the longestCNV was 34MB.Therewere 20 CNVs shorter than 200 kb,34 CNVs between 200 kb and 1Mb,26 CNVs between 1Mb and 5Mb,and 29 CNVs between 5Mb and 34Mb.NGS identified 68 CNVs,of them 3 were shorter than 200 kb(15%),22 between 200 kb and 1 Mb(64.7%),20 between 1 Mb and 5 Mb (76.9%),29 between 5Mb and 34Mb(100%).Conclusion NGS has good consistency w ith aCGH in detect⁃ing CNVs longer than 5 Mb.Therefor it can be a reliablemethod to test the CNV.The performance of NGS in testing the short CNVs,especially the CNVs shorter than 200 kb w ill have a better resultby increasing the read
Copy number variation;Nextgeneration sequencing;Array based comparative genom ic hybridization
广州市科技计划产学研协同创新重大专项(201604020104);广东省科技计划项目公益研究与能力建设专项(2015A030401040);广州市重大科技攻关项目子课题(2014Y2-00220)
1.广州市达瑞生物技术股份有限公司,广东,广州510665 2.南方医科大学检验与生物技术学院,广东,广州510515
★通讯作者:李粉霞,E⁃mail:lifenxia123@qq.com
data.
