利用CRISPR/Cas9技术敲除水稻Pi21基因的效率分析
2016-10-25王芳权范方军李文奇朱金燕王军仲维功杨杰
王芳权 范方军 李文奇 朱金燕 王军 仲维功 杨杰,*
(1江苏省农业科学院 粮食作物研究所/国家水稻改良中心南京分中心/江苏省优质水稻工程技术研究中心, 南京 210014;2扬州大学 江苏省粮食作物现代产业技术协同创新中心, 江苏 扬州 225009; *通讯联系人, E-mail: yangjie168@aliyun.com)
利用CRISPR/Cas9技术敲除水稻Pi21基因的效率分析
王芳权1,2范方军1,2李文奇1,2朱金燕1,2王军1,2仲维功1,2杨杰1,2,*
(1江苏省农业科学院 粮食作物研究所/国家水稻改良中心南京分中心/江苏省优质水稻工程技术研究中心, 南京 210014;2扬州大学 江苏省粮食作物现代产业技术协同创新中心, 江苏 扬州 225009;*通讯联系人, E-mail: yangjie168@aliyun.com)
WANG Fangquan, FAN Fangjun, LI Wenqi, et al. Knock-out efficiency analysis ofPi21 gene using CRISPR/Cas9 in rice. Chin J Rice Sci, 2016, 30(5): 469-478.
利用CRISPR/Cas9技术,针对Pi21基因的两个靶位点(靶位1和靶位2),构建基因敲除载体,转化水稻品种南粳9108。经PCR鉴定,获得了28株T0代阳性转基因植株。对靶位点酶切检测发现,靶位 1突变效率为78.57%,靶位 2突变效率为92.86%;靶位 1和靶位 2同时突变的效率为78.57%,突变效率较高。通过对靶位点进行测序,发现靶位点突变类型较多,包括碱基缺失、碱基插入、碱基缺失后插入其他碱基和大片段DNA缺失等类型。对突变株系进行氨基酸预测,发现大部分株系都存在移码突变现象而使基因突变彻底,少数株系表现为部分氨基酸的缺失或变异。成功敲除了水稻Pi21基因,并对突变效率和类型进行了分析,为进一步验证Pi21基因功能、培育广谱抗稻瘟病的水稻新品系奠定了基础。
CRISPR/Cas9; 稻瘟病;Pi21; 基因敲除
CRISPR/Cas9是近几年发展起来的一类基因编辑技术,它能对特定的靶位点进行基因编辑,技术门槛低,操作简单,试验周期短,成本低廉[1-4]。随着CRISPR/Cas9技术的不断完善和成熟,该技术已经在微生物[5,6]、动物[7-9]和植物[10-13]等领域广泛应用。利用CRISPR/Cas9技术,科研工作者已经在水稻[12-14]、拟南芥[15]、烟草[15,16]、玉米[17]、小麦[13,18]、高粱[11]、大豆[19-21]、番茄[22]等植物上实现了基因组编辑。Shan等[13]利用CRISPR/Cas9技术对水稻OsPDS-P1和OsBADH2基因进行编辑,开启了CRISPR/Cas9技术在水稻基因编辑中的先河。目前,CAO1[23]、PDS[24]、PMS3[14]、AOX1[25]、Waxy[26]等水稻重要功能基因已经成功实现编辑。
表1本研究所用的引物
Table 1. Primers used in this research.

引物名称Primername引物序列(5'→3')Primersequence(5'→3')Pi21g-FACCAAAGCCTGTCTATCTGCPi21g-RCACATCGATCAGCCTCGTGPi21UT1-FGGCAGAAGCTGTGCAAGAAGATCPi21UT1-RAAACGATCTTCTTGCACAGCTTCPi21UT2-FGCCGCCGCTGCGATGCCAAGATCPi21UT2-RAAACGATCTTGGCATCGCAGCGGpU-FCTCCGTTTTACCTGTGGAATCGgRNA-RCGGAGGAAAATTCCATCCACPi21T1L-FTTCAGAGGTCTCTCTCGCACTGGAATCGGCAGCAAAGGPi21T1L-RAGCGTGGGTCTCGTCAGGGTCCATCCACTCCAAGCTCPi21T2L-FTTCAGAGGTCTCTCTGACACTGGAATCGGCAGCAAAGGPi21T2L-RAGCGTGGGTCTCGACCGGGTCCATCCACTCCAAGCTCSP1CCCGACATAGATGCAATAACTTCSP2GCGCGGTGTCATCTATGTTAPi21T1-FAGGCTAATCAGCAGTGTTCCTPi21T1-RCAGCTTGCACTCCGGCTTCGPi21T2-FATTGGTAACATTCGGCAAATTPi21T2-RGTTCTTCACGTCGTACTCCA
稻瘟病是水稻生产上危害性极大的病害之一,目前已经报道了很多主效稻瘟病抗性基因,但大多数都是小种特异性基因[27]。Pi21基因是一个广谱抗稻瘟病基因,其抗性不因稻瘟病菌生理小种的变化而改变,具有很好的应用前景[28-30]。在Pi21基因多个编码区缺失的复等位基因中,只有其功能缺失型等位基因pi21才具有稻瘟病抗性,而且该抗病基因型pi21与高垩白相关基因紧密连锁[29],因而用传统杂交方法获得的抗稻瘟病株系稻米品质往往较差,为培育优质的抗稻瘟病水稻品系增加了很大困难。
为了获得广谱抗稻瘟病的水稻遗传材料,本研究构建了含有Pi21基因两个靶位点的CRISPR/Cas9突变载体,以优良食味水稻品种南粳9108为受体材料,获得了一系列Pi21基因突变株系,并对其突变情况进行了分析。本研究成功实现了水稻Pi21基因的单基因双靶点同时敲除,有利于进一步验证Pi21基因功能,为水稻稻瘟病抗病育种提供了新思路。
1 材料与方法
1.1植物材料、载体和菌株
转基因受体材料为江苏省农业科学院粮食作物研究所选育的优良食味水稻品种南粳9108。
本研究所用引导序列载体gRNA-U3、gRNA-U6a和转基因载体CRISPR/Cas9由华南农业大学刘耀光教授课题组开发[26]。pMD19-T购自TaKaRa公司,大肠杆菌DH5α、农杆菌EHA105由本实验室保存。
1.2主要试剂
限制性内切酶BsaⅠ购自NEB公司,T4 DNA连接酶、PrimeSTAR HS DNA聚合酶购自TaKaRa公司。卡那霉素等其他分子生物学试剂购自南京天为生物科技有限公司。
1.3引物
本研究所设计的引物见表1。引物由南京锐真生物科技有限公司合成。
1.4靶位点的选择与验证
根据CRISPR/Cas9系统识别原型间隔序列毗邻基序(Protospacer adjacent motif, PAM)上游约20 nt核酸序列的特点设计2个靶位点(靶位1和靶位2),靶位点的特异性通过Cas-OFFinder(http://www.rgenome.net/cas-offinder/)分析和水稻基因组BLAST分析进行验证。
1.5CRISPR/Cas9表达载体构建
利用合成引物Pi21UT1-F/Pi21UT1-R和Pi21UT2-F/Pi21UT2-R,分别制成靶点接头Pi21T1(1 μmol/L)和Pi21T2(1 μmol/L)。分别将接头Pi21T1和Pi21T2连接gRNA-U3和gRNA-U6a载体,反应体系包括gRNA载体 1 μL,接头1 μL,10 × DNA连接缓冲液1 μL,BsaⅠ (5 U) 0.5 μL,T4 DNA连接酶0.2 μL,去离子水6.3 μL。反应程序如下:37℃下5 min,20℃下 5 min;5个循环。
获得的产物进行2轮巢式PCR。第一轮PCR体系包括2 × PrimeSTAR GC缓冲液7.5 μL,dNTP 混合液1.5 μL,引物pU-F、gRNA-R各1.5 μL,PrimeSTAR HS DNA聚合酶0.2 μL,gRNA连接产物2 μL,去离子水0.8 μL。PCR程序如下:95℃下1 min;95℃下10 s,60℃下15 s,68℃下20 s,10个循环;95℃下10 s,60℃下15 s,68℃下30 s,22个循环;4℃下保存。第二轮PCR体系与第一轮PCR类似,将扩增引物换成Pi21T1L-F/Pi21T1L-R和Pi21T2L-F/Pi21T2L-R,扩增模板为第一轮PCR产物稀释50倍。
将Pi21T1和Pi21T2扩增产物同时连接CRISPR/Cas9载体。反应体系包括Pi21T1、Pi21T2扩增产物各1 μL,CRISPR/Cas9载体5 μL,BsaⅠ 1 μL,10 × DNA连接缓冲液2 μL,T4 DNA 连接酶0.5 μL,去离子水4.5 μL。反应程序如下:37℃下5 min,10℃下5 min,20℃下5 min;15个循环。
连接产物用热激法转化大肠杆菌DH5α,菌液涂布于含有50 mg/L卡那霉素的LB平板上,培养约12 h。挑取平板上长出的单菌落,摇菌扩繁。以菌液为模板,以引物组合SP1/Pi21T1-R和Pi21T2-F/SP2进行PCR验证。提取阳性菌株的质粒,再以上述引物进行PCR验证。质粒送往南京锐真生物科技有限公司测序验证。将确认无误的阳性质粒转入农杆菌EHA105。
1.6阳性转基因植株的获得及验证
利用构建好的阳性农杆菌EHA105转化水稻品种南粳9108的愈伤组织,用潮霉素筛选获得再生组培苗,即为T0代植株。组培苗经炼苗后,移栽盆钵,按转基因水稻种植要求严格管理。提取T0代植株的基因组DNA,用T-DNA特异引物SP1和Pi21T1L-R进行PCR扩增,理论产物长度约为525 bp,能扩增出目的片段的植株即为阳性转基因植株。
1.7酶切检测靶位点
为了检测靶位点的突变情况,跨越靶位 1设计引物Pi21T1-F和Pi21T1-R(表1),理论产物长度为494 bp;野生型水稻的扩增产物能被BglⅡ (A/GATCT)识别并完全酶切成181和313 bp条带,突变基因型则不能被酶切。跨越靶位 2设计引物Pi21T2-F和Pi21T2-R(表1),理论产物长度为654 bp,野生型水稻的扩增产物能被MboⅠ(/GATC)识别并完全酶切成450和204 bp条带,突变基因型则不能被酶切。
1.8测序检测Pi21基因型和靶位点
以南粳9108品种基因组为模板,用Pi21g-F和Pi21g-R引物组合扩增Pi21基因,回收扩增产物,送往南京锐真生物科技有限公司测序,用DNAMAN软件进行核酸序列比对分析。
利用Pi21T2-F和Pi21T1-R引物组合,能同时扩增出包含靶位 1和靶位 2的DNA序列。将扩增产物连接pMD19-T载体后,转化大肠杆菌DH5α,挑取的单菌落摇菌后用PCR检测,阳性菌液送往南京锐真生物科技有限公司测序。每个株系检测2~4个单菌落。测序结果用FinchTV软件读取,核酸序列分析、氨基酸序列预测和比对采用BioXM 2.6软件。
2 结果与分析
2.1南粳9108的Pi21基因型及靶位点设计
接种鉴定试验表明,南粳9108苗期对江苏省稻瘟病B、C、D生理小种都表现为高感[31]。对水稻品种南粳9108的Pi21基因测序分析表明,其富脯氨酸编码区的部分碱基缺失,未造成移码突变(图1)。Pi21基因由2个外显子和1个内含子构成,其功能区域为两段富含脯氨酸的氨基酸基序,该富脯氨酸元件编码序列完全缺失导致Pi21基因丧失功能而具有稻瘟病抗性,而另外11种富脯氨酸元件编码序列部分缺失类型(缺失碱基数都为3的倍数)均未造成Pi21基因功能丧失,不具有稻瘟病抗性[29]。利用RNA干扰Pi21基因,能显著提高水稻对稻瘟病的抗性[29]。因此,南粳9108的Pi21基因属于感稻瘟病基因型,通过基因突变有望提高对稻瘟病的抗性。
根据CRISPR/Cas9系统识别特点设计两个靶位点,靶位点位于富脯氨酸编码序列之前(图2)。靶位 1在编码区+158到+180处,其理论突变位点的碱基序列AGATCT能被BglⅡ识别并切割;靶位2在编码区+38到+60处,其理论突变位点的碱基序列GATC能被MboⅠ识别并切割。选择能被限制性内切酶识别并切割的位点进行突变,有利于对转基因植株突变情况进行检测。将设计的靶序列进行Cas-OFFinder分析和BLAST比对分析,发现靶位 1和靶位 2特异匹配目标位点,不会或不易引起脱靶效应。靶位 1和靶位 2的命名是根据它们在载体上的前后顺序来确定的,而不是它们在基因位点的顺序。

南粳9108感稻瘟病, 为Pi21感病基因型; Owarihatamochi抗稻瘟病, 为pi21抗病基因型。
Nanjing 9108 is a blast sensitive variety, containing the susceptible allelePi21. Owarihatamochi is a blast resistant variety, containing the resistant allelepi21.
图1Pi21基因编码序列比对
Fig. 1. Coding sequences co-linearity analysis of Pi21 gene.

加粗的碱基为PAM,下划线标出的碱基为限制性内切酶识别位点,黑色区域为外显子,白色区域为内含子,灰色方框标识区域为富脯氨酸元件。
The base in bold indicates PAM; The restriction endonuclease recognized site is underlined; The black region indicates exon; The white region indicates intron; The gray box region indicates proline-rich motifs.
图2Pi21基因结构及gRNA靶点位置
Fig. 2. Schematic diagram of Pi21 gene structure and gRNA targets.
2.2表达载体的构建及转化水稻
采用酶切连接的方法构建Pi21突变载体,Target 1-gRNA由OsU3基因启动子驱动,Target 2-gRNA由OsU6a基因启动子驱动,构建好的载体命名为CRISPR/Cas9-Pi21载体(图3)。经过PCR、酶切和测序验证无误后(数据未显示),转入农杆菌EHA105。以阳性农杆菌EHA105菌株为介体转化水稻品种南粳9108的愈伤组织,筛选获得抗潮霉素的再生T0代组培苗。用载体特异性引物(SP1和Pi21T1L-R)鉴定组培苗是否携带目的基因。如图4所示,野生型植株不能扩增出目的片段;42株组培苗中,28株能扩增出目的片段,为阳性转基因苗;14株不能扩增出目的片段,为假阳性苗。

图3CRISPR/Cas9-Pi21载体结构
Fig. 3. Schematic diagram of the CRISPR/Cas9-Pi21 vector construction.

M-DL 2000 DNA标记; 1~42-T0代转基因株系; WT-南粳9108(野生型)。
M, DL 2000 DNA marker; 1-42, T0transgenic lines; WT, Nanjing 9108 (wild-type).
图4PCR鉴定转基因阳性植株
Fig. 4. PCR identification of the positive transgenic plants.
2.3酶切法分析Pi21基因突变效率
利用引物组合Pi21T1-F和Pi21T1-R,分析靶位1突变情况,结果如图5所示。从PCR扩增产物来看,株系10、14和20的PCR产物明显小于野生型的条带,可初步判定这3个株系发生突变。野生型扩增出494 bp条带,其产物能被BglⅡ完全酶切成181 bp和313 bp条带。转基因植株酶切分析发现,42个T0代株系中,19个株系(1、3、7、10、13~16、19~22、24、25、35、37、39、41和42)不能被酶切,为两个等位基因都发生突变的株系;3个株系(4、11和17)只能被部分酶切,为只有一个等位基因发生突变的株系;20个株系(2、5、6、8、9、12、18、23、26~34、36、38和40)能被完全酶切,为未发生突变的株系。从PCR产物大小推断株系10、14和20发生突变的结果得到酶切结果的验证。经PCR验证为假阳性植株的14个株系(9、18、23、26~30、32~34、36、38、40),在酶切检测靶点突变的结果中都表现为未突变。有6个株系(2、5、6、8、12和31)虽然为阳性转基因植株,但靶位1未发生突变。
利用引物组合Pi21T2-F和Pi21T2-R,分析靶位 2突变情况。如图6所示,野生型扩增出654 bp条带,其产物能被MboI完全酶切成450和204 bp条带。从PCR产物来看,株系14和41的PCR产物小于野生型的条带,可初步判定这两个株系发生了突变。酶切后发现,42个T0代株系中,24个株系(3、4、6~8、10~17、19~22、24、25、35、37、39、41和42)不能被酶切,1个株系(株系31)酶切后大小与野生型不同,为两个等位基因都发生突变的株系;1个株系(株系1)只能被部分酶切,为只有一个等位基因发生突变的株系;16个株系(2、5、9、18、23、26~30、32~34、36、38和40)能被完全酶切,为未发生突变的株系。利用PCR产物大小推断株系14和41发生突变的结果得到酶切结果的验证。经PCR验证为假阳性植株的14个株系,靶位 2均未发生突变。有2个株系(2和5)尽管携带CRISRP/Cas9-Pi21结构,但靶位 2未发生突变。

A-酶切前; B-酶切后。 1~42-T0代转基因株系; WT-南粳9108(野生型)。
A, Before digestion; B, After digestion. 1~42, T0transgenic lines; WT, Nanjing 9108 (wild-type).
图5酶切鉴定靶位 1突变情况
Fig. 5. Mutation identification at Target 1 by enzyme digestion.
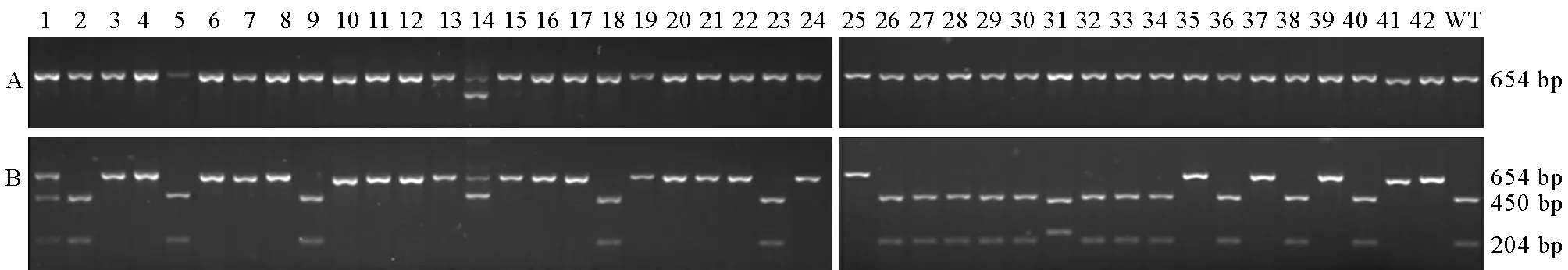
A-酶切前; B-酶切后。 1~42-T0代转基因株系; WT-南粳9108(野生型)。
A, Before digestion; B, After digestion. 1~42, T0transgenic lines; WT, Nanjing 9108 (wild-type).
图6酶切鉴定靶位 2突变情况
Fig. 6. Mutation identification at Target 2 by enzyme digestion.
因此,经过酶切法分析,在28株阳性转基因植株中,22株靶位 1发生突变,突变效率为78.57%;26株靶位 2发生突变,突变效率为92.86%;靶位 1和靶位 2同时发生突变的株系有22株,突变的效率为78.57%。
2.4靶位点突变类型分析
基于酶切鉴定的结果,我们对靶位 1、靶位 2都发生突变的8个株系进行了测序分析,进一步研究了靶位点突变情况,结果如图7和图8所示。与酶切检测结果一致,所测序的8个株系的靶位 1和靶位 2均发生了不同类型的碱基变异。其中,4个株系(16、21、22和41),靶位 1和靶位 2的两个等位基因突变类型均相同,为突变纯合系。3个株系(13、20和39),靶位 1和靶位 2的两个等位基因同时发生突变,但突变类型不一致,为杂合突变系。株系10的酶切鉴定结果为突变杂合型(PCR产物长度呈多种带型),但3个独立的单克隆测序都只检测到一种突变类型,可能另一种突变类型PCR扩增效率低,测序时没有检测到。因此,测序的8个株系中,靶位 1和靶位 2都检测到11个突变类型。
靶位 1和靶位 2突变均主要以碱基缺失为主。对于靶位 1,在检测的11个突变类型中,有3个为缺失1 bp的突变类型,占27.27%;其他还包括缺失4、15、23、34和180 bp等突变类型。对于靶位 2,在检测的11种突变型中,缺失2或4 bp为主要的突变类型,占63.64%。另外,本研究还发现,20-2突变系存在DNA大片段(559 bp)缺失,靶位 1和靶位 2中间的序列全部删除,这可能是靶位 1和靶位 2的突变事件同时发生的结果。
已有研究发现,CRISPR/Cas9介导的双链DNA断裂修复过程中,插入突变的碱基通常为A或T[26]。我们也发现了靶位 1和靶位 2存在着碱基插入的现象。株系16的靶位 1有一个碱基A插入;株系20-1的靶位 1和株系10的靶位 2均在缺失部分碱基后,又插入了一个碱基(T/A);株系13-2的靶位 2则在缺失19 bp后,又插入了15 bp,插入的碱基中,有12 bp(GTATAATAAATT)可能来源于与突变靶位点相邻的位置(图8)。

图7野生型、转基因株系16和21靶位测序结果
Fig. 7. Sequencing results of target sites from wild-type, transgenic lines 16 and 21.

“-1”和“-2”表示不同突变类型,括号内的数值为测序的克隆数,箭头所指的位置为Cas9剪切位点,加粗碱基为前间区序列邻近基序(PAM),红色字体的碱基为靶序列,蓝色字体的碱基为插入碱基。WT, 野生型;#, 转基因株系; d, 缺失; i, 插入。
“-1” and “-2” indicate different mutation type; The numbers in brackets are the numbers of sequenced clone; The arrows indicate the digestion sites of Cas9; The base in bold indicates protospacer adjacent motif(PAM); The base in red is the target sequence; The base in blue is the insert base; WT, Wild-type; #, Transgenic lines; d, Deletion; i, Insert.
图8靶位点突变序列分析
Fig. 8. Analysis of mutation sequences of targets.

图9突变株系的氨基酸序列共线性分析
Fig. 9. Co-linearity analysis of amino acid sequences of mutant lines.
2.5突变植株的氨基酸序列分析
进一步对突变株Pi21基因表达氨基酸序列进行分析(图9),发现株系13、16、20-1、21、22和39的表达均由于靶位 2变异引发的移码突变而提前终止;株系20-2中DNA大片段缺失引起基因起始密码子丢失,Pi21基因不表达;株系41中靶位 2缺失12个氨基酸,靶位 1造成移码突变;株系10中靶位 2突变造成11个氨基酸缺失和1个氨基酸改变,靶位 1缺失60个氨基酸。表明本研究所获得的转基因植株,Pi21基因编码的氨基酸都存在不同程度的变异。
3 讨论
CRISPR/Cas9技术是近年来发展起来的一种基因定点编辑技术,具有简单、快速、高效等特点,目前已经在微生物、动物和植物中获得了广泛应用[5-13]。本研究利用CRISPR/Cas9技术,针对水稻Pi21基因设计两个靶位点(靶位 1和靶位 2),实现了Pi21基因的敲除。
根据CRISPR/Cas9系统识别特点,选择能被限制性内切酶识别并切割的位点进行突变,有利于快速且较准确地检测转基因植株靶位点是否发生突变。但CRISPR/Cas9系统基因编辑后突变情况较复杂,酶切检测可能无法包含所有突变类型;因此,实际突变率可能略高。
本研究设计的两个靶位点虽然位于同一基因,但它们的突变效率存在差异,靶位 1突变效率为78.57%,靶位 2突变效率为92.86%。在CRISPR/Cas9系统中,gRNA引导Cas9核酸酶识别并剪切靶位点是实现基因编辑的关键,gRNA识别、结合靶位点的能力直接影响基因编辑的效率[26]。GC含量较高的靶序列,基因突变的效率往往也较高。我们选择的两个靶点中,靶位 1的GC含量为45%,突变效率为78.57%;靶位 2的GC含量为65%,突变效率为92.86%,与前人报道结果类似[26]。两个靶位点表现出不同的突变效率,可能与gRNA对靶序列的结合能力有关。
CRISPR/Cas9系统对靶位点的编辑会引发基因多种形式的突变,例如碱基的缺失、插入或替换等[13,15,16,32]。本研究中出现的突变类型主要是少数碱基缺失,部分株系也存在碱基插入、碱基缺失和插入同时发生、大片段DNA缺失等突变现象。在突变株系中,39-1的靶位 1和41的靶位 2出现的缺失碱基数为3的整数倍,导致基因在相应位点直接删除若干个氨基酸而不引起移码突变。因此,若选择适当位点进行突变,可能获得部分靶基因表达产物活性减弱的株系,可以为某些必需基因的突变、改变蛋白某个活性中心、改变蛋白空间结构等研究提供突变材料。
Ma等[26]利用CRISPR/Cas9技术对水稻Os03g0126800和Os03g0409500基因进行双靶点编辑时发现,部分突变株系的两个靶位点之间的序列出现删除。本研究中,我们也发现株系20-2的靶位 1和靶位 2中间的序列全部缺失,可能是由于两个位点的突变事件同时发生引起,为敲除两个位点中间一段序列提供了一定的依据。株系13-2的靶位 2出现了19个碱基缺失后15个碱基的插入,插入碱基以T/A为主,与以前人报道的结果类似[14,26];分析发现,插入碱基中的12个碱基可能来源于PAM位点下游的一段序列,这可能为CRISPR/Cas9系统对编辑基因机理的探索提供部分依据。
利用CRISPR/Cas9突变载体对靶序列的编辑时,通常会得到多种类型的T0代株系,包含无突变型、杂合突变型、突变纯合型、突变杂合型等[25]。由于等位基因突变类型不同,杂合突变型和突变杂合型株系自交后代存在功能丧失、功能减弱或无义突变等类型的分离,不利于基因功能分析或稳定新品系选育。突变纯合型后代则不会出现分离,能从T1代植株中直接选育到基因型纯合、性状稳定的株系,有利于加快新品系选育。在本研究测序的8个株系都能检测到突变发生,其中有4个株系为突变杂合型,4个株系为突变纯合型,纯合突变比例较高,能快速获得遗传稳定的株系。
传统的回交遗传改良,周期长,工序繁琐,连锁累赘难以打破,因而较难达到理想的效果[33]。CRISPR/Cas9技术可以特异地对一个基因或多个基因进行定点编辑,实现对一种或多种特定性状的定向改变,大大缩短了作物品系改良的周期,且不影响作物的其他性状。通过后代的自交分离,能获得剔除外源基因的株系,在作物育种和遗传改良方面具有重要的应用前景[25]。
稻瘟病是水稻主要病害之一,近年来在全国范围内均有发生,严重影响水稻产量和品质[34]。培育水稻抗稻瘟病品种是防治稻瘟病最有效的手段,但由于稻瘟病菌生理小种多样,进化速度快,导致这些抗病品种推广3~5年后即失去抗性。目前,已报道的抗稻瘟病基因中,大多数是垂直抗性基因,而pi21为水平抗性基因。本研究针对粳稻品种南粳9108Pi21的感病基因型,利用CRISPR/Cas9技术,实现了Pi21基因的定点突变,但Pi21基因突变的遗传性及突变株系是否提高了对稻瘟病的抗性,还需进一步研究。
[1]Belhaj K, Chaparro-Garcia A, Kamoun S, et al. Editing plant genomes with CRISPR/Cas9.CurrOpinBiotech, 2015, 32: 76-84.
[2]Baltes N J, Voytas D F. Enabling plant synthetic biology through genome engineering.TrendsBiotechnol, 2015, 33(2): 120-131.
[3]解莉楠, 宋凤艳, 张旸. CRISPR/Cas9 系统在植物基因组定点编辑中的研究进展. 中国农业科学, 2015, 48(9): 1669-1677.
Xie L N, Song F Y, Zhang Y. Progress in research of CRISPR/Cas9 system in genome targeted editing in plants.SciAgricSin, 2015, 48(9): 1669-1677. (in Chinese with English abstract)
[4]李君, 张毅, 陈坤玲, 等. CRISPR/Cas 系统: RNA 靶向的基因组定向. 遗传, 2013, 35(11): 1265-1273.
Li J, Zhang Y, Chen K L, et al. CRISPR/Cas: A novel way of RNA-guided genome editing.Hereditas, 2013, 35(11): 1265-1273. (in Chinese with English abstract)
[5]Jiang W, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems.NatBiotechnol, 2013, 31(3): 233-239.
[6]DiCarlo J E, Norville J E, Mali P, et al. Genome engineering inSaccharomycescerevisiaeusing CRISPR-Cas systems.NucleicAcidsRes, 2013, 41(7): 4336-4343.
[7]Cong L, Ran F A, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems.Science, 2013, 339(6121): 819-823.
[8]Mali P, Yang L, Esvelt K M, et al. RNA-guided human genome engineering via Cas9.Science, 2013, 339(6121): 823-826.
[9]Wang H, Yang H, Shivalila C S, et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering.Cell, 2013, 153(4): 910-918.
[10]Feng Z, Zhang B, Ding W, et al. Efficient genome editing in plants using a CRISPR/Cas system.CellRes, 2013, 23(10): 1229-1232.
[11]Jiang W, Zhou H, Bi H, et al. Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification inArabidopsis, tobacco, sorghum and rice.NucleicAcidsRes, 2013, 41(20): e188.
[12]Mao Y, Zhang H, Xu N, et al. Application of the CRISPR-Cas system for efficient genome engineering in plants.MolPlant, 2013, 6(6): 2008-2011.
[13]Shan Q, Wang Y, Li J, et al. Targeted genome modification of crop plants using a CRISPR-Cas system.NatBiotechnol, 2013, 31(8): 686-688.
[14]Zhang H, Zhang J, Wei P, et al. The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation.PlantBiotechnolJ, 2014, 12(6): 797-807.
[15]Li J F, Norville J E, Aach J, et al. Multiplex and homologous recombination-mediated genome editing inArabidopsisandNicotianabenthamianausing guide RNA and Cas9.NatBiotechnol, 2013, 31(8): 688-691.
[16]Nekrasov V, Staskawicz B, Weigel D, et al. Targeted mutagenesis in the model plantNicotianabenthamianausing Cas9 RNA-guided endonuclease.NatBiotechnol, 2013, 31(8): 691-693.
[17]Liang Z, Zhang K, Chen K, et al. Targeted mutagenesis inZeamaysusing TALENs and the CRISPR/Cas system.JGenetGenom, 2014, 41(2): 63-68.
[18]Wang Y, Cheng X, Shan Q, et al. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew.NatBiotechnol, 2014, 32(9): 947-951.
[19]Jacobs T B, LaFayette P R, Schmitz R J, et al. Targeted genome modifications in soybean with CRISPR/Cas9.BMCBiotechnol, 2015, 15(1): 16.
[20]Li Z, Liu Z B, Xing A, et al. Cas9-guide RNA directed genome editing in soybean.PlantPhysiol, 2015, 169(2): 960-970.
[21]Sun X, Hu Z, Chen R, et al. Targeted mutagenesis in soybean using the CRISPR-Cas9 system.SciRep-UK, 2015, 5: 10342.
[22]Brooks C, Nekrasov V, Lippman Z B, et al. Efficient gene editing in tomato in the first generation using the clustered regularly interspaced short palindromic repeats/CRISPR-associated9 system.PlantPhysiol, 2014, 166(3): 1292-1297.
[23]Miao J, Guo D, Zhang J, et al. Targeted mutagenesis in rice using CRISPR-Cas system.CellRes, 2013, 23(10): 1233-1236.
[24]Shan Q, Wang Y, Li J, et al. Genome editing in rice and wheat using the CRISPR/Cas system.NatProtec, 2014, 9(10): 2395-2410.
[25]Xu R F, Li H, Qin R Y, et al. Generation of inheritable and “transgene clean” targeted genome-modified rice in later generations using the CRISPR/Cas9 system.SciRep-UK, 2015, 5: 11491.
[26]Ma X, Zhang Q, Zhu Q, et al. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants.MolPlant, 2015, 8(8): 1274-1284.
[27]鄂志国, 王磊. 水稻抗病性基因的克隆和功能研究进展. 遗传, 2009, 31(10): 999-1005.
E Z G, Wang L. Advance on the cloning and functional analysis of disease resistance genes in rice.Hereditas, 2009, 31(10): 999-1005. (in Chinese with English abstract)
[28]Fukuoka S, Okuno K. QTL analysis and mapping ofpi21, a recessive gene for field resistance to rice blast in Japanese upland rice.TheorApplGenet, 2001, 103(2): 185-190.
[29]Fukuoka S, Saka N, Koga H, et al. Loss of function of a proline-containing protein confers durable disease resistance in rice.Science, 2009, 325(5943): 998-1001.
[30]朱金燕, 王军, 范方军, 等. 水稻稻瘟病广谱抗病新等位基因pi21t的鉴定及其抗性应用. 华北农学报, 2014, 29(6): 11-15.
Zhu J Y, Wang J, Fan F J. et al. Identification and application of one new rice blast broad-spectrum resistance allelepi21t.ActaAgricBor-Sin, 2014, 29(6): 11-15. (in Chinese with English abstract)
[31]王才林, 张亚东, 朱镇, 等. 优良食味粳稻新品种南粳9108的选育与利用. 江苏农业科学, 2013, 41(9): 86-88.
Wang C L, Zhang Y D, Zhu Z, et al. Breeding and application of Nanjing 9108, a new japonica variety with excellent eating quality.JiangsuAgricSci, 2013, 41(9): 86-88.
[32]Xie K, Yang Y. RNA-guided genome editing in plants using a CRISPR-Cas system.MolPlant, 2013, 6(6): 1975-1983.
[33]朱立宏. 关于我国水稻高产育种的我见. 南京农业大学学报, 2007, 30(1): 129-135.
Zhu L H. Some critical considerations on rice high-yielding breeding in China.JNanjingAgricUniv, 2007, 30(1): 129-135. (in Chinese with English abstract)
[34]凌忠专, 雷财林, 王久林. 稻瘟病菌生理小种研究的回顾与展望. 中国农业科学, 2004, 37(12): 1849-1859.
Ling Z Z, Lei C L, Wang J L. Review and prospect for study of physiologic races on rice blast fungus (Pyriculariagrasea).SciAgricSin, 2004, 37(12): 1849-1859. (in Chinese with English abstract)
Knock-out Efficiency Analysis of Pi21 Gene Using CRISPR/Cas9 in Rice
WANG Fang-quan1,2, FAN Fang-jun1,2, LI Wen-qi1,2, ZHU Jin-yan1,2, WANG Jun1,2, ZHONG Wei-gong1,2,YANG Jie1,2,*
(1Institute of Food Crops, Jiangsu Academy of Agricultural Sciences/Nanjing Branch of Chinese National Center for Rice Improvement/Jiangsu High Quality Rice R & D Center, Nanjing 210014, China;2Jiangsu Co-Innovation Center for Modern Production Technology of Grain Crops, Yangzhou University, Yangzhou 225009, China;*Corresponding author, E-mail: yangjie168@aliyun.com)
A CRISPR/Cas9 vector, which contains two targets (Target 1 and Target 2) ofPi21gene, was constructed and transformed to rice variety Nanjing 9108. Twenty-eight T0transgenic lines were obtained and confirmed by T-DNA specific PCR. Restriction digestion analysis revealed 78.57% mutant ratio in Target 1 site, 92.86% mutant ratio in Target 2 site, 78.57% ratio of lines containing both Target 1 and Target 2 mutant in transgenic lines. Through sequencing analysis, we found the mutant types of targets, such as deletion of bases, insertion of bases, insertion behinds deletion of bases, and long fragment deletion of DNA, etc. Moreover, except amino acid deletion, most transgenic lines displayed frameshift mutation,which caused function loss ofPi21. In this way, a series ofPi21-knock-out rice lines were obtained, and further, could be used for functional analysis ofPi21 and rice blast broad-spectrum resistant breeding.
CRISPR/Cas9; rice blast;Pi21; gene knock-out
2016-01-18; 修改稿收到日期: 2016-03-03。
江苏省现代农业重点研发项目(BE2015355); 国家重大农技推广项目(NG[15]003); 国家自然科学基金资助项目(31301652); 江苏省自然科学基金资助项目(BK20130723)。
Q755; S511.01
A
1001-7216(2016)05-0469-10
王芳权, 范方军, 李文奇, 等. 利用CRISPR/Cas9技术敲除水稻Pi21基因的效率分析. 中国水稻科学, 2016, 30(5): 469-478.
