文本挖掘在药物靶位研究中的应用
2017-03-21,
,
现代新药开发与研究的关键,首先是寻找和筛选药物靶位(drug target)。药物靶位是指机体内具有药效功能并且能被药物作用的生物大分子物质,如某些蛋白质和核酸等。文本挖掘是目前发现潜在药物靶位的新兴手段之一,目前大多数文章都是通过定性和举例来阐述文本挖掘技术在药物靶位领域的研究成果。本文通过构建词篇矩阵等数学模型,以聚类方式更加直观和科学地定量阐述了自1999-2015年该领域的发展情况,希望对相关领域的研究人员选择参考文献和研究方向有所帮助。
1 资料来源与方法
首先对该领域高被引论文进行同被引聚类分析。按照图1所示流程,以((TS=drug target*) OR (TS=drug delivery system)) AND (TS=text mining))为检索式在Web of Science中SCI核心文献集进行检索,共下载88篇相关文献,里面包含了4 415篇参考文献。

图1 利用文本挖掘技术进行药物靶位研究进展的定量分析流程
利用书目共现分析系统(Bibliographic Item Co-Occurrence Matrix Builder2.01, BICOMB2.01)对参考文献被引频次进行统计后,将被引频次高于5次的文献作为高被引论文,使用gCLUTO聚类软件对高被引论文进行同被引聚类分析,得到5个类别(图2)
综合评估文献数量和同类文献的紧密程度,对高被引论文的标题和摘要进行了人工分析和归纳(表1),总结出自1999-2015年该领域的研究状况,并就每类文献语义内容进行了分析和梳理。
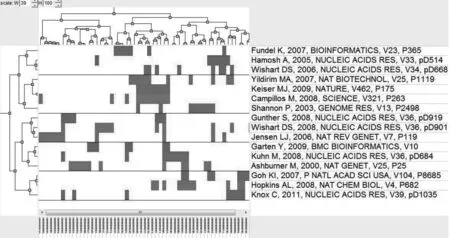
图2 高被引论文聚类分析结果

表1 高被引论文聚类结果

(续表1)
2 结果与分析
结合软件输出内容,我们将各类别按照学科领域的知识体系重新排列,将文本挖掘在药物靶位研究中的应用分成总体趋势、理论基础、主要方法和主要资源4个方面,具体内容如下。
2.1 当前药物靶位研究的总体趋势
综合分析cluster 0后,发现当前药物靶位研究有两个明显的趋势:一是通过新药检测分析抗肿瘤药的相关靶位,即将各种化学物质与标准的肿瘤治疗药物放在一起,通过它们之间的联系进行靶位挖掘;二是多学科结合,通过计算机和专家配合进行药物研究[2]。Yoo Kyung Jeong等人使用第一种途径,结合PubMed和ClinicalTrials数据库中的抗肿瘤药物相关文献进行综合分析,发现不同的化学物质和标准治疗药物放在一起时,可以发现新的药物靶位[1]。该方法很大程度上节约了药物研究的成本和降低了开发的风险。
2.2 文本挖掘预测药物靶位的理论基础
基于cluster 1中Predicting new molecular targets for known drugs(预测已知药物的新分子靶位)[3]和Drug target identification using side-effect similarity(基于药物副作用相似性的靶位识别)[4]两篇高被引文献的关注方向,总结出利用文本挖掘预测药物靶位有两种常用的方法:一是通过化学结构相似性进行预测,二是通过药物副作用相似性进行靶位识别。如Fechete, R等人研究糖尿病肾病的分子途径、生物标记物和识别药物靶位时,就基于第一种理论基础进行了深入研究。他们构建了人类相关蛋白、基因的结构相似性网络,分析后得出了约1 000个基因与糖尿病肾病病理和临床特征相关[17]。
2.3 利用文本挖掘技术预测药物靶位的主要方法
针对cluster 2分析后,我们发现网络分析是预测药物靶位的主要方法,通过网络的构建可以更加清晰、有效地揭示药物靶位和致病基因间可能存在的联系,从而预测新的药物靶位,为进一步靶向治疗提供理论支持。其中,网络药理学(Network pharmacology)是常用的药物发掘范式,多用于寻找新的药物靶位和发掘相应药物结构、活性间的关系。也可用于保持药物属性前提下新主导范式的构建,进而实现药物的概念性设计[5]。在人类疾病研究过程中,人类疾病网络常用于挖掘疾病和基因间的潜在联系[7]。同时,网络分析依赖于一些平台提供的数据,如化合物-蛋白质交互平台STITCH[8]中提供的超过30,000个小分子化合物和来自近1 133个物种的260万个蛋白质之间的相互作用的数据,就可用于交互式检索或大规模数据分析。Vazquez等人开发了可用于研究药物毒性和蛋白-疾病-化合物网络分析的程序[18],使网络分析更加智能化和高效化。
2.4 文本挖掘在药物靶位研究领域主要利用的资源
2.4.1 常用数据库
在cluster 3 中,我们总结出两个研究常用的数据库Drugbank和OMIM。其中Drugbank是药物学领域十分重要的数据库,提供了丰富的生物学信息和化学信息、药物数据(如药理和制药信息)与药物靶位(即序列、结构和通路)的详细信息,包含有8 206条药物条目(涵盖了美国食品和药物管理局批准的小分子药物、生物技术药品(如蛋白质/肽)、保健品和超过6 000个实验药物)[19]。
人类孟德尔遗传数据库OMIM(网络版)是一个关于人类基因和遗传变异的数据库,主要着眼于人类遗传疾病[12]。包括了文本信息和相关参考信息、序列纪录、图谱,提供了相关数据库的跨库检索功能,内容上涵盖了已知的遗传病、相应遗传基因及其决定的性状。除了简略描述各种疾病的临床特征、鉴别诊断、治疗与预防外,还提供了已知的相关致病基因的连锁关系、染色体定位、组成结构、功能和一些实验动物模型的数据。其中以OMIM数据库、基因图谱、OMIM 疾病基因图谱利用最为广泛。蛋白质-蛋白质相互作用的信息,可用来研究癌症的体细胞突变、遗传重叠现象和预测癌症的高风险位点。
2.4.2 重要软件工具
经过对cluster 4中文献标题、摘要的总结,得出常用的软件工具主要有Cytoscape[13],Pharmspresso[14],RelEx[16],SuperTarget and Matador[15]。Cytoscape用于构建生物分子相互作用的网络模型,它能将生物分子的相互作用图形化显示并进行分析和编辑,多用于研究基因表达、DNA损伤、基因关联性等[13];Pharmspresso是一个提取药物基因组学概念和关系的文本挖掘工具,可协助识别重要的药物基因组学概念等相关信息,侧重于基因组学概念的命名体识别和抽取[14];一站式数据仓库SuperTarget集成了药品相关的医疗信息,包括药物副作用、药物代谢通路和药物靶位、临床指征等,方便了研究者进行查询;Matador是一个蛋白-化学物资源交互平台。相比之下,DrugBank通常只包含蛋白质或药物相互作用的主要模式,而Matador则包含尽可能多的蛋白、药物间直接和间接的交互信息。例如,在探究一个药物代谢物和基因表达之间的关系时,利用Matador中相应的交互信息可以将尽可能多的相关研究对象组合在一起进行讨论[15]。
3 讨论
随着算法和技术的不断发展,文本挖掘技术在药物靶位领域的应用也更加广泛和深入。结合高被引论文的最新引用文献,我们对上述研究领域的未来可能发展方向进行如下预测。
药物研究在未来可能趋向于利用整合后的临床试验数据和现有软件处理后的二次信息,即通过多个软件的相互结合来抽取更加贴切的信息集。例如,实验人员利用临床试验数据结合文本挖掘,对激酶抑制剂的不良事件进行预测,通过挖掘抑制效力不同的多个激酶和人类疾病间潜在联系,从而为临床诊疗做出一定指导[20]。
目前药物靶位挖掘主要使用网络分析的方法。未来研究主要有3个可能发展方向:一是通过分子分类来探索潜在药物靶位[21];二是根据蛋白可以直接或间接影响药物间相互作用的理论基础,探索脱靶药物和其下游药物的反应[22]以及它们之间的潜在联系,这可能成为个性化医疗发展的又一新方向;三是将文本挖掘的信息与化学结构信息进行整合,实现全面的可视化表达。如通过由高到低的分子建模来搭建分子相互作用框架,以此来揭示信号分子、代谢通路的层次结构,从而获得新的范式[23],或是构建药物靶位图谱,使人类疾病的潜在靶位得以更加直观和准确的呈现[24]。
我们推测今后几年数据库的数据优化也将成为研究者关注的一大方向,人们将更加致力于提高数据的质量、降低噪音。DrugBank数据库中提供了药物/化学数据、药物靶位和蛋白质数据的字段检索,近年又添加了许多新的数据字段,如食物-药物相互作用、药物之间的相互作用等[9],未来也将不断更新数据库中的字段以满足研究需要。
药物靶位挖掘的研究工具的开发也将随着学科发展逐步细化。当前研究分析主要依赖于一些平台提供的数据,如STITCH就是一个典型的用于检索化合物-蛋白质交互关系的平台[8];接下来更多具有针对性的工具和平台将应用于药物靶位领域,如专门针对肠道药物[25]或者针对于心脏疾病药物的靶位预测工具。此外,将有更多科学家致力于开发用于临床药物-基因相互作用的软件[26],以满足临床工作者进行精准治疗的需求。
4 结论
当前该领域的研究主要以药物靶位间化学结构相似性和副作用相似性作为理论基础,通过复杂网络分析的方法,结合大量药物数据资源来进行靶位预测。研究人员主要利用Cytoscape[13]、Pharmspresso[14]、RelEx[16]、SuperTarget and Matador[15]几个软件,以DrugBank、OMIM为主要数据来源进行潜在药物靶位的发掘。
通过分析推测该领域未来的发展方向将文本挖掘的结果与临床实践相互结合,综合利用各类挖掘软件工具,通过范式、网络的可视化分析,对相应药理实验结果做出合理解释,预测潜在的药物靶位,为药物研制提供指导性意见,切实为药物生物学和临床医学提供帮助,以真正推动精准医疗的进一步发展。
