干旱胁迫下杂草稻和栽培稻根系基因表达差异研究
2016-10-25丁国华孙健杨光张凤鸣白良明孙世臣姜树坤王彤彤郑洪亮夏天舒沈希宏马殿荣陈温福
丁国华 孙健 杨光 张凤鸣 白良明 孙世臣 姜树坤 王彤彤 郑洪亮夏天舒 沈希宏 马殿荣 陈温福
(1 黑龙江省农业科学院博士后科研工作站/黑龙江省农业科学院 耕作栽培研究所/中国科学院 北方粳稻分子育种联合研究中心, 哈尔滨 155086;2 沈阳农业大学 水稻研究所, 沈阳110866; 3 辽宁省经济作物研究所, 辽宁 辽阳 111000; 4中国水稻研究所, 杭州 310006;#共同第一作者;*通讯联系人, E-mail: madianrong@163.com; wfchen5512@126.com)
干旱胁迫下杂草稻和栽培稻根系基因表达差异研究
丁国华1,2,#孙健2,#杨光3张凤鸣1白良明1孙世臣1姜树坤1王彤彤1郑洪亮1夏天舒1沈希宏4马殿荣2,*陈温福2,*
(1黑龙江省农业科学院博士后科研工作站/黑龙江省农业科学院 耕作栽培研究所/中国科学院 北方粳稻分子育种联合研究中心, 哈尔滨 155086;2沈阳农业大学 水稻研究所, 沈阳110866;3辽宁省经济作物研究所, 辽宁 辽阳 111000;4中国水稻研究所, 杭州 310006;#共同第一作者;*通讯联系人, E-mail: madianrong@163.com; wfchen5512@126.com)
DING Guohua, SUN Jian, YANG Guang, et al. Global genome expression analysis of root genes under drought stress in weedy rice and up-land rice. Chin J Rice Sci, 2016, 30(5): 458-468.
利用Affymetrix水稻表达谱芯片(GeneChip Rice Genome Array)研究抗旱杂草稻HEB07-2与巴西陆稻(IAPAR9)在PEG模拟干旱胁迫下根系基因表达变化。结果表明,杂草稻HEB07-2转录组对干旱信号的响应程度与方向均与巴西陆稻存在很大差异,HEB07-2以正向调控为主,而巴西陆稻以负调控为主。干旱胁迫下杂草稻HEB07-2与巴西陆稻分别有6878个和2923个基因表达,其中HEB07-2受干旱胁迫诱导上调的基因有4693个,下调的基因有2185个;而巴西陆稻则分别有983个和1940个。在差异基因表达的倍数上也是HEB07-2高于巴西陆稻。进一步的GO分析表明,在干旱胁迫下,HEB07-2和IAPAR9根系基因响应途径的差异表现为HEB07-2在钾离子转运(GO:0006813)、次生物质代谢(GO:0019748)、细胞生长(GO:0016049)、葡萄糖代谢(GO:0006006)、跨膜离子转运器活性(GO:0015075)、亚铁血红素结合体(GO:0020037)、氧化还原酶活性(GO:0016491)等方面基因显著上调,这与生理数据和表型数据一致。
杂草稻; 根系; 表达谱芯片; 耐旱
水稻(Oryzasativa)是重要的粮食作物,全世界一半以上的人口都将稻米作为主食[1]。我国是水稻生产大国,稻谷总产量世界第一,水稻生产耗水量大,占农业用水的60%左右,水资源已经成为限制水稻发展的一个潜在因素[2]。因此,培育抗旱节水的水稻新品种是本领域亟待解决的问题之一,而植物的耐旱性是典型的数量性状,由众多基因控制,十分复杂。关于作物耐旱的QTL研究较多[3-6],但到目前为止没有克隆一个与作物耐旱性相关的QTL,而基于耐旱性QTL进行的分子辅助育种效果并不能令人满意。
基因芯片使研究人员能够快速、高通量地了解植物在特定环 境条件下的基因表达水平及变化[7-11]。Thomas等[9]利用芯片技术进行研究,发现耐旱性不同的水稻品种干旱胁迫下基因表达模式不同,长期干旱胁迫下干旱敏感品种表达发生变化的基因数量较多。李永春等[10]用Affymetrix基因芯片研究小麦在模拟干旱胁迫(20% PEG-6000溶液)下根系基因表达谱,结果表明干旱胁迫后下调表达的基因4573个(5043个探针组),上调表达的基因3743个(4074个探针组)。Zhang 等[11]利用表达谱芯片研究了干旱胁迫下巴豆苗期根系和叶片中基因表达状况,干旱胁迫下有关生物合成、转运、细胞蛋白修饰、脱落酸合成、脱落酸信号转导、棉籽糖合成、海藻糖合成相关基因显著上调。
杂草稻是亚洲栽培稻的一级基因源,具有较强的抗逆性,是改良栽培稻的优良基因库[12-13]。利用基因芯片技术检测杂草稻与栽培稻全基因组基因在干旱胁迫下的表达变化情况,并进一步分析数据,对比杂草稻与栽培稻全基因组在特定条件下对外界环境的响应状况,从转录和表达的层面揭示杂草稻和栽培稻在干旱胁迫下的基因组差异,进一步发掘杂草稻可能存在的特殊抗旱机理,对于拓宽水稻耐旱遗传基础,培育节水抗旱水稻新品种具有重要意义。以往关于抗旱表达谱的研究往往基于抗旱样本与干旱敏感样本的相互比较。本研究将极抗旱种质资源杂草稻HEB07-2与抗旱的陆稻样本进行表达谱差异分析,试图从一个新的高度进一步理解稻属植物抗旱的机制。
1 材料与方法
1.1植物材料与试验设计
供试材料为北方杂草稻HEB07-2和巴西陆稻。选取饱满的种子,经10%次氯酸钠消毒后,于30℃下恒温催芽3 d,之后选取长势一致的种子播于96孔PVP板中,并置于培养容器(长24 cm×宽24 cm×高10 cm)中。在RXZ-500C人工气候箱中进行幼苗培养。培养前2周放入水中培养,于第3周时置于木村B半营养液中,于第4周开始时放入木村B全营养液中,将生长4周的幼苗置于20%PEG-6000中模拟干旱胁迫,处理24 h,以不进行干旱胁迫营养液培养的幼苗为对照(CK)。人工气候箱白天温度设定为29℃,夜间设定为24℃,14 h光照/12 h黑暗,光强为22 000 lx。胁迫结束后马上取样,取幼苗所有根,用铝箔纸包好,放入液氮中迅速冷冻,然后于-80℃超低温冰箱中保存,用于提取总RNA和测定相关生理指标。试验设置3次重复,取样时每个品种取一盘,每20株为一次重复。
本研究采用Affymetrix水稻表达谱芯片(GeneChip Rice Genome Array)分析干旱胁迫24 h后HEB07-2与巴西陆稻表达谱差异。芯片覆盖了约51 279个转录本,代表两个水稻亚种,48 564个粳稻亚种转录本和1260个籼稻亚种转录本。序列信息来源于GenBank mRNAs,TIGR基因预测和国际水稻基因组序列计划。分析干旱胁迫24 h后杂草稻HEB07-2与巴西陆稻根系转录丰度的差异,通过计算杂交信号的比值和统计分析,获得差异表达基因的信息,研究在功能或表达调控上具有相关性的基因。
1.2项目测定
1.2.1 叶片总RNA的提取
按Trizol试剂盒(康为公司,中国)说明书提取总RNA。用紫外分光光度计测定所抽RNA样品的A260、A280值,根据测定的浓度,每个RNA样品稀释成相同浓度,取1 μg RNA用0.8%变性琼脂糖凝胶电泳检测。
1.2.2生理指标测定
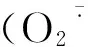
1.2.3实时荧光定量PCR分析
将HEB07-2和IAPAR9各样品RNA反转录为cDNA,以反转录产物为模板,以水稻Actin为内参基因,采用QIAGEN公司的SYBR Green RT-PCR 试剂盒及ABI7500荧光定量PCR仪进行检测。随机选择在HEB07-2和IAPAR9基因芯片检测表现为差异表达的4个基因(LOC_Os03g37840,LOC_Os04g52390,LOC_Os08g32840,LOC_Os02g02400),用Primer Premier 5.0软件设计引物,进行实时荧光定量PCR分析,以验证基因芯片的分析结果。
表1实时荧光定量PCR分析所用的引物
Table 1. Primers used for real-time quantitative PCR.

基因Gene正向引物Forwardprimer(5'-3')反向引物Reverseprimer(5'-3')LOC_Os03g37840CGAAATAAGAAGGACGGATGGTGATGGGAGCAAGACAAAGGTALOC_Os04g52390TCAAGAACCAAAGTCAGATAGGAAGAAAGCACAATAAAGGTGACAACTAGLOC_Os08g32840CTCAACTACACCTACCGAAACGCTCCAACTTGTGCAGGAACTCATLOC_Os02g02400GAGGCAGAAGGCGACGATAGAGGTAGTTGACCCAGATGGCActinGGAACTGGTATGGTCAAGGCAGTCTCATGGATACCCGCAG
1.3数据处理
1.3.1芯片数据分析
本研究应用MAS 5.0软件(Affymetrix MicroArray Suite 5.0 software)分析扫描芯片上的每一基因的信号强度,之后利用GCOS(Affymetrix GeneChip Operating Software 4.0)建立每个样品生物学重复内扩增样品的信号数据库,进行信号的背景校正与数据的标准化分析。进而比较杂草稻与巴西陆稻干旱胁迫下两种背景的基因表达谱的变化。进一步采用Gene Spring 7.1进行标准化芯片数据的统计分析。根据标准化后的均值来计算每个探针的表达变化。用t检验(one-way)筛选差异显著基因,筛选标准为差异两倍的探针位点,方差分析的显著性差异为0.005。
1.3.2生理数据分析
试验数据用SPSS 19.0进行统计分析。试验数据均为平均值±标准差(n=3)。每个供试材料不同处理间进行方差分析(ANOVA),采用Duncan法(P<0.05,0.01)以0 h数据为对照。利用GraphPad Prism 6作图。
1.3.3实时荧光定量PCR数据分析
各样品每个基因重复3次,按相对定量法进行定量计算,目的基因相对定量计算公式为Rel.Exp=2-△△Ct,其中△△Ct=△Ct未知样品-△CtCalibrator,△Ct未知样品=Ct内参基因-Ct目的基因,△CtCalibrator=Ct参比样内参基因-Ct参比样目的基因。
2 结果与分析
2.1HEB07-2和巴西陆稻差异基因倍数变化及表达模式
杂草稻HEB07-2与巴西陆稻分别有6878个和2923个基因表达,根据RICE芯片的诠释可知,在干旱胁迫下HEB07-2表达的基因中5410个基因有明确功能注释,1468个基因无确定功能,占21.3%;巴西陆稻干旱胁迫下表达的基因中,2076个基因有确定功能,847个基因无明确功能注释,占表达基因总数的29.0%;2个供试材料差异表达基因有6579个,其中4799个基因功能已知,1780个表达基因无明确注释,占总数的27.1%。
HEB07-2和巴西陆稻表达重叠的基因有1329个。其中HEB07-2受干旱胁迫诱导上调的基因有4693个,下调的基因有2185个。而巴西陆稻受干旱胁迫诱导上调基因远小于HEB07-2,有983个,受干旱胁迫下调的基因与HEB07-2相当,有1940个。上调的最大倍数在44.2到58.4倍之间,平均变化2.9~3.4倍;下调最大倍数在20.6~167.5倍,平均下调2.9~5.3倍。干旱胁迫下HEB07-2和巴西陆稻相比有差异表达的基因数量为6579个,无差异的基因有48 246个,其中有2546个基因下调,4033个基因上调。
2.2干旱胁迫下HEB07-2根系差异表达基因GO分析
对差异表达基因进行GO (GENE ONTOLOGY)分析,按照GO分类法则,对差异基因进行功能分类。干旱胁迫处理下,HEB07-2与对照比发生变化的基因,经过GO分析后按分子功能、生物过程和细胞组成进行诠释(详见在线辅助性表S1,http://www.ricesci.cn/),涉及分子功能基因有2841个,这些基因涉及阳离子结合、离子结合、金属离子结合、DNA结合、铁离子结合、钙离子结合、四吡咯结合亚铁血红素结合、碳水化合物结合、维生素结合、糖结合、磷酸吡哆醛结合、维生素B6结合、糖脂类结合,分别占14.5%、14.5%、13.3%、7.2%、3.6%、2.8%、2.4%、2.4%、1.0%、0.9%、0.8%、0.8%、0.8%、0.12%;此外,还有蛋白激酶活性、蛋白组氨酸/丝氨酸激酶活性、转录调节因子活性、电子载体活性、转录因子活性、氧化还原酶活性、几丁质酶活性、脂氧合酶活性、半胱氨酸活性、谷氨酸脱羧酶活性、糖脂转运器活性,各占5.5%、4.8%、4.2%、3.6%、3.1%、1.0%、0.3%、0.2%、0.2%、0.1%、0.1%。
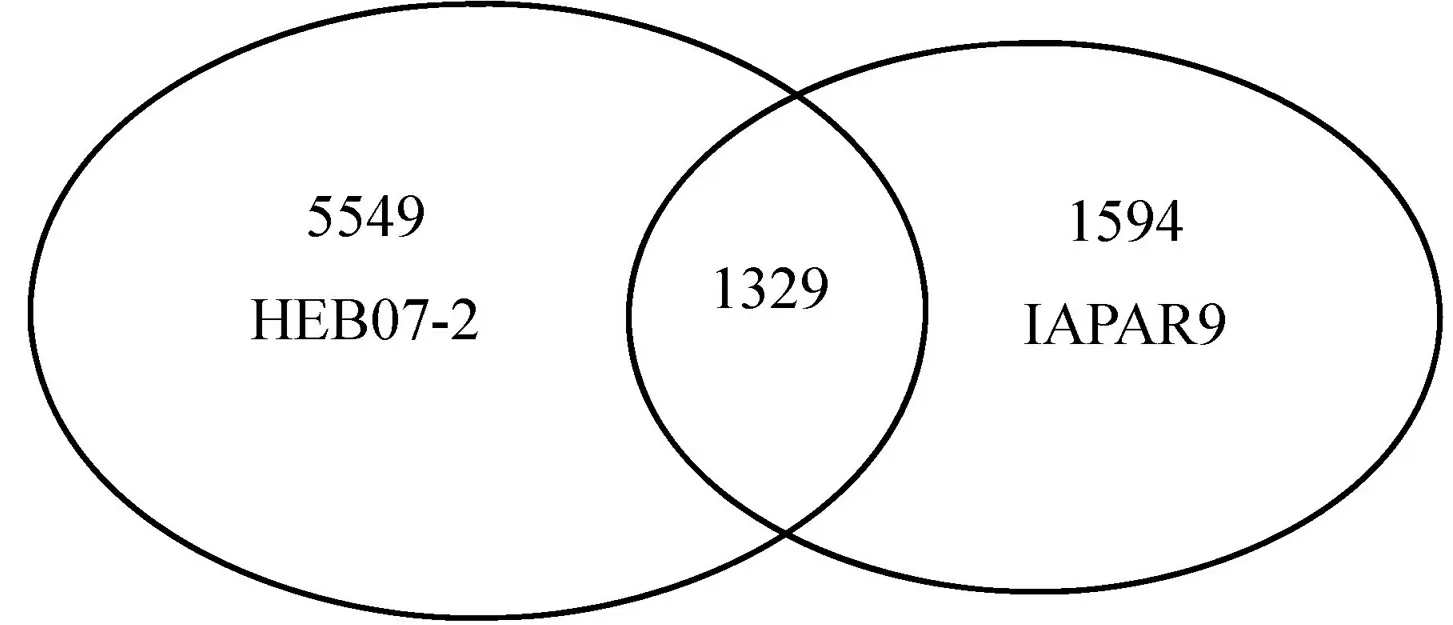
图1HEB07-2和IAPAR9响应干旱胁迫基因比较的韦恩图
Fig. 1. Ven figure of HEB07-2 and IAPAR9 regulated genes under drought.
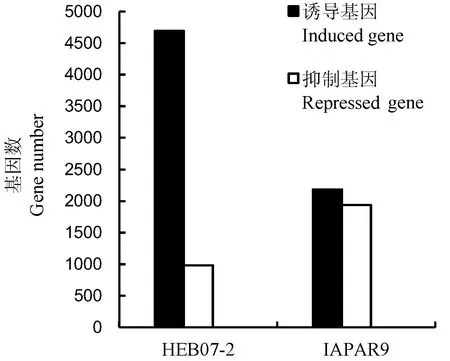
图2HEB07-2和IAPAR9干旱胁迫诱导和抑制的基因
Fig. 2. Gene numbers of HEB07-2 and IAPAR9 induced and repressed by drought.
按生物过程分有1286个基因表达发生了变化,这些基因的功能包括代谢过程(RNA代谢调节过程、脂质合成过程、氨基酸衍生物代谢等)、转录(转录调控等)、蛋白氨基酸磷酸化、氧化还原、转运(金属离子转运、无机离子转运等)、光合捕光、细胞运动及定位、响应(水响应等),分别占总数的12.2%、14.1%、5.5%、5.0%、1.7%、0.2%、0.7%、0.5%。
按细胞组成分有1283个基因表达发生了变化,主要是细胞器(膜结合细胞器、非膜结合细胞器)、细胞、蛋白复合体(泛素连接酶复合体)、细胞外区域(胞外区域和质外体),分别占24.1%、13.1%、0.5%、2.3%。
2.3干旱胁迫下巴西陆稻根系差异基因GO分析
干旱胁迫下,巴西陆稻根系与对照比基因组发生表达变化,经GO分析后,涉及分子功能的基因共有1059个,包括离子结合(阳离子结合、金属离子结合等)、DNA结合、转录因子活性、激酶活性、转运载体活性、氧化还原酶活性,所占比例分别为46.1%、12.2%、10.4%、6.0%、3.0%、1.2%。干旱胁迫期间和生物过程相关的基因共有505个基因表达发生变化,这些基因涉及转录调节、代谢过程、离子转运、响应反应,所占比例分别为18.9%、8.9%、8.7%、1.2%。按细胞组成分,发生表达的基因有512个,基因功能涉及细胞组成(细胞膜、细胞壁)、细胞器(膜结合细胞器,非膜结合细胞器)、细胞外区域,所占比例分别为13.6%、23.2%、1.3%(详见在线辅助性表S2,http://www.ricesci.cn/)。
表 2HEB07-2和IAPAR9差异表达基因数及倍数变化
Table 2. Up- and down-regulated fold of genes in HEB07-2 and IAPAR9.
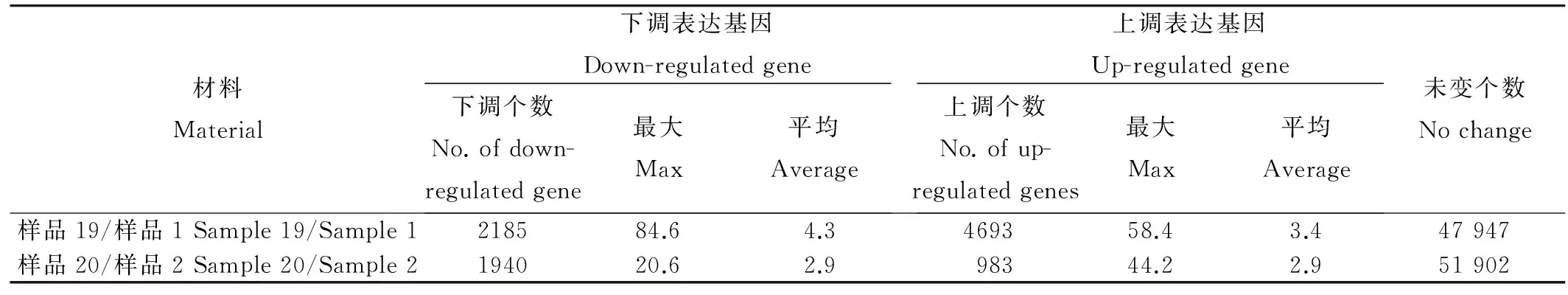
材料Material下调表达基因Down-regulatedgene下调个数No.ofdown-regulatedgene最大Max平均Average上调表达基因Up-regulatedgene上调个数No.ofup-regulatedgenes最大Max平均Average未变个数Nochange样品19/样品1Sample19/Sample1218584.64.3469358.43.447947样品20/样品2Sample20/Sample2194020.62.998344.22.951902
样品1为HEB07-2正常条件下样品,样品19为HEB07-2干旱处理后样品;样品2为巴西陆稻正常条件下样品,样品20为巴西陆稻干旱处理后样品。
Sample 1 is from HEB07-2 under normal condition, sample 19 is collected from HEB07-2 under drought, Sample 2 is from IAPAR9 under normal condition, sample 20 is collected from IAPAR9 under drought treatment.
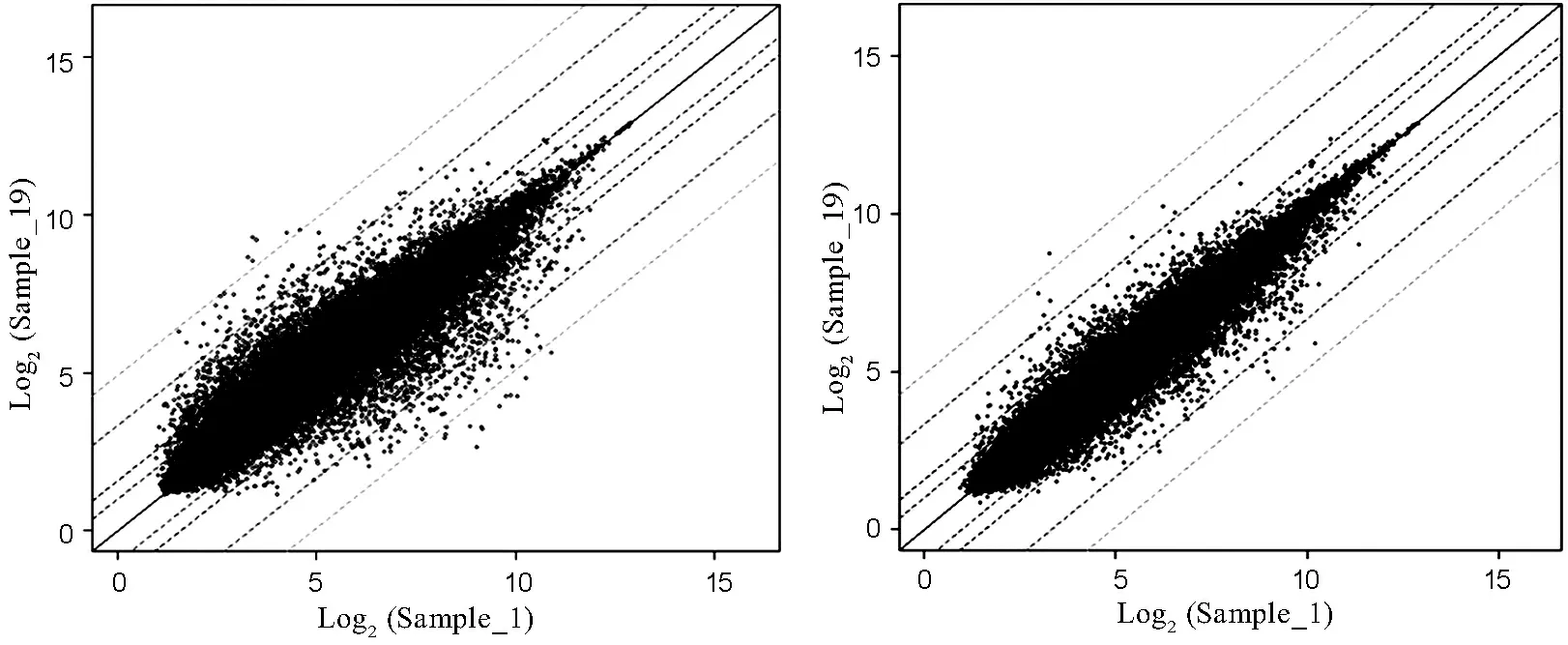
图3干旱胁迫下HEB07-2和IAPAR9差异表达倍数分布
Fig. 3. Change folds of HEB07-2 and IAPAR9 under drought condition.
2.4干旱胁迫下HEB07-2和巴西陆稻根系差异表达基因GO分析
干旱胁迫下,对HEB07-2与巴西陆稻根系差异表达基因进行GO分析后可知(详见在线辅助性表S3,http://www.ricesci.cn/),共有4799个已知功能基因表达,其中涉及分子功能的基因共有2248个,包括离子结合 、DNA连接、血红素结合、叶绿体结合、转录调控子活性、电子载体活性及酶活性,分别占46.7%、7.4%、5.9%、2.0%、12.0%、3.9%、1.1%。
生物过程方面已知功能的差异基因有974个,涉及转录、氧化还原、防御反应、光合作用、蛋白质泛素化、胞外分泌、代谢过程、植物型细胞壁组织、蛋白质发色团、细胞运动及定位,分别占18.1%、5.3%、2.2%、2.5%、0.6%、0.4%、3.7%、0.3%、0.2%、0.8%。
涉及细胞组成有功能的差异基因有1577,功能涉及细胞器(囊泡、质体、叶绿体等)、细胞组成(细胞膜、细胞壁)、胞外区域、光合系统(光系统Ⅰ、光系统Ⅱ)、泛素连接酶复合体、细胞皮层及鞭毛,所占比例分别为23.7%、24.2%、2.4%、2.8%、0.5%、1.8%。
2.5干旱胁迫下HEB07-2和巴西陆稻根系响应基因的染色体定位
为了分析干旱胁迫下HEB07-2和巴西陆稻根系响应基因在水稻染色体上的分布,我们比较了供试材料在干旱胁迫下总响应基因及诱导、抑制基因在各染色体上的分布个数(图4)。结果表明,HEB07-2总响应基因在各染色体上分布个数从多到少排序依次为第1、3、2、5、6、4、7、8、11、12、10和9染色体。其中,第1、3、2、5、4染色体上受干旱胁迫抑制基因多于受胁迫诱导基因,而这几条染色体也是总响应基因最多的几条染色体;其余染色体上受干旱胁迫诱导基因数量大于受干旱胁迫抑制基因的数量,这几条染色体上总响应基因数量小于其他染色体。而巴西陆稻在干旱胁迫下染色体上总响应基因数量在各染色体上分布个数从多到少排序依次是第1、3、2、5、6、4、7、11、8、9、12、10染色体。除第6、11染色体外,巴西陆稻其他染色体上均是受干旱抑制的基因数量多于诱导基因数量。在未知功能基因中HEB07-2受干旱胁迫诱导的基因远远大于受干旱胁迫抑制的基因数量,而巴西陆稻则是受抑制的基因数量远大于受诱导的数量(图5)。
2.6干旱胁迫下HEB07-2和巴西陆稻表型和根系生理变化
由图6-A~B可知,干旱胁迫下IAPAR9根系超氧阴子产生速率和丙二醛含量显著提高,与对照比达到极显著水平,而HEB07-2上升幅度小,说明根系受伤害程度轻。干旱胁迫下HEB07-2根系可溶性糖含量显著增高,与对照差异极显著。
由图6-D~F可知干旱胁迫下HEB07-2根系超氧化物歧化酶、过氧化氢酶活性显著增高,而IAPAR9只有过氧化物酶活性显著增大。
图6-G~I显示干旱胁迫下HEB07-2根系抗坏血酸过氧化物酶、谷胱甘肽还原酶、脱氧抗坏血酸还原酶活性均显著增高,且抗坏血酸过氧化物酶、谷胱甘肽还原酶增高幅度都大于IAPAR9。
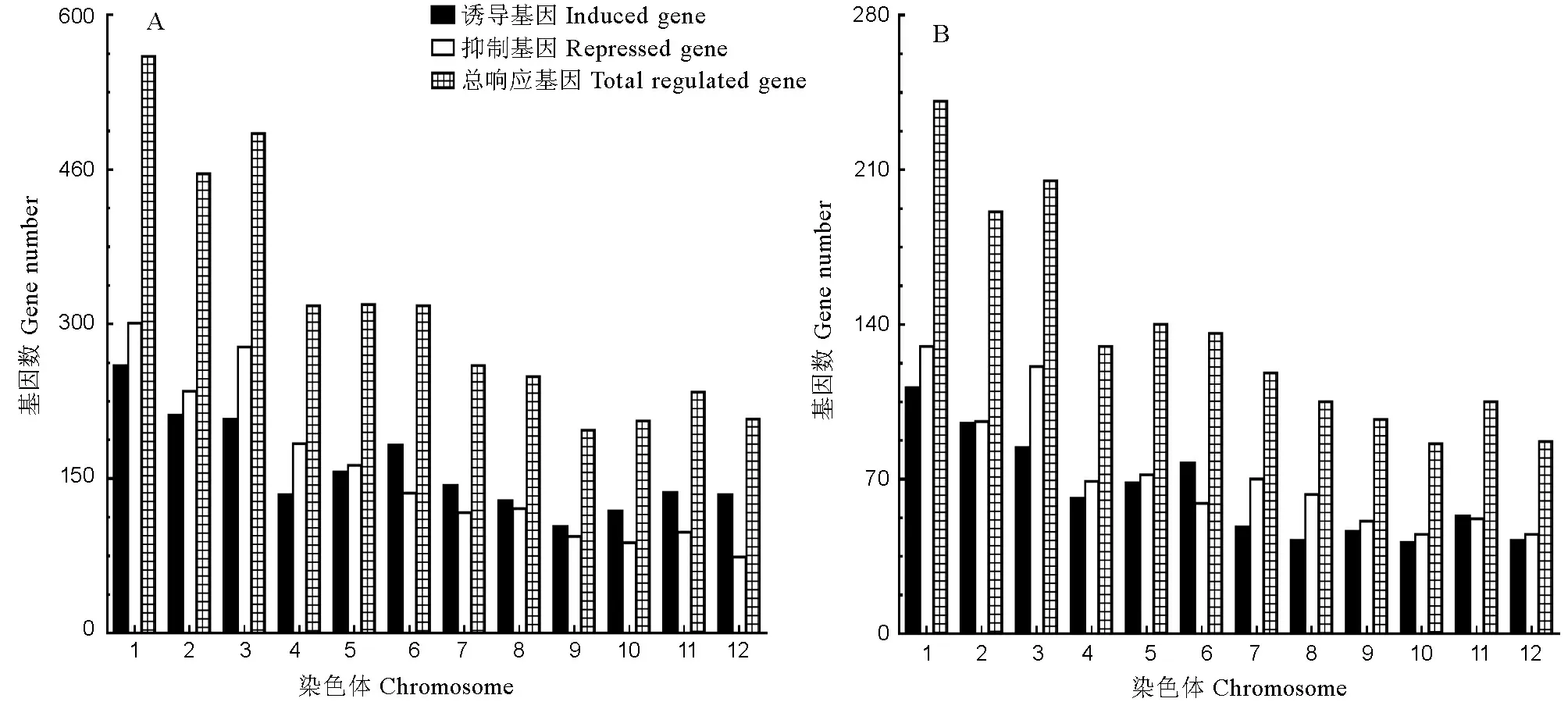
A, HEB07-2; B, IAPAR9.
图4HEB07-2和IAPAR9干旱胁迫下响应基因染色体分布
Fig. 4. Distribution of regulated genes on rice chromosome under drought stress.
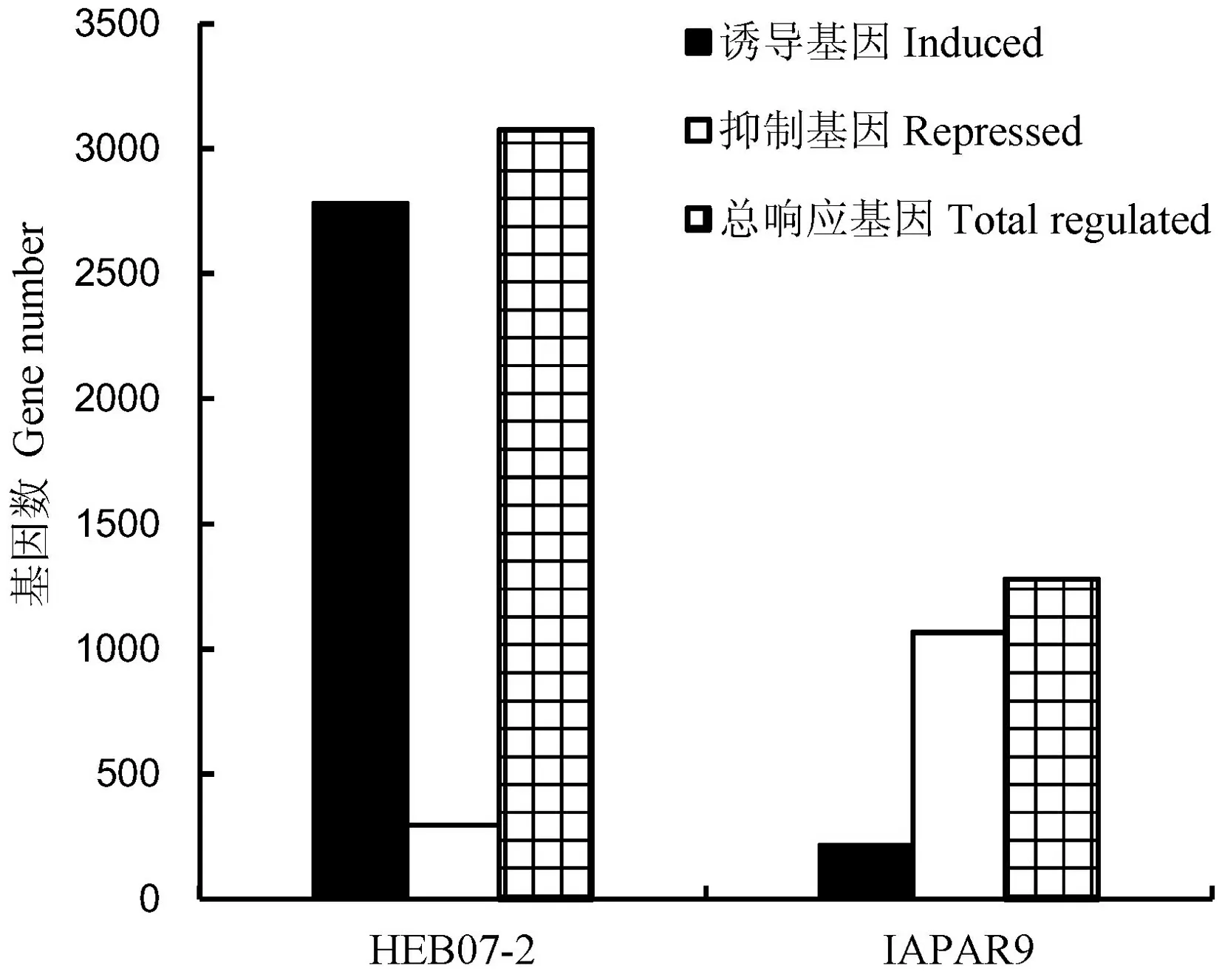
图5HEB07-2和IAPAR9响应干旱胁迫的未知基因数
Fig. 5. Unknow genes of HEB07-2 and IAPAR9 under drought stress.
2.7实时荧光定量PCR分析
为验证芯片数据准确与否,随机选取HEB07-2和IAPAR9中表达发生变化的4个基因设计引物,进行实时荧光定量PCR分析。其中,2个基因与抗氧化物酶相关,为LOC_Os08g32840、LOC_Os02g02400;2个基因与钾离子转运相关,为LOC_Os03g37840、LOC_Os04g52390。结果如图8-A~B所示,在HEB07-2的实时荧光定量分析结果中,LOC_Os03g37840和LOC_Os04g52390略低于芯片分析的结果,LOC_Os08g32840和LOC_Os02g02400结果高于芯片分析的结果;在IAPAR9中,LOC_Os08g32840、LOC_Os02g02400的实时荧光定量分析结果略高于芯片分析的结果,LOC_Os03g37840、LOC_Os04g52390结果低于芯片分析的结果。从几个基因的表达趋势来看,实时荧光定量分析结果与基因芯片分析结果基因的表达趋势基本一致。证明芯片分析结果是可靠的。
3 讨论
近年来研究人员利用表达谱技术、转基因技术等在鉴定植物耐旱基因方面做了大量工作[10,11, 14-18]。侯欣等[17]利用基因芯片筛选到了一个候选基因OsSKIP,转入该基因的水稻幼苗在缺水条件下生存能力明显增强。熊立仲等[18]在水稻中鉴定了一个控制水稻叶片蜡质积累的基因DWA1,缺失该基因的dwa1突变体对干旱十分敏感。在本研究中,杂草稻以基因表达量上调这种主动的调控形式来避害,而巴西陆稻的负调控水平高于正向的调控则表明其避害的方式与杂草稻可能存在本质的区别。从胁迫后的两种类型水稻生理指标与生长指标角度分析,杂草稻HEB07-2对于干旱胁迫有更强的耐受程度,其响应程度体现出了生物学效应与遗传学效应,是一类可以应用于抗旱遗传改良的优异种质资源。干旱胁迫下两种抗旱类型水稻重叠的转录本的比例较小,表明稻属植物应对干旱胁迫的机制十分复杂。值得一提的是,从遗传背景角度分析,杂草稻HEB07-2具有古老粳稻的基因组遗传组成,而巴西陆稻属于具有较高籼稻血缘的外来粳稻品种。因此,这种遗传背景的差异同样可以在表达谱中体现出来。
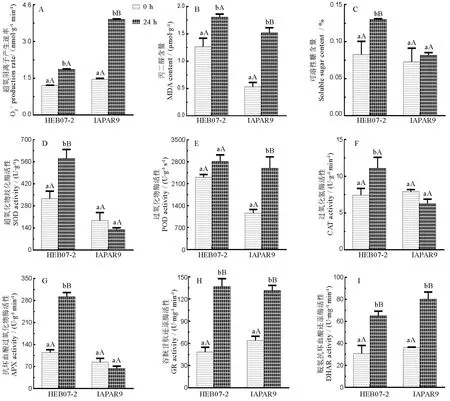
图6干旱胁迫下HEB07-2和IAPAR9根系生理指标变化
Fig. 6. The physiological index change in HEB07-2 and IAPAR9 under drought condition.
干旱胁迫下HEB07-2生物过程相关基因中表达数量最多的几类基因涉及转录调控(215个)、转录(116个)、依据DNA转录调节(122个)、RNA代谢(122个)、金属离子转运(40个)。当外界环境发生变化时,由转录因子控制的基因会对环境做出应答。Ali Moumeni等[19]研究表明在干旱胁迫中有2336个基因表达发生变化,其中1461个基因来自于转录因子基因家族,占变化基因总数的62.5%。本研究发现干旱胁迫下杂草稻差异表达基因中,转录家族基因也占很大比例,这有利于提高植物的耐旱性[20]。本研究中,离子转运基因表达数量也较多,其中钾离子转运相关基因上调表达幅度较大,LOC_Os03g37840上调表达8.3倍、LOC_Os04g52390上调表达3.8倍,钾是植物体内活细胞进行新陈代谢的重要阳离子,一切生理生化反应差不多都有钾参加,是植物需要最多的阳离子,它能够促进根系吸水,提高植物水分利用效率,并能够活化多种酶,促进蛋白质的合成,从而提高氮素的利用率,所以干旱胁迫下钾离子转运活性相关基因上调表达,有利于植物体内钾离子的积累,提高水分利用效率,促进功能蛋白合成,提高肥料利用效率,从而帮助植物渡过逆境胁迫[21-24]。除此以外,涉及细胞过程相关基因上调表达的还有几丁质代谢相关基因、次生代谢过程相关基因、细胞氨基酸衍生物合成相关基因。这些基因的表达与HEB07-2和巴西陆稻相应的抗旱生理指标可以很好地印证。因此,这些生物过程相关基因对于杂草稻抗旱具有重要的贡献。

图7干旱胁迫下HEB07-2和IAPAR9表型变化
Fig. 7. Phenotype of HEB07-2 and IAPAR9 under drought condition.
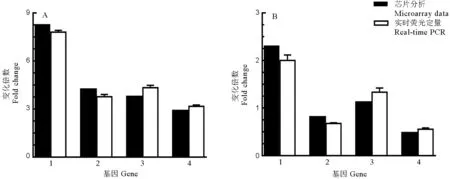
A, HEB07-2; B, IAPAR9; 1, LOC_Os03g37840; 2, LOC_Os04g52390; 3, LOC_Os08g32840; 4, LOC_Os02g02400.
图8HEB07-2和IAPAR9实时荧光定量PCR结果与芯片数据的比较
Fig. 8. Comparison of microarray data with real-time PCR results in HEB07-2 and IAPAR9.
干旱胁迫下杂草稻HEB07-2涉及最多的细胞组成差异表达基因为膜系统组成相关基因(184个)、内在膜蛋白(187个)、细胞质囊泡(193个)以及细胞质膜结合囊泡(193个)等。本研究发现,杂草稻之所以能很好地抵御干旱胁迫,保持膜系统的完整性是其中最重要的原因之一。这与基因组表达谱高度吻合,这种吻合性也同样体现在细胞壁的合成上。研究表明几丁质是植物细胞壁重要组成部分,干旱胁迫可以通过影响几丁质代谢进而影响植物细胞壁的合成,而在几丁质代谢相关基因的表达上杂草稻HEB07-2同样体现出很高的正向调控水平。因此,这些细胞组成相关基因对于杂草稻出众的抗旱能力有着很好的解释。
干旱胁迫期间,HEB07-2有关分子功能差异基因,主要有离子结合(466个)、金属离子结合(428个)、蛋白激酶活性(178个)、蛋白质丝氨酸/组氨酸激酶活性(154个)、转录调控因子活性(136个)、电子载体活性等。其中,金属离子结合、电子载体活性与氧化还原酶合成有关,这些基因的表达有利于提高拟南芥耐旱和耐高温的能力[25],以及水稻对长期干旱胁迫的适应[26]。本研究表明,质膜较高的抗氧化能力是杂草稻耐旱的重要原因,这一过程中相关的酶活性提高是关键所在,干旱胁迫促使HEB07-2超氧化物歧化酶(SOD)、过氧化物酶(POD)、过氧化氢酶(CAT)、抗坏血酸过氧化物酶(APX)、谷胱甘肽还原酶(GR)、脱氢抗坏血酸还原酶(DHAR)的活性上升,维持活性氧代谢平衡,提高抗旱能力,同样在表达谱中与氧化还原相关基因的表达水平处于非常突出的地位。
巴西陆稻在干旱胁迫下涉及生物过程的基因主要包括转录调控(126个)、依赖DNA的转录控制(74个)、RNA代谢调控过程(74个)、转录(53个)、离子转运(34个)。其下调表达基因主要有钾离子转运、谷氨酸家族氨基酸代谢过程、细胞氨基酸合成过程。干旱胁迫下钾离子通道蛋白基因表达受抑,说明干旱胁迫在一定程度上影响了巴西陆稻细胞内钾离子转运,加剧了细胞的渗透胁迫,导致干旱损伤。同时干旱胁迫导致细胞氨基酸合成减少,进而影响蛋白质的功能,这也是干旱损伤的表现。生物过程中上调表达的主要与抵抗细菌的基因相关,属于OsPR4家族,受干旱胁迫等非生物胁迫诱导,过表达OsPR4家族基因有利于植物提高耐旱性[27]。巴西陆稻细胞组成相关基因发生表达变化的主要涉及内在膜蛋白(81个)、膜完整性(79个)、细胞质膜结合囊泡(78个)等。其中上调表达涉及的基因有胞外区域调节基因,下调表达的基因主要有细胞壁。有研究表明轻度干旱胁迫下根系仍能维持生长[28],在本研究中涉及细胞壁合成的几个基因中,CSLC-1(LOC_Os01g56130)、XG_FTase(LOC_Os06g10980)上调表达,而CSLE-6(LOC_Os09g30130)、XG_FTase(LOC_Os02g52640)、CSLA-9(LOC_Os06g42020)、CSLC2(LOC_Os09g25900)均下调表达。众多研究表明CslA亚家族编码(1,4)-β-D-甘露聚糖合成酶,而CslC-1家族编码(1,4)-β-D-葡聚糖合成,其是木葡聚糖的骨架组成部分[29-31]。较低的细胞水势使玉米根系木葡聚糖活性上升,是为维持根系在胁迫条件下的生长。本研究结果表明,干旱胁迫下巴西陆稻根系与细胞壁合成相关基因既有上调表达又有下调表达,但以下调表达为主,说明干旱已经影响了其细胞壁的合成,是遭受干旱胁迫伤害的一种表现。巴西陆稻涉及分子功能的基因发生表达变化的主要有离子结合(203个)、DNA结合(123个)、转录调节因子活性(80个)、转录因子活性(59个)、特异序列DNA结合(41个)等。大多以下调表达为主,涉及碳水化合物激酶活性、钾离子跨膜转运体活性、金属离子跨膜转运体活性、氧化还原酶活性等。这些都说明与杂草稻相比巴西陆稻易受干旱胁迫的危害。
HEB07-2与巴西陆稻干旱胁迫下响应途径的差异表现为HEB07-2在钾离子转运、次生物质代谢、细胞生长、葡萄糖代谢、跨膜离子转运器活性、亚铁血红素结合体、氧化还原酶活性等方面基因显著上调。研究结果表明控制SOD(LOC_Os07g46990)、POD(LOC_Os01g18970)、CAT(LOC_Os02g02400)、APX(LOC_Os07g49400)、MDHAR(LOC_Os08g32840)、GR(LOC_Os02g56850)酶活性的基因与巴西陆稻比分别上调表达1.9倍、6.43倍、5.20倍、2.39倍、6.07倍、1.14倍,这与生理研究结果一致,即干旱胁迫下杂草稻抗氧化酶活性更高,能够及时清除ROS,保持细胞内ROS代谢平衡,减轻干旱胁迫对细胞产生的伤害,表明HEB07-2与巴西陆稻比在生理生化代谢方面更适应干旱胁迫。前人研究表明干旱胁迫下耐旱品种抗氧化酶系基因表达显著上调[19],与本研究结果一致。维持植物根系在干旱胁迫下的生长十分重要,这需要协调细胞内众多过程[32]。本研究结果表明HEB07-2和巴西陆稻比关于细胞生长(cell growth)的差异表达基因有29个,平均上调4.1倍,这说明在干旱胁迫下HEB07-2根系受到胁迫较轻,有更强的生长势,耐旱性强于巴西陆稻,这与Bartels研究[28]结果一致。此外,HEB07-2与钾离子转运相关基因表达活性也显著高于巴西陆稻,这有利于提高HEB07-2的水分利用率及渗透调节能力,进而提高耐旱能力。
本研究通过对干旱胁迫后北方杂草稻HEB07-2与巴西陆稻表达谱的比较研究,对不同稻属植物的抗旱分子机制有了进一步的认识。另外,本研究虽然对差异表达基因进行了GO分析,但仍然缺乏系统解析不同基因家族对于杂草稻在更高抗旱水平上的表达调控的特点,这有待于进一步研究。
水稻抗旱的分子机制被普遍认为是所有非生物胁迫中最复杂的,虽然前人有过许多正向遗传学研究的尝试,也取得了一定的进展,然而并没有克隆到主效的抗旱相关基因。关于水稻耐旱反向遗传学的研究也有一定的进展,但鉴定到的抗旱位点很难确定遗传效应,在不同的遗传背景或不同的生物学重复中很难重复。鉴于抗旱稻属植物分子遗传机制的复杂性,在今后的研究中将正向遗传学与反向遗传学相互结合,即对可以定位到数量性状基因座中具有大的遗传贡献的表达差异位点进行着重分析,有望大大简化抗旱分子遗传机制的复杂程度,定位、克隆到可以在育种中应用的具有遗传效应的主效基因。
在线辅助信息:有3个在线辅助性表S1、S2和S3放在中国水稻科学网站上,网址为http://www.ricesci.cn/。
[1]Miura K, Ikeda M, Matsubara A, et al. OsSPL14 promotes panicle branching and higher grain productivity in rice.NatGenet, 2010, 42(6): 545-549.
[2]Lafitte H R, Li Z K, Vijayakumar C H M. Improvement of rice drought tolerance through backcross breeding: Evaluation of donors and selection in drought nurseries.FieldCropsRes, 2006, 97: 77-86.
[3]Lilley J M, Ludow T J, McCouch S R, et al. Locating QTL for osmotic adjustment and dehydration tolerance in rice.ExpBot, 1996, 47(302): 1427-1436.
[4]Courtois G M, Shinha P K, Prasad K, et al. Mapping QTLs associated with drought avoidance in up lands rice.MolBreeding, 2000, 6: 55-66.
[5]徐吉臣, 李晶昭, 郑先武, 等. 苗期水稻根部性状的QTL定位. 遗传学报, 2001, 28(5): 433-438.
Xu J C, Li J Z, Zheng X W, et al. QTL mapping of the root traits in rice seeding.ActaGenetSin, 2001, 28(5): 433-438.(in Chinese with English abstract)
[6]Kumar R, Venuprasad R, Atlin G N. Genetic analysis of rainfed lowland rice drought tolerance under naturally-occurring stress in eastern India: Heritability and QTL effects.FieldCropsRes, 2007, 103: 42-52.
[7]赵宝存, 赵芊, 葛荣朝, 等. 利用基因芯片研究小麦耐盐突变体盐胁迫条件下基因的表达图谱. 中国农业科学, 2007, 40(10): 2355-2360.
Zhao B C, Zhao Q, Ge R C, et al. Study on the expression profile of salt-tolerance mutant under salt-stress in wheat using gene microarray.SciAgricSin, 2007, 40(10): 2355-2360. (in Chinese with English abstract)
[8]Jung C, Lyou S H, Yeu S Y, et al. Microarray-based screening of jasmonate responsive genes inArabidopsisthaliana.PlantCellRep, 2007, 26: 1053-1063.
[9]Degenkolbe T, Do P T, Zuther E, et al. Expression profiling of rice cultivars differing in their tolerance to long-term drought stress.PlantMolBiol, 2009, 69: 133-153.
[10]李永春, 孟凡荣, 王潇, 等. 干旱胁迫条件下“洛旱2号”小麦根系的基因表达谱. 作物学报, 2008, 34(12): 2126-2133.
Li Y C, Meng F R, Wang X, et al. Gene expression profiling in roots of wheat cultivar “Luohan 2” under water stress.ActaAgronSin, 2008, 34(12): 2126-2133.(in Chinese with English abstract)
[11]Zhang C, Zhang L, Zhang S, et al. Global analysis of gene expression profiles in physic nut (JatrophacurcasL.) seedlings exposed to drought stress.BMCPlantBiol, 2015, 15(1): 17.
[12]Wang G J, Miao W, Wang J Y, et al. Effects of exogenous abscisic acid on antioxidant system in weedy and cultivated rice with different chilling sensitivity under chilling stress.JAgronCropSci, 2013, 199(3): 200-208.
[13]Tang L, Ma D R, Xu Z J. Utilization of weedy rice for development of japonica hybrid rice(OryzasativaL.).PlantSci, 2011, 180: 733-740.
[14]Huang D, Wu W, Abrams S R, et al. The relationship of drought-related gene expression inArabidopsisthalianato hormonal and environmental factors.JExpBot, 2008, 59: 2991-3007.
[15]Manavalan L P, Guttikonda S K, Tran L S, et al. Physiological and molecular approaches to improve drought resistance in soybean.PlantCellPhysiol, 2009, 50: 126-127.
[16]Gong P, Zhang J, Li H, et al. Transcriptional profiles of drought-responsive genes in modulating transcription signal transduction, and biochemical pathways into tomato.JExpBot, 2010, 61: 3563-3575.
[17]Hou X, Xie K, Yao J, et al. A homolog of human ski-interacting protein in rice positively regulates cell viability and stress tolerance.PNAS, 2009, 106(15): 6410-6415.
[18]Zhu X, Xiong L. Putative megaenzyme DWA1 plays essential roles in drought resistance by regulating stress-induced wax deposition in rice.PNAS, 2013, 110(44): 17790-17795.
[19]Moumeni A, Satoh K , Kondoh H, et al. Comparative analysis of root transcriptome profiles of two pairs of drought-tolerance and susceptible rice near-isogenic lines under different drought stress.BMCPlantBiol, 2011, 11: 174-191.
[20]Wang H G, Zhang H L, Li Z C. Analysis of gene expression profile induced by water stress in upland rice (OryzasativaL.var.IRAT109) seedlings using subtractive expressed sequence tags library.JIntegrPlantBiol, 2007, 49(10): 1455-1463.
[21]Maathuis F J M, Sanders D. Energization of potassium uptake inArabidopsisthaliana.Planta, 1993, 191: 302-307.
[22]Walker D J, Leigh R A, Miller A J. Potassium homeostasis in vacuolate plant cells.PNAS, 1996, 93: 10510-10514.
[23]Britto D T, Kronzucker H J. Cellular mechanisms of potassium transport in plants.PhysiolPlant, 2008, 133: 637-650.
[24]Mahouachi J, Socorro A R, Talon M. Responses of papaya seedlings (CaricapapayaL.) to water stress and rehydration: growth, photosynthesis and mineral nutrient imbalance.PlantSoil, 2006, 281: 137-146.
[25]Rizhsky L, Liang H, Shuman J, et al. When defense pathways collide: The response ofArabidopsisto a combination of drought and heat stress.PlantPhysiol, 2004, 134: 1683-1696.
[26]Degenkolbe T, Do P T, Zuther E, et al. Expression profiling of rice cultivars differing in their tolerance to long-term drought stress.PlantMolBiol, 2009, l69: 133-153.
[27]Wang N L, Xiao B Z, Xiong L Z. Identification of a cluster ofPR4-like genes involved in stress responses in rice.JPlantPhysiol, 2011, 168(18): 2212-2224.
[28]Bartels D, Sunkar R. Drought and salt tolerance in plants.CritRevPlantSci, 2005, 24: 23-58.
[29]Liepman A H, Nairn C J, Willats W G, et al. Functional genomic analysis supports conservation of function among cellulose synthase-like a gene family members and suggests diverse roles of mannans in plants.PlantPhysiol, 2007, 143: 1881-1893.
[30]Cocuron J C, Lerouxel O, Drakakaki G, et al. A gene from the cellulose synthase like C family encodes a beta-1, 4 glucan synthase.PNAS, 2007, 104: 8550-8555.
[31]Burton R A, Jobling S A, Shirley N J, et al. The genetics and transcriptional profiles of the cellulose synthase-like HvCsIF gene family in barley.PlantPhysiol, 2008, 146: 1821-1833.
[32]Dolan L, Davies J. Cell expansion in roots.CurrOpinPlantBiol, 2004, 7: 33-39.
Global Genome Expression Analysis of Root Genes under Drought Stress in Weedy Rice and Up-land Rice
DING Guo-hua1,2,#, SUN Jian2,#, YANG Guang3, ZHANG Feng-ming1, BAI Liang-ming1, SUN Shi-chen1, JIANG Shu-kun1, WANG Tong-tong1, ZHENG Hong-liang1, XIA Tian-shu1, SHEN Xi-hong4, MA Dian-rong2, *, CHEN Wen-fu2,*
(1Postdoctoral Scientific Research Station of HAAS/Cultivation and Farming Research Institute of HASS/ Northern Japonica Rice Molecular Breeding Joint Research Center, Harbin 150086, China;2Shenyang Agricultural University, Shenyang 110866, China;3Institute of Economic Crop of Liaoning, Liaoyang 111000, China;4China National Rice Research Institute,Hangzhou 310006,China;#These authors contributed equally to this work;*Corresponding author, E-mail: madianrong@163.com, wfchen5512@126.com)
The expression changes of root genes in drought-resistance weedy rice HEB07-2 (Oryzasativaf.spontanea) and up-land rice IAPAR9 (Oryzasativa) were analyzed under polyethylene glycol(PEG)-simulated with drought stress condition with Affymetrix GeneChip rice genome array. The results indicated that the extent and direction of transcriptome response of HEB07-2 and IAPAR9 to drought differed greatly. For HEB07-2, among 6878 expressed genes, 4693 were up-regulated and 2185 were down-regulated under drought condition. For IAPAR9, among 2923 expressed genes,983 were up-regulated and 1940 were down-regulated. Analysis of differentially expressed genes in HEB07-2 and IAPAR9 showed that the weedy rice HEB07-2 had a higher changing fold than the up-land rice IAPAR9. Gene ontology analysis revealed that genes of HEB07-2 in potassium ion transporting(GO: 0006813), secondary metabolite(GO: 0019748), cell growth(GO: 0016049), glucose metabolism(GO:0006006), transmembrane ion transporter activity(GO:0015075), ferroheme coalition(GO:0020037), oxidordeuctase activity(GO:0016491)were significantly up-regulated in comparing with IAPAR9. These were consistent with the physiological and phenotypic data.
weedy rice; root; gene expression profiling; drought resistance
2015-12-09; 修改稿收到日期: 2016-01-23。
黑龙江省农业科学院博士后工作站;哈尔滨市科技局项目(2014RFQYJ125);黑龙江省农业科学院创新工程项目(2014QN006);黑龙江省农业科学院博士人员科研启动金项目(201507-16);国家水稻产业技术体系项目(CARS-01-43); 北方寒地粳稻资源研究与新品种选育项目(2014BAD01B03-02)。
Q786; Q945.78
A
1001-7216(2016)05-0458-11
丁国华, 孙健, 杨光, 等. 干旱胁迫下杂草稻和栽培稻根系基因表达差异研究. 中国水稻科学, 2016, 30(5): 458-468.
