谷氨酸合成途径基因缺失对大肠杆菌发酵L-色氨酸的影响
2016-10-13赵春光魏洪波徐庆阳张成林陈宁
赵春光,魏洪波,徐庆阳,张成林,陈宁
(天津科技大学 生物工程学院,天津,300457)
谷氨酸合成途径基因缺失对大肠杆菌发酵L-色氨酸的影响
赵春光,魏洪波,徐庆阳,张成林,陈宁*
(天津科技大学 生物工程学院,天津,300457)
降低谷氨酸的积累可提高L-色氨酸产量及糖酸转化率。敲除EscherichiacoliTRTH中的谷氨酸脱氢酶及谷氨酸合成酶编码基因gdhA、gltB,构建TRTHA(TRTH,ΔgdhA)、TRTHB(TRTH,ΔgltB),考察gdhA、gltB缺失对L-色氨酸发酵的影响。结果表明,gdhA及gltB缺失能有效降低谷氨酸的积累,但会降低细胞生长及色氨酸合成;培养基中谷氨酸的添加可恢复TRTHA及TRTHB的生长及色氨酸合成能力。在含1 g/L谷氨酸培养基中,利用TRTHB发酵L-色氨酸,L-色氨酸产量(41.23 g/L)及糖酸转化率(15.45%)最高,较TRTH分别提高了10.92%和7.89%;谷氨酸生成量(5.72 g/L)及乙酸积累量(1.73 g/L)分别较TRTH降低了25.23 %及提高了10.19%。TRTH和TRTHB代谢流分析结果表明,gltB缺失会降低谷氨酸合成代谢流并提高乙酸合成代谢流;TRTHB的色氨酸合成代谢流(11.4%)较TRTH提高了40.74%。
大肠杆菌;L-色氨酸;谷氨酸;gdhA;gltB;代谢流分析
L-色氨酸作为人与动物的必需氨基酸,在食品、医药及饲料等行业得到广泛应用[1]。目前,科研人员已对Escherichiacoli发酵生产L-色氨酸进行了深入研究[1-2]。研究表明,谷氨酸是L-色氨酸发酵中的重要代谢副产物,且降低L-色氨酸发酵中的谷氨酸积累量可提高L-色氨酸产量及糖酸转化率[3]。通过代谢流分析表明,还原力不平衡是导致谷氨酸积累的主要原因[4]。在细胞生长阶段,细胞生长可消耗大量的NADPH;而当细胞生长速率降低或者停止生长时对NADPH需求下降,则过剩的NADPH导致谷氨酸的积累[5]。
在E.coli中,谷氨酸的合成途径主要有两条。一条是由gdhA基因编码的谷氨酸脱氢酶(glutamate dehydrogenase, GDH)催化的由α-酮戊二酸(α-Ketoglutaric acid, α-KG)与游离氨发生还原氨基化形成谷氨酸;另一条途径是通过glnA基因编码的谷氨酰胺合成酶(glutamine synthetase, GS)和由gltB/gltD基因编码的谷氨酸合酶(glutamate synthase, GOGAT)合成[6]。通过敲除或弱化GDH、GOGAT编码基因可以减少谷氨酸的积累。在利用谷氨酸棒状杆菌生产α-KG时,通过敲除谷氨酸脱氢酶基因gdh及谷氨酸合成酶基因gltB可降低谷氨酸积累,提高α-KG产量[7]。在L-色氨酸发酵中,通过优化葡萄糖补料策略,使谷氨酸的积累量由83 g/L降至3 g/L以下,L-色氨酸对葡萄糖的得率从10.5%提升至18.5%,达到了理论得率的78%[3]。
在本研究中,利用Red重组技术敲除基因gdhA、gltB,构建缺失菌株TRTHA(TRTH,ΔgdhA)及TRTHB(TRTH,ΔgltB),考察gdhA及gltB缺失对L-色氨酸发酵及代谢流分布的影响,以降低L-色氨酸发酵中的谷氨酸积累,提高L-色氨酸产量及糖酸转化率。
1 材料与方法
1.1材料与试剂
1.1.1菌株、质粒及引物
本研究所用菌株、质粒及引物参见表1,其中菌株E.coliTRTH(trpEDCBA+TetR, Δtna)由天津科技大学工业微生物菌种保藏室提供(保藏号:TCCC 27003)。
1.1.2培养基
LB培养基、2- YT培养基及SOC培养基,参见参考文献[2]。
种子培养基(g/L):葡萄糖 20,酵母粉 7.5,KH2PO41.5,KCl 1.9,(NH4)2SO44,MgSO4·7H2O 3.15,FeSO4·7H2O 75.6 mg/L。
发酵培养基(g/L):葡萄糖 10,酵母浸粉 1,KH2PO41.5,KCl 1.9,(NH4)2SO44,MgSO4·7H2O 3.15,FeSO4·7H2O 75.6 mg/L。
1.1.3主要试剂
TaqDNA聚合酶、限制性内切酶(大连宝生物工程有限公司);PCR引物(北京博迈德科技有限公司);1 kb Marker(Fermentas);DNA片段回收试剂盒、基因组DNA提取试剂盒、质粒小样快速提取试剂盒(北京天恩泽基因科技有限公司);IPTG、X-gal、溶菌酶、氨苄青霉素、氯霉素、四环素(北京索莱宝公司)。
1.2主要仪器
PCR仪、电转化仪、核酸分析仪,BIO-RAD;10 L、50 L发酵罐,上海保兴生物设备工程有限公司;pH、溶氧电极,METTLER TOLEDO;生物传感分析仪(SBA-40C),山东省科学院生物研究所;高效液相色谱系统(Agilent 1200),Agilent Technologies;BioProfile 300A Nova,美国Nova Biomedical。
1.3方法
1.3.1培养方法
基因gdhA及gltB敲除过程中,细菌培养方法参见文献[2]。
一级种子培养:用接种环挑取L-色氨酸菌落接种于装有50 mL一级种子培养基的三角瓶中,置于摇床,36 ℃、200 r/min,培养时间8 h。
二级种子培养:采用压差法按照10%(v/v)的接种量将一级种子液接种于装有二级种子培养基的发酵罐中,培养温度为35 ℃,通过添加氨水将pH维持在7.0,调节通风量及搅拌转速将溶氧水平维持在20%,培养12 h,作为二级种子。
发酵培养:发酵罐接种量10%,培养温度为36 ℃,pH通过流加氨水维持在7.0,通过调节通风量及搅拌转速将溶氧水平维持在20%。当初始葡萄糖耗尽时,采用溶氧反馈控制补料策略流加葡萄糖溶液,培养40 h。
1.3.2Red技术敲除gdhA及gltB基因
利用Red重组系统敲除E.coliTRTH中的gdhA及gltB基因,具体操作步骤参见文献[2]。
1.3.3谷氨酸脱氢酶及谷氨酸合酶活性测定
将菌株TRTH、TRTHA和TRTHB于50 mL LB液体培养基培养至对数期后,4 ℃ 8 000 r/min离心10 min。菌体经Tris-HCl 缓冲液(100 mmol/L,pH 7.2)洗涤并用1 mL相同缓冲液重悬后利用超声破碎仪裂解细胞(功率 350 W,工作时间 5 s,间隔时间 10 s, 100个循环,于冰上操作)。将上述细胞破碎液于4℃ 10 000 r/min离心30 min后取上清液并定容至10 mL,即为粗酶液。采用BCA蛋白定量试剂盒测定总蛋白浓度。
谷氨酸脱氢酶及谷氨酸合酶反应体系见表2和表3,反应180 s后加入10μL 10%(v/v)乙酸终止反应,利用NADP/NADPH定量分析试剂盒测定NADPH浓度。单位酶活定义为在标准酶活性测定条件下(34 ℃,pH 7.2),单位体积的反应体系中单位蛋白质量(mg)每分钟所消耗的NADPH量(mmol)。
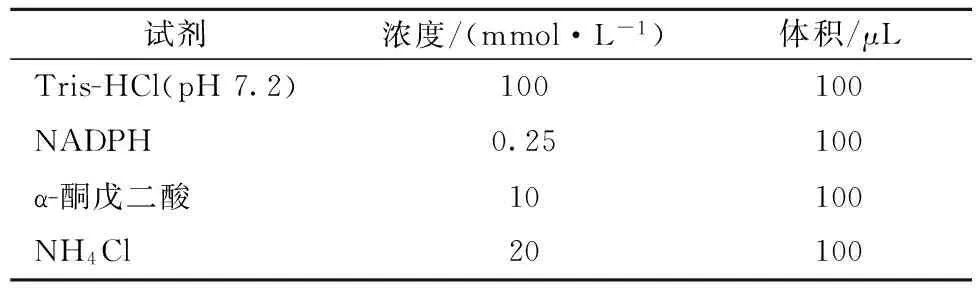
表2 谷氨酸脱氢酶酶促反应体系

表3 谷氨酸合酶酶促反应体系
1.3.4分析方法
生物量及L-色氨酸含量测定参见文献[1];葡萄糖及谷氨酸浓度采用生物传感分析仪测定;乙酸、乳酸及丙氨酸浓度采用BioProfile 300A分析仪测定;建立代谢网络及代谢流平衡模型[2],利用MATLAB软件linprog 函数计算代谢流分布。
1.4动力学参数分析
1.4.1菌体生长速率
通过测定菌体的生物量,计算菌体每小时的生长速率,单位为g/(L·h)。
1.4.2产物合成速率
通过测定产物浓度,计算产物每小时的合成速率,单位为g/(L·h)。
1.4.3显著性分析
每个试验重复3次,且求其数据的平均值,并用±表示数据的标准误差。利用Origin 8.0对数据进行显著性分析,显著性误差为P<0.05。
2 结果与分析
2.1缺失菌株TRTHA及TRTHB的鉴定及其谷氨酸脱氢酶和谷氨酸合酶活性分析
根据参考文献[2]敲除TRTH基因gdhA及gltB,得到缺失菌株TRTHA、TRTHB;分别利用鉴定引物gdhA-P3-gdhA-P4、gltB-P3-gltB-P4进行PCR鉴定,将扩增片段纯化后测序并利用DNAMAN软件进行序列比对,结果与预测值相符,表明gdhA和gltB被成功敲除。分别测定菌株TRTHA和TRTHB的谷氨酸脱氢酶及谷氨酸合酶活性,出发菌株TRTH的谷氨酸脱氢酶及谷氨酸合酶活性分别为1.22 mmol/(min·mg)和1.78 mmol/(min·mg)而TRTHA和TRTHB未检测到相应敲除基因的酶活性(表4),表明敲除gdhA和gltB基因能够使得其编码产物失活。
2.2TRTHA及TRTHB发酵L-色氨酸的过程分析
谷氨酸为细胞生长及色氨酸合成的必需物质,gdhA、gltB基因缺失会影响菌株生长与色氨酸合成。因此,在利用TRTHA及TRTHB发酵L-色氨酸发酵时,在发酵培养基中添加1 g/L谷氨酸(以不含谷氨酸的发酵培养基为对照)。

表4 菌株TRTH、TRTHA和TRTHB谷氨酸脱氢酶及谷氨酸合酶活性
注:*:未检测到;**:未检测。
2.2.1生物量及细胞生长速率
分别利用gdhA缺失菌TRTHA、gltB缺失菌TRTHB在不含及含有谷氨酸的培养基中发酵生产L-色氨酸,生物量及细胞生长速率如图1所示。

图1 基因缺失对生物量及细胞生长速率的影响Fig.1 Effect of gene deletion on biomass and cell growth rate(注:实心图标表示在不含谷氨酸培养基;空心图标表示在含1g/L谷氨酸培养基)
由图1可知,在不含谷氨酸的培养基发酵L-色氨酸时,TRTHA及TRTHB的细胞生长速率及生物量较低,二者生物量分别为41.78 g/L和37.56 g/L,均低于TRTH(44.78 g/L)。而在含有谷氨酸的培养基中,TRTHA及TRTHB的生长速率及生物量均得到提高,其生物量分别为45.32 g/L和46.23 g/L,与TRTH的生物量无显著性差异。
2.2.2L-色氨酸产量及其合成速率
在TRTHA、TRTHB在不含或者含有谷氨酸培养基中发酵L-色氨酸时,L-色氨酸产量及其合成速率如图2所示。
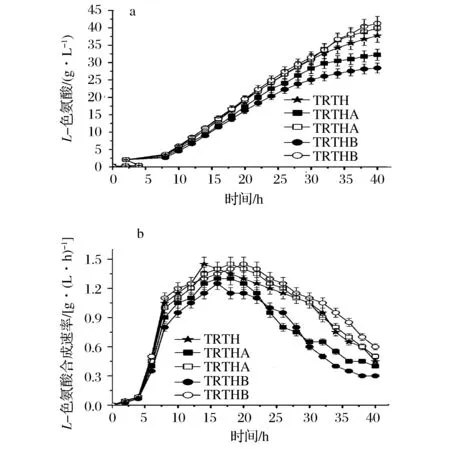
图2 基因缺失对L-色氨酸产量及其合成速率的影响Fig.2 Effect of gene deletion on production and production rate of L-tryptophan(注:实心图标表示在不含谷氨酸培养基;空心图标表示在含1 g/L谷氨酸培养基)
由图2可知,TRTHA及TRTHB在不含谷氨酸培养基中的L-色氨酸产量及其合成速率均低于含有谷氨酸培养基的;TRTHA及TRTHB的L-色氨酸产量分别为32.23 g/L和28.43 g/L。在含谷氨酸培养基中,TRTHA及TRTHB的L-色氨酸合成速率较高,其L-色氨酸产量分别为39.89 g/L和41.23 g/L,较TRTH(37.74 g/L)分别提高了5.69%、10.92%。
2.2.3谷氨酸及乙酸积累量
菌株TRTHA、TRTHB发酵L-色氨酸时,发酵过程中谷氨酸及乙酸浓度如图3所示。
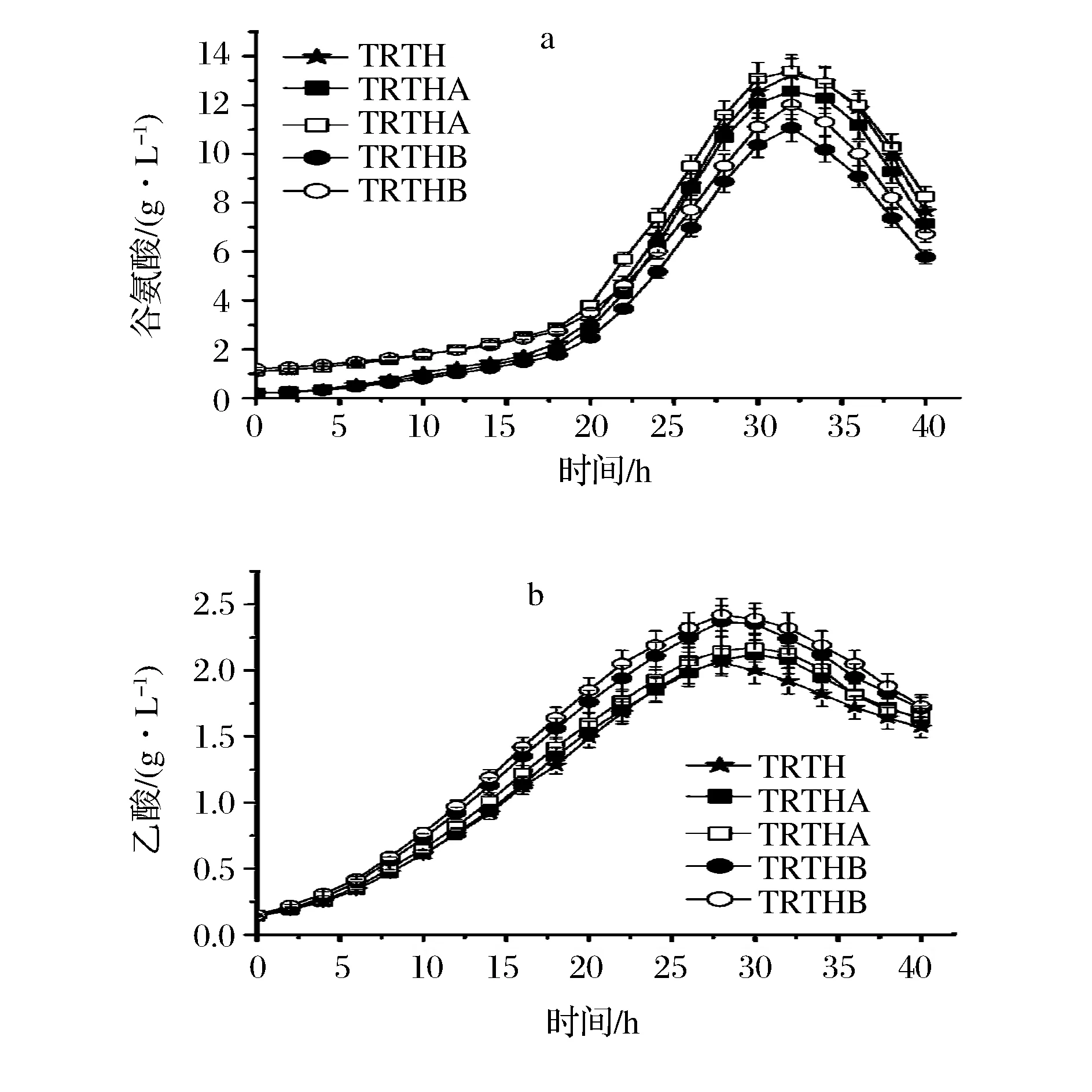
图3 基因缺失对谷氨酸及乙酸积累量的影响Fig.3 Effect of gene deletion on accumulation of glutamate and acetate(注:实心图标表示在不含谷氨酸的培养基;空心图标表示在含1 g/L谷氨酸培养基)
基因gdhA及gltB缺失降低发酵过程中谷氨酸的积累,且TRTHB的谷氨酸生成量低于TRTHA;TRTHA、TRTHB在不含或者含谷氨酸培养基中的谷氨酸生成量分别为7.15 g/L、7.25 g/L、5.78 g/L及5.72 g/L(表5),均低于TRTH(7.56 g/L)。TRTHA及TRTHB的乙酸积累量均高于TRTH(1.57 g/L),且其在含谷氨酸培养基的乙酸积累量高于不含谷氨酸培养基的(无显著差异)。TRTHA、TRTHB在不含或者含谷氨酸培养基中的乙酸积累量分别为1.63、1.65、1.71及1.73 g/L(表5)。此外,于发酵结束后测定了L-色氨酸发酵过程中常见副产物丙氨酸和乳酸的生成量,结果表明在添加谷氨酸条件下敲除gdhA和gltB均能显著降低丙氨酸积累,3菌株发酵液中均未检测到乳酸(表5)。

表5 菌株TRTH、TRTHA和TRTHB副产物生成量
注:*未检测到。
2.2.4葡萄糖消耗速率及糖酸转化率
在L-色氨酸发酵中,葡萄糖消耗速率及糖酸转化率如图4所示。
图4 基因缺失对葡萄糖消耗速率及糖酸转化率的影响Fig.4 Effect of gene deletion on glucose consumption and conversion rate(注:实心图标表示在不含谷氨酸的培养基;空心图标表示在含1 g/L谷氨酸培养基)
由图4可知,菌株TRTHA及TRTHB在不含谷氨酸培养基中葡萄糖消耗速率均低于TRTH;而其在含谷氨酸培养基中的葡萄糖消耗速率高于TRTH,这表明在含谷氨酸培养基中细胞具有较高的生长能力。从整个发酵过程来看,TRTHA及TRTHB在含谷氨酸培养基的糖酸转化率均高于不含谷氨酸培养基的;TRTHA及TRTHB在含谷氨酸的总体糖酸转化率分别为15.05%和7.89%,较TRTH(14.32%)分别提高了5.01%和7.89%;TRTHA及TRTHB在不含谷氨酸的总体糖酸转化率分别14.45%和14.02%。
2.2.5TRTH及TRTHB的L-色氨酸合成途径代谢流分布
利用TRTH及TRTHB(含1 g/L谷氨酸的培养基)生产L-色氨酸的发酵过程可知,在28 h后菌体生物量无明显变化,则假设此阶段细胞处于拟稳态。测定发酵后期(28~40 h)的葡萄糖、色氨酸、乙酸、乳酸、丙氨酸及谷氨酸的胞外浓度,并计算其消耗或积累速率。利用MATLAB软件计算利用TRTH及TRTHB发酵L-色氨酸时,发酵中后期L-色氨酸生物合成途径的代谢流分布情况,如图5所示。

图5 菌株TRTH及TRTHB的L-色氨酸发酵后期的代谢流分布情况Fig.5 The metabolic flux distribution of TRTH and TRTHB during the later fermentation period of L-tryptophan production(括号内数值表示TRTHB的代谢流)
由图5可知,基因gltB缺失会影响整个代谢流的分布情况,碳代谢流由糖酵解途径向磷酸戊糖途径发生迁移;gltB缺失使进入糖酵解途径的碳代谢流由87.2%降至78.9%,而进入磷酸戊糖途径的代谢流由12.8%提高至21.1%。TRTHB中的谷氨酸合成及排除细胞的代谢流均低于TRTH,其中TRTHB中排除细胞的谷氨酸代谢流为1.2%,较TRTH降低了36.84%。gltB缺失引起乙酸及乳酸合成代谢流的提高,其中乙酸合成代谢流为13.7%,较TRTH提高了23.42%。TRTHB中色氨酸合成的代谢流为14.4%,较TRTH提高了40.74%。
3 讨论
基因gdhA、gltB在E.coli的谷氨酸合成途径中起着重要作用,且gdhA及gltB基因缺失会降低菌株的谷氨酸积累量[8]。与TRTH相比,TRTHA及TRTHB的谷氨酸生成量分别降低了6.54%和25.23%。在E.coli的谷氨酸合成途径中,gltB对谷氨酸合成的作用大于gdhA,这可能是由于gdhA编码的谷氨酸脱氢酶在高浓度的NH4+环境下才具有较高活性造成的[9]。谷氨酸是细胞生长过程中所需的前体物,则谷氨酸合成量不足会影响菌体的生长及生物量[10]。TRTHA及TRTHB在不含谷氨酸培养基中的生长速率及生物量均低于其在含谷氨酸的培养基中。gdhA及gltB基因缺失会降低谷氨酸的积累,但会提高其他代谢副产物合成的代谢流(如:丙酮酸、乳酸、乙酸等),从而会提高其它代谢副产物的积累量[11]。TRTHA及TRTHB的乙酸积累量均高于菌株TRTH。TRTHA及TRTHB在含谷氨酸培养基发酵L-色氨酸,其L-色氨酸产量及糖酸转化率均高于TRTH。这表明基因gdhA、gltB的缺失降低了谷氨酸的积累,使L-色氨酸产量及糖酸转化率得到了提高[3]。对于L-色氨酸生产菌株而言,gltB缺失菌株的L-色氨酸产量及糖酸转化率均高于gdhA缺失菌株,则表明缺失gltB基因更有利于L-色氨酸发酵。
由TRTH及TRTHB的代谢流分布可知,TRTHB中进入磷酸戊糖途径的代谢流提高,这会增加色氨酸合成前体物4-磷酸赤藓糖及NADPH的供给,从而促进L-色氨酸合成[12]。TRTHB中谷氨酸合成代谢流的降低,会引起乙酸及乳酸等其它代谢副产物的合成代谢流的提高[13]。因此,应进一步研究TRTHB中的乙酸合成机制,降低其乙酸积累,以提高L-色氨酸产量。谷氨酸合成途径基因的缺失在一定程度上降低了谷氨酸合成,但从谷氨酸合成的原因分析,其主要是由于能量代谢和还原力代谢不平衡造成的,因此针对与能量和还原力代谢有关的ATP和NADPH等辅因子的调控可作为降低谷氨酸积累更有效的手段[14],如克隆由NADPH到NADP的脱氢酶;增加从NADPH到NADP净转化的无效循环的代谢流[3];通过改变磷酸烯醇式丙酮酸代谢,限制通过异柠檬酸脱氢酶生产NADPH以增加色氨酸合成的代谢流;通过改变异柠檬酸脱氢酶对NADP的特异性也能解决此问题[7]。
[1]WANG Jian, CHENG Li-kun, WANG Jing, et al. Genetic engineering ofEscherichiacolito enhance production ofL-tryptophan[J]. Applied Microbiology and Biotechnology, 2013, 97(17):7 587-7 596.
[2]黄静, 史建明, 刘倩, 等.pta基因敲除对L-色氨酸发酵的影响[J]. 微生物学报, 2011, 51(4):480-487.
[3]DODGE TC, GERSTNER JM. Optimization of the glucose feed rate prole for the production of tryptophan from recombinantE.coli[J]. Journal of Chemical Technology & Biotechnology, 2002, 77(11):1 238-1 245.
[4]王镜岩, 朱圣庚, 徐长法. 生物化学第三版下册[M]. 北京: 高等教育出版社, 2002:153.
[5]张伟国, 徐建中.L-赖氨酸合成代谢中NADPH代谢的研究进展[J]. 食品与生物技术学报, 2012, 31(10):1 009-1 017.
[6]李文滨, 周失, 李永光. 大肠杆菌谷氨酸脱氢酶基因的克隆及功能分析[J]. 东北农业大学学报, 2014, 45(1):53-58.
[7]LEE YB, JO JH, KIM MH, et al. Enhanced production of α-ketoglutarate by fed-batch culture in the metabolically engineered strains ofCorynebacteriumglutamicum[J]. Biotechnology and Bioprocess Engineering, 2013, 18(4):770-777.
[8]SSHAFER A, KALINOWSKI J, SIMON R, et al. High frequency conjugal plasmid transfer from gram-negativeEscherichiacolito various gram-positive coryneform bacteria[J]. Journal of Bacteriology, 1990, 172(3):1 663-1 666.
[9]GUTIERREZ-MENDEZ N, VALLJO-CORDOBA B, GONZALEZ-CORDOVA A F, et al. Evaluation of aroma generation ofLactococcuslactiswith an electronic nose and sensory analysis[J]. Journal of Dairy Science, 2008, 91(1):49-57.
[10]SILBERBACH M, SCHAFER M, HUSER A T, et al. Adaptation ofCorynebacteriumglutamicumto ammonium limitation: a global analysis using transcriptome and proteome techniques[J]. Applied & Environmental Microbiology, 2005, 71(5):2 391-2 402.
[11]MARIENHAGEN J, KENNERKNECHT N, SAHM H, et al. Functional analysis of all aminotransferases inferred from the genomic sequence ofCorynebacteriumglutamicum[J]. Journal of Bacteriology, 2005, 187(22):7 639-7 646.
[12]BAEZ-VIVEROS JL, FLORES N, JUAREZ K, et al. Metabolic transcription analysis of engineeredEscherichiacolistrains that overproduceL-phenylalanine[J]. Microb Cell Factories, 2007, doi:10.1186/1475-2859-6-30
[13]ZHAO Chun-guang, CHENG Li-kun, WANG Jian, et al. Impact of deletion of the genes encoding acetate kinase on production ofL-tryptophan byEscherichiacoli[J]. Annals of Microbiology, 2016, 66(1):261-269
[14]TJENER K, STAHNKE LH, ANDERSEN L, et al. Addition of α-ketoglutarate enhances formation of volatiles byStaphylococcuscarnosusduring sausage fermentation[J]. Meat Science, 2004, 67(4):711-719.
Effect of deletion of genes in glutamate biosynthesis pathway onL-tryptophan fermentation byEscherichiacoli
ZHAO Chun-guang, WEI Hong-bo, XU Qing-yang,ZHANG Cheng-lin, CHEN Ning*
(College of Biotechnology, Tianjin University of Science and Technology, Tianjin 300457,China)
The production ofL-tryptophan and glucose conversion rate was increased by decreasing the accumulation of glutamate.gdhA encoding glutamate dehydrogenase andgltB encoding glutamate decarboxylase inE.coliTRTH were deleted to construct TRTHA (TRTH, ΔgdhA) and TRTHB (TRTH, ΔgltB). The effect of deletion ofgdhA andgltB onL-tryptophan fermentation was subsequently investigated. The results indicated that the disruption ofgdhA orgltB decreased the excretion of glutamate as well as the cell growth andL-tryptophan biosynthesis. While the cell growth rate andL-tryptophan biosynthesis of TRTHA and TRTHB were recovered by adding glutamate in media. WithL-tryptophan fermentation using TRTHB, the highest production ofL-tryptophan (41.23 g/L) and glucose conversion rate (15.45%) were increased by 10.92% and 7.89%, glutamate production (5.72 g/L) was decreased by 25.23% and acetate accumulation was increased by 10.19% compared with TRTH. Analysis on metabolic flux distribution of TRTH and TRTHB showed that the flux of glutamate was decreased and the flux of acetate was increased with deletion ofgltB, and the flux of tryptophan of TRTHB (11.4%) was increased by 40.74%.
Escherichiacoli;L-tryptophan; glutamate;gdhA;gltB; analysis of metabolic flux
10.13995/j.cnki.11-1802/ts.201609002
在读博士(陈宁教授为通讯作者,E-mail: ningch@tust.edu.cn)。
国家高技术研究发展计划(863计划):2015AA021003
2016-04-22,改回日期:2016-06-07
