乳酸菌中乳酸脱氢酶的研究进展
2016-09-14黄艳娜许光涛游春苹乳业生物技术国家重点实验室光明乳业股份有限公司乳业研究院上海00436邯郸钢铁集团职工医院河北邯郸05600
黄艳娜,许光涛,游春苹,*(.乳业生物技术国家重点实验室,光明乳业股份有限公司乳业研究院,上海00436;.邯郸钢铁集团职工医院,河北邯郸05600)
乳酸菌中乳酸脱氢酶的研究进展
黄艳娜1,许光涛2,游春苹1,*
(1.乳业生物技术国家重点实验室,光明乳业股份有限公司乳业研究院,上海200436;2.邯郸钢铁集团职工医院,河北邯郸056001)
乳酸脱氢酶(Lactate dehydrogenases,LDHs)是乳酸菌利用碳源转化丙酮酸为乳酸的关键酶。本文从乳酸脱氢酶的分类、结构与功能、合成乳酸的微生物属别及代谢途径等方面,综述了乳酸菌中乳酸脱氢酶的研究进展,以期为微生物合成乳酸的代谢调控提供一定的理论指导。
乳酸菌,L-乳酸脱氢酶,D-乳酸脱氢酶,分类,结构与功能
乳酸是一种重要的生物基平台化合物,广泛应用于农业、食品、医药、化工和环保等领域。根据其旋光性的不同可分为D-乳酸和L-乳酸,分别由D-乳酸脱氢酶(D-LDH,EC:1.1.1.28)和L-乳酸脱氢酶(LLDH,EC:1.1.1.27)催化[1-3]。近年来,随着人们对利用L-乳酸聚合生成聚L-乳酸(PLA)以合成生物降解塑料的重视,乳酸菌也随之得到了广泛关注[4]。
乳酸菌是一类发酵糖类合成乳酸的革兰氏阳性细菌的总称,广泛存在于动物、植物和乳制品中。乳酸脱氢酶(Lactate dehydrogenases,LDHs)作为乳酸菌合成乳酸的关键酶,目前已研究的较为透彻。尽管该酶分布十分广泛,但不同的LDHs性质存在较大的差异。因此,本文将主要围绕乳酸脱氢酶的分类、种群分布、结构与功能以及乳酸合成的代谢调控等方面,综述乳酸菌中乳酸脱氢酶的研究进展,以解析其在乳酸合成中的作用机制。
1 乳酸脱氢酶的分类
根据结合辅酶的不同,微生物体一般包含两种乳酸脱氢酶,NAD-依赖型乳酸脱氢酶(NAD-dependent lactate dehydrogenases,nLDHs)和NAD-非依赖型乳酸脱氢酶(NAD-independent lactate dehydrogenases,iLDHs)两大类。iLDHs,也被称作呼吸链乳酸脱氢酶,在微生物体内通常被认为是负责乳酸氧化的酶类。它们编码的基因与乳酸利用相关的乳酸透性酶和调节蛋白编码的基因较近[5]。相较于iLDHs,nLDHs分布范围更广泛,在人体、动物以及微生物中都普遍存在。在微生物中,nLDHs存在于其细胞质中,体外实验证实:nLDHs能够以NADH为辅因子催化丙酮酸还原生成乳酸,并且氧化NADH为NAD+,同时该酶在特定的条件下可以催化逆反应的进行,但是活力非常弱,不容易检测。一般说来,与nLDHs以NADH为辅因子不同的是iLDHs主要利用黄素[6-7]。nLDHs也可以在
体内催化乳酸的氧化,但体外实验可能需要较高的pH和底物浓度[8]。
按其催化底物的构型不同,nLDHs可以分为NAD依赖型-L-乳酸脱氢酶(L-nLDHs)和NAD依赖型-D-乳酸脱氢酶(D-nLDHs)两大类,分别催化丙酮酸合成L-乳酸和D-乳酸[6]。研究表明,虽然L-nLDHs和D-nLDHs均催化丙酮酸合成乳酸,但二者的基因序列不具有相似性或一致性。已知的不同乳杆菌的D-nLDHs之间存在40%~50%的氨基酸同源性,而D-nLDHs和L-nLDHs之间则只存在10%左右的同源性,表明两种脱氢酶分别由两个完全不相关的家族进化而来。不仅如此,酶之间的差异也表现在动力学参数差异明显,比如,两者催化反应的反应常数kcat和米氏常数Km不同。
根据天然电子受体的不同,可以将iLDHs分为三类。第一类为膜蛋白,利用膜醌类作为外部的电子受体[7,9];第二类直接利用O2作为电子受体,根据氧化终产物的不同,又将其细分为乳酸氧化酶(Lactate oxidase,LOX)和乳酸单氧酶(Lactate monooxygenases,LMO),其中前者产生丙酮酸和H2O2,而后者产生乙酸、CO2和H2O[10-11];第三类是硫胺b2(flavocytochrome b2),存在于真菌中,它天然的电子受体为细胞色素c。大多数L-iLDHs属于α-羟酸氧化黄素蛋白家族,利用还原型黄素单核苷酸(Flavin mononuleotide,FMN),而所有的D-iLDHs均可利用黄素腺嘌呤二核苷酸(FAD)作为电子受体,而不是FMN,它们具有FAD结合的氧化还原酶/转移酶4的家族特征[12-13]。此外,D-iLDHs还可以利用醌类或细胞色素c作为电子受体,但是迄今为止,还没有D-iLDHs直接利用O2的相关报道。Wolin[14]的研究表明,一些链球菌LDHs的催化活性绝对依赖于果糖-1,6-二磷酸(Fructose-1,6-Diphosphate,FDP)。其他链球菌、双歧杆菌属的所有种、干酪乳杆菌和木糖乳杆菌等都发现了相似的iLDHs。
2 乳酸脱氢酶的结构与功能
伴随着全基因组测序技术的发展,科学家已发现很多乳酸菌中存在多个乳酸脱氢酶基因[15],这些基因表达的乳酸脱氢酶由于基因序列和氨基酸序列的不同,在酶学性质上有很大的差异,甚至在乳酸菌中的功能都有所差异[16]。研究表明,L-LDH和L-苹果酸脱氢酶属于同一家族,而D-LDH属于D-2-羟酸脱氢酶家族[8]。
2.1 D-乳酸脱氢酶(D-LDH)的结构和功能
大多数D-LDH催化是可逆反应,极少数不可逆;对于可逆的D-LDH来说,只有环境中乳酸浓度较高时才催化逆反应,即催化乳酸合成丙酮酸,来参与细菌的代谢。
D-LDH的一级结构比对表明,不同种属的D-LDH的氨基酸序列存在较大的差异,但是参与丙酮酸的结合与催化的氨基酸残基却十分保守。Kochhar等[17]的研究显示L.bulgaricus的D-LDH是一个由相同亚基组成的二聚体,每个亚基由332个氨基酸残基组成,分子量为36.8 ku,经化学修饰发现位于催化中心的三个组氨酸(His205、His296和His303)和Asp259对底物的催化具有十分重要的作用。Kochhar等还发现,His289也参与了底物结合,但His289不是D-2-羟基酸脱氢酶家族中保守的氨基酸残基。另外,通过对Cys残基的化学修饰发现Cys234可能参与底物结合和催化作用,这在L.helveticus的羟基丙酮酸还原酶中也能得以证实[17]。研究还发现L.helveticus CNRZ32 的D-LDH氨基酸序列和D-2-羟基酸脱氢酶家族的D-3-磷酸甘油酸脱氢酶、羟基丙酮酸还原酶和D-2-羟基己酸脱氢酶存在很高的同源性,这也暗示了该酶是D-2-羟基酸脱氢酶家族的一个成员[18]。Razeto等[19-20]对L.bulgaricus的D-LDH构象进行了研究,结果发现L.bulgaricus的D-LDH是一个由两个相同的亚基构成的不对称酶,A亚基的酶蛋白是典型的“开放”构象,而B亚基是典型的“闭合”构象。其中NADH的结合位点主要存在于B亚基中,而在A亚基中仅有30%,同时在底物结合口袋中有一个硫酸根离子。另外,该研究还建立了丙酮酸分子在活性位点的模型。在闭合域中,存在一簇疏水的氨基酸残基紧紧围绕着丙酮酸分子的甲基,在这个疏水氨基酸簇中至少有三个氨基酸残基(Tyr52、Phe299和Trp135’)决定底物的专一性。研究表明该底物结合位点有利于闭合域的稳定和酶的激活。
2.2 L-乳酸脱氢酶(L-LDH)的结构与功能
大多数乳酸菌中不仅存在D-LDH也存在L-LDH,L-LDH催化丙酮酸还原生成L-乳酸。L-LDH分为两型:一类可被FDP激活,属于别构酶;另一类不需要FDP激活,不具有别构效应。据Hiroyuki Uchikoba报道,L.pentosus的L-LDH是一个非异构酶,但是它的氨基酸序列和一些细菌的别构LDH具有很高的相似性[21]。该酶的四聚体由相同亚基组成对称的酶结构。它的活性构象和别构LDH的构象相似。
Kazuhito Arai等[22]的研究表明,Lactobacillus的L-LDH氨基酸序列中,Glu102、Asp197和Thr246是高度保守的,它们参与了底物的识别;另外,98-110氨基酸残基构成了活性位点环,它们参与催化反应。Arg171的胍基和丙酮酸的羰基形成双氢键,从而使丙酮酸在催化位点中处于正确的结合方向[23]。研究表明,在L-LDH的氨基酸序列中同样存在一个与辅酶NADH结合的结构域Gly-Xaa-Gly-Xaa-Xaa-Gly-(17Xaa)-Asp,其中该结构域中的Asp决定了L-LDH的辅酶是NADH,而不是NADPH[24]。L.pentosus中L-LDH的活性位点和其他LDH相似,包括了参与催化与底物结合的保守氨基酸残基,如Aspl68、Argl71和Hisl95[25]。其中,Argl71在底物的结合中起着重要作用;另外,Hisl95主要在催化过程中作为质子供体和受体[26]。值得注意的是,虽然Bacillus 和Lactobacillus的L-LDH在进化关系上存在很近的亲缘关系,但是活性位点环的氨基酸残基也存在差异,如101位Bacillus是Asn,而Lactobacillus是Pro。这表明L-LDH是否来自好氧或厌氧的革兰氏阳性菌可以由这个位置的氨基酸残基是Asn101还是Pro101来识别[27]。
3 合成乳酸的微生物属别
3.1 合成L-乳酸的微生物属别
产光学纯的L-乳酸的乳酸菌主要有以下四个属中的四十余种[24,28-29],包括乳杆菌属(Lactobacillus)、链球菌属(Streptococcus)、明串珠菌属(Leuconostoc)和片球菌属(Pediococcus),如L.casei、L.paracasei、L.mali、L.alimentarius、Lactococcus lactis、Streptococcus thermophilus和Pediococcus pentosaceus等均合成L-乳酸,也有利用嗜热脂肪芽孢杆菌(Bacillus stearothermophilus)和 凝 结 芽 孢 杆 菌(Bacillus coagulans)生产L-乳酸的报道。
3.2 合成D-乳酸的微生物属别
产光学纯的D-乳酸的乳酸细菌主要分布在乳杆菌属(Lactobacillus)[24,30]、芽孢杆菌属(Bacillus)、芽孢乳杆菌属(Sporolactobacillus)和明串珠菌属(Leuconostoc)4个属[31-32]。除此以外,还分布在肉杆菌属(Carnobacterium)[33]、肠球菌属(Enterococcus)[34]、乳球菌属(Lactococcus)[35]、片球菌属(Pediococcus)[36]、链球菌属(Streptococcus)[37]、四联球菌属(Tetragenococcus)[38]、漫游球菌属(Vagococcus)和魏斯氏菌属(Weissella)等中[32]。目前国内外研究较多的D-乳酸生产菌主要集中在乳杆菌属和芽孢乳杆菌属,这两类菌都是同型发酵菌,以葡萄糖为碳源,通过糖酵解途径生成丙酮酸,然后丙酮酸经D-乳酸脱氢酶作用生成D-乳酸。
4 D/L-乳酸的合成
乳酸是由丙酮酸还原而成,由于菌体内酶系统的差异,其代谢途径分为三类,即同型发酵(Homofermentation)途径、异型发酵(Heterofermentation)途径和双歧(Bifidum Pathway)途径,其中以前两种为主(图1)。工业生产乳酸的菌种为同型乳酸发酵类型,其理论转化率为100%,发酵产物中乳酸占80%,其余为乙酸等[39]。
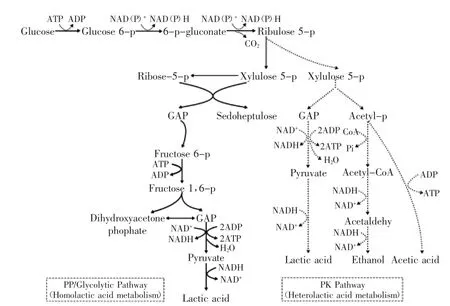
图1 乳酸菌利用葡萄糖合成乳酸的代谢途径[2]Fig.1 Metabolic pathways for lactic acid production from glucose by the lactic acid bacteria[2]
乳酸菌细胞内乳酸脱氢酶的立体特异性决定了形成乳酸的立体构型,如L.delbrueckii、L.bulgaricus、L.jensenii和L.coryniforis发酵产物以D-乳酸为主[11-12,31,40],有的菌株含有两种乳酸脱氢酶或存在乳酸消旋酶,最终形成DL-乳酸。而有些乳酸菌产生的是L-乳酸,当L-乳酸积累时,则诱导产生乳酸消旋酶,该酶将L-乳酸转变为D-乳酸,直至平衡。有研究显示,改变pH或营养物质,D-乳酸或L-乳酸的比例将发生变化。
光学纯的同分异构体可以通过表达光学特异活性的D或L-乳酸脱氢酶获得。对于合成D-乳酸的乳酸菌来说,D-LDHs具有较L-LDHs更高的丙酮酸催化效率。采用D-LDHs或者L-LDHs生物转化丙酮酸可产生有光学特性的D-乳酸或者L-乳酸。通过过量表达ldhD或ldhL基因,在合适的宿主中高效表达来实现相应构型乳酸产量的提高。杨登峰等[41]在大肠杆菌中过量表达L.rhamnosus的ldhL基因,成功生产获得纯度为99%的L-乳酸;Zhou等通过将乳酸片球菌Pediococcus acidilactici的L-LDH基因ldhL替换大肠杆菌中的D-LDH基因ldhA,从而得到的工程菌SZ85可生产产率为95%的高光学纯的L-乳酸[42]。Okano等敲除L.plantarum NCIMB 8826的ldhL基因,表达来自牛链球菌Streptococcus bovis148的α-淀粉酶基因,得到的工程菌可有效利用淀粉合成高光学纯的D-乳酸[43]。
乳酸的合成受到发酵温度的影响,这与乳酸脱氢酶受到温度的调控有关。Doo等[44]筛选到一株D-乳酸的高产菌株L.coryniformis subsp.torquens KCTC 3535,其合成D-乳酸的浓度可高达131.7 g/L,提高发酵温度,L-乳酸的合成迅速增加。随后他们开展了温度相关的乳酸脱氢酶的研究,研究显示L.coryniformis在培养温度为30℃时发酵产物以合成D-乳酸为主,发酵温度提高到40℃可合成大量的L-乳酸,这表明L-LDHs较高的热稳定性可能是L.coryniformis在高温条件下合成光学纯D-乳酸产率降低的关键因素[40]。
较高的底物和乳酸浓度会影响细胞的渗透压,从而抑制细胞的生长和乳酸的合成。Zou等在干酪乳杆菌L.casei摇瓶发酵过程中添加2 g/L甜菜碱代替Tween 80,可提高L-乳酸脱氢酶的活性,对该菌进行补料分批发酵,L-乳酸的浓度提高到190 g/L[45]。
乳酸的合成还受到氧的调控。Collins和Lascelles的研究显示,厌氧条件下LDHs的酶活性是好氧条件的10倍左右[46],因此不同的发酵条件,如通气量和搅拌转速等的不同也会影响乳酸的合成。除此以外,还可以通过代谢调控的手段控制乳酸的合成。有研究表明,在链球菌中,FDP通过影响别构酶L-LDH的活力,可改变发酵代谢途径的方向和通量:在细胞内FDP充足时,发酵产物主要是乳酸;而细胞内FDP缺乏时,发酵产物主要是乙酸、甲酸和乙醇[47]。适当调节葡萄糖碳源流向糖酵解途径和磷酸戊糖途径,使之积累更多的FDP,可达到积累乳酸的目的。所以如何调控胞内FDP浓度、能量水平、还原力以及几种酶激活剂浓度,是研究的下一个目标。
5 展望
乳酸脱氢酶是一类分布非常广泛的脱氢酶类,该酶是生物法合成乳酸的关键酶,尤其在乳酸菌中。
本文从乳酸脱氢酶的分类、结构和功能以及合成乳酸的种属分布等方面,综述了目前D-乳酸脱氢酶和L-乳酸脱氢酶在乳酸菌中的研究现状。鉴于其在合成乳酸中的关键作用以及其清楚的蛋白结构和功能,可对该酶进行理性改造和定向进化,不仅可以拓宽其底物范围、提高其酶活力,还可以赋予乳酸脱氢酶其他重要的生物功能。
[1]Abdel-Rahman M A,Tashiro Y,Sonomoto K.Lactic acid production from lignocelluloses derived sugars using lactic acid bacteria:overview and limits[J].J Biotechnol,2011,156:286-301.
[2]Abdel-Rahman M A,Tashiro Y,Sonomoto K.Recent advances in lactic acid production by microbial fermentation processes[J].Biotechnol Adv,2013,31:877-902.
[3]Gasser F.Electrophoretic characterization of lactic dehydrogenases in the genus Lactobacillus[J].J Gene Microbiol,1970,62:223-239.
[4]Kinnersley A M,Scott T C,Yopp J H.Promotion of plant growth by polymers of lactic acid[J].Plant Growth Regul,1990,9:137-146.
[5]Zhao R,Zheng S,Duan C C,et al.NAD-dependent lactate dehydrogenase catalyses the first step in respiratory utilization of lactate by Lactococcus lactis[J].FEBS Open Bio,2013,3:379-386.
[6]Jiang T Y,Gao C,Ma C Q,et al.Microbial lactate utilization:enzymes,pathogenesis,and regulation[J].Trends in Microbiol,2014,22:589-599.
[7]Garvie E I.Bacterial lactate dehydrogenases[J].Microbiol Rev,1980,44:106-139.
[8]Goffin P,Lorquet F,Kleerebezem M,et al.Major role of NAD-dependent lactate dehydrogenases in aerobiclactate utilization in Lactobacillus plantarum during early stationary phase[J].J Bacteriol,2004,186:6661-6666.
[9]Stansen C,Uy D,Delaunay S,et al.Characterization of a Corynebacterium glutamicum lactate utilization operon induced during temperature triggered glutamate production[J].Appl Environ Microbiol,2005,71:5920-5928.
[10]Furuichi M,Suzuki N,Dhakshnamoorhty B,et al.X-ray structures of Aerococcus viridans lactate oxidase and its complex with D-lactate at pH4.5 show an alpha-hydroxyacid oxidation mechanism[J].J Mol Biol,2008,378:436-446.
[11]Maeda-Yorita K,Aki K,Sagai H,et al.L-Lactate oxidase and L-lactate monooxygenase:mechanistic variations on a common structural theme[J].Biochimie,1995,77:631-642.
[12]Cristescu M E,Egbosimba E E.Evolutionary history of D-lactate dehydrogenases:a phylogenomic perspective on functional diversity in the FAD binding oxidoreductase/transferase type 4 family[J].J Mol Evol,2009,69:276-287.
[13]Fraaije M W,van Berkel W J,Benen J A,et al.Anovel oxidoreductase family sharing a conserved FAD-binding domain [J].Trends Biochem Sci,1998,23:206-207.
[14]Wolin M J.Fructose 1,6-diphosphate requirement of streptococcal lactic dehydrogenases[J].Science,1964,146:775-777.
[15]Cristescu M E,Innes D J,Stillman J H,et al.D-and L-lactate dehydrogenases during invertebrate evolution[J].BMC Evol Biol,2008,8:268.
[16]Denni D,Kaplan N O.D-and L-lactic acid dehydrogenases in Lactobacillus plantarum[J].J Biol Chem,1960,235:810-818.
[17]Kochhar S,Hunziker P E,Leong-Morgenthaler P,et al.Evolutionary relationship ofNAD+-dependent D-lactate dehydrogenase:comparison of primary structure of 2-hydroxy acid dehydrogenases[J].Biochem Biophy Res Commun,1992,184:60-66.
[18]Arthur M,Molinas C,Dutka-Malen S,et al.Structural relationship between the vancomycin resistance protein VanH and 2-hydroxycarboxylic acid dehydrogenases[J].Gene,1991,103:133-134.
[19]Razeto A,Kochhar S,Hottinger H,et al.Domain closure,substrate specificity and catalysis of D-lactate dehydrogenase from Lactobacillus bulgaricus[J].J Mol Biol,2002,318:109-119.
[20]Holton S J,Anandhakrishnan M,Geerlof A,et al.Structural characterization ofa D-isomer specific 2-hydroxyacid dehydrogenase from Lactobacillus delbrueckii ssp.bulgaricus[J].J Struct Biol,2013,181:179-184.
[21]Uchikoba H,Fushinobu S,Wakagi T,et al.Crystal structures of non-allosteric L-lactate dehydrogenase from Lactobacillus pentosus at 2.3 A resolution:specific interactions at subunit interfaces[J].Protein,2002,46:206-214.
[22]Arai K,Kamata T,Ucbikoba H,et al.Some Lactobacillus L-lactate dehydrogenases exhibit comparable catalytic activities for pyruvate and oxaloacetate[J].J Bacteriol,2001,183:397-400.
[23]Hart K W,Clarke A R,Wigley D B,et al.A strong carboxylatearginine interaction is important in substrate orientation and recognition in lactate dehydrogenase[J].Biochem Biophys Acta,1998,914:294-298.
[24]Bernard N,Ferain T,Garmyn D,et al.Cloning of the D-lactate dehydrogenase gene from Lactobcillus delbrueckii subsp.bulgaricus by complementation in Escherichia coli[J].FEBS Lett,1991,290:61-64.
[25]Taguchi H,Ohta T.D-Lactate dehydrogenase is a member of the D-isomer-specific 2-hydroxyacid dehydrogenase family [J].Biol Chem,1993,268:l8030-18034.
[26]Hart K W,Clarke A R,Wigley D B,et al.The importance of arginine 171 in substrate binding by Bacillus stearothermophilus lactate dehydrogenase isozymes[J].Biochem Biophys Res Comm,1987,146:346-353.
[27]Griffin H G,Swindell S R,Gasson M J.Cloning and sequence analysis of the gene encoding L-lactate dehydrogenase from Lactococcus lactis:evolutionary relationships between 21 different LDH enzymes[J].Gene,1992,122:193-197.
[28]Laopaiboon P,Thani A,Leelavatcharamas V,et al.Acid hydrolysis of sugarcane bagasse for lactic acid production[J].Bioresour Technol,2010,101:1036-1043.
[29]Wessels S,Axelsson L,Hansen B E,et al.The lactic acid bacteria,the food chain,and their regulation[J].Trends Food Sci Tech,2004,15:498-505.
[30]Lapierre L,Germond J E,Ott A,et al.D-Lactate dehydrogenase gene(ldhD)inactivation and resulting metabolic effects in the Lactobacillus johnsonii strains La1 and N312[J].Appl Environ Microbiol,1999,65:4002-4007.
[31]Savijoki K,Palva A.Molecular genetics characterization of theL-lactatedehydrogenasegene(ldhL)of Lactobacillus helveticus and biochemical characterization of the enzyme[J].Appl Environ Microbiol,1997,63:2850-2856.
[32]Li L,Eom H J,Park J M,et al.Characterization of the major dehydrogenase related to D-lactic acid synthesis in Leuconostoc mesenteroides subsp.mesenteroides ATCC 8293[J].Enzyme Microb Technol,2012,51:274-279.
[33]Leisner J J,Hansen M A,LarsenM H,et al.The genome sequence of the lactic acid bacterium,Carnobacterium maltaromaticum ATCC 35586 encodes potential virulence factors [J].Int J Food Microbiol,2011,152:107-115.
[34]Shibata K,Flores D M,Kobayashi G,et al.Direct L-lactic acid fermentation with sago starch by a novel amylolytic lactic acid bacterium,Enterococcusfaecium[J].Enzyme Microbiol Technol,2007,41:149-155.
[35]Wu C D,Zhang J,Du G C,et al.Heterologous expression of Lactobacillus casei RecO improved the multiple-stress tolerance and lactic acid production in Lactococcus lactis NZ9000 during salt stress[J].Bioresour Technol,2013,143:238-241.
[36]Zhao K,Qiao Q G,Chu D Q,et al.Simultaneous saccharification and high titer lactic acid fermentation of corn stover using a newly isolated lactic acid bacterium Pediococcus acidilactici DQ2[J].Bioresour Technol,2013,135:481-489.
[37]Gezginc Y,Topcal F,Comertpay S,et al.Quantitative analysis of the lactic acid and acetaldehyde produced by Streptococcus thermophilus and Lactobacillus bulgaricus strains isolated from traditional Turkish yogurts using HPLC[J].Bioresour Technol,2013,135:481-489.
[38]Masuda S,Yamaguchi H,Kurokawa T,et al.Immunomodulatory effect of halophilic lactic acid bacterium Tetragenococcus halophilus Th221 from soy sauce moromi grown in high-salt medium[J].Int J Food Microbiol,2008,121:245-252.
[39]杨洁彬.乳酸菌-生物学基础及应用[M].北京:中国轻工业出版社,1996:203-204.
[40]Gu SA,Jun C,Joo J C,et al.Higher thermostability of L-lactate dehydrogenases is a key factor indecreasing the optical purity of D-lactic acid produced from Lactobacillus coryniformis [J].Enzyme Microb Tech,2014,58-59:29-35.
[41]杨登峰,潘丽霞,关妮,等.产高纯度L-乳酸大肠杆菌基因工程菌的初步研究[J].现代食品科技,2010,26:126-128.
[42]Zhou S,Shanmugam K T,Ingram L O.Functional replacement of the Escherichia coli D-(-)-lactate dehydrogenase gene (ldhA)with the L-(+)-lactate dehydrogenase gene(ldhL)from Pediococcus acidilactici[J].Appl Environ Microbiol,2003,69:2237-2244.
[43]Okano K,Zhang Q,Shinkawa S,et al.Efficient production of optically pure D-Lactic acid from raw corn starch by using a genetically modified L-Lactate dehydrogenase gene-deficient and α-amylase-secreting Lactobacillus plantarum strain[J].App Environ Microb,2009,75:462-467.
[44]Doo E H,Kim H H,Oh J Y,et al.Mutants of Lactobacillus coryniformis producing d-lactic acid with high concentration,high yield,and high purity and their uses(for production of d-lactic acid)[J].Republic of Korea Patent,2013,KR2013001780.
[45]Zou H B,Wu Z Q,Xian M,et al.Not only osmoprotectant:Betaine increased lactate dehydrogenase activity andL-lactate production in lactobacilli[J].Bioresour Technol,2013,148:591-595.
[46]Collins F M,Lascelles J.The effect of growth conditions on oxidative and dehydrogenase activity in Staphylococcus aureus [J].J Gen Microbiol,1962,29:531-535.
[47]Thomas T D,Turner K W,Crow V L.Galactose fermentation by Streptococcus lactis and Streptococcus cremoris:pathways,products,and regulation[J].J Bacteriol,1980,144:672-682.
The research progress of the lactate dehydrogenases in lactic acid bacteria
HUANG Yan-na1,XU Guang-tao2,YOU Chun-ping1,*
(1.State Key Laboratory of Dairy Biotechnology,Dairy Research Institute,Bright Dairy&Food Co.,Ltd.,Shanghai 200436,China;2.Handan Steel&Iron Group Staff Hospital,Handan 056001,China)
The lactate dehydrogenase was a key enzyme which converts pyruvic acid to lactic acid in lactic acid bacteria.In this paper,the research progresses of the classification,structures and functions of the lactate dehydrogenases,as well as the microbial genuses and synthetic metabolic pathway of the lactic acid production were summarized.This review will assist in understanding the metabolic regulation of lactic acid synthesis in many different species and genera,hoping for theoretical guidance for others.
lactic acid bacteria;L-lactate dehydrogenase;D-lactate dehydrogenase;classification;structure and function
TS201.1
A
1002-0306(2016)08-0369-05
10.13386/j.issn1002-0306.2016.08.069
2015-09-08
黄艳娜(1980-),女,博士,工程师,研究方向:益生菌代谢工程及代谢调控,E-mail:huangyanna@brightdairy.com。
*通讯作者:游春苹(1981-),女,博士,高级工程师,研究方向:益生菌功能研究,E-mail:youchunping@brightdairy.com。
国家“十二五”科技支撑计划(2013BAD18B01,2012BAD12B08)。
