Bt Cry1类毒素共性结构域的分析、表达及鉴定
2016-09-09刘贝贝谢雅晶焦凌霞张存政赵岩岩武爱华刘贤金
刘贝贝,张 霄,谢雅晶,焦凌霞,刘 媛,张存政,赵岩岩,武爱华,刘贤金
(1南京农业大学植物保护学院,南京 210095;2江苏省农业科学院食品质量安全与检测研究所/江苏省食品质量安全重点实验室-省部共建国家重点实验室培育基地/农业部农产品量安全控制技术与标准重点实验室,南京 210014;3河南科技学院食品学院,河南新乡 453003)
Bt Cry1类毒素共性结构域的分析、表达及鉴定
刘贝贝1,2,张 霄2,谢雅晶2,焦凌霞3,刘 媛1,2,张存政2,赵岩岩2,武爱华2,刘贤金1,2
(1南京农业大学植物保护学院,南京 210095;2江苏省农业科学院食品质量安全与检测研究所/江苏省食品质量安全重点实验室-省部共建国家重点实验室培育基地/农业部农产品量安全控制技术与标准重点实验室,南京 210014;3河南科技学院食品学院,河南新乡 453003)
【目的】分析定位Bt Cry1类毒素Cry1Ab、Cry1Ac、Cry1B、Cry1C、Cry1F的共性结构域,克隆并表达共性结构域蛋白,为筛选Bt毒素广谱抗体及建立广谱检测方法打下基础。【方法】利用生物信息学和分子模拟技术,通过SWISS-MODEL同源建模分别对5种Cry1类毒素进行三维建模,并结合Ramachandran plot、ERRAT和Verify3D方法评价模型构象的合理性。通过分析比对5种Cry1类毒素的三维结构,确定Domain I 区域作为5种Cry1毒素的共性结构域。以含Cry1Ac基因的苏云金芽孢杆菌库斯塔克亚种为模板设计引物,PCR扩增获得共性结构域Domain I基因,将其经Nco I和Not I双酶切连接至原核表达载体pET-26b(+),构建原核表达载体pET-26b-Domain I。重组质粒经菌液PCR、双酶切以及测序鉴定验证正确后,转化至E.coli BL21 (DE3),经终浓度为1 mmol·L-1的IPTG在20℃下诱导表达16 h后检测共性结构域蛋白的表达情况。离心收集诱导表达的大肠杆菌菌液,进行超声波破碎处理,收集上清及沉淀,采用 SDS-PAGE 分析融合蛋白的表达。利用His-Trap HP镍亲和柱纯化上清中的可溶性融合蛋白,经SDS-PAGE电泳、Western blot和ELISA试验验证纯化的共性结构域蛋白的生物活性。【结果】基于氨基酸序列及三维空间比对分析,发现 5种 Cry1类毒素的Domain I 的序列一致性最高,而且它们的DomainⅠ三维结构几乎完全重合,确定Domain I 区域作为5种Cry1毒素的共性结构域;通过PCR、双酶切及测序鉴定成功构建原核表达载体pET-26b-Domain I,经IPTG诱导表达、His-Trap HP镍亲和柱纯化获得了可溶性的Domain I共性结构域蛋白;SDS-PAGE和Western blot证实表达的共性结构域蛋白的分子量约为33.4 kD,且能与抗His标签鼠单克隆抗体发生特异性反应;ELISA试验证实共性结构域蛋白与5种Cry1类毒素特异性抗体均具有很强的结合能力,抗原表位分析结果显示共性结构域蛋白具有和完整的 Cry蛋白存在多个潜在抗原表位位点的特征,抗原表位区域所占的比例分别为 48.4%和63.6%,表明共性结构域蛋白具有良好的免疫原性和免疫反应性。【结论】基于分子模拟与分子克隆技术,成功定位及表达纯化获得共性结构域蛋白,为下一步利用共性结构域为靶标分子制备广谱特异性识别Cry1类毒素抗体打下基础。
Bt Cry1类毒素;共性结构域;原核表达;纯化
0 引言
【研究意义】随着转Bt Cry基因在抗虫性作物中大面积的推广及应用,在给人类带来极其显著的经济、社会和生态效益的同时,其应用存在的生态安全风险以及对人类和其他非靶标生物的安全隐患也备受关注[1-3],越来越多的国家针对转基因产品建立了标识系统[4],因此对 Cry毒素表达产物安全筛查检测就尤显迫切。目前报道对转基因产品中毒素蛋白的检测方法和市场应用的ELISA试剂盒主要是单一毒素的抗体检测[5-7],而且不同的毒素需要制备相应抗体,周期长、成本高,难以满足实际应用时一种抗体检测多种毒素的需求。因此,凭借Bt Cry毒素三维结构具有较高相似性的特点,开发基于共性结构的Bt Cry毒素广谱特异性检测技术是一项非常有应用价值的工作。【前人研究进展】目前,针对小分子有害物质多残留免疫分析技术主要利用小分子的共性结构作为半抗原制备广谱特异性抗体用于检测[8-10]。研究发现尽管Cry毒素的氨基酸序列相似性很低,但是它们的三维结构均非常相似,都是由3个结构域组成,且存在大量的保守区域[11]。赵新民等[12]在对Cry1Aa、Cry2Aa、Cry3Aa 和Cry4Aa 4类具有代表性毒素三维结构的预测和对比分析中发现它们的三级结构模型非常相似,其中DomainⅠ几乎完全相同,结构的相似性高于其序列的相似性;吴洪福等[13]对已解析的Bt晶体蛋白的三维结构进行了比较分析,发现它们都有类似的结构,而且单独比较各种Cry蛋白的DomainⅠ,发现它们的DomainⅠ差别最小。而针对多种Cry毒素的广谱检测目前还未见报道。目前,美国Envirologix 公司生产的Cry1Ab/Cry1Ac平板试剂盒,能够同时检测Cry1Ab/Cry1Ac两种毒素[6,14],这是基于它们的三维结构具有高度相似性的特点,也为后续研究提供了依据。【本研究切入点】基于Cry毒素具有共性结构域,以及抗体对共性结构域存在很强的交叉反应[15-17],分析定位Cry1类毒素的共性结构域,通过对其成功表达及生物活性验证,制备广谱识别Cry1类毒素的抗体,应用于Cry毒素的广谱检测。【拟解决的关键问题】基于序列及空间比对分析,定位共性结构域;表达纯化共性结构域蛋白,并验证其生物活性,为下一步制备广谱抗体打下基础。
1 材料与方法
试验于2014年5月至2015年11月在江苏省农业科学院食品质量安全与检测研究所完成。
1.1 材料
1.1.1 菌株及质粒 苏云金芽孢杆菌库斯塔克亚种(Bacillus thuringiensis ssp. kurstaki 2671)为徐健博士提供(江苏里下河地区农业科学研究所);表达载体pET-26b(+)购于Novagen公司;E. coli Trans10、E. coli BL21(DE3)感受态细胞购自北京全式金生物技术有限公司。
1.1.2 试验材料与试剂 引物合成由上海生工生物工程有限公司完成;PCR产物回收试剂盒购自Promega公司;质粒提取试剂盒购自 Axygen公司;Taq DNA聚合酶、Nco I、Not I内切酶、T4 DNA连接酶购自美国NEB公司;His-Trap HP纯化柱购自GE Healthcare公司;其他试剂均为分析纯级试剂。
1.2 方法
1.2.1 序列比对、同源建模及模型评价 从 NCBI数据库检索5种Cry1类毒素(Cry1Ab、Cry1Ac、Cry1B、Cry1C、Cry1F)的氨基酸序列(登录号分别为ALJ10947.1、KF148682.1、ABQ23438.1、BAM67040.1、ACD50893.1),进行多序列比对分析。然后分别以5 种 Cry1类毒素的 Domain I作为目标蛋白序列,用BLAST中的 blastp suite对 PDB蛋白结构数据库(http://blast.ncbi.nlm.nih.gov/Blast.cgi)分别进行多序列同源性搜索,取同源性最高的晶体结构作为同源建模的模板。其中 Cry1Ac已经获得晶体结构模型[18-19],无需建模。利用Swiss-Model(http://swissmodel. expasy.org/)蛋白质结构同源建模服务器[20-21],分别预测了4种Cry1类毒素(Cry1Ab、Cry1B、Cry1C、Cry1F)的 Domain I三维结构,模建结构采用PROCHECK[22]、ERRAT[23]以及Verify3D[24]进行评估,检测其合理性。
1.2.2 共性结构域基因克隆 通过对5种Cry1类毒素(Cry1Ab、Cry1Ac、Cry1B、Cry1C、Cry1F)DomainⅠ的核苷酸序列及三维结构比对,以其中任意一种毒素的DomainⅠ扩增表达共性结构域,是可行的。因此以笔者实验室现有的含 Cry1Ac的苏云金芽孢杆菌库斯塔克亚种为模板来设计其DomainⅠ引物。设计引物时在DomainⅠ氨基酸序列C端加长6个氨基酸,并插入限制性内切酶切位点,以方便后续试验中原核表达载体的构建以及共性结构域蛋白表达、纯化研究。上游引物为5′-CATGC/CATGGATAACAATCCGAA CAT-3′,下划线部分为NcoⅠ酶切位点;下游引物为5′-ATAAGAATG/CGGCCGCGGATCCTCGAATTGG ATATCTTC-3′,下划线部分为NotⅠ酶切位点。PCR反应条件为95℃预变性5 min;94℃变性1 min,52℃退火1 min,72℃延伸1 min,共30个循环;最后72℃终延伸10 min,4℃保存。扩增产物经1.0%的琼脂糖凝胶电泳验证后,PCR产物回收试剂盒回收、纯化目的片段。
1.2.3 pET-26b-DomainⅠ重组质粒的构建 DomainⅠ基因PCR回收、纯化产物和表达载体pET-26b同时进行NcoⅠ和NotⅠ双酶切处理,纯化回收后,T4 DNA连接酶连接过夜,连接产物转化E. coli Trans10感受态细胞。转化的菌液涂布于 LB-Kan(含 50 µg·mL-1Kanamycin)平板上,挑取阳性转化子培养后提取质粒,对质粒进行酶切、PCR扩增鉴定及序列测定(由上海生物工程公司完成)。将测序结果提交到GenBank进行序列同源性比对。
1.2.4 DomainⅠ共性结构域的原核表达及纯化 通过测序及双酶切验证后,将pET-26b-DomainⅠ重组质粒转化至E. coli BL21(DE3),筛选阳性菌株。挑取阳性克隆,接种于含50 µg·mL-1卡那霉素的LB培养基中过夜培养,次日按 1%比例转接于含 50 µg·mL-1Kanamycin的LB液体培养基中,37℃,250 r/min振荡培养至 OD600约为 0.6—0.8,加入终浓度为 1 mmol·L-1IPTG,20℃诱导表达16 h,以空载菌株为对照。4℃ 10 000×g离心15 min,收集菌体称量湿重,以100 mg·mL-1比例用磷酸缓冲液(PBS,pH 7.4)重悬菌体,加入溶菌酶(终浓度1 mg·mL-1),PMSF(100 mmol·L-1)混匀后,于冰浴中超声破碎菌体,12 000 ×g离心收集上清液,SDS-PAGE分析目的蛋白的表达情况。
将收集的 DomainⅠ上清液上样于经 Binding Buffer A(20 mmol·L-1磷酸盐,500 mmol·L-1NaCl,10 mmol·L-1咪唑,pH 7.4)预平衡的HisTrap HP 1 mL预装柱中,反复上样,用5—10倍柱体积Binding Buffer A洗去未与柱子结合的蛋白,再用分别含20、50、100、200、300、400、500 mmol·L-1咪唑的Elution Buffer B(其他成分同Binding Buffer A)洗涤柱子,收集各个梯度的洗脱液,SDS-PAGE分析蛋白纯化效果。
1.2.5 Western blot和ELISA试验鉴定 纯化的共性结构域蛋白经SDS-PAGE电泳分离后,用湿转法将目的蛋白转移至PVDF膜上,5% MPBS(含5%脱脂奶粉的PBS)室温封闭过夜,洗膜后,加入HRP标记的抗His标签鼠单克隆抗体(1∶5 000)作为二抗,室温孵育2 h,洗膜后用DAB底物显色试剂盒显色,至目的条带清晰终止反应。
分别取100 µL 4 µg·mL-1的纯化的共性结构域蛋白和5种Cry1类毒素(Cry1Ab、Cry1Ac、Cry1B、Cry1C、Cry1F)的标准蛋白包被于96孔酶标板上,同时设置阴性对照组,4℃包被过夜。次日用洗涤缓冲液PBST(含0.05% Tween 20的PBS)洗板3次,每次3 min。每孔加入200 µL 3% MPBS封闭,37℃孵育2 h后洗涤,然后每孔分别对应加入100 µL 1∶1 000稀释的纯化的抗Cry1Ab、Cry1Ac、Cry1B、Cry1C、Cry1F多克隆抗体,37℃孵育2 h后洗涤,再加入1∶5 000稀释的辣根过氧化物酶(horseradish peroxidase,HRP)标记的山羊抗兔IgG,37℃孵育1 h。PBST洗涤4—5次后加TMB显色15 min,每孔加50 μL的2 mol·L-1H2SO4终止反应后于450 nm 测OD值。
2 结果
2.1 序列比对、同源建模及模型评价
5种 Cry1类毒素(Cry1Ab、Cry1Ac、Cry1B、Cry1C、Cry1F)的3个结构域的氨基酸序列比对如图1-A,可以看出Domain I 的序列一致性最高达到71%。分别以Cry1Ab、Cry1B、Cry1C和Cry1F的DomainⅠ为目标蛋白序列,对PDB蛋白结构数据库分别进行序列同源性搜索,结果显示Cry1Ab、Cry1C和Cry1F 的 Domain I同源性最高的模版均是Cry1Ac(PDB编号为4arx.A),而Cry1B的 Domain I选定的模板为Cry3Bb1(PDB编号为1ji6.A)。
经Swiss-Model构建的4种毒素Domain I三维结构模型,通过Ramachandran plot(图1-B)、ERRAT 和Verify3D(表1)评价构象合理性,从结果可以看出所构建的4种DomainⅠ结构模型是合理可信的,可以用于后续研究。对5种Cry1类毒素的DomainⅠ三维结构模型进行重合比对(图 1-C),发现它们的三维结构几乎完全重合,因此以任意的DomainⅠ扩增表达共性结构域是可行的。
2.2 共性结构域基因的克隆与序列分析


图1 5种Cry1类毒素氨基酸序列多重比对(A)、三维模型的拉氏图分析(B)及三维结构比对(C)Fig. 1 Multiple alignment of amino acid sequences (A), Ranmachandan plot (B) and comparison of the tertiary structures (C) of five toxins
以徐健博士提供的 Bt菌株提取的质粒为模板,PCR扩增获得了大小约为 800 bp的扩增条带(图2-A)。扩增出的条带与预期大小一致。然后将纯化后的PCR产物连接至pET-26b载体中并进行测序,序列分析结果表明,该基因编码区序列为880 bp,编码293个氨基酸。经NCBI网站Blastp搜索比对后发现与Cry1Ac Domain I序列一致性为100%,序列比对得到蛋白的系统进化树(图2-B)。
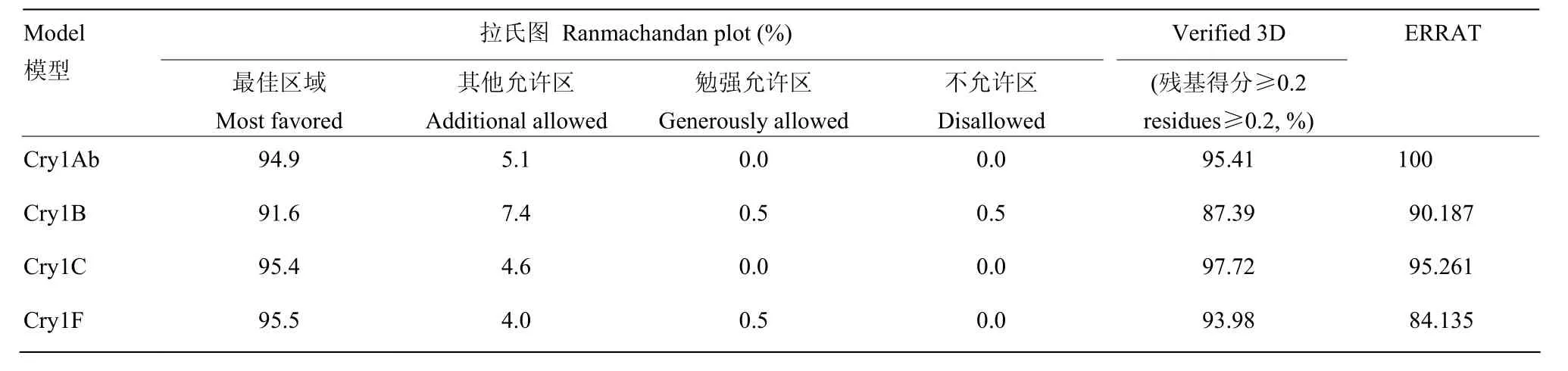
表1 模型评价Table 1 Evaluation of model structures

图2 DomainⅠ基因的PCR扩增(A)及其序列同源性分析(B)Fig. 2 PCR amplification (A) and sequence homologous analysis (B) of DomainⅠ
2.3 pET-26b-DomainⅠ表达载体的构建及鉴定
通过 pET-26b-DomainⅠ化转至 E. coli BL21 (DE3)以后,挑取阳性单菌落进一步PCR扩增、酶切鉴定。PCR电泳结果(图3-A)显示,阳性菌落扩增片段大小与预计大小(约1 039 bp)一致。提取阳性菌落的重组质粒,经NcoⅠ和NotⅠ酶切得到800 bp左右的目的片段(图3-B),说明DomainⅠ的表达载体pET-26b-DomainⅠ构建成功。
2.4 共性结构域蛋白的表达与纯化
通过测序及双酶切验证后,将阳性克隆在20℃培养 16 h条件下表达,离心分别收集上清和菌体,加PBS重悬菌体后冰上超声破碎菌体,分别收集上清和沉淀。经SDS-PAGE电泳发现,破碎上清在33.4 kD处出现明显的特异性蛋白条带,与理论分子量大小一致(图4-A)。将诱导的Domain I上清经HisTrap HP预装柱梯度洗脱纯化后,进行SDS-PAGE分析,结果见图4-B,经梯度洗脱后,最终Domain I(约33.4 kD)在咪唑浓度为300 mmol·L-1时被洗脱下来,纯化的目的蛋白条带单一。
2.5 Western blot、ELISA和抗原性分析
Western blot结果证实,纯化的 Domain I(含6×His标签)对抗His标签鼠单克隆抗体有免疫反应,且蛋白分子量大小在33.4 kD左右,同预期结果一致(图5-A)。ELISA结果发现纯化的Domain I分别与5种Cry1类毒素(Cry1Ab、Cry1Ac、Cry1B、Cry1C、Cry1F)特异性抗体均具有很强的结合能力(图5-B)。通过Jameson-Wolf法分别预测Cry1Ac和DomainⅠ氨基酸的抗原性指数(图5-C),结果显示DomainⅠ蛋白和 Cry1Ac蛋白均存在多个潜在的抗原表位位点,抗原表位区域所占的比例分别为48.4%和63.6%。说明Domain I区域存在较强的免疫原性,能够在在免疫过程中产生针对Domain I的特异性抗体。

图3 pET-26b-Domain I 重组质粒菌液PCR(A)及双酶切(B)验证Fig. 3 PCR amplification (A) and restricted enzymes digestion (B) of pET-26b-Domain I plasmid
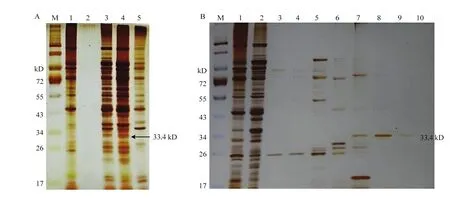
图4 共性结构域蛋白表达与纯化的SDS-PAGE分析Fig. 4 SDS-PAGE analysis of the common structural domain expression and purification
3 讨论
本研究对5种Cry1类毒素(Cry1Ab、Cry1Ac、Cry1B、Cry1C、Cry1F)的氨基酸全序列进行了比对,发现它们的 DomainⅠ序列 Identity最高。利用Swiss-Model对DomainⅠ进行同源建模分析,发现5种毒素的DomainⅠ三维结构几乎完全重合。赵新民等[12]对Cry1Aa、Cry2Aa、Cry3Aa和Cry4Aa 4类具有代表性的毒素结构进行了系统比较,发现它们的三级结构模型非常相似,而且所有的3个结构域中结构域I中的 α螺旋几乎完全重叠,结构差异最小,只有结构域 II、III有细微差别;吴洪福等[13]对已解析的 Bt晶体蛋白的三维结构进行了比较分析,发现它们都有类似的结构,而且单独比较各种Cry蛋白的结构域Ⅰ,发现它们的结构域Ⅰ差别最小。本研究结果与前人的研究结论完全一致,这也是笔者选取DomainⅠ作为共性结构域的依据。
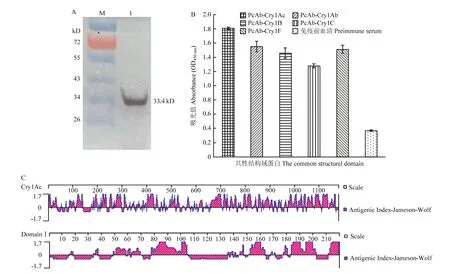
图5 共性结构域蛋白的Western blot(A)、ELISA(B)和抗原性(C)分析Fig. 5 Western blot (A), ELISA (B) and antigenicity (C) analysis of the common structural domain protein
目前,关于DomainⅠ单独表达纯化也有文献报道[25],本文在前人研究的基础上对共性结构域DomainⅠ进行表达,但前期纯化及 ELISA、Western blot验证均没有结果,分析原因可能是目的蛋白在折叠时由于空间位阻使His标签没有充分暴露。根据融合蛋白连接肽的设计原则[26-27],笔者对DomainⅠ蛋白与His标签蛋白之间的连接肽长度进行适当优化,使其在优化前的5个氨基酸基础之上增加至11个氨基酸,使目的蛋白在正常折叠的同时,His标签也能充分暴露在其C端,最终纯化得到共性结构域蛋白。
抗原表位是蛋白质抗原性的基础,在蛋白质二级结构分析中,α螺旋、β折叠的结构规则不易形变,多位于蛋白质内部形成构象表位,不易与配体结合,一般不作为抗原表位,而β转角结构和无规则卷曲比较松散,易形成线性表位,有利于抗体结合,符合作为抗原表位的特性[28-29]。本研究中的共性结构域蛋白主要是一个由7个α螺旋组成的螺旋束,但从其三维结构观察发现也具有作为连接链的无规则卷曲(图1-C),通过ELISA试验也证明它具有与抗Cry毒素抗体特异结合的能力,说明其共性结构域蛋白中存在的无规则卷曲可以形成抗原表位,使其具有免疫原性和免疫反应性。此研究结果为下一步利用共性结构域蛋白制备其单、多克隆抗体或者通过时菌体展示技术制备广谱抗体进行多种Cry毒素筛选提供理论基础。
4 结论
分析并定位了共性结构域,克隆、表达及纯化共性结构域蛋白;证实DomainⅠ区域存在抗原表位并具有很强的免疫原性及反应性;经Western blot和ELISA鉴定了共性结构域蛋白的生物活性。试验结果可为Cry1类毒素共性结构域广谱抗体的制备及建立多种Cry毒素广谱筛选提供理论依据及技术指导。
References
[1] HAMMOND B G, KOCH M S. A review of the food safety of Btcrops//SANSINENEA E. Bacillus thuringiensis Biotechnology, Springer,2012: 305-325.
[2] KAUR S. Risk assessment of Bt transgenic crops//SANSINENEA E. Bacillus thuringiensis Biotechnology, Springer, 2012: 41-85.
[3] YU H L, LI Y H, WU K M. Risk assessment and ecological effects of transgenic Bacillus thuringiensis crops on non-target organisms. Journal of Integrative Plant Biology, 2011, 53(7): 520-538.
[4] 连丽君, 王雷, 张可炜. 转基因食品安全性的争论与事实. 食品与药品, 2006, 8(11): 12-15. LIAN L J, WANG L, ZHANG K W. Debate and fact on safety of genetically modified foods. Food and Drug, 2006, 8(11): 12-15. (in Chinese)
[5] GUERTLER P, PAUL V, ALBRECHT C, MEYER H D. Sensitive and highly specific quantitative real-time PCR and ELISA for recording a potential transfer of novel DNA and Cry1Ab protein from feed into bovine milk. Analytical and Bioanalytical Chemistry, 2009, 393(6/7):1629-1638.
[6] SAXENA D, FLORES S, STOTZKY G. Transgenic plants:Insecticidal toxin in root exudates from Bt corn. Nature, 1999,402(6761): 480-481.
[7] 王广印, 范文秀, 陈碧华, 张建伟, 韩世栋. 转基因食品检测技术的应用与发展 I. 主要检测技术及其特点. 食品科学, 2008, 29(10):698-705. WANG G Y, FAN W X, CHEN B H, ZHANG J W, HAN S D. Application and development of detection technology of genetically modified foods (GMFs)Ⅰ. main detection technologies of GMFs and their characteristics. Food Science, 2008, 29(10): 698-705. (in Chinese)
[8] SPINKS C A. Broad-specificity immunoassay of low molecular weight food contaminants: new paths to Utopia! Trends in Food Science & Technology, 2000, 11(6): 210-217.
[9] 刘媛, 余向阳, 梁颖, 祝金凤, 李顺玲, 张存政, 刘贤进. 农药广谱特异性抗体制备技术研究进展. 江苏农业学报, 2009, 25(2):428-432. LIU Y, YU X Y, LIANG Y, ZHU J F, LI S L, ZHANG C Z, LIU X J. Recent advances in development of broad specificity antibodies for pesticides. Jiangsu Journal of Agricultural Sciences, 2009, 25(2):428-432. (in Chinese)
[10] LI Y L, ZHAO F C, ZHAO L Y, YANG Z Y. Development of a broad-specificity immunoassay for determination of organophosphorus pesticides using dual-generic hapten antigens. Food Analytical Methods, 2015, 8(2): 420-427.
[11] PARDO-LOPEZ L, SOBERON M, BRAVO A. Bacillus thuringiensis insecticidal three-domain Cry toxins: mode of action, insect resistance and consequences for crop protection. FEMS Microbiology Review,2013, 37(1): 3-22.
[12] 赵新民, 夏立秋, 王发祥, 丁学知, 单世平, 张友明. 苏云金芽孢杆菌毒素Cry1Aa, Cry2Aa, Cry3Aa和Cry4Aa结构的计算机对比分析. 化学学报, 2008, 66(1): 108-111. ZHAO X M, XIA L Q, WANG F X, DING X Z, SHAN S P, ZHANG Y M. Comparison and analysis of Cry1Aa, Cry2Aa, Cry3Aa and Cry4Aa of Bacillus thuringiensis toxins with computer. Acta Chimica Sinica, 2008, 66(1): 108-111. (in Chinese)
[13] 吴洪福, 郭淑元, 李海涛, 高继国. 苏云金芽孢杆菌杀虫晶体蛋白结构和功能研究进展. 东北农业大学学报, 2009, 40(2): 118-122. WU H F, GUO S Y, LI H T, GAO J G. Progress on structure and function of insecticidal crystal proteins from Bacillus thuringiensis. Journal of Northeast Agricultural University, 2009, 40(2): 118-122. (in Chinese)
[14] 谢小波, 舒庆尧. 用 Envirologix Cry1Ab/Cry1Ac试剂盒快速测定转基因水稻Bt杀虫蛋白含量的研究. 中国农业科学, 2001, 34(5):465-468. XIE X B, SHU Q Y. Studies on rapid quantitative analysis of Bt toxin by using envirologix kits in transgenic rice. Scientia Agricultura Sinica, 2001, 34(5): 465-468. (in Chinese)
[15] ETESHOLA E. Isolation of scFv fragments specific for monokine induced by interferon-gamma (MIG) using phage display. Journal of Immunology Methods, 2010, 358(1/2): 104-110.
[16] RODA A, MIRASOLI M, GUARDIGLI M, MICHELINI E, SIMONI P, MAGLIULO M. Development and validation of a sensitive and fast chemiluminescent enzyme immunoassay for the detection of genetically modified maize. Analytical and Bioanalytical Chemistry,2006, 384(6): 1269-1275.
[17] ZHANG X, LIU Y, ZHANG C Z, WANG Y, XU C X, LIU X J. Rapid isolation of single-chain antibodies from a human synthetic phage display library for detection of Bacillus thuringiensis (Bt) Cry1B toxin. Ecotoxicology and Environmental Safety, 2012, 81: 84-90.
[18] GROCHULSKI P, MASSON L, BORISOVA S, PUSZTAI-CAREY M,SCHWARTZ J L, BROUSSEAU R, CYGLER M. Bacillus thuringiensis CrylA (a) insecticidal toxin: crystal structure and channel formation. Journal of Molecular Biology, 1995, 254(3):447-464.
[19] 刘卓明, 谢柳, 叶大维. 苏云金芽孢杆菌 Cry1Ac杀虫晶体蛋白及其分子设计. 基因组学与应用生物学, 2009, 28(2): 356-364. LIU Z M, XIE L, YE D W. Bt Cry1Ac insecticidal crystal protein family and its molecular design. Genomics and Applied Biology, 2009,28(2): 356-364. (in Chinese)
[20] ZHAO X M, ZHOU P D, XIA L Q. Homology modeling of mosquitocidal Cry30Ca2 of Bacillus thuringiensis and its molecular docking with N-acetylgalactosamine. Biomed Environmental Science,2012, 25(5): 590-596.
[21] SCHWEDE T, KOPP P, GUEX N, PEITSCH M C. SWISS-MODEL:an automated protein homology-modeling server. Nucleic Acids Research, 2003, 31(13): 3381-3385.
[22] LASKOWSKI R A, MACARTHUR M W, MOSS D S, THORNTON J M. PROCHECK: a program to check the stereochemical quality of protein structures. Journal of Applied Crystallography, 1993, 26(2):283-291.
[23] COLOVOS C, YEATES T O. Verification of protein structures:patterns of nonbonded atomic interactions. Protein Science, 1993, 2(9):1511-1519.
[24] EISENBERG D, LOTHY R, BOWIE J U. VERIFY3D: Assessment of protein models with three dimensional profiles. Methods in Enzymology, 1997, 277: 396-404.
[25] GUO S Y, LI J, CHEN Z, HE K L. Penetration of a single domain of Bacillus thuringiensis Cry1Ie-domain I to a lipid membrane in vitro. Journal of Integrative Agriculture, 2014, 13(5): 1043-1050.
[26] GEORGE R A, HERINGA J. An analysis of protein domain linkers:their classification and role in protein folding. Protein Engineering Design and Selection, 2003, 15(11): 871-879.
[27] 闫璐颖, 陈建华, 张新国. 融合蛋白连接肽的研究进展. 生物技术,2008, 18(3): 92-94. YAN L Y, CHEN J H, ZHANG X G. Research progress in the linker of fusion protein. Biotechnology, 2008, 18(3): 92-94. (in Chinese)
[28] BARLOW D J, EDWARDS M S, THORNTON J M. Continuous and discontinuous protein antigenic determinants. Nature, 1986, 322(6081):747-748.
[29] JAMESON B A, WOLF H. The antigenic index: a novel algorithm for predicting antigenic determinants. Bioinformatics, 1988, 4(1):181-186.
(责任编辑 岳梅)
Analysis, Expression and Identification of the Common Structural Domain of Bacillus thuringiensis (Bt) Cry1 Toxins
LIU Bei-bei1,2, ZHANG Xiao2, XIE Ya-jing2, JIAO Ling-xia3, LIU Yuan1,2, ZHANG Cun-zheng2,ZHAO Yan-yan2, WU Ai-hua2, LIU Xian-jin1,2
(1College of Plant Protection, Nanjing Agricultural University, Nanjing 210095;2Institute of Food Quality Safety and Detection Research, Jiangsu Academy of Agricultural Science/Key Laboratory of Food Quality and Jiangsu Province-State Key Laboratory Breeding Base/Key Laboratory of Control Technology and Standard for Agro-product Safety and Quality, Ministry of Agriculture,
Nanjing 210014;3School of Food Science, Henan Institute of Science and Technology, Xinxiang 453003, Henan)
【Objective】The objective of this study is to express the optimal common structural domain through analyzing and locating the common structure of five Bacillus thuringiensis Cry1 toxins. This research will lay a foundation for producing the generic antibody and developing the detection method for Cry1 toxins. 【Method】Through bioinformatics, molecular simulation technique and homology modeling to build the three-dimensional structure models of five Cry1 toxins. The structures were evaluated using three programmes, Ramchandran plot, Verify3D and ERRAT. Domain I was identified as the common structure domain of five Cry1 toxins finally. In order to construct the pET-26b-Domain I vector, primers were designed according to the Cry1Ac gene of Bacillus thuringiensis ssp. kurstaki. As well as insert with Nco I and Not I digestion sites. When it was identified by PCR, restriction enzyme digestion and sequencing, the recombinant plasmid was transformed into E. coli BL21 (DE3) which was induced with 1 mmol·L-1IPTG, 20for 16 h. The supernatant and precipitate were collected and verified by SDS-PAGE after E. coli BL21 (DE3)cells were collected and crushed by ultrasonic wave. The soluble fusion protein was purified by His-Trap HP nickel affinity column and verified by SDS-PAGE, Western blot and ELISA. 【Result】 According to the analysis of amino acid sequences and threedimensional structures of the five Cry1 toxins, the sequences of Domain I were the highest identity part and its three-dimensional structure was very similar and then the Domain I was chosen as the common structure domain of the five Cry1 toxins. The expression vector pET-26b-Domain I was constructed successfully, and soluble Domain I protein was expressed and purified. The molecular weight of the fusion protein was confirmed to be 33.4 kD by SDS-PAGE and Western blot, which also showed specific activity to anti-6×His monoclonal antibody. The ELISA assay showed that the Domain I protein had a good sensitivity with the specific antibodies of the five Cry1 toxins, and the epitope prediction results showed that both the Domain I protein and the complete Cry protein existed multiple potential epitopes, and the percentage of their antigenic peptides were 48.4% and 63.6%, respectively. These results indicate that the Domain I protein has good immunogenicity and immune response. 【Conclusion】Based on molecular simulation and molecular cloning technology, the conserved structural domain protein was successfully expressed and purified. This study has established a foundation for the following studies and the common structural domain protein would as target molecule for the production of the generic antibody of Cry1 toxins in further research.
Bacillus thuringiensis Cry1 toxins; common structural domain; prokaryotic expression; purification
2016-03-18;接受日期:2016-04-25
国家自然科学基金(31371778、31301703)、国家公益性行业(农业)科研专项(201303088)、江苏省自主创新项目(CX(14)5068)
联系方式:刘贝贝,E-mail:2013102120@njau.edu.cn。通信作者刘贤金,E-mail:jaasliu@jaas.ac.cn
