巴马香猪周身皮肤厚度的测量及其与7号染色体候选SNPs位点的关联分析
2016-09-09黄晓畅邱恒清严国荣黄贻忠张弋峰江嘉程周李生麻骏武肖石军黄路生艾华水
黄 涛,黄晓畅,邱恒清,严国荣,黄贻忠,张弋峰,江嘉程,周李生,任 军,麻骏武,肖石军,黄路生,杨 斌,艾华水
(江西农业大学省部共建猪遗传改良与养殖技术国家重点实验室,南昌 330045)
巴马香猪周身皮肤厚度的测量及其与7号染色体候选SNPs位点的关联分析
黄 涛,黄晓畅,邱恒清,严国荣,黄贻忠,张弋峰,江嘉程,周李生,任 军,麻骏武,肖石军,黄路生,杨 斌,艾华水
(江西农业大学省部共建猪遗传改良与养殖技术国家重点实验室,南昌 330045)
【目的】 测定中国地方小型猪品种巴马香猪成年时周身9个典型部位的皮肤厚度,揭示巴马香猪不同部位皮肤厚度变化规律;进行9个部位皮肤厚度与候选SNPs位点的关联分析,在巴马香猪群体中验证影响皮肤厚度的7号染色体主效QTL,为进一步在巴马香猪群体中大规模开展皮肤厚度等形态变化的分子遗传控制机理及其相关基因功能研究奠定基础,从而增强人们对猪皮肤的认知。【方法】从一个由319头300日龄巴马香猪组成的成年屠宰群体中,随机选取50头,包括27头母猪和23头阉割公猪,分别取头脸、肩、背、肷、臀、胸、下腹、腋下和管等9个部位的皮肤,利用电子游标卡尺对这些不同部位皮肤厚度进行精确测量,利用R语言基本统计包进行不同部位和不同性别间皮肤厚度的差异分析以及不同部位间皮肤厚度的相关分析。在猪7号染色体34.5—36.2 Mb的区域选取46个SNPs位点,利用MassARRAY时间飞行质谱技术进行基因分型,结合上述测定的皮厚表型,利用广义混合线性模型及 R 语言SNPassoc软件包进行目标候选区域的关联分析。根据关联分析结果和基因的生物学功能确定可能的位置候选基因。【结果】(1)单因素方差分析表明巴马香猪9个部位的皮肤厚度存在极显著差异(P = 2.95×10-117),肷部和背部的皮肤最厚,分别为(5.15 ± 0.92)和(4.97 ± 0.85)mm,下腹和腋下的皮肤最薄,分别为(1.77 ± 0.36)和(1.97 ± 0.68) mm。皮肤厚度从厚到薄依次是肷部、背部、肩部、头脸、臀部、管部、胸部、腋下和下腹。(2)阉割公猪腋下皮肤厚度显著小于母猪的(P = 0.021),其它部位皮肤厚度在母猪和阉割公猪间差异均不显著。(3)除了下腹皮肤厚度与背部、肩部、头脸部的不相关(P>0.05)外,巴马香猪不同部位两两之间皮肤厚度均呈现不同程度地显著或极显著正相关。(4)关联分析结果表明,9个不同部位的皮肤厚度表型均与候选区域的某些SNPs存在极显著的关联,从强到弱依次是肷部、肩部、背部、腋下、臀部、胸部、头脸、管部和下腹。从而,证实了巴马香猪群体存在影响猪皮肤厚度的7号染色体主效QTL。(5)3个与皮肤厚度关联性最强的SNPs值得进一步关注,分别位于7号染色体的34856565、35543837和35573869位置。肷部的皮肤厚度与SNP(chr7:34856565)的关联显著性最强(Pcor= 5.15×10-6),这个SNP也是肩部皮肤厚度最关联(Pcor= 5.75×10-6)的位点。SNP(chr7:35543837)是腋下(Pcor= 3.05×10-5)、臀部(Pcor= 0.010)、胸部(Pcor= 0.013)和头脸(Pcor= 0.025)皮肤厚度的最关联位点,也是肩部皮肤厚度的次最关联位点。SNP(chr7:35573869)则是背部皮肤厚度的最关联位点(Pcor= 1.17×10-5),SNPs(chr7:35543837和chr7:34856565)次之。(6)根据最强关联SNPs所在基因及基因生物学功能,初步推测ANKS1A和HMGA1基因可能是影响皮肤厚度的候选因果基因。【结论】较全面地测量了中国小型地方猪品种巴马香猪周身皮肤厚度,揭示了巴马香猪皮肤厚度在不同部位之间的变化规律。在巴马香猪群体中验证了影响皮肤厚度的7号染色体主效QTL位点,肷部、背部和肩部皮肤厚度表型性状与候选SNPs位点关联性更强,可能适合下一步大规模深入分析。ANKS1A和HMGA1基因可能是影响皮肤厚度的候选因果基因,但需要进一步生物学功能试验证明。
巴马香猪;皮肤厚度;候选SNPs位点;关联分析
0 引言
【研究意义】巴马香猪原产于广西巴马瑶族自治县,是中国优良的小型地方猪品种之一[1]。既有独特的肉用经济价值[2],又可用作良好的实验动物模型[3-4]。皮肤是动物外皮系统中最大的器官,在保护机体内环境和抵御外界环境病原菌侵入等方面起着重要屏障作用[5]。同时,猪皮是皮革产业的重要原料之一[6],也是现代医学异种皮肤移植的理想材料[7]。测量巴马香猪周身皮肤厚度,了解巴马香猪皮肤厚度的变化规律,可增加人们对巴马香猪皮肤的认知。在巴马香猪群体中验证 7号染色体影响皮肤厚度的主效QTL,可为进一步在巴马香猪群体中大规模开展皮肤厚度等形态变化的分子遗传控制机理及其相关基因功能研究奠定基础,为更好地开发利用巴马香猪提供参考。【前人研究进展】巴马香猪作为人类疾病模型已在皮肤创伤移植、心血管疾病和肌肉萎缩等疾病研究中应用。例如:2006年陈俊颖和魏泓用巴马香猪皮制作无细胞真皮,可作为创伤修复的真皮支架[8];2008年刘宇以洛伐他汀为典型药物研究巴马香猪和人代谢的异同[9]。目前,涉及皮肤厚度的研究主要集中在皮肤科学的医药研究和皮肤病理治疗上,而针对皮肤厚度的分子遗传控制机理研究鲜见报道。比如,SCHMOOK 等 2001 年报道了皮肤厚度对不同药物的体外渗透力的影响[10];SETO 等 2010 年报道了不同方法估计人皮肤厚度的研究[11];WONG等2011 年报道了乳腺癌患者在乳房切除术后放射治疗的皮肤厚度变化情况[12];YONDEDA等2011 年报道了在成年女性中骨密度与皮肤厚度的相关性[13]。在猪中,有少量文献报道了有关猪皮肤厚度的研究,比如猪皮肤厚度与瘦肉率的关系研究[14],云南本地猪及其杂交猪的皮肤厚度变化情况[15],猪皮部位差异的基本规律及对制革工艺的影响[16]等。目前尚没有关于巴马香猪的皮肤厚度研究报道。而从分子遗传学角度研究猪皮肤厚度只有AI等2014年报道的一篇关于猪皮厚性状连锁分析和全基因组关联分析的研究。该研究在二花脸×白色杜洛克F2资源群体和苏太猪群体中,利用194个微卫星标记和猪60K SNP芯片,发现猪4、7和15号染色体存在影响皮肤厚度主效QTL,其中7号染色体~35Mb处的主效QTL效应最强[17]。【本研究切入点】以中国地方小型猪品种巴马香猪为研究素材,随机选取了50头300日龄的成年猪,测量猪只周身9个典型部位的皮肤厚度,统计皮肤厚度在不同部位和不同性别间的差异性以及不同部位间的相关性。在巴马香猪群体中进行了皮肤厚度与7号染色体34.5—36.2 Mb候选区域46个SNPs位点的关联分析,分析不同部位皮肤厚度与这个区域SNPs的关联性,根据强关联的SNPs及其物理位置上所在或临近基因,结合基因的生物学功能,推测影响皮肤厚度变化的位置候选基因。【拟解决的关键问题】全面地了解巴马香猪周身皮肤厚度的变化情况,揭示其变化规律。同时在巴马香猪群体中验证在SSC7影响皮肤厚度变化的主效QTL,探讨影响猪皮肤厚度的遗传控制机理,增强人们对猪皮肤的认知。
1 材料与方法
1.1 试验动物
试验猪品种为巴马香猪,2014年购自广西巴马自治县巴马香猪繁育基地,统一饲养于江西南昌蒋巷镇国鸿生态园猪场,供猪只自由采食和饮水。购买时,巴马香猪的日龄为40—50日龄,其中公猪于30—35日龄被阉割。所有猪只饲养至(300±5)日龄,运至附近的国鸿生猪屠宰场,共屠宰了319头巴马香成年猪,其中阉割公猪160头,母猪159头,为43头父本和61头母本的后代。
1.2 方法
1.2.1 表型测定 巴马香猪宰前禁水禁食 24h后进行屠宰。在剔毛、去除内脏后,随机抽取50头(公猪23头,母猪27头,为17头父本和20头母本的后代)进行皮肤组织取样。用手术刀和剪刀,分别在头脸部、肩部、背部、肷部、臀部、胸部、下腹部、腋下和前管等9个部位(图1)处,取约5 cm2大小的完整皮肤组织样品。然后用手术刀垂直割取皮肤形成平整的切面,立刻用精度为0.01mm的电子游标卡尺垂直卡在皮肤切割面上进行测量,并读取、记录皮肤厚度的数据。
1.2.2 DNA提取及SNPs位点判型 采集0.5—1.0 g猪耳组织,按照标准酚/氯仿法提取 DNA。其A260/A280比值在1.8—2.0且琼脂糖电泳检测片段长度大于10 kb的,视为合格DNA,储存于-30℃冰箱待用。
使用Sequenom公司的MassARRAY®时间飞行质谱分型技术,进行7号染色体34.5—36.2 Mb内的46个SNPs位点的基因分型。即根据SNPs碱基分子量存在差异这一特性,通过PCR扩增得到一定数量级的包含SNPs在内的目的片段,再利用质谱仪测定不同基因型目的片段的质荷比,对比片段分子量差异,从而实现SNPs位点基因分型。

图1 巴马香猪皮肤取样的9个躯体部位Fig. 1 The 9 body sites where skin were sampled in Bamaxiang pig
1.2.3 统计分析 巴马香猪周身 9个部位皮肤厚度的描述性统计均使用R语言的相关统计函数进行计算分析。用 boxplot绘制箱式分布图,使用 shapiro.test进行正态分布检验,使用单因素方差分析aov检验不同部位皮肤厚度的总差异性,使用t.test检验两两不同部位间皮肤厚度的差异显著性,使用cor.test进行相关系数计算和显著性分析。
运用R语言SNPassoc 软件包中的association函数进行不同部位皮肤厚度与46个位点候选基因型的相关性分析。建立的广义混合线性模型如下:y=u+geno+ sex+batch+e。其中y为测定的皮肤厚度表型值,u表示皮肤厚度的平均数,geno表示SNPs的基因型,sex为性别效应,batch为批次效应,e为残差。
2 结果
2.1 皮肤厚度的描述性统计分析
精确测量了50头300日龄成年巴马香猪的头脸、肩、背、肷、臀、管、胸、下腹和腋下等9个典型部位的皮肤厚度(表1,图2)。从厚到薄依次是肷、背、肩、头脸、臀、管、胸、腋下和下腹,其中肷部和背部的皮肤厚度分别为(5.15±0.92)和(4.97±0.85) mm,下腹和腋下的皮肤厚度分别为(1.77±0.36)和(1.97±0.68)mm。背部的皮肤厚度变异度最小,为17.1%,腋下的最大,为34.8%。
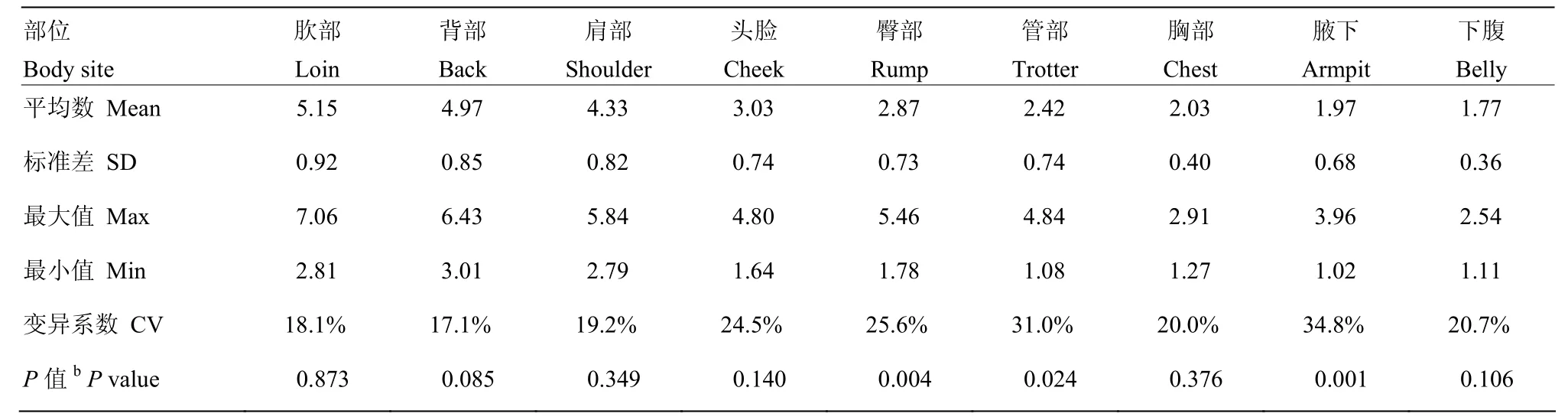
表1 巴马香猪周身9个部位皮肤厚度描述性统计量aTable 1 Descriptive statistics for skin thickness at 9 body sites in Bamaxiang pig
正态分布检验表明,头脸、肩、肷、胸、背和下腹皮厚的P值均大于0.05,符合正态分布;而腋下、管和臀的P值小于0.05,说明这个3个部位的皮肤厚度不符合正态分布。腋下的可能是由于不同性别间的皮肤厚度存在显著性差异(P = 0.021);管部皮肤厚度在母猪和阉割公猪间的差异接近显著水平(P = 0.072),可能是造成管部正态检验显著的原因之一;臀部皮肤厚度可能是由于测量的个体数偏少,测量个体的随机性造成的。
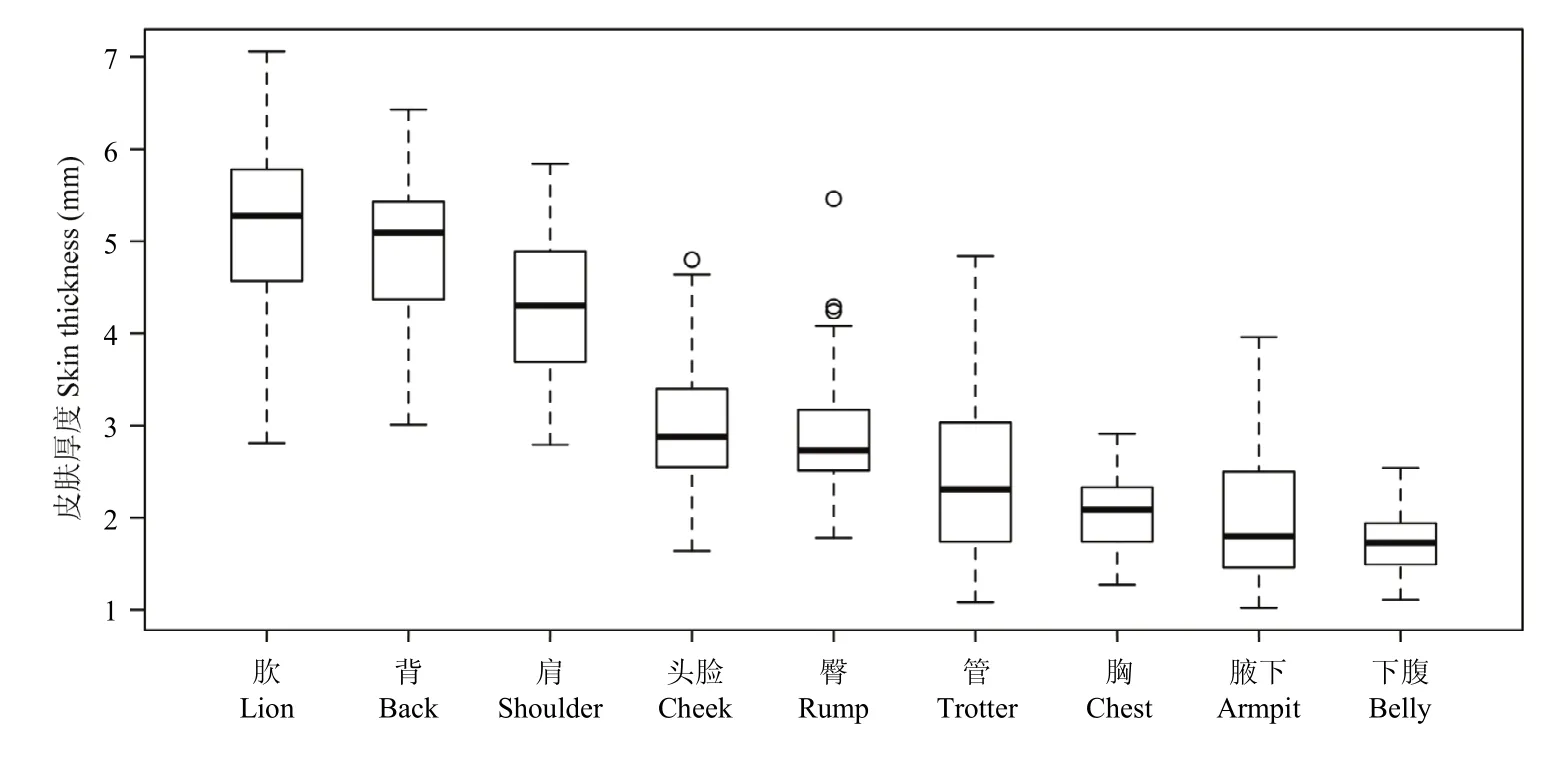
图2 巴马香猪不同躯体部位的皮肤厚度Fig. 2 skin thickness at different body sites in Bamaxiang pig
2.2 不同部位皮肤厚度的差异分析
巴马香猪 9个部位皮肤厚度在整体上差异极显著,P值为2.95×10-117,表2列出了它们两两之间相互比较的差异显著性。由此可知,除了背部与肷部、头脸部与臀部、胸部与腋下以及下腹与腋下的皮肤厚度差异不显著(P>0.05)以外,其余各个部位之间的皮肤厚度均存在极显著差异(P<0.01),其中以背部、肷部与下腹、腋下之间的皮肤厚度差异最为显著。
性别可能是影响皮肤厚度的影响因素之一,因此本文以性别作为分类依据进行皮肤厚度的显著性检验。检验结果表明,阉割的公猪个体腋下皮肤厚度显著小于母猪的皮肤厚度(P = 0.021),其它部位均不显著,但管部皮肤和下腹皮肤在母猪和阉割公猪间的厚度差异接近显著水平,P值分别为0.072和0.054(图3)。
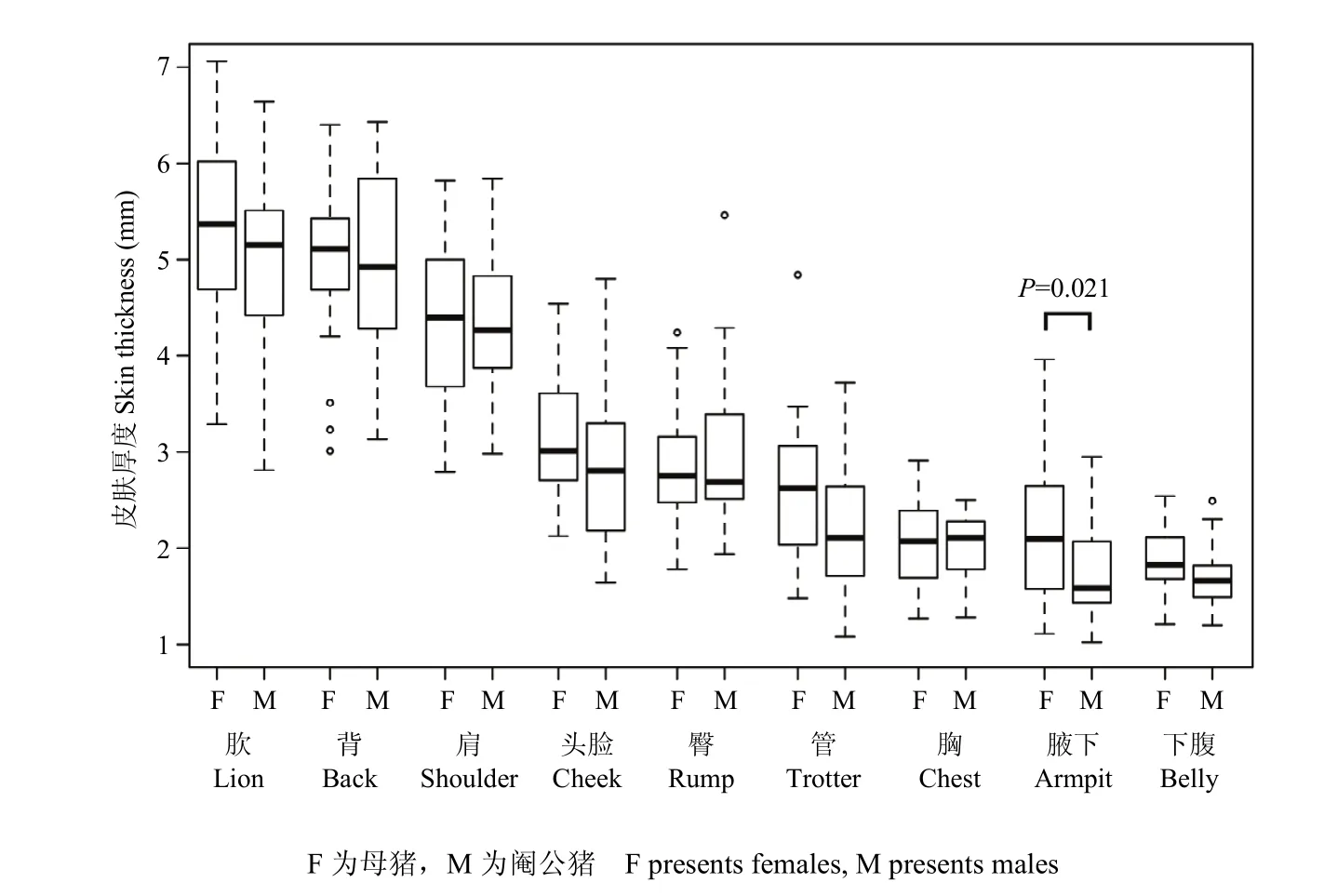
图3 巴马香猪不同躯体部位皮肤厚度在不同性别间的差异Fig. 3 Difference of skin thickness between two gander groups at 9 body sites in Bamaxiang pigs
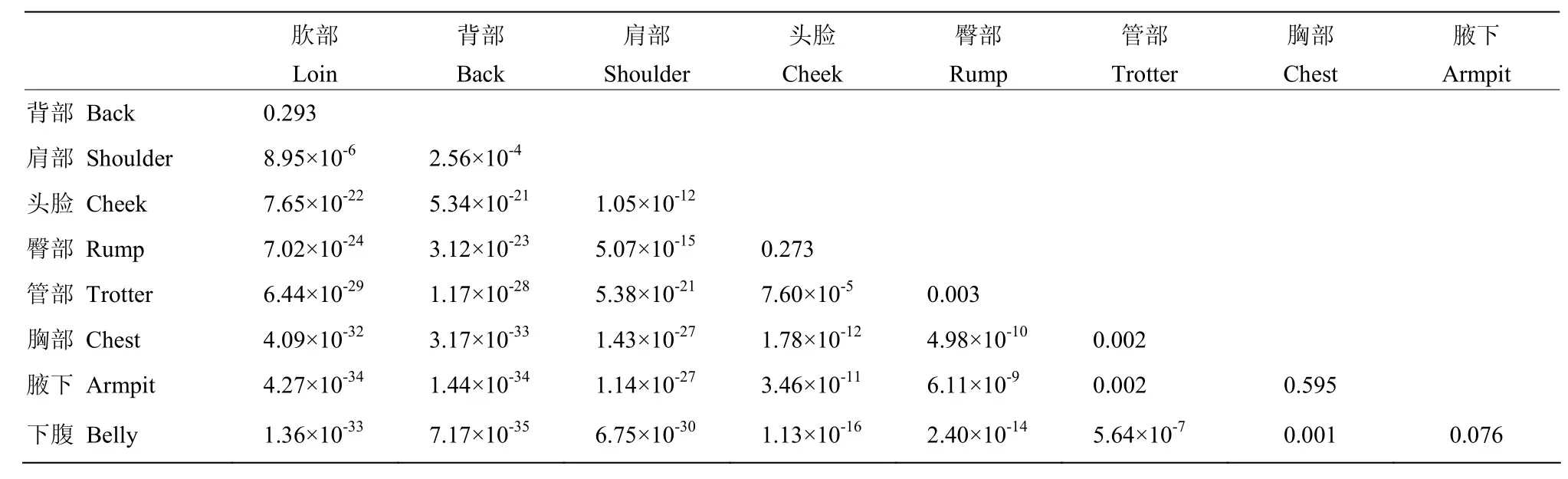
表2 巴马香猪9个部位皮肤厚度差异显著性P值Table 2 P value of t-test for skin thickness at 9 body sites in Bamaxiang pigs
2.3 不同部位皮肤厚度的相关性分析
巴马香猪9个部位之间的皮肤厚度除了下腹部与背部、肩部和头脸部的不相关外,其余部位两两之间均呈正相关(P<0.05,表3)。肩、背和肷三个部位的皮肤厚度两两之间都呈极显著正相关,管与腋下也呈现极显著正相关,它们之间的相关系数均大于0.6, P值均远小于0.01。其余部位两两之间的皮肤厚度相关性在0.28—0.6之间,P值也都小于0.05,均呈极显著正相关或显著正相关。
2.4 皮肤厚度表型与候选SNPs位点的关联分析
根据此前2014年AI等[17]的研究,在7号染色体的目标QTL区域34.5—36.2 Mb内选取和设计了46个SNPs位点,并进行MassARRAY的质谱基因分型,图4显示了这些SNPs在基因组中的物理位置。利用1.2.3方法中线性加性模型进行这46个SNPs位点的基因分型数据与9个部位皮肤厚度表型的关联分析。这9个皮肤厚度表型均与这个区域的某些 SNPs存在极显著的相关,与这个区域SNPs位点的相关性从强到弱依次是:肷部>肩部>背部>腋下>臀部>胸部>头脸>管部>下腹(图4)。
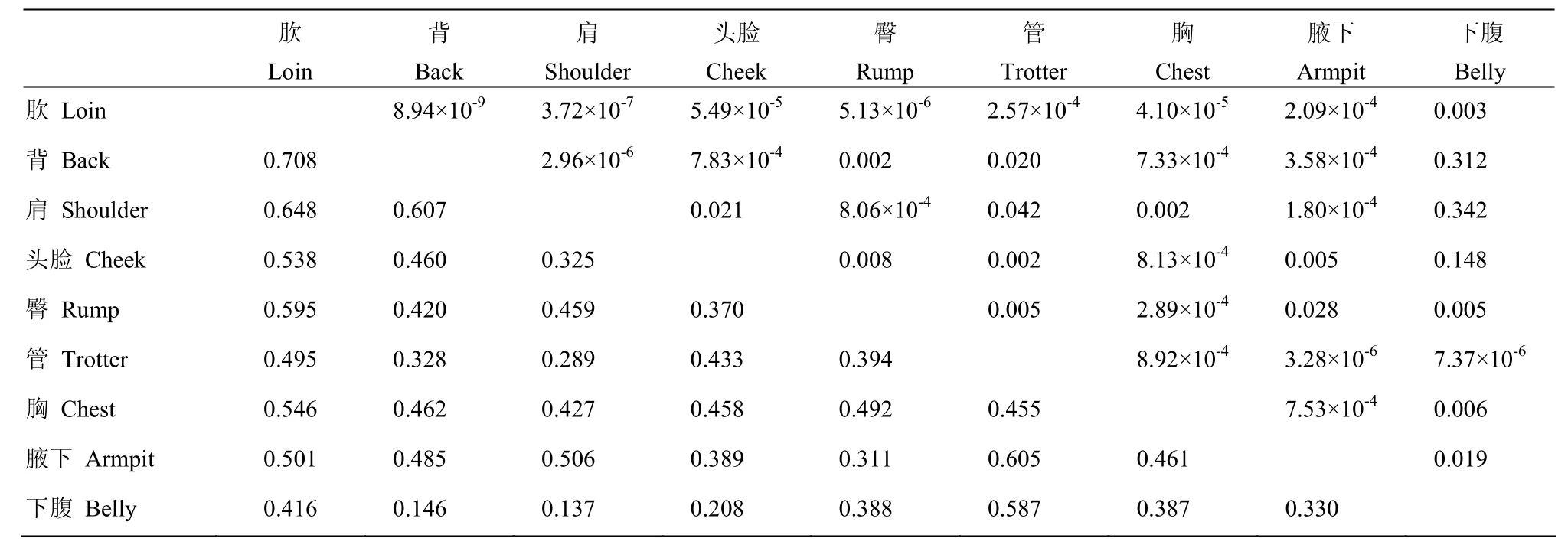
表3 巴马香猪9个部位皮肤厚度的相关性Table 3 Correlation of skin thickness between pairwise different body sites in Bamaxiang pigs
肷部的皮肤厚度与SNP(chr7: 34856565)的相关显著性最强(Pcor= 5.15×10-6),这个SNP同时也是肩部皮肤厚度最相关(Pcor=5.75×10-6)的位点;SNP(chr7: 35543837)是腋下(Pcor= 3.05×10-5)、臀部(Pcor= 0.010)、胸部(Pcor= 0.013)和头脸部(Pcor= 0.025)皮肤厚度的最相关位点,也是肩部皮肤厚度的次最相关位点。SNP(chr7: 35573869)则是背部皮肤厚度的最相关位点(Pcor=1.17×10-5),SNPs(chr7: 35543837和chr7: 34856565)次之(表4)。这3个与皮肤厚度强关联的SNPs分别位于GRM4,TAF11和ANKS1A等3个基因中(表4)。
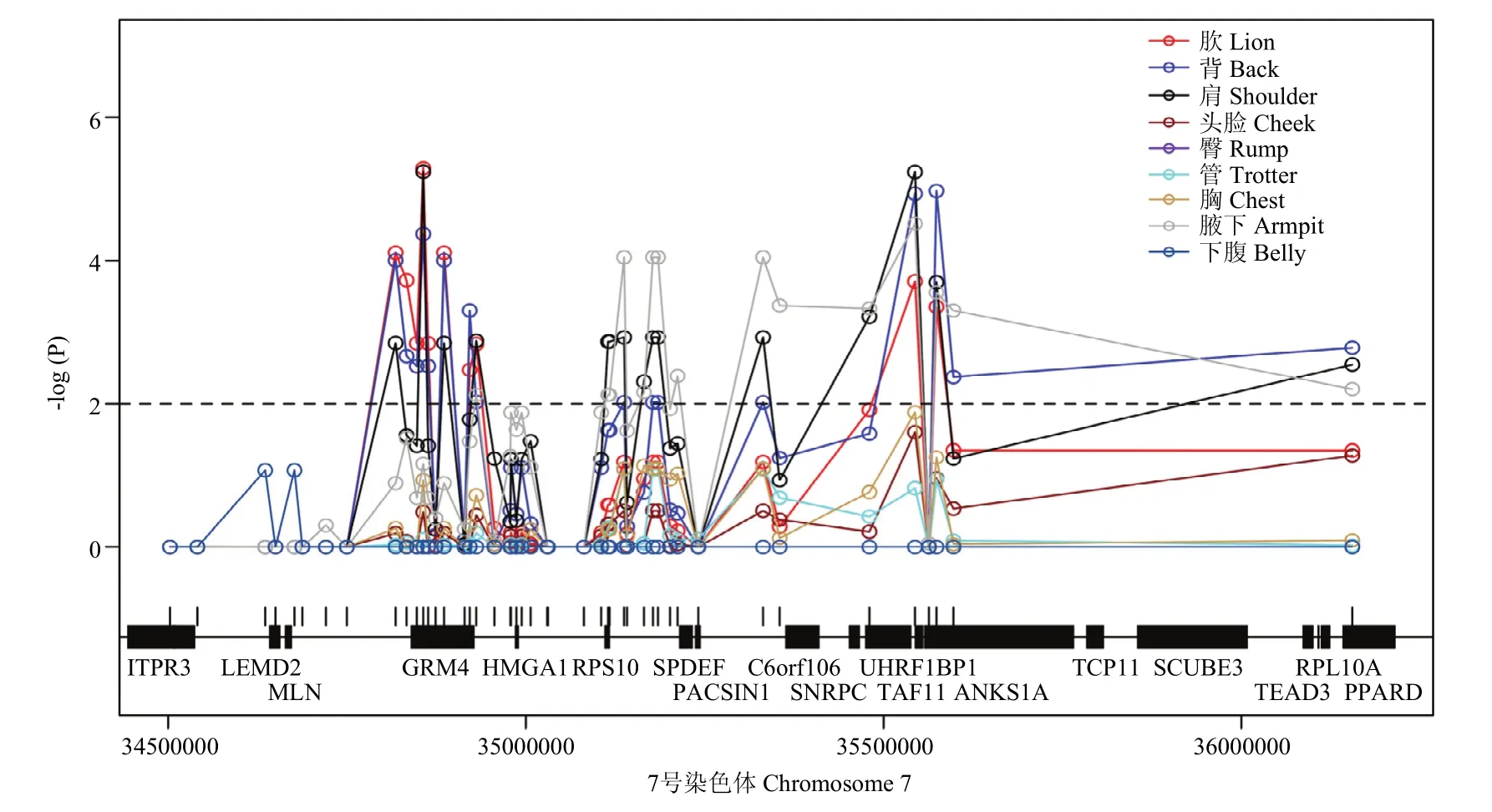
图4 巴马香猪9个不同部位皮肤厚度与7号染色体候选区域46个SNPs位点的关联分析Fig. 4 Association assay between skin thickness at 9 body sites and 46 candidate SNPs on SSC7 in Bamaxiang pig
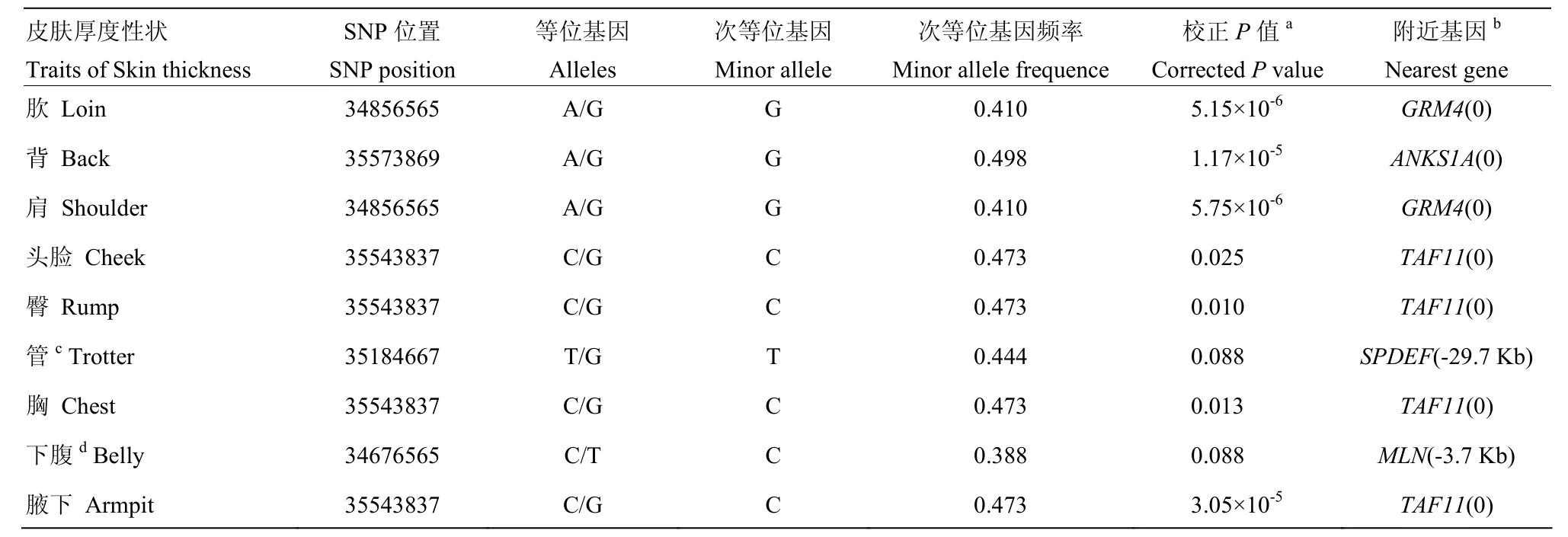
表4 关联分析中P值最高SNPs位点的信息Table 4 Information of the most significant SNPs in association assay
3 讨论
研究猪及其各类器官的形态结构变化、生长发育和病理变化等生命规律可为人类的相关研究提供重要借鉴。猪皮肤在形态结构上跟人类皮肤相近,猪皮可作为人类皮肤替代物进行药物试验或模拟治疗[18],利用猪皮作为人类烧伤皮肤的异种植皮来源也得到深入研究[7]。为了更好地了解猪皮肤的形态构造变化和生长发育规律,本文以深入了解巴马香猪皮肤厚度的变化规律和探究其分子遗传控制机理为出发点,开展相关猪皮肤厚度的研究工作,即对中国地方小型猪种巴马香猪进行了 9个典型部位皮肤厚度的表型统计分析,及其与7号染色体影响皮肤厚度主效QTL区域46个SNPs位点的关联分析。
有研究表明,在哺乳动物中皮肤厚度的变化因个体或部位而异,比如在人中一般为 0.5—4.0 mm[19],在马中为1.5—4.6 mm[20]。针对猪皮肤厚度变化规律的研究,有两篇较为经典的文献报道。1993年陈德端和王瑞华研究了云南当地猪和各杂交组合猪只的肩、背、臀和腹部4个部位皮肤厚度变化,发现猪皮肤厚度在躯体的变化通常为1.1—5.8 mm[15]。1994年王全杰比较全面地介绍了中国国产猪皮的厚度变化规律:整张猪皮以尾根部为底脊背为中线,呈现长把梨状三角形,厚度由底部沿着中垂线向顶角逐级递减,直到其2/3处不再明显下降,基本保持稳定[16]。笔者经过精确测量,发现巴马香猪的皮肤厚度也因个体不同或部位不同而不一样,变化范围为 1.02—7.06 mm。巴马香猪周身9个部位皮肤从薄到厚依次为:下腹<腋下<胸部<管部<臀部<头脸<肩部<背部<肷部。由此可见,巴马香猪躯体背侧朝上的部位皮肤较厚,而躯体两端和肢蹄端的皮肤厚度相对背侧较薄,而躯体的腹侧朝下或朝内的部位皮肤最薄。
巴马香猪躯体各部位的皮肤厚度变化的差异非常大,尤其是背侧与腹侧皮肤相比较;不同部位间的皮肤厚度呈现不同程度的正相关,同侧皮肤厚度的相关性强或较强,不同侧皮肤厚度相关性较弱或无相关,如背部皮肤厚度与肷部、肩部的呈现极显著正相关,而背部皮肤与下腹皮肤不呈现显著相关(P>0.05)。性别可能是影响皮肤厚度的一个重要影响因素。DERRAIK等2014年研究发现成年男性大腿上的皮肤显著地厚于女性的皮肤,但在腹部的皮肤男女性的差别不显著[21]。本研究发现在大部分躯体部位的皮肤厚度在母猪和阉割公猪间没有显著差别,但腋下皮肤在两者之间存在显著差异,这也可能是造成腋下的皮肤厚度不呈正态分布的原因之一。
2014年AI等利用二花脸×白色杜洛克的F2代资源群体,使用194个微卫星标记进行了猪皮肤厚度性状的连锁分析,发现了3个主效QTL位点,分别位于7号染色体的58 cM、15号的78 cM 和4号的107 cM上,其中7号染色体的QTL 解释了23.9%遗传变异。同时,新增苏太猪群体,使用猪60K SNP芯片,进行了两个群体猪皮肤厚度性状的全基因组水平关联分析,将验证了这3个主效QTL位点。将7 号染色体主效QTL置信区域缩短到683kb,将15 号染色体主效QTL 区域也缩短到2.2 Mb[17]。本研究则利用中国南方地方小型猪巴马香猪群体,进行了躯体9个部位皮肤厚度与7号染色体34.5—36.2Mb候选区域46个SNPs位点的关联分析。首先验证了巴马香猪群体也存在影响猪皮肤厚度的7号染色体主效QTL,为今后利用巴马香猪群体进行皮肤厚度的全基因组水平关联分析奠定了基础。其次,发现背侧(如:背部、肷部和肩部)的皮肤厚度与候选SNPs位点的关联性比躯体两端或腹侧(如:管部、胸部、臀部、下腹和头脸部)的更高。这提示以下两点:(1)在今后的皮肤厚度关联分析研究中,肩、背和肷部的皮肤取样部位可能会更为合适,在统计上造成的误差更小;(2)腹侧皮肤与背侧皮肤的皮肤厚度有可能存在某些不同的内在分子遗传控制机理,这一点值得今后更深入的研究和探讨。
在7号染色体34.5—36.2Mb的46个SNPs中,有3个与皮肤厚度关联性最强的SNPs值得进一步关注(表4),分别是SNP(Chr7: 34856565),SNP(Chr7: 35543837)和 SNP(Chr7: 35573869),它们分别位于GRM4,TAF11和ANKS1A基因中。GRM4基因编码一种代谢性谷氨酸受体,参与抑制神经系统中的cAMP级联反应(http://www.ncbi.nlm.nih.gov/gene/ 2914)。TAF11基因编码TF11D的小亚基,是一种转录因子,参与 RNA聚合酶 II的转录起始过程(http://www.ncbi.nlm.nih.gov/gene/6882)。ANKS1A基因编码一种锚蛋白重复及 SAM 结构域蛋白(http://www.ncbi.nlm.nih.gov/gene/23294),也称作为奥丁(Odin)。PANDEY等2002年克隆了奥丁,发现奥丁是一种含有酪氨酸磷酸化结合域的蛋白,揭示出奥丁可能在生长因子受体信号通路起着重要的负调控作用[22]。2013年TANG等进一步证实奥丁在表皮生长因子受体的循环利用上行使着效应物功能,通过贩运作用回收或降解表皮生长因子受体的方式来稳定表皮生长因子受体在细胞中的剂量[23]。从生物学功能上看,GRM4和TAF11这两个基因似乎与皮肤生长发育并不相关,而ANKS1A基因编码的蛋白与表皮生长因子受体有非常密切的相互作用,值得深入研究。7号染色体34.5—36.2Mb区域是一个基因富含的区间,包含有ITPR3,UQCC2,LEMD2,MLN,GRM4,HMGA1,RPS10,SPDEF,PACSIN1,C6orf106,SNRPC,UHRF1BP1,TAF11,ANKS1A,TCP11,SCUBE3,TEAD3,RPL10A,FANCE和PPARD等20个基因(图4)。这些基因均可能是影响皮肤厚度的因果基因,需要进一步做相关的生物学功能实验来验证。其中,HMGA1基因也值得关注。HMGA1基因编码一种参与多个细胞过程的非组蛋白转录因子,常被乙酰化存在于细胞核内,优先结合在双链 DNA的 AT富集区的小沟上(http://www.ncbi.nlm.nih.gov/ gene/3159)。有文献报道,在哺乳动物中HMGA1基因的突变影响着身高和体型的变化[24-25]。据此可初步推断,ANKS1A和HMGA1基因为影响巴马香猪皮肤厚度变化的候选因果基因,但均需进行生物学功能试验的深入研究,加以证实。
4 结论
首次较全面地测量了中国小型地方猪品种巴马香猪周身皮肤厚度,揭示了巴马香猪皮肤厚度在不同部位之间的变化规律。在巴马香猪群体中验证了影响皮肤厚度的7号染色体主效QTL位点,肷部、背部和肩部皮肤厚度表型性状与候选SNPs位点关联性更强,可能适合下一步大规模深入分析。ANKS1A和 HMGA1基因可能是影响皮肤厚度的候选因果基因,但需要进一步生物学功能试验证明。
References
[1] 国家畜禽遗传资源委员会. 中国畜禽遗传资源志:猪志. 北京:中国农业出版社,2011. National Commission on Livestock and Poultry Genetic Resources. Livestock and Poultry Genetic Resources in China: Pigs. Beijing:China Agricultural Press, 2011. (in Chinese)
[2] 张自勤. 长寿之乡的巴马香猪. 中国牧业通讯,1998(5): 59. ZHANG Z Q. Bamaxiang pigs in the longevity hometown. China Animal Husbandry Bulletin, 1998(5):59. (in Chinese)
[3] LIU Y, ZENG B H, SHANG H T, CEN Y Y, WEI H. Bama miniature pigs (Sus scrofa domestica) as a model for drug evaluation for humans:comparison of in vitro metabolism and in vivo pharmacokinetics of lovastatin. Comparative Medicine, 2008, 58(6):580.
[4] 陈玉龙. 巴马香猪肝硬化模型的建立及其肠道菌群演替的研究[D].重庆:西南大学, 2010. CHEN Y L. Develop an animal model of liver cirrhosis in Bama miniature pig and research its succession of intestinal flora [D]. Chongqing: Southwest University, 2010. (in Chinese)
[5] PROKSCH E, BRANDNER J M, JENSEN J M. The skin: an indispensable barrier. Experimental Dermatology, 2008, 17(12):1063-1072.
[6] 罗建勋,李书卿. 对猪皮革产品的思考与对策. 西部皮革,2008,30(8): 3-5. LUO J X, LI S Q. Consideration on pigskin leather products. West Leather, 2008, 30(8):3-5. (in Chinese)
[7] 陈炯,韩春茂,张力成. 异种 (猪) 脱细胞真皮基质与自体皮复合移植的临床应用. 中华整形外科杂志,2002,18(5):271-272. CHEN J, HAN C M, ZHANG L C. The clinical application of xenogenic (porcine) acellular dermal matrix grafting with thin autogenic skin on deep wounds. Chinese Journal Plastic Surgery,2002, 18(5):271-272. (in Chinese)
[8] 陈俊颖,魏泓. 巴马香猪无细胞真皮制备及其生物学特性. 中国比较医学杂志,2006, 16(11): 671-674. Chen J Y, Wei H. Preparation of BAMA miniature pis's acellular dermal matrix and its biological characteristics. Chinese Journal of Comparative Medicine, 2006, 16(11): 671-674. (in Chinese)
[9] 刘宇. 以洛伐他汀为典型药物研究巴马香猪和人代谢的异同[D].重庆:第三军医大学,2008. LIU Y. A comparative study on the differences and similarities of lovastatin metabolism between the Bama miniature pigs and human[D]. Chongqing: Third Military Medical University, 2008. (in Chinese)
[10] SCHMOOK F P, MEINGASSNER J G, BILLICH A. Comparison of human skin or epidermis models with human and animal skin in in vitro percutaneous absorption. International Journal of Pharmaceutics,2001, 215(1): 51-56.
[11] SETO J E, POLAT B E, LOPEZ R F, BLANKSCHTEIN D, LANGER R. Effects of ultrasound and sodium lauryl sulfate on the transdermal delivery of hydrophilic permeants: Comparative in vitro studies with full-thickness and split-thickness pig and human skin. Journal of Controlled Release, 2010, 145(1):26-32.
[12] WONG S, KAUR A, BACK M, LEE K M, BAGGARLEY S, LU J J. An ultrasonographic evaluation of skin thickness in breast cancer patients after postmastectomy radiation therapy. Radiation Oncology,2011, 6(9):1-10.
[13] YONEDA P D P, BIANCOLIN S E, GOMES M S M, MIOT H A. Association between skin thickness and bone density in adult women. Anais Brasileiros de Dermatologia, 2011, 86(5):878-884.
[14] 邹铭之. 猪皮肤厚度与瘦肉率的关系. 养猪,1989(3):26-27. ZOU M Z. The Relationship between swine skin thickness and the ratio of lean meat. Swine, 1989(3):26-27. (in Chinese)
[15] 陈德端, 王瑞华. 云南本地猪与各杂交组合猪皮厚度的研究. 云南畜牧兽医,1993(3): 7-9. CHEN D D, WANG R H. Study on skin thickness in Yunnan local pigs and hybrid pigs. Yunnan Journal of Animal Science and Veterinary Medicine, 1993(3): 7-9. (in Chinese)
[16] 王全杰. 猪皮部位差异的基本规律及对制革工艺的影响. 中国皮革,1993, 22(2):17-20. WANG Q J. The basic variation law of pig skin at different body sites and its effect on the tanning process. China Leather, 1993, 22(2):17-20. (in Chinese)
[17] AI H, XIAO S J, ZHANG Z Y, YANG B, LI L, GUO Y M, LIN G S,REN J, HUANG L S. Three novel quantitative trait loci for skin thickness in swine identified by linkage and genome-wide association studies. Animal Genetics, 2014, 45(4): 524-533.
[18] SULLIVAN T P, EAGLSTEIN W H, DAVIS S C, MERTZ P. The pig as a model for human wound healing. Wound Repair and Regeneration,2001, 9(2):66-76.
[19] SANDBY-MØLLER J, POULSEN T, WULF H C. Influence of epidermal thickness, pigmentation and redness on skin autofluorescence. Photochemistry and Photobiology, 2003, 77(6): 616-620.
[20] VOLKERING M. Variation of skin thickness over the equine body and the correlation between skin fold measurement and actual skin thickness [D]. Utrecht, Netherlands: University Utrecht, 2009.
[21] DERRAIK J G, RADEMAKER M, CUTFIELD W S, PINTO T E,TREGURTHA S, FAHERTY A, PEART J M, DRURY P L, HOFMAN P L. Effects of age, gender, BMI, and anatomical site on skin thickness in children and adults with diabetes. PloS One, 2014, 9(1): e86637.
[22] PANDEY A, BLAGOEV B, KRATCHMAROVA I, FERNANDEZ M,NIELSEN M, KRISTIANSEN T Z, OHARA O, PODTELEJNIKOV A V, ROCHE S, LODISH H F, MANN M. Cloning of a novel phosphotyrosine binding domain containing molecule, Odin, involved in signaling by receptor tyrosine kinases. Oncogene, 2002, 21(52):8029-8036.
[23] TONG J, SYDORSKYY Y, ST-GERMAIN J R, TAYLOR P, TSAO M S, MORAN M F. Odin (ANKS1A) modulates EGF receptor recycling and stability. PloS One, 2013, 8(6):e64817.
[24] GUDBJARTSSON D F, WALTERS G B, THORLEIFSSON G,STEFANSSON H, HALLDORSSON B V, ZUSMANOVICH P,SULEM P, THORLACIUS S, GYLFASON A, STEINBERG S,HELGADOTTIR A, INGASON A, STEINTHORSDOTTIR V,OLAFSDOTTIR E J, OLAFSDOTTIR G H, JONSSON T,BORCH-JOHNSEN K, HANSEN T, ANDERSEN G, JORGENSEN T,PEDERSEN O, ABEN K K, WITJES J A, SWINKELS D W, DEN HEIJER M, FRANKE B, VERBEEK A L, BECKER D M, YANEK L R, BECKER L C, TRYGGVADOTTIR L, RAFNAR T, GULCHER J,KIEMENEY L A, KONG A, THORSTEINSDOTTIR U, STEFANSSON K. Many sequence variants affecting diversity of adult human height. Nature Genetics, 2008(40):609-615.
[25] ZHANG L C, LI N, LIU X, LIANG J, YAN H, ZHAO K B, PU L,SHI H B, ZHANG Y B, WANG L X. A genome-wide association study of limb bone length using a Large White × Minzhu intercross population. Genetics Selection Evolution, 2014(46):56.
(责任编辑 林鉴非)
Measurement of Skin Thickness Along the Whole Body Sites and Association Study with Candidate SNPs on Sus Scrofa Chromosome 7 in Bamaxiang Pigs
HUANG Tao, HUANG Xiao-chang, QIU Heng-qing, YAN Guo-rong, HUANG Yi-zhong, ZHANG Yi-feng,JIANG Jia-cheng, ZHOU Li-sheng, REN Jun, MA Jun-wu, XIAO Shi-jun,HUANG Lu-sheng, YANG Bin, AI Hua-shui
(National Key Laboratory for Pig Genetic Improvement and Production Technology, Jiangxi Agricultural University,Nanchang 330045)
【Objective】Skin thickness at 9 typical sites of the whole pig body were measured in Bamaxiang pig breed, one of the Chinese local miniature pig breeds. There are 46 candidate SNPs located in a major QTL affecting skin thickness on sus scrofa chromosome 7 (SSC7) were genotyped in the Bamaxiang pig population. One aim of this study is to make clear variation rule of skin thickness at different body sites in Bamaxiang pigs. The other aim is to verify the major QTL affecting skin thickness on SSC7 in Bamaxiang pig population by association study in a specific genomic region. Result of this study will contribute to further revealing the genetic mechanism of swine skin thickness in a larger pig population at whole genome level, and increase our understanding about pig skin. 【Method】A totle of 50 Bamaxiang pigs, including 27 females and 23 castrated males, were randomly selected from a Bamaxiang pig population consisting of 319 adult pigs, which were slaughtered at 300 days. The skin with 5 cm2area were cut from 9 body sites of cheek, shoulder, back, loin, rump, chest, belly, armpit and trotter. Skin thickness at these 9 different body sites were accurately measured using digital caliper. The difference of skin thickness between pairwise body sites and the difference between females and castrated males were tested using the basic statistic package of R language. Pairwise Pearson's correlation coefficients were calculated between skin thickness at different body sites, meanwhile their significant tests were done. A total of 46 SNPs located in the region from 34.5 to 36.2 Mb on SSC7 were selected and designed for genotyping on a SEQUENOM MassARRAY SNP Genotyping platform. Combined with the above phenotypes of skin thickness, association studies on the target candidate region were performed by a general mixed linear model using SNPasscoc package of R language. Based on the association results and biological function of genes, positional candidate genes were determined. 【Result】One-way analysis of variance showed that an extreme significant difference with a 2.95×10-117probability existed among skin thickness at 9 body sites in Bamaxiang pigs. The thickest body sites were loin and back with skin thickness of (5.15 ± 0.92) and (4.97 ± 0.85) mm, respectively, while the thinnest sites were belly and armpit with skin thickness of (1.77 ± 0.36) and (1.97 ± 0.68) mm, respectively. The body sites with skin thickness from thick to thin in turn were loin, back, shoulder, cheek, rump, trotter, chest, armpit and belly. Skin thickness of castrated male pig at armpit was significantly thinner than female pig with a 0.021 probability. When compared between other pairwise body sites, skin thickness had no significant differences. There was no significant correlation between skin thickness at belly and back,between belly and shoulder, and between belly and cheek. Other pairwise combinations had a significant positive correlation. Association analysis between skin thickness at 9 different body sites and the above candidate SNPs suggested that the major QTL on SSC7 affecting skin thickness was also identified in Bamaxiang pig population. Three most promising SNPs were worthy of being further focused on. SNP (chr7:34856565) showed most significant association with skin thickness at loin (Pcor= 5.15×10-6), which was also the most significant site associated with skin thickness at shoulder (Pcor= 5.75×10-6). SNP (chr7:35543837) was the most significant site associated with skin thickness at armpit (Pcor= 3.05×10-5), at rump (Pcor= 0.010), at chest (Pcor= 0.013) and cheek(Pcor= 0.025), also was the second most significant site with shoulder. SNP (chr7:35573869) was the top significant site associated with back skin thickness (Pcor= 1.17×10-5), and followed by SNPs (chr7:35543837 and chr7: 34856565). Based on nearest genes of top SNPs and their biological function, ANKS1A and HMGA1 were preliminarily suggested to be positional candidate genes possibly affecting skin thickness in pigs.【Conclusion】It was the first time to measure skin thickness at 9 different body sites and report the variation rule of skin thickness along the whole body in Bamaxiang pig breed, one of the Chinese local miniature pig breed. Meanwhile the major QTL affecting skin thickness on SSC7 was verified in Bamaxiang pig population, it provided an important clue and base to reveal the genetic mechanism of pig skin thickness in future. ANKS1A and HMGA1 might be positional causal genes affecting skin thickness in pigs, and it needs further verification of biological functional test.
Bamaxiang pigs; skin thickness; candidate SNP sites; association study
2015-08-19;接受日期:2016-07-05
国家自然科学基金(31460283)
联系方式:黄涛,Tel:0791-83813080;E-mail:taohuang@live.cn。通信作者杨斌,Tel:0791-83813080;E-mail:binyang@live.cn。通信作者艾华水,Tel:0791-83813080;E-mail:aihsh@hotmail.com
