159株志贺菌耐药及非典型1类整合子基因盒携带情况分析
2016-09-05刘奉云王俊董丽娟吴晓妹祁伟
刘奉云,王俊,董丽娟,吴晓妹,祁伟
(天津医科大学第二医院,天津300211)
159株志贺菌耐药及非典型1类整合子基因盒携带情况分析
刘奉云,王俊,董丽娟,吴晓妹,祁伟
(天津医科大学第二医院,天津300211)
目的观察志贺菌临床耐药情况及非典型1类整合子基因盒的携带情况。方法采用K-B纸片法对天津地区3所三级甲等医院分离的159株志贺菌进行药敏试验,分析其耐药情况。采用RT-PCR法检测志贺菌1类整合子基因盒携带情况,并进行基因测序分析其耐药基因盒类型。结果159株志贺菌中,对3种及3种以上抗菌药耐药148株(93.1%),抗生素耐药情况分别为四环素85.5%、复方新诺明82.4%、氨苄西林82.4%;携带1类整合子基因盒134株,其中非典型1类整合子106株(66.7%);非典型1类整合子可变区携带的耐药基因盒 blaox a-30-aadA1(6.6%)、aadA5R(4.7%)、dfrA12R(57.5%)。 结论志贺菌多药耐药现象较明显,菌株携带非典型1类整合子比例较高,其主要可变区耐药基因盒种类为dfrA12R。
非典型1类整合子;志贺菌;多重耐药;基因盒;耐药性
引起细菌性痢疾的志贺菌属是发展中国家感染性腹泻主要致病菌之一[1],近年广谱抗生素的滥用导致志贺菌耐药情况逐年增多,甚至出现多重耐药性,造成治疗效果不佳。非典型1类整合子耐药基因盒可能与细菌耐药性有关。2014年8月~2015年4月,我们观察了天津地区临床分离的159株志贺菌中非典型1类整合子基因盒携带情况,并分析志贺菌的多重耐药情况,分析其在临床分离株中的分布及与多重耐药的关系,旨在为临床用药提供参考依据。
1 资料与方法
1.1临床资料临床分离的志贺菌159株为本研究所保存,来自天津医科大学第二医院、天津市儿童医院和天津医科大学总医院3所三级甲等医院肠道门诊非重复腹泻患者粪便。患者中男75例、女84例,年龄17~38岁;均经常规生化以及血清凝集试验鉴定分型证实,其中福氏志贺菌(B群)63株(39.6%)、粗糙型宋内志贺菌(R群)96株(60.3%)。
1.2志贺菌临床株耐药情况观察参照美国2013年临床实验室标准化研究所(CISI)的规定,采用改良K-B纸片扩散法进行药敏试验。质控菌株为大肠埃希菌ATCC25922。抗生素药敏纸片包括:氨苄西林、头孢噻肟、头孢曲松、亚胺培南、庆大霉素、链霉素、四环素、阿米卡星、氧氟沙星、诺氟沙星、左氧氟沙星、磺胺甲恶唑/甲氧苄啶、头孢哌酮、氯霉素共计14种,购自北京天坛生物制品研究所。观察149株志贺菌临床株的耐药情况,计算耐药率。耐药率=耐药菌株/总菌株×100%。所有操作严格按照使用说明书进行。
1.3志贺菌非典型1类整合子基因盒携带情况观察采用煮沸法提取志贺菌总DNA[2],RT-PCR法检测志贺菌1类整合子基因盒携带情况。引物设计及反应所需体系条件参照文献[3]进行,引物合成以及PCR产物测序由生工生物工程(上海)股份有限公司完成。扩增产物经1%琼脂糖凝胶电泳,溴化乙锭染色后,电压220 V、功率2 W条件下进行30 min的脉冲场凝胶电泳,凝胶成像系统下观察结果,采用BLAST程序(美国BIO-RAD 公司)和GenBank数据库比对分析非典型1类整合子基因盒携带情况[4,5]。
2 结果
2.1志贺菌耐药情况159株志贺菌耐药情况见表1。耐药情况依次为四环素 85.5%、复方新诺明 82.4%、氨苄西林82.4%,未见亚胺培南耐药。 159株志贺菌中多重耐药菌148株,占全部菌株的93.1%。
2.2志贺菌非典型1类整合子基因盒携带情况159株志贺菌中典型1类整合子阳性志贺菌24株,其中福氏志贺菌20株,宋内志贺菌4株;非典型1类整合子志贺菌106株,其中福氏志贺菌20株,宋内志贺菌86株。携带非典型1类整合子的106株志贺菌中,可变区耐药基因阳性志贺菌70株。其中3株可变区可同时检测到耐药基因dfrA12R与 aadA5R。106株非典型1类整合子基因盒携带情况为:blaoxa-30-aadA1 7株(6.6%)、aadA5R0 5株(4.7%)、dfrA12R 61株(57.5%)。
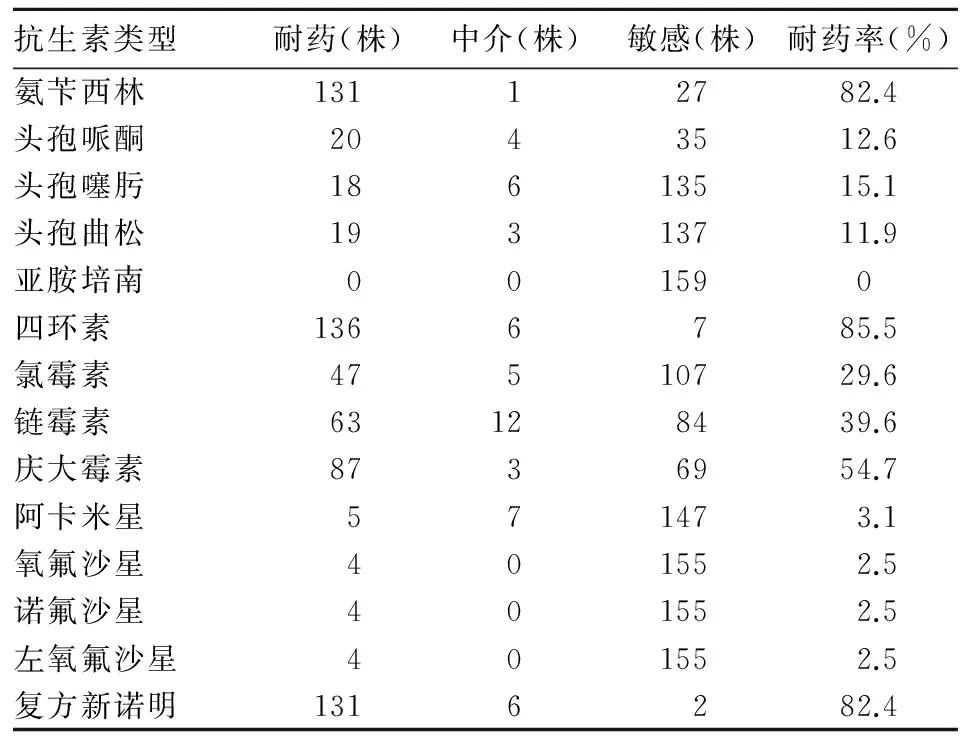
表1 159株志贺菌药敏结果
3 讨论
志贺菌是细菌性痢疾的主要致病菌,本研究所收集天津地区3家大型综合医院临床非重复分离的159株志贺菌中,宋内志贺菌株比例(60.3%)高于福氏志贺菌株(39.6%),与本所上世纪90年代及新世纪的研究结果[6]不一致,说明天津地区导致细菌性痢疾的志贺菌血清型发生变化。
目前研究表明,横向获得外源性耐药基因是大多数细菌产生耐药性特别是多重耐药的主要原因之一。而整合子是天然的克隆和表达的工具,可携带开放阅读框(ORF) 并将其转化为功能性基因,主要包括3部分:5′保守末端、可变区和3′保守末端。根据5′保守末端所含整合酶种类分为10种类型,常见为1类、2类、3类整合子,而非典型1类整合子是指应用常规PCR扩增,1类整酶呈阳性,而3′保守末端由于插入或者删除某片段呈阴性的整合子。非典型1 类整合子位于细菌染色体的Tn21转座子上[7]。本研究中,106株志贺菌携带非典型1类整合子,其中福氏志贺菌20株、宋内志贺菌86株。耐药基因盒dfrA12R编码叶酸二氢还原酶,导致对甲氧苄啶的耐药。本研究中,非典型1类整合子blaoxa-30-aadA1基因盒的阳性率(6.6%)相比较菌株对氨苄西林和链霉素耐药率较低,同样,dfrA12R耐药基因盒的阳性率与复方磺胺甲恶唑耐药率亦不相符,即实验菌株对相关抗菌药的耐药率高,而耐药基因盒的检出率低,可能是耐药基因位于整合子可变区之外的原因。国内也有学者报道,不同耐药基因可编码同一类抗生素耐药[8]有关,例如aphA1和aphA15均编码乙酰基转移酶,也介导对氨基糖苷类抗生素的耐药。典型1类整合子基因盒blaoxa-1、blaoxa-10(介导β-内酰胺类抗菌药耐药),dfrI(介导磺胺类抗菌药耐药)亦存在此种现象。而其介导氨基糖苷类抗菌药耐药的基因盒aadA1、aadA2、aadA5R 阳性率依次为20.8%、25.0%、12.5%与庆大霉素(54.7%)、链霉素(39.6%)、氯霉素(29.6%)的耐药率基本相符。在Peirano等[9]学者研究发现,编码对甲氧苄啶和大观霉素/链霉素耐药的耐药基因dfr17和aadA5R主要存在于福氏志贺菌典型1类整合子中;编码对大观霉素/链霉素的耐药的耐药基因aadA1主要存在于宋内志贺菌的典型1类整合子可变区。而本研究中,针对非典型1类整合子可变区耐药基因筛查发现,携带基因盒aadA5R志贺菌5株,基因盒dfrA12R志贺菌61株,同时检测到耐药基因dfrA12R与 aadA5R的志贺菌3株。推测可能是同种基因盒在不同类型整合子之间传递,或由于志贺菌株中典型1类整合子和非典型1类整合子的共存[10]存在一定困难,具体机制尚有待于进一步研究。
[1] Guang Y, Ligui W, Yong W, et al. Hfq regulates acid tolerance and virulence by responding to acid stress in Shigella flexneri [J].Res Microbiol, 2015,1(10):1-10.
[2] Richa J, Aruna M, Jonas S, et al. Serotype-conversion in Shigella flexneri:identification of a novel bacteriophage, Sf101,from aserotype 7astrain [J]. BMC Genomics, 2014,15(742):1471-2164.
[3] 董利娟,杨贤,王俊,等. 志贺菌 1、2 类整合子及 ISCR1 携带情况与耐药性的关系[J].天津医药,2015,43(4):400-403.
[4] 王俊,祁伟,王淑香,等.天津地区福氏志贺菌整合子携带及耐药性研究[J].中国病原生物学杂志,2014,9(2):146-149.
[5] Ud-Din AI,Wahid SU,Latif HA , et al. Changing trends in the prevalence of shigella species: emergence of multi-drug resistant shigella sonnei biotype g in bangladesh [J]. PLoS One, 2013,12(8):1-6.
[6] 梁帆,程玉谦,张娜. 志贺菌临床株耐药性分析及对氟喹诺酮类抗生素耐药机制的研究[J]. 天津医药,2011,39(11):1009-1012.
[7] 朱静媛,段广才,范清堂,等.志贺菌属非典型1类整合子与耐多药关系[J].中国公共卫生, 2011,7(5):554-556.
[8] 梁蓓蓓,邱少富,宋宏彬,等.志贺菌属整合子系统研究进展[J].传染病信息,2014,7(6):377-381.
[9] Peirano G, Agerso Y, Aarestrup FM, et al. Occurrence of integrons and resistance genes among sulphonamide-resistant Shigella spp. from Brazil[J]. J Antimicrob Chemother, 2005,55(3):301-305.
[10] 闻新棉,边锋芝,郑秀峰,等. 整合子及其耐药基因盒在临床分离多重耐药志贺菌中的分布[J].中华传染病杂志,2013,31(6):321-326.
10.3969/j.issn.1002-266X.2016.18.035
R183.4
B
1002-266X(2016)18-0090-02
2015-08-28)
