O1群E1Tor型霍乱弧菌溶原性测定的机理研究
2016-08-19申小娜张京云付秀萍李杰梁未丽阚飙
申小娜 张京云 付秀萍 李杰 梁未丽 阚飙
102206北京,中国疾病预防控制中心传染病预防控制所传染病预防控制国家重点实验室
·噬菌体研究·
O1群E1Tor型霍乱弧菌溶原性测定的机理研究
申小娜 张京云 付秀萍 李杰 梁未丽 阚飙
102206北京,中国疾病预防控制中心传染病预防控制所传染病预防控制国家重点实验室
目的 确定我国O1群E1 Tor生物型霍乱弧菌“噬菌体-生物分型”方案中“溶原性测定”中检测噬菌体的种类,研究溶原性测定的原理。方法 选择O1群E1 Tor型霍乱弧菌76株、O1群古典株8株、O139群菌株34株,利用溶原性测定方法检测菌株溶原性,检测菌株自发产生的溶原性噬菌体种类,检测阳性和阴性菌株染色体中该前噬菌体基因组,并检测这些菌株对溶原性噬菌体919TP(弧菌噬菌体K139家族)的敏感性。结果 O1群E1 Tor型菌株溶原性测定为阳性的19株菌,其释放的噬菌体主要为K139家族,具有溶原化的K139噬菌体,并对溶原性噬菌体919TP的敏感性为阴性;O1群E1 Tor型菌株22株对溶原性噬菌体919TP的敏感性为阳性,溶原性测定全部为阴性,基因组中不含有K139噬菌体;6株O1群古典型菌株溶原性检测为阳性株,但产生的噬菌体不是K139类;0139群菌株中5株溶原性测定为阳性,能检测到溶原性K139噬菌体基因组,并且34株菌全部不能被919TP感染。结论 O1群E1 Tor型霍乱弧菌“噬菌体-生物分型”方案中溶原性测定指标主要检测的是菌株释放K139类噬菌体的能力,即是否有K139噬菌体的溶原化并释放子代,“对溶原性噬菌体的敏感性”检测的是菌株能否被K139家族噬菌体(919TP)感染,因此生物分型中“溶原性测定”与“对溶原性噬菌体的敏感性”两个指标是相关联的。
【主题词】 霍乱弧菌;噬菌体-生物分型;溶原性噬菌体
【Key words】Vibrio cholerae;Phage-Biotyping Scheme;1ysogenic bacteriophage Fund programs:China Mega-Project for Infectious Diseases Grants from the Ministry of Sceince and Techno1ogy and the Ministry of Hea1th(2012ZX10004215)
霍乱是由霍乱弧菌引起的急性肠道传染病,在我国是传染病防治法规定报告管理的甲类传染病。霍乱弧菌按照菌体脂多糖抗原的不同,已分出210余个血清群,其中仅O1血清群或O139血清群霍乱弧菌的产毒株会导致霍乱流行。历史记载已有七次霍乱全球大流行,目前仍未停息的第七次霍乱大流行,是由O1群E1 Tor生物型引起,该型在1961年由东南亚传入我国并在我国引起流行。由于环境中存在大量对人不致病的非产毒霍乱弧菌菌株,而在六七十年代对霍乱毒素基因的确认和检测还缺乏特异和方便的方法,因此,对来自患者和环境监测分离E1 Tor型菌株进行亚型分析、尤其鉴定是否为能够引起发病和流行的菌株,成为霍乱防控与研究急需的技术。高守一等建立了“噬菌体-生物分型”方案,根据5株分型弧菌噬菌体(VP1-VP5)将O1群E1 Tor型霍乱弧菌分成32个(1-32)噬菌体型[1]。根据菌株的溶原性、对溶原噬菌体的敏感性、山梨醇发酵试验和溶血实验等4个生物学试验,将E1 Tor型菌株分成12个(a-1)生物型,再组合这些指标形成更细致的分型[2,3]。根据噬菌体-生物分型方案,可以将埃尔托型菌株分为流行株和非流行株,这对我国的霍乱防治发挥了重要作用。
对噬菌体-生物分型的相关机制进行研究,能够有助于从表型差异入手,分析霍乱弧菌的分型机理、生物学变异和基因组变异。我们已经对分型噬菌体VP3[4,5]、VP4[6]、VP5[7]的基因组特征和其受体进行了相关研究,发现霍乱弧菌菌株中这些分型噬菌体受体基因变异会导致菌株产生抗性,即裂解表型为阴性。在生物分型指标中,“溶原性测定”是指某些E1 Tor型菌株在繁殖过程中自发地释放溶原性噬菌体,用菌株16141 SM6作为指示菌、可使之形成半透明噬斑[3]。在E1 Tor型菌株中,存在溶原性测定阳性和阴性菌株。本研究对霍乱弧菌的溶原性测定进行了研究,分析其释放的溶原性噬菌体种类、以及与另一个生物分型指标“对溶原性噬菌体919TP的敏感性”之间的关联,研究其用于分型的分子机理。同时,也分析了O1群古典型和O139群携带类似溶原性噬菌体的特征。
1 材料与方法
1.1菌株来源 O1群E1 Tor霍乱弧菌76株,其中按照《霍乱防治手册》(第6版)[3]划分,属于噬菌体1-6型并生物分型a-f型的流行株45株,其余生物型即非流行株31株。O1群古典株8株,O139群菌株34株,共118株菌,具体见表1。

表1 菌株来源信息Tab.1 Strains used in this study
1.2溶原性测定和对溶原性噬菌体919TP敏感性的测定 依据《霍乱防治手册》(第6版)[3]进行。
1.3溶原性噬菌体919TP基因及霍乱毒素基因检测 在919TP噬菌体上设计5对基因引物(表2)包括整合酶基因、噬菌体排斥基因、衣壳蛋白基因、核酸内切酶基因和尾丝蛋白基因(int、glo、orf15、orf19 和orf35),检测这些基因是否存在。霍乱毒素基因按照《霍乱防治手册》(第 6版)[3]提供引物序列进行。
1.4溶原性菌株产生噬菌体确定 对溶原性测定阳性菌株,取待检菌LB液体培养物1 m1放入离心管中,12 000 rpm离心1 min,取上清用0.22 μm过滤器过滤,滤后液处理参照试剂盒(M6101,Promega)说明,加入DNase I放入37℃消化30 min,加入终止液放入65℃水浴10 min以终止反应。将处理过的液体100℃水煮10 min以释放噬菌体DNA后,作为核酸模板检测霍乱弧菌管家基因(recA,扩增引物见表2)和919TP基因(orf15、orf19、int、glo和orf35),其中recA作为检测上清经DNase I处理后霍乱弧菌染色体被去除的程度,这个基因扩增阴性可以判断处理过的上清中除尽了霍乱弧菌残余、仅存在菌株释放了的K139家族噬菌体。
2 结果
2.1O1群埃尔托型霍乱弧菌溶原性测定阳性菌株主要释放K139噬菌体 从本实验室收集的O1群埃尔托型霍乱弧菌随机挑选了76株进行噬菌体-生物分型方案中的溶原性测定,以菌株16141 SM6作为指示菌,检测这些菌株的溶原性。在42株产毒菌株中,有18株溶原性测定阳性。在34株非产毒菌株中,溶原性测定阳性1株。溶原性测定和对溶原性噬菌体的敏感性结果示例如图1。

表2 实验所用的引物序列Tab.2 Primers used in this study

A四株O1埃尔托型霍乱弧菌溶原性测定结果:1052(-)2866(+)218(-)2369(-);B对溶原性噬菌体的敏感性阳性结果;C对溶原性噬菌体的敏感性阴性结果图1 菌株的溶原性测定和溶原性噬菌体的敏感性的检测结果A The resu1ts of“1ysogenicity”in O1 E1 Tor Vibrio cholerae strains:1052(-),2866(+),218(-),2369(-);B The positive resu1t of“sensitivity to 1ysogenic phage”;C The negative resu1t of“sensitivity to 1ysogenic phage”Fig.1 The resu1ts of“1ysogenicity”and“sensitivity to 1ysogenic phage”
在生物分型方案的“对溶原性噬菌体的敏感性”指标中,我们发现使用的该溶原性噬菌体(命名为919TP)经基因组序列测定(GenBank KU504502)并比对,实际为已报道的霍乱弧菌K139[8]噬菌体家族(文章尚未发表)。919TP在被检测的敏感菌株上表现为半透明噬斑,与“溶原性测定”实验中阳性菌株产生的噬斑形态很相似,并且919TP能够感染的被测菌株在“溶原性测定”中均为阴性,即不释放溶原性噬菌体,所以我们推测溶原性测定阳性的菌株产生的噬菌体很可能为K139家族。为了验证假设,对本研究中溶原性测定阳性的19株菌(产毒株18株、及非产毒株1株),分别将其培养物上清进行离心、过滤后DNA酶消化,去除上清中残余的细菌基因组DNA,水煮裂解噬菌体后获得含溶原性噬菌体DNA的细菌培养液。经霍乱弧菌1个管家基因和噬菌体919TP序列设计的K139家族5个基因扩增检测,所有19株菌上清制备的核酸模板中霍乱弧菌recA基因扩增检测均为阴性,说明上清中霍乱弧菌染色体DNA已去除,而5对K139家族噬菌体的基因扩增均为阳性,提示这些溶原性阳性菌株释放了类似919TP的K139家族噬菌体。
为进一步检测K139前噬菌体在O1群E1 Tor型霍乱弧菌中的分布情况,我们又提取了霍乱弧菌染色体DNA作为模板检测K139噬菌体相关基因,检测到溶原性测定阳性的19株菌株中K139基因扩增都为阳性(表3)。同时检测到溶原性测定阴性的E1 Tor型菌株中有4株K139噬菌体基因检测阳性(表3),其余菌株均未检测到K139基因。
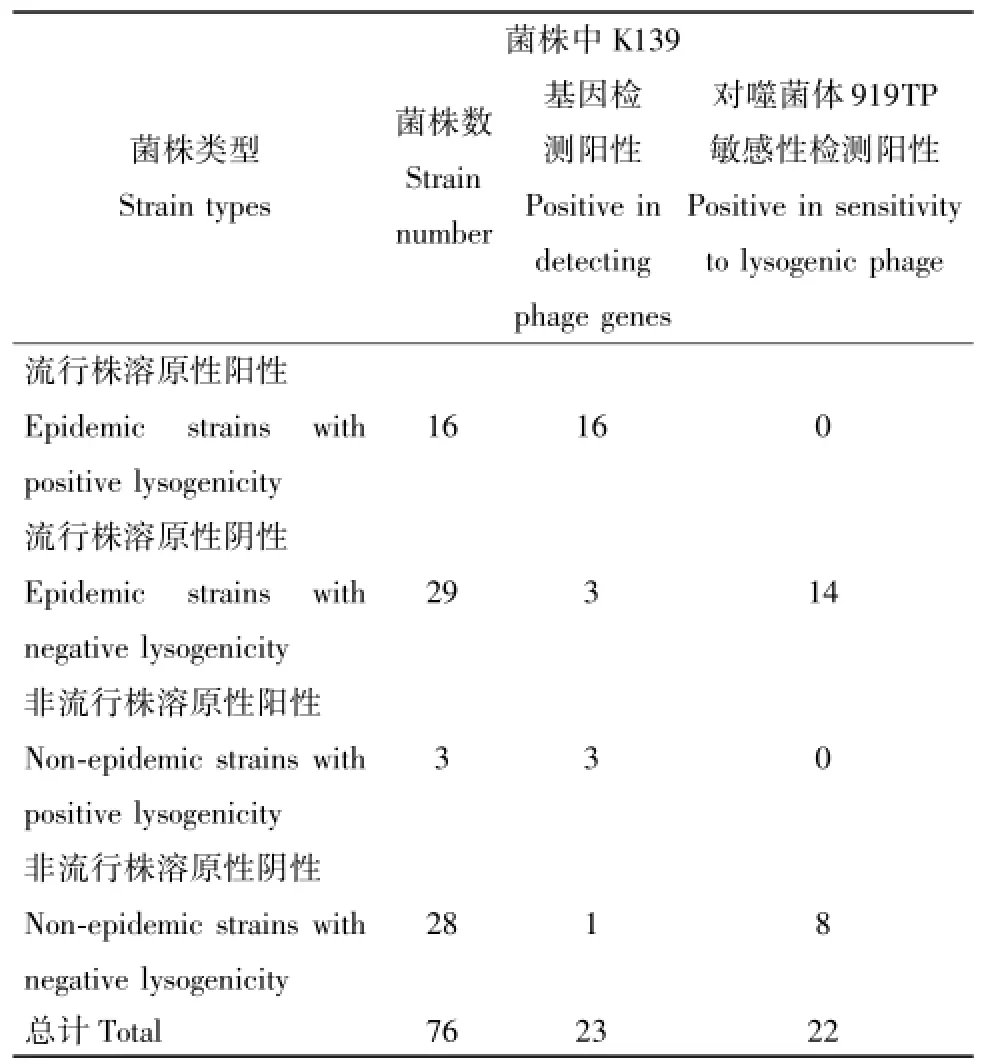
表3 O1群埃尔托型霍乱弧菌检测结果Tab.3 Detection resu1ts of the O1 E1 Tor strains
2.2O1群El Tor型菌株溶原化测定阳性菌株对噬菌体919TP的感染具有抗性 在针对O1群E1 Tor型霍乱弧菌噬菌体-生物分型方案的生物分型中,除了溶原性测定,第二个指标为对溶原性噬菌体(919TP)的敏感性测定。而经以上研究,发现“溶原性测定”阳性菌株主要释放的是K139家族噬菌体,因此可能这两个检测指标存在关联性,即对于被检测的一株E1 Tor型菌株来说,如果溶原性测定为阳性、即存在溶原化的K139噬菌体,则其“对溶原性噬菌体919TP的敏感性”会因为噬菌体超感染免疫而为检测阴性。
为分析这种可能性,对19株溶原性测定阳性株,又分别进行了对溶原性噬菌体919TP敏感性的测定,发现该19株菌株未产生噬斑,不能被919TP所感染,即对溶原性噬菌体敏感性均为阴性(表3)。另外,对其他O1群E1 Tor型霍乱弧菌“对溶原性噬菌体敏感性”检测后发现,22株 “对溶原性噬菌体敏感性”检测结果为阳性的E1 Tor菌株,其溶原性测定都为阴性,即这些菌株不释放K139家族噬菌体,能够被K139家族噬菌体919TP所感染。
2.3部分O1群古典型霍乱弧菌释放溶原性噬菌体但不属于K139家族 虽然噬菌体-生物分型不用于古典型和O139群株的分群,但鉴于O1群E1 Tor型菌株释放K139噬菌体,我们也进一步检测了导致第六次大流行的古典型菌株是否也溶原了K139噬菌体并能够释放子代。选取现有古典型菌株8株,对其进行溶原性测定,受体指示菌仍为生物分型方案中的16141 SM6,实验发现其中6株溶原性测定阳性。对这6株菌株,我们对其产生的噬菌体上清进行了K139基因组的检测,发现5对K139基因引物扩增全部为阴性,提示古典株产生的噬菌体不是919TP噬菌体。进一步对该8株古典株染色体DNA进行K139噬菌体基因组的检测,有2株(4477和浙66)其K139特异基因检测全部阴性,其余6株检测的5对引物中,其中噬菌体尾丝蛋白基因orf35引物扩增阳性、其余4对引物检测阴性,说明这6株菌中溶原的K139基因组仅存在部分基因,应为噬菌体基因组残余。
2.4仅少量 O139群霍乱弧菌菌株有溶原化的K139前噬菌体并可释放子代 我们同样筛选了O139血清群菌株34株,利用16141 SM6作为指示菌进行溶原性测定,发现阳性菌株仅5株。对于该5株菌株,将细菌过夜培养的待测上清去除细菌基因组DNA后检测,发现其产生的噬菌体是K139类噬菌体。对O139群菌株染色体进行K139基因扩增,发现此5株菌中含有K139噬菌体的所有检测基因,说明染色体中存在溶原化的K139噬菌体基因组。对溶原性测定阴性的29株菌检测K139的特异基因,发现都为阴性,说明这些菌株未含有K139噬菌体基因组。
另外,我们对此34株O139群菌株进行了对噬菌体919TP的敏感性测定,发现全部为阴性,说明O139群菌株难以被K139噬菌体感染。
3 讨论
病原菌的血清分型、生化特征分型以及噬菌体分型等,以及当前正在发展的分子分型和基于全基因组序列信息的分型比对,在区分细菌微生物学和遗传学特征、分析进化、以及作为流行病学分析的依据指标等,发挥了重要的作用。引起第七次全球流行的O1群E1 Tor型霍乱弧菌在1961年也传入我国并引起流行。我国自1970年以后开始使用噬菌体-生物分型方案对E1 Tor型霍乱弧菌进行分型,一方面能够鉴别流行的主要型别,另外在当时还不能检测毒素基因时,能够发现具有一些特定噬菌体-生物分型型别的菌株,对人体不致病或可能性很低,从而用于对菌株流行威胁的分析。对噬菌体-生物分型的检测指标分型原理开展研究,能够揭示出不同噬菌体-生物型别菌株之间的遗传差异本质。
在本研究中,我们对生物分型方案中的菌株溶原性测定进行了研究,分析了E1 Tor型菌株释放的溶原性噬菌体,确定了溶原性测定阳性的E1 Tor型菌株产生的噬菌体主要为K139家族噬菌体。对这些菌株的染色体 DNA进行分析,也证实其含有K139家族前噬菌体基因组。因此,基于目前对E1 Tor型菌株的溶原性指标分析,我们认为在我国霍乱防控中沿用多年的“噬菌体-生物分型方案”中,生物分型指标内的“溶原性测定”,主要检测了E1 Tor型菌株是否具有释放溶原性K139家族噬菌体的能力,这些检测为阳性的菌株,含有K139前噬菌体。
同时通过本研究发现,E1 Tor型菌株的生物分型方案中“溶原性测定”结果和“对溶原性噬菌体的敏感性”分析结果具有关联性,即溶原性测定为阳性的菌株、其对溶原性噬菌体敏感性的实验结果为阴性。对溶原性噬菌体敏感的菌株(即“对溶原性噬菌体的敏感性”为阳性),其“溶原性测定”结果为阴性。我们已发现“对溶原性噬菌体的敏感性”指标中,使用的溶原性噬菌体919TP实际上就是K139家族噬菌体,当被检测的菌株含有并释放K139噬菌体、即“溶原性测定”结果为阳性,该菌株因含有K139家族前噬菌体,因为同型噬菌体超感染的免疫机制,在对溶原性噬菌体(919TP)的敏感性指标测定中,919TP难以再感染该菌株,因此导致该指标检测为阴性。同理,如果检测的E1 Tor型菌株“对溶原性噬菌体的敏感性”分析结果为阳性,能够被919TP感染,则不含有K139前噬菌体,溶原性测定结果为阴性。在研究中还发现4株菌株基因组中含有K139家族前噬菌体,而“溶原性测定”阴性的菌株,可能是前噬菌体基因存在变异失去了进入烈性周期的能力,或者其不能自发释放需要诱导才能检测到噬菌体,这需要进一步的研究。
据此,我们对霍乱弧菌溶原性噬菌体的分析集中于K139家族噬菌体。虽然“噬菌体-生物分型”不用于O1群古典型和O139群菌株,我们也分析了这些菌株中是否含有K139家族噬菌体。我们发现在检测的古典型菌株中,大多溶原性测定为阳性,但通过对其产生的噬菌体进行检测,可以确认其产生的溶原性噬菌体不是919TP,这些溶原性噬菌体的具体特征还需进一步研究。但发现在古典株中存在不完整的K139前噬菌体基因组序列,结合我们溶原性测定结果,可以判断这个原噬菌体已经失去了进入烈性周期的能力,不能包装生成正常的噬菌体颗粒形成具有感染性的子代。
O139群菌株中少数含有K139前噬菌体,并能在溶原性测定阳性的菌株培养液上清中检测到噬菌体919TP基因,确定其产生的噬菌体是919TP。已知K139噬菌体家族受体为O1群脂多糖抗原[9],因O139群与O1群脂多糖结构的明显不同[10],O139群脂多糖不能作为噬菌体K139的受体,造成该类噬菌体不能在O139群菌株中传播,所以O139群菌株中含有K139前噬菌体基因组的菌株数量较少。但有意思的是K139噬菌体是首先在O139群菌株中发现的[11],这些少量的O139群菌株中K139前噬菌体的来源还不明确。
在本研究中,我们确定了霍乱弧菌E1 Tor型菌株的“噬菌体-生物分型”方案中检测的溶原性、实际主要是检测了菌株是否含有溶原化的K139噬菌体,并发现溶原性测定的指标和对溶原性噬菌体敏感性检测的指标具有关联性,解释了在我国应用数十年的噬菌体-生物分型中两个生物分型指标的原理,使得能够深入分析霍乱弧菌E1 Tor型菌株中一些表型差异的机制,并能够为分析菌株的遗传差异提供基因组水平的特征依据。
[1] 高守一,吴顺娥,刘秉金.O1埃尔托型霍乱弧菌分型噬菌体特征分析.副霍乱资料汇编,1984,4:237-245.
[2] 高守一.埃尔托霍乱弧菌流行株和非流行株两类菌株的研究及应用.中华流行病学杂志,1988,9:10-26.
[3] 肖东楼.《霍乱防治手册》第6版.人民卫生出版社,2013:5.
[4] Li W,Zhang J,Chen Z,et a1.The genome of VP3,a T7-1ike phage used for the typing of Vibrio cho1erae.Arch Viro1,2013,158:1865-1876.doi:10.1007/s00705-013-1676-9.
[5] Zhang J,Li W,Zhang Q,et a1.The core o1igosaccharide and thioredoxin of Vibrio cho1erae are necessary for binding and propagation of its typing phage VP3.J Bacterio1,2009,191:2622-2629.doi:10.1128/JB.01370-08.
[6] Xu J,Zhang J,Lu X,et a1.O antigen is the receptor of Vibrio cho1erae serogroup O1 E1 Tor typing phage VP4.J Bacterio1,2013,195:798-806.doi:10.1128/JB.01770-12.
[7] Xu D,Zhang J,Liu J,et a1.Outer membrane protein OmpW is the receptor for typing phage VP5 in the Vibrio cho1erae O1 E1 Tor biotype.J Viro1,2014,88:7109-7111.doi:10.1128/JVI. 03186-13.
[8] Kapfhammer D,B1ass J,Evers S,et a1.Vibrio cho1erae phage K139:comp1ete genome sequence and comparative genomics of re1ated phages.J Bacterio1,2002,184:6592-6601.doi:10.1128/JB.184.23.6592-6601.2002.
[9] Nesper J,Kapfhammer D,K1ose KE,et a1.Characterization of vibrio cho1erae O1 antigen as the bacteriophage K139 receptor and identification of IS1004insertions aborting O1antigen biosynthesis.JBacterio1,2000,182:5097-5104.doi:10.1128/JB.182.18.5097-5104.2000.
[10] Nesper J,Kraiss A,Schi1d S,et a1.Comparative and genetic ana1yses of the putative Vibrio cho1erae 1ipopo1ysaccharide core o1igosaccharide biosynthesis(wav)gene c1uster.Infect Immun,2002,70:2419-2433.doi:10.1128/IAI.70.5.2419-2433. 2002.
[11] Reid1 J,Meka1anos JJ.Characterization of Vibrio cho1erae bacteriophage K139 and use of a nove1 mini-transposon to identify a phage-encoded viru1ence factor.Mo1 Microbio1,1995,18:685-701.doi:10.1111/j.1365-2958.1995.mmi_18040685.
The mechanism of applying lysogenicity in phage-biotyping scheme for subtyping O1 E1 Tor Vibrio cholerae strains
Shen Xiaona,Zhang Jingyun,Fu Xiuping,Li Jie,Liang Weili,Kan BiaoNational Institute for Communicable Disease Control and Prevention,State Key Laboratory of Infectious Disease Control and Prevention,Chinese Center for Disease Control and Prevention,Beijing 102206,China
Kan Biao,Email:kanbiao@icdc.cn
ObjectiveTo determine the princip1e of app1ying 1ysogenicity in Phage-Biotyping Scheme deve1oped for the subtyping of O1 E1 Tor Vibrio cholerae strains.Methods 118 V.cholerae strains inc1uding 76 E1 Tor strains,8 c1assica1 strains and 34 serogroup O139 were se1ected to ana1yze the 1ysogenicity and sensitivity to 1ysogenic phage 919TP as described in the Manua1s of cho1era prevention and contro1,the genes of this phage were a1so determined among the genome sequences of these strains and the phages produced by them.Results A11 O1 E1Tor Vibrio cholerae 19 strains that produced positive resu1ts in 1ysogenicity,had the 1ysogenic K139 phage in genome and cou1d both resist to 1ysogenic phage 919TP and re1ease the K139 fami1y phage.A11 the O1 E1Tor Vibrio cholerae 22 strains that produced positive resu1ts in sensitivity to the 1ysogenic phage,had no K139 fami1y phage genes and got negative resu1ts in 1ysogenicity. However,the phages of this fami1y were not re1eased from 6 c1assica1 strains with positive 1ysogenicity resu1t. Five serogroup O139 strains were detected re1easing temperate phages K139 without the sensitivity to phage 919TP.Conclusions App1ying the 1ysogenicity in Phage-Biotyping Scheme for subtyping O1 E1 Tor Vibrio cholerae strains is based on the abi1ity to produce 1ysogenic bacteriophage K139.The index of“sensitivity to the 1ysogenic phage”was a1so associated with this abi1ity.
国家传染病防治科技重大专项(2012ZX10004215)
阚飙,Emai1:kanbiao@icdc.cn
10.3760/cma.j.issn.1003-9279.2016.02.022
2016-03-14)
