河南省一起斯坦利沙门菌聚集性病例的病原学研究及溯源分析
2016-07-29赵嘉咏郝宝林谢志强黄学勇穆玉娇夏胜利
赵嘉咏,郝宝林, 谢志强,黄学勇,穆玉娇,夏胜利
河南省一起斯坦利沙门菌聚集性病例的病原学研究及溯源分析
赵嘉咏,郝宝林, 谢志强,黄学勇,穆玉娇,夏胜利
河南省疾病预防控制中心,郑州450016
“十二五”国家科技重大专项(No.2012ZX10004201和No.2013ZX10004203)联合资助
摘要:目的分析2013年1-7月份河南省2地市3家医院分离自婴幼儿患者粪便中的斯坦利(S.stanley)沙门菌的病原学特征及分子流行病学关联,为食源性疾病聚集性及暴发病例的调查及院感防控提供新的思路和模式。方法采集河南省3家医院婴幼儿住院病例的腹泻粪便,SBG增菌,沙门菌科玛嘉选择性培养基分离培养,API20E肠杆菌科系统生化板条鉴定后使用丹麦SSI分型血清进行沙门菌O抗原及H1/2相鞭毛诱导血清凝集试验。根据PulseNet China病原体分子分型网络公布的沙门菌脉冲场凝胶电泳(PFGE)操作指南与美国临床实验室标准化协会(CLSI)沙门菌K-B法药敏测试方案,对其进行PFGE分子分型与聚类分析及17种抗生素药敏测试。结果分离自15位婴幼儿住院病例粪便中的沙门菌经鉴定均为斯坦利沙门菌(S.stanley),药敏测试显示其对环丙沙星、左氧氟沙星、复方新诺明、四环素、亚胺培南等8种抗生素敏感,对氨苄西林、头孢噻肟、头孢吡肟、阿莫西林等9种抗生素耐药,其中12株菌耐药表型完全一致。15株斯坦利沙门菌经XbaI与BlnI限制性双酶切和PFGE电泳后分为STN1和STN2两种带型,其中STN1带型包含14株菌,与另1株(STN2)具有流行病学关联度的菌株带型差异度较大。STN1经“PulseNet China”数据库比对证实为斯坦利沙门菌河南省独有带型。结论病原学诊断及PFGE分子分型结果证实了15例斯坦利沙门菌感染病例间可能存在高度的分子流行病学关联和聚集性,为进一步的追踪溯源和调查取证提供了技术支撑。
关键词:斯坦利沙门菌; 聚集性病例;药敏测试;PFGE
沙门菌属是一种广泛存在于自然界的重要人兽共患病原菌,也是近几年来全球范围内食源性疾病病原学监测和暴发疫情常见的主要病原,准确快速的病原学诊断能为临床治疗和疫情处置提供必需的参考依据。2012年1月起,河南省2地市斯坦利沙门菌(S.stanley)感染病例异常增多,怀疑为散发性暴发事件,随即河南省CDC传染病实验室开展了病原学诊断与脉冲场凝胶电泳(PFGE)分子分型与聚类分析,为临床治疗和及时启动流行病学调查及防控措施的制定提供病原学依据。
1材料与方法
1.1材料
1.1.1腹泻样本2012年1~7月间采集自河南省平顶山市与郑州市3家医院婴幼儿住院患者的腹泻粪便。病例年龄主要集中于0~1.5岁: 出生4天-30天9例,1个月~2个月 4例,3个月~18个月2例;男性7例,女性8例,男女性别比0.88:1。病例分别来自于平顶山市A、B两家医院和郑州市C医院,8例出生后即入住ICU病房。患者普遍存在腹泻、发热、呕吐等症状,其粪便性状以黄绿色水样便为主,一共11例,占腹泻总数的73.3%。流行病学调查显示,B到C医院、A到C医院,AB医院间发生过病例转院事件,其中A、B两个医院各向C医院转院4例病人,A、B医院间相互转院2例病人,见表1,图1。
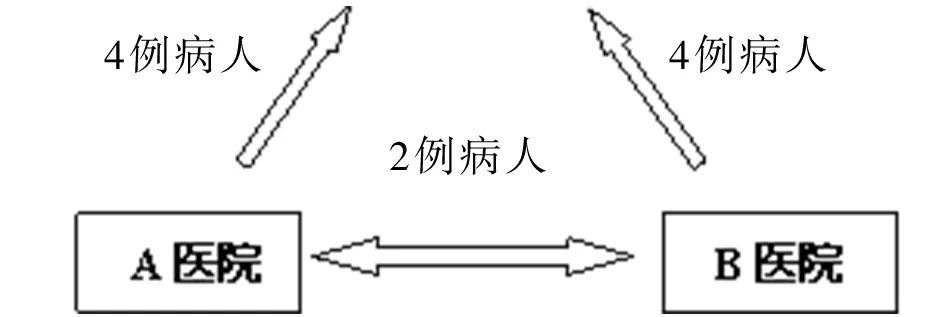
图1 斯坦利沙门菌感染病例转院示意图Fig.1 The transfer map of S. stanley infected patient
表115例婴幼儿病例临床表征统计
Tab.1Clinical characterization of 15 infant cases
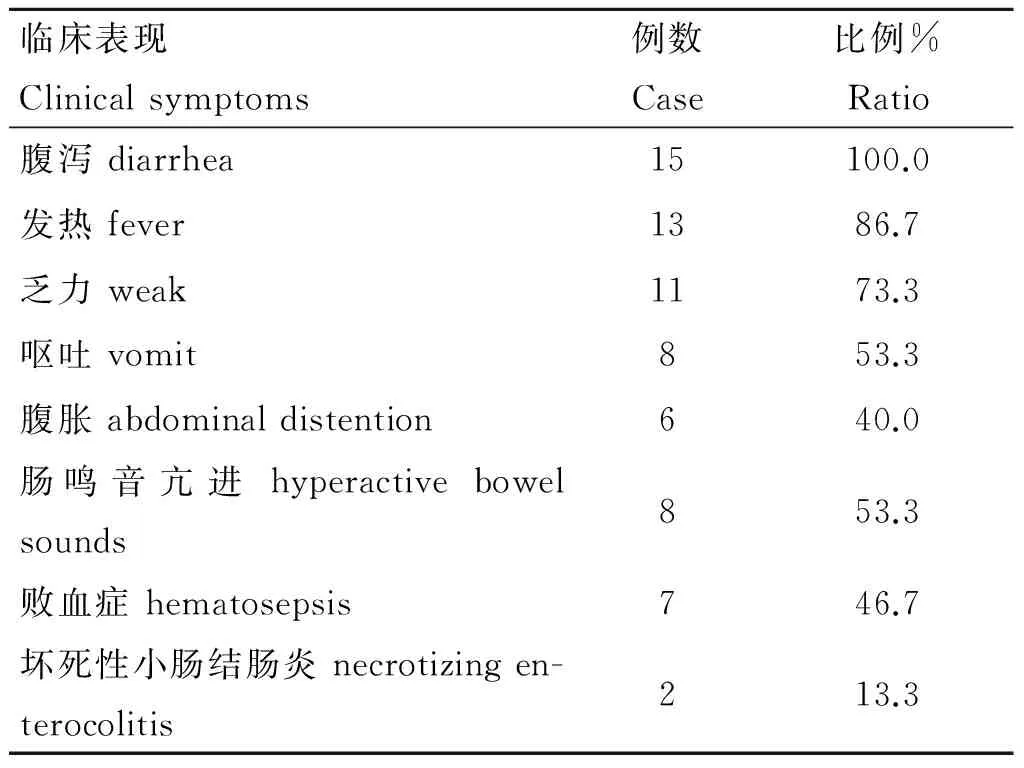
临床表现Clinicalsymptoms例数Case比例%Ratio腹泻diarrhea15100.0发热fever1386.7乏力weak1173.3呕吐vomit853.3腹胀abdominaldistention640.0肠鸣音亢进hyperactivebowelsounds853.3败血症hematosepsis746.7坏死性小肠结肠炎necrotizingen-terocolitis213.3
1.1.2主要试剂和仪器1)试剂:沙门菌亚硒酸盐煌绿增菌液(SBG)、沙门菌科玛嘉选择性培养基购自法国CHROMAgar公司;克氏双糖(KIA)、动力吲哚尿素琼脂(MIU)购自北京陆桥技术公司;脑心浸液、M-H琼脂及药敏纸片购自英国OXOID公司;API20E肠杆菌科细菌鉴定板条购自法国bioMérieux公司;沙门菌诊断血清购自丹麦SSI公司; Seakem Glod琼脂糖购自美国LONZA公司;蛋白酶K购自英国Roche公司;限制性内切酶XbaI购自大连宝生物公司; Tris-Hcl(1 mol/L, pH8.0),EDTA(0.5 mol/L,pH8.0),5×TBE购自solarbio公司。2)仪器:去离子水系统购自美国Milipore公司;凝胶成像系统购自美国Bio-Rad公司;VITEK2浊度仪购自法国bioMérieux公司;脉冲场凝胶电泳系统购自美国Bio-Rad公司。
1.2方法
1.2.1沙门菌分离培养、生化鉴定与血清学分型将0.5克(稀便)或100 μL(水样便)腹泻粪便接种于5 mL亚硒酸盐煌绿增菌液,37 ℃培养16 h后用无菌接种环三区划线接种于沙门菌科玛嘉选择性培养基,37 ℃培养16~18 h后挑取边缘整齐、光滑、凸起的淡紫色菌落穿刺接种于克氏双糖/动力吲哚尿素培养基,37 ℃培养16~18 h后挑取发酵葡萄糖不发酵乳糖、产气产硫化氢、动力阳性尿酶阴性靛基质阴性的菌株转种脑心浸液平板,37 ℃培养16~18 h,0.85%生理盐水制备0.5麦氏浊度菌液,API20E肠杆菌科系统生化板条鉴定,SSI沙门菌诊断血清进行O相玻片凝集分型,再利用0.4%营养软琼脂进行H相(1/2相)鞭毛诱导与血清凝集,生理盐水做自凝对照。根据卡芙曼-怀特(Kauffmann-White)血清分型表确定对应血清型别。
1.2.2脉冲场凝胶电泳分型按照国际Pulsenet China网络实验室公布的沙门菌脉冲场凝胶电泳标准分型方法,对沙门菌株进行分子分型,标准株H9812作为分子量标记。限制性内切酶XbaI/BlnI(50U),37 ℃酶切2.5 h。电泳参数:电压6 V/cm,脉冲时间2.2~63.8 s,线性转换,电场夹角120°,电泳时间19 h,电泳液温度14 ℃。电泳结束后胶块用GelRed染料染色20 min后纯水清洗30 min后凝胶成像仪拍照(曝光时间3.0 s,去过饱和成像)。
1.2.3药敏试验参照WHO推荐的Kirby-Bauer法,将沙门菌转种脑心浸液琼脂平板,37 ℃培养16 h后0.85%生理盐水制成0.5麦氏单位悬液。用无菌棉拭子蘸取菌液均匀涂布于M-H平板表面,用分配器将药敏纸片压盖其上,15 min内送37 ℃温箱培养18 h以后测量各药敏纸片抑菌圈直径,参照美国临床实验室标准化协会(CLSI)2009年标准进行耐药表型判定,大肠杆菌ATCC25922作为质控菌株。
1.2.4数据分析用BioNumerics6.0软件对电泳图谱进行数据分析,绘制聚类分析树状图。聚类算法为非加权配对平均法(UPGMA,Dice),电泳条带位置优化度(Position tolerance)1.5%,相似度100%认定为同一PFGE带型。
2结果
2.1沙门菌鉴定与血清学分型结果15株沙门菌在沙门菌科玛嘉培养基上均为紫红色、边缘整齐、光滑凸起小菌落;KIA/MIU小生化反应为:发酵葡萄糖不发酵乳糖,产酸产气产硫化氢;尿酶阴性,动力阳性,靛基质阴性。API20E肠杆菌科系统生化鉴定为沙门菌(ID%99.2%),血清抗原式为1,4,5,12:d:1,2,参照卡芙曼-怀特沙门菌血清分型手册判定均为斯坦利沙门菌。
2.2药敏测试结果15株斯坦利沙门菌对环丙沙星(CIP)、左氧氟沙星(LEV),复方新诺明(SXT),四环素(TET),亚胺培南(IPM),美罗培南(MEM),头孢哌酮(SCF),哌拉西林(TZP)敏感;对氨苄西林(AMP),头孢他啶(CAZ)、头孢噻肟(CTX),阿莫西林(AML),头孢吡肟(FEP),萘啶酸(NAL),诺氟沙星(NOR),链霉素(STR)及增效磺胺类抗生素甲氧苄氨嘧啶(TMP)耐药。15株菌中有12株耐药表型一致,耐药谱为AMP-CAZ-AML-FEP-NAL-NOR-STR-TMP。
2.3PFGE分子分型结果15株斯坦利沙门菌经XbaI/BlnI双酶切后分为2种带型,分别命名为STN1与STN2,其中STN1包含14株菌,双酶切带型完全一致;STN2仅有1株菌,与STN1带型相似度38.9%。STN1带型图谱递交至“PulseNet China”中心实验室数据库进行比对,未发现与其他省份斯坦利沙门菌带型存在同源性,证实为河南省斯坦利沙门菌独有带型(图2,图3)。

图2 河南省斯坦利沙门菌XbaI/BlnI双酶切PFGE与聚类分析图谱Fig.2 XbaI/BlnI PFGE cluster analysis of S. stanley in Henan Province

图3 河南省斯坦利沙门菌STN1带型(红框内)与“PulseNet China”中心实验室数据库比对结果Fig.3 Comparison results of STN1 belt type with “PulseNet China” data base
3讨论
能引起人类食源性疾病的非伤寒沙门菌血清型众多。从河南省近4年的监测数据和其他省份的报道看,位居前列的血清型主要是鼠伤寒(S.typhimurium)、肠炎(S.enteritidis)、阿贡那(S.agona)、德尔比(S.derby)等[1-2]。斯坦利沙门菌在我省属于罕见血清型,2009-2011年共计分离到2株,但从2012年2月开始,菌株数突然增多,至7月份已增至15株,且病例主要集中于2个地市的3家医院,具有初步的聚集性特征,尽管时空跨度比较大,但足以引起我们的重视。从国内外的报道看,斯坦利沙门菌在病人,食品及外环境中均有检出,主要引起成人或儿童散发性腹泻,偶有小规模爆发[3-4],临床症状轻、病程短,但此次河南省2地市的感染病例主要集中于出生3个月以内的婴幼儿,且8例出生后即入住ICU病房,除腹泻、呕吐、发热等症状外,还出现了败血症和坏死性小肠结肠炎(NEC),通过访谈没有发现居住地、出生医院、使用奶粉、发病前用药、喂养、饮水等共同的商品化暴露因素,这在河南省以往的沙门菌感染病例中是非常罕见的。结合病原学诊断和流行病学资料综合分析,我们高度怀疑这是一起由斯坦利沙门菌院内交叉感染引起的散发性暴发事件,即由一代病例产生交叉传染,继而伴随病人的转院形成传播和产生后续病例。
脉冲场凝胶电泳(PFGE)不仅能够通过日常监测积累的病原菌分子流行病学数据建立基线和设定阈值,为及时发现聚集性病例或暴发疫情提供早期预测预警,而且能够对类似本案例这种以高度散发形式存在的、在较长时间范围内跨地域传播的病例进行溯源调查分析,查找出各病例间的流行病学关联,这是以往靠传统流行病学调查方法无法实现的,是现代分子生物学技术与现场流行病学结合的产物。在美国“鼠伤寒沙门菌污染花生酱事件”、英国“吉百利巧克力事件”、欧洲“O104出血性大肠杆菌”暴发疫情及国内类似疫情的处置过程中,以PFGE技术为代表的网络化监测溯源平台就发挥了不可替代的重要作用[5-7]。在本研究中,我们就利用该技术将分布于不同地区3家医院的斯坦利沙门菌散发性感染病例成功建立了流行病学关联。PFGE聚类分析技术虽然是比较成熟的技术体系,但是想要充分发挥它的作用,仍需要在试剂、方法、人员操作、分析算法等各个环节做好质量控制和标准化工作,否则就容易在病例间建立错误的关联,将流行病学调查指向错误的方向[8]。本研究中,STN2带型尽管和STN1带型差异度较大,但流行病学调查显示该病例与STN1带型病例间存在密切关联性,需要我们做进一步的研究分析。这不仅说明PFGE技术在分辨度和识别力方面仍具有一定的技术局限性,更提醒我们在实践中要充分结合和深入分析病原学与流行病学信息,发挥各自优势的同时紧密合作,才能更好把握疾病传播特点,发挥PFGE技术在疾病防控、突发公卫事件/疫情处置中的作用。
本研究中,15株斯坦利沙门菌药敏测试显示其对临床常用的三代、四代头孢类抗生素,三代氟喹诺酮类抗生素和增效磺胺类等9种抗生素均呈耐药性。从河南省近5年的沙门菌药敏测试数据看,耐9种以上抗生素的多重耐药菌一般只会出现在鼠伤寒、肠炎、阿贡那、德尔比等常见沙门菌血清型上,类似斯坦利沙门菌这种罕见血清型别的沙门菌株对绝大部分抗生素都是敏感的,因此我们认为这种耐药表型与PFGE带型聚类一致性的特点,更好的说明了基于共同暴露源的菌株往往具有相同的耐药基因和表型特征,这种特征尽管存在变异性和不确定性,但在一定范围内也可以为及早发现聚集性病例提供重要线索。
由于客观原因在调查过程中我们没能从ICU病房内采集环境样本进行分离检测,本聚集性病例的调查没有形成完整的病原学证据链,只能推论为医院ICU病房消毒不彻底和转院导致的院内继发性感染。这是河南省首次利用分子分型网络技术平台开展食源性疾病病例调查和危险因素分析研究,为今后相关工作的开展奠定了基础。
参考文献:
[1] Wang LL,Ma XC,Teng RM,et al.An etiological survey and traceability analysis on a foodborne illness epidemic outbreak caused by Salmonella enteritidis[J].Chin J of Prevent Medi, 2015.49(1):60-62.(in chinese)
王丽丽, 马晓晨, 滕仁明,等.一起由肠炎沙门菌所致食源性疾病暴发疫情的病原学研究及溯源分析[J].中华预防医学杂志,2015.49(1):60-62
[2] Li BS,Ke BX,He DM,et al.Antimicrobial resistance patterning and pulsed field gel electrophoresis (PFGE) typing for non -typhoidalSalmonellaisolated from diarrhea cases in Guangdong province, China[J]. Chin J of Microbiol and Immunol, 2012, 32(06):542-528.(in chinese)
李柏生,柯碧霞,何冬梅,等.广东省腹泻病例非伤寒沙门菌耐药谱和pfge分型研究[J]. 中华微生物学和免疫学杂志, 2012, 32(06):542-528.
[3] Yang X,Kuang D,Meng J, et al. Antimicrobial resistance and molecular typing ofSalmonellastanleyisolated from humans, foods, and environment[J]. Foodborne Pathog Dis, 2015, (10):945-949.DOI: 10.1089/fpd.2015.2010.
[4] Springer B,Allerberger F,Kornschober C. Letter to the editor:Salmonellastanleyoutbreaks--a prompt to reevaluate existing food regulations[J]. Euro Surveill, 2014, 19(22) pii:20818. DOI:10.2807/1560-7917.ES2014.19.22.20818.
[5] Laconcha I,Baggesen DL,Rementeria A,et al. Genotypic characterisation by pfge of Salmonella enterica serotype enteritidis phage types 1, 4, 6, and 8 isolated from animal and human sources in three european countries[J]. Vet Microbiol, 2000, 75(2): 155-165. DOI:10.1016/S0378-1135(00)00208-X
[6] Xu XB,Chen M,Tu LH,et al. Atypical molecular traceability ofSalmonellasenftenbergstrains of diarrhea[J].Chin J Labmed,2009, 32(11):1239-1241.(in chinese)
许学斌,陈敏,屠丽红,等.不典型山夫登堡沙门菌腹泻株的分子溯源研究[J]. 中华检验医学杂志, 2009, 32(11):1239-1241
[7] Löfström C,Eriksson J, Aspan, et al. Aimprovement and validation of rapd in combination with pfge analysis ofSalmonellaentericassp. enterica serovar senftenberg strains isolated from feed mills[J]. Vet Microbiol, 2006, 114(3): 345-351. DOI:10.1016/j.vetmic.2005.12.015
[8] Chen MH, Hwang WZ, Wang SW, et al. Pulsed field gel electrophoresis (pfge) analysis for multidrug resistantSalmonellaentericaserovar schwarzengrund isolates collected in six years (2000-2005) from retail chicken meat in Taiwan[J]. Food Microbiol, 2011, 28(3): 399-405. DOI: 10.1016/j.fm.2010.10.002
DOI:10.3969/j.issn.1002-2694.2016.05.016
通讯作者:夏胜利,Email:xiasl@hncdc.com.cn
中图分类号:R378
文献标识码:A
文章编号:1002-2694(2016)05-497-05
Corresponding author:Xia Sheng-li, Email: xiasl@hncdc.com.cn
收稿日期:2015-10-25修回日期:2016-03-11
Pathogenic investigation and molecular tracing analysis ofSalmonellastanleyclusters cases in Henan Province
ZHAO Jia-yong1, HAO Bao-lin2, XIE Zhi-qiang3,HUANG Xue-yong4, MU Yu-jiao5, XIA Sheng-li6
(InstituteforInfectiousDiseaseControlandPrevention,HenanProvincialCenterforDiseaseControlandPrevention,Zhengzhou450016,China)
Abstract:In order to analyze the etiological characteristics and molecular epidemiological relationship of Salmonella stanley (S. stanley) strains isolated from infant patients in three hospitals of Henan Province from January to July in 2013 and provide new ideas and models for foodborne illness's clustered and outbreak cases investigations and hospital infection prevention and control, we collected and isolated diarrhea stool of hospitalized infants cases in three hospitals of two cities. By using SBG enrichment and Salmonella CHROMagar selective media, suspicious strains were identified and been made Salmonella O antigen and H1/ 2 phase flagellum-induced serum agglutination test by API20E biochemical systems and SSI Salmonella typing sera. According to molecular typing and Salmonella K-B drug susceptibility testing method published by the international PulseNet bacterial infectious disease monitoring network and USA clinical and Laboratory Standards Institute (CLSI), we analyzed drug sensitivity to 17 kinds antibiotics and PFGE molecule characteristics.Fifteen strains of S. stanley were identified in this research and antibiotics susceptibility testing showed they were sensitive to ciprofloxacin, levofloxacin, cotrimoxazole, tetracycline, imipenem and other 8 kinds of antibiotics and resistant to 9 kinds of antibiotics with 12 strains having the same AST phenotypes. Fifteen strains of S. stanley were divided into 2 molecular patterns by digestion with XbaI/BlnI and pulsed field gel electrophoresis (PFGE), STN1 pattern contains 14 strains and STN2 pattern contains 1 strains. There was a bigger difference between STN1 and STN2 belts in similarity though they existed epidemiological link. STN1 was confirmed as a unique S. stanley type in Henan Province by "PulseNet China" database comparison. The PFGE technology confirmed the molecular epidemiological relevance among S. stanley infection cases, providing the technical support for further epidemiological tracing and investigation.
Key words:S. stanley; clusters cases; AST; PFGE Supported by the National Science and Technology Major Project(Nos. 2012ZX10004201& 2013ZX10004203)
