结核分枝杆菌rpoB基因突变特征与利福平耐药水平关系的研究
2016-07-28胡族琼刘燕文高璐璐陈俊宇谢伟胜周德旺曾少芳钟炳棠
胡族琼,刘燕文,周 文,高璐璐,陈俊宇,谢伟胜,周德旺,曾少芳,钟炳棠
结核分枝杆菌rpoB基因突变特征与利福平耐药水平关系的研究
胡族琼1,刘燕文1,周文2,高璐璐3,陈俊宇1,谢伟胜1,周德旺1,曾少芳1,钟炳棠1
1.国家呼吸疾病重点实验室结核病室,广州市胸科医院检验科,广州510095;2.广州市越秀区疾病预防控制中心,广州510513;3.广州空军后勤部,广州510523
摘要:目的了解结核分枝杆菌rpoB基因突变特征与利福平耐药水平的关系。方法测定266株(109株利福平耐药株,157株利福平敏感株)结核分枝杆菌利福平MIC值及其rpoB基因序列,分析不同突变特征的菌株其利福平MIC值的差异。结果109株利福平耐药株rpoB基因皆发生错义突变,常见突变密码子531与526的突变频率分别为69.7%与17.4%。利福平耐药株中单密码子突变株占82.6%,多重密码子突变株占17.4%。531单密码子突变株与526单密码子突变株之间平均利福平MIC值差异无统计学意义(P=0.87),但两独立样本T检验分析显示,多重密码子突变株平均利福平MIC值(115.8 μg/mL)显著高于单密码子突变株平均利福平MIC值(48.4 μg/mL)(t=2.659,P=0.016)。利福平敏感株未发生错义突变。5.5%(6/109)的利福平耐药株仅在rpoB基因起始端发生突变,突变密码子分别是247,251和253。结论531与526是最常见突变密码子,且以单密码子突变为主;531与526单密码子突变株之间利福平耐药水平差异无统计学意义,但多重密码子突变株利福平耐药水平显著高于单密码子突变株利福平耐药水平;rpoB基因起始端突变可能是除利福平耐药决定区域之外导致利福平耐药的第2个耐药决定区;rpoB基因DNA序列分析有助于结核分枝杆菌利福平耐药表型和耐药水平的预测。
关键词:结核分枝杆菌;rpoB突变;利福平;耐药水平;最低抑菌浓度
Supported by the Research Project on Medical Science in Guangdong Province(No. A2013530)
利福平是一线抗结核药物中的关键药物,其耐药机制与编码RNA聚合酶β-亚基的rpoB基因突变有关,特别与该基因中利福平耐药决定区(rifampicin resistance-determining region, RRDR)的突变有关[1-2]。利福平耐药结核分枝杆菌通常同时对异烟肼耐药,从而容易形成耐多药结核病(multidrug-resistant tuberculosis, MDR-TB)。有效控制耐多药结核病的传播是降低结核病致死率的有效措施,但是耐多药结核病难以治愈,费用昂贵,常依赖于毒副作用更大的二线抗结核药物[3-7]。在全球抗结核药物匮乏的现况下,高剂量莫西沙星治愈低水平氟喹诺酮类耐药结核病和高剂量异烟肼治疗耐多药结核病疗效显著的报道[8-9]提示,提高利福平剂量有可能治愈低水平利福平耐药的结核病。利福平耐药水平是通过测定利福平最低抑菌浓度(minimal inhibitory concentration, MIC)值来表示,而利福平MIC值的测定依赖于结核分枝杆菌的生长,结核分枝杆菌生长缓慢延迟了利福平耐药水平的获得。
了解rpoB基因突变特征与利福平耐药水平的关系有利于快速获得利福平耐药水平以便于制定治疗方案和阐明利福平耐药机制。目前为止,仅少数研究人员报道过利福平耐药水平与rpoB基因突变特征之间的关系[10-18]。但可能由于资源不足,他们多采用小样本研究而达不到统计分析的样本量要求,且研究结果常因地域不同而发生分歧[10-18]。为此,本研究采用266株结核分枝杆菌测定其利福平MIC值和rpoB基因核苷酸序列,以探索rpoB基因突变特征与利福平耐药水平之间有无相关性。
1材料与方法
1.1菌株266株结核分枝杆菌分离自2012年8月至2013年11月来广州市胸科医院就诊的门诊患者和住院患者,包括初诊患者和复诊患者,菌株无重复收集。患者年龄12~79岁,平均年龄46.5岁,男性190人,女性76人。患者来自广东194人,广西14人,海南16人,福建17人,四川16人,江西9人。所有患者在本次研究进行之前已用MGIT 960(Becton & Dickinson)完成常规一线药物的敏感性测定,为了保证研究结果的准确性,在本研究进行之前又采用同样方法单独对利福平敏感性进行测定。标准株H37Rv作利福平敏感株对照加入至本研究中。
1.2MIC值测定先配制7H9肉汤(4.7 g 7H9琼脂粉+蒸馏水900 mL+甘油2 mL),高压10 min,使用前加入100 mL MGIT生长添加剂OADC,以此肉汤现用现配制利福平溶液与细菌悬液。取96孔板1个,第1孔与第3~12孔加入100 μL 7H9肉汤,取200 μL配制好的利福平药液至第2孔,再从第2孔取100 μL至第3孔混匀后再取100 μL至第4孔,以此类推至第11孔混匀后弃去100 μL含利福平的7H9混合液,第12孔作无菌无药的阴性对照,再加入6×106CFU/ML的菌液100 μL至第1到第11孔。考虑到利福平耐药株的MIC值可能比较高,敏感株的MIC值可能比较低,因此分别配制不同浓度的利福平溶液。最终用于测定耐药株MIC值的96孔板上的利福平终浓度分别为640、320、160、80、40、20、10、5、2.5、1.25 μg/mL;测定敏感株的利福平终浓度分别为80、40、20、10、5、2.5、1.25、0.625、0.312、0.156 μg/mL。封板后置37 ℃孵育4周观察结果,读取最低抑菌浓度值。
1.3引物设计根据基因库记载的rpoB基因(Rv0677)序列,利用软件Primer Premier 5.0(Premier, Canada) 设计引物KH-12035(5′-CGAGGACTTGACGGCAGACGCT-3′)和KH-12036(5′-AACCACGCCGTCGACCACCT-3′),由上海生工合成。PCR产物全长1 924 bp包括rpoB起始密码子上游-370 nt至+1554 nt。其余测序引物(HC-11070-F1: 5′-CCGATGATGACCGAGAA-3′和HC-11072-F2: 5′-CCACGATGACCGTTCCG-3′由测序公司华大基因公司合成。
1.4DNA提取DNA提取试剂采用德国进口的GenoLyse VER 1.0(Hain Lifescience GmbH, Nehren, Germany)试剂盒,具体操作过程详见说明书。
1.5扩增目的基因PCR反应体系50 μL,分别由0.25 μL PrimerSTAR HSpolymerase(TaKaRa)、5 μL 10×Primer STAR buffer(TaKaRa)、8 μL 2.5 mmol/L dNTP mixture(TaKaRa)、0.2 μL引物KH-12035(20 μmol/L)、0.2 μL引物KH-12036(20 μmol/L)、31.35 μL灭菌水、5 μL DNA模板组成50 μL总反应体系。PCR反应条件:94 ℃,变性3 min;94 ℃,1 min, 60 ℃,1 min , 72 ℃,1 min,30个循环;最后72 ℃延伸5 min。PCR产物全长1 924 bp。
1.6DNA测序及序列分析PCR产物经琼脂糖凝胶电泳后送华大基因公司测序,华大基因公司先对PCR产物纯化后再测序。采用标准Sanger DNA测序法在3730XL(Applied Biosystems)测序仪上进行测定,所测序列由生物信息软件Clustalx 和 DANstar联合分析,分析结果见表1和表2。
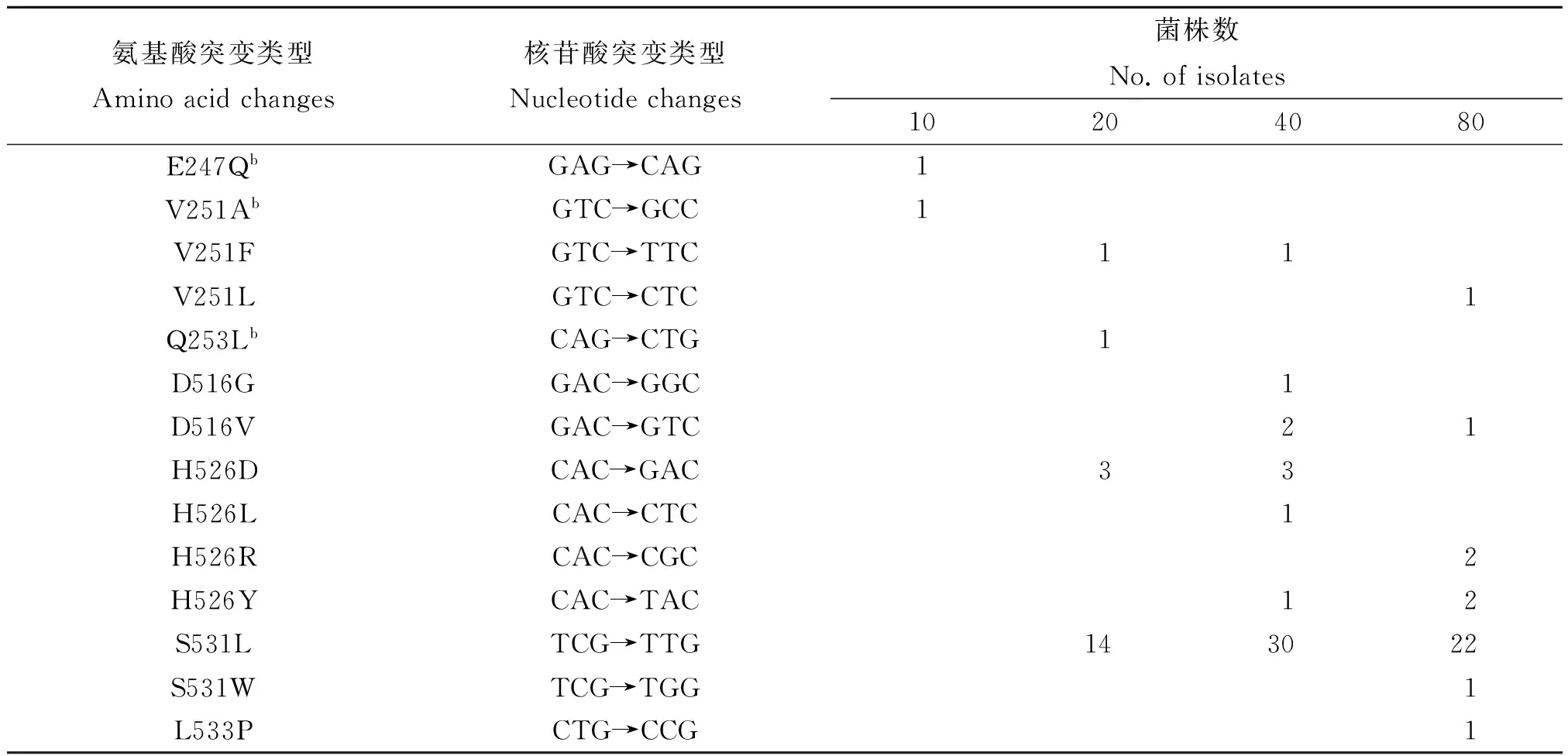
表1 90株利福平耐药结核分枝杆菌的单密码子突变类型及其利福平MIC值(μg/mL)
b.本次研究中的新发突变类型。
b. The novel mutations in this study.
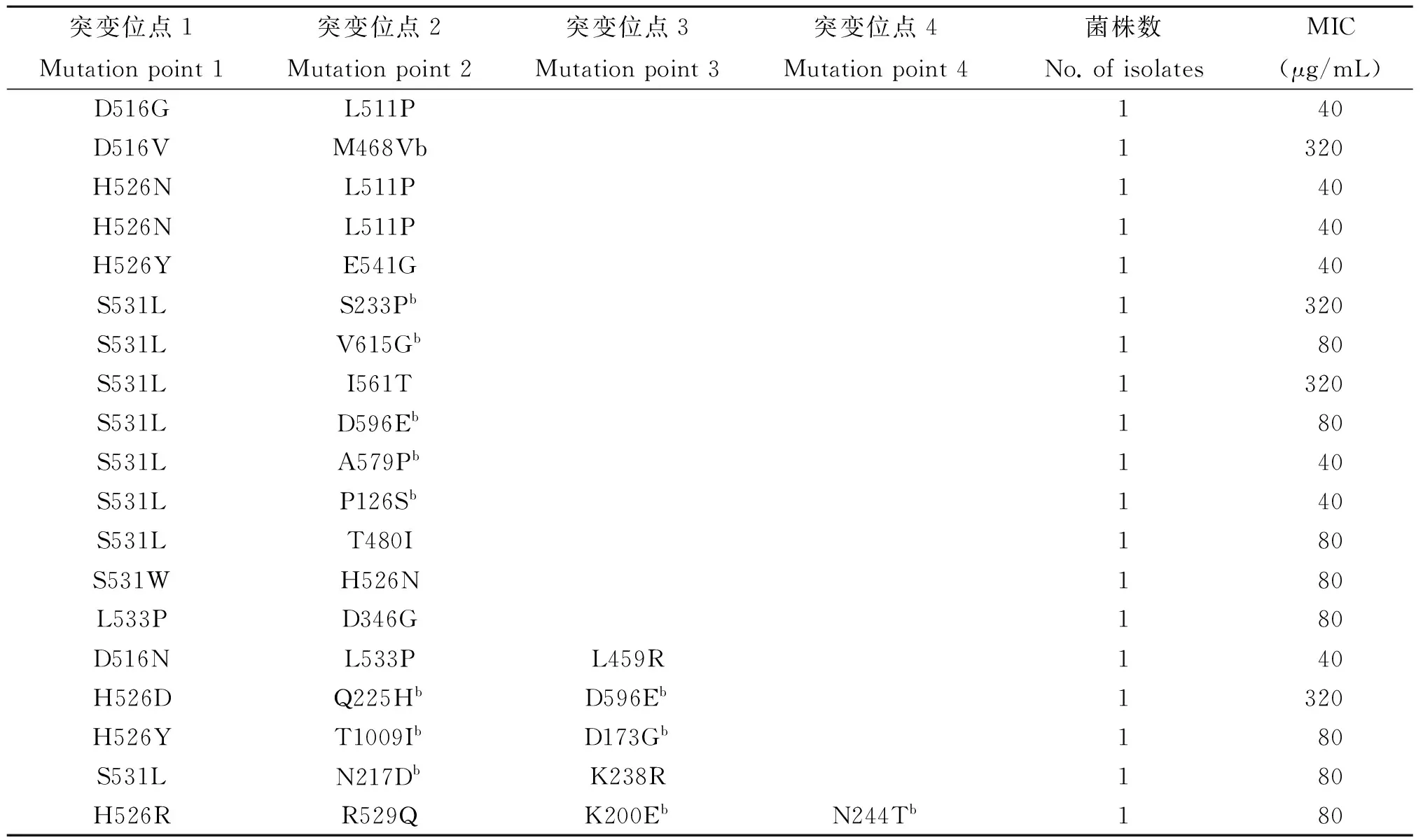
表2 19株利福平耐药结核分枝杆菌rpoB基因的多重突变类型及其利福平MIC值
b.本次研究中的新发突变类型。
b. The novel mutations in this study.
1.7统计学分析使用SPSS 18.0软件(SPSS Inc., Chicago, IL, USA)t检验分析rpoB单重突变和多重突变菌株之间利福平MIC值差异,以及常见526单密码突变株和531单密码突变株之间利福平MIC值差异,P<0.05为差异有统计学意义。
2结果
2.1利福平药敏结果266株临床菌株中,利福平敏感株157株,利福平耐药株109株。标准株H37Rv为利福平敏感株。全部利福平耐药菌株的MIC值见表1和表2,敏感株的MIC值见表3。
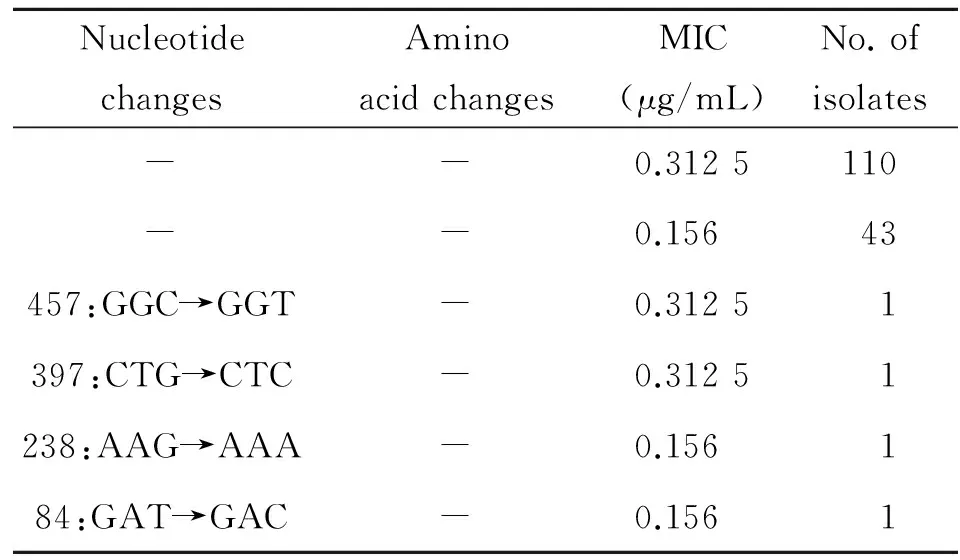
表3 157株利福平敏感株rpoB基因突变类型及其利福平MIC值
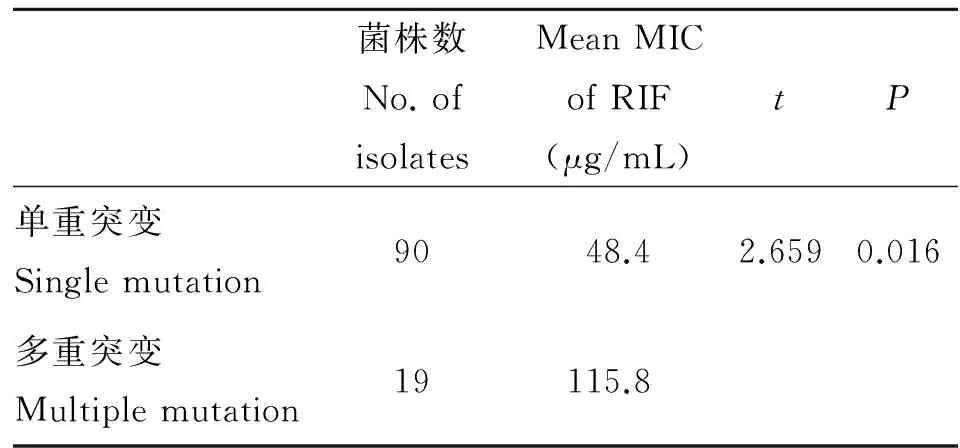
表4 单重突变株与多重突变株RIF平均MIC值比较
2.2rpoB基因的突变类型与分布情况根据我们之前的研究结果以及本研究进行前对16株菌株的全序列测定结果显示,绝大部分单密码子突变发生在rpoB基因前半部分,主要发生在第593位密码子之前[19]。为了降低研究成本,本研究主要对第593位密码子之前的序列进行分析。结果显示,一共发现36种错义突变形式,其中15种突变是本次研究新发现的突变类型,所有突变形式中14种突变类型为单密码子突变(n=90),多重突变类型有19种,分别发生于19株菌株中,见表1和表2。利福平耐药株rpoB基因中531(69.7%)与526(17.4%)密码子依然是最常见的突变密码子。94.5%(103/109)的利福平耐药株至少有1个突变位点发生在RRDR之内,其余5.5%(6/109)利福平耐药株所有突变位点都发生在RRDR之外(V251A [n=1], V251F [n=2], V251L [n=1], Q253L [n=1], E247Q [n=1])。有2株利福平耐药株的同义突变发生在RRDR之内(L533, 533:CTG→CTC和T525, 525: ACC→ACG),还有2株利福平耐药株的同义突变发生在RRDR之外(P480, 480:CCA→CCG和I569, 569: ATC→ATT)。H37Rv无任何突变发生。4株利福平敏感株在RRDR之外发生同义突变(G457,457:GGC→GGT; L397, 397:CTG→CTC; K238, 238:AAG→AAA; D84, 84: GAT→GAC),其余利福平敏感株的rpoB基因未发现任何突变。
2.3利福平耐药株MIC值与rpoB基因突变类型的关系本研究中,109株利福平耐药株的耐药水平在10~320 μg/mL之间,其平均MIC值为60.15 μg/mL(见表1和表2);157株利福平敏感株的利福平最低抑菌浓度值为0.156~0.312 5 μg/mL之间,其平均MIC值为0.27 μg/mL。利福平耐药株的平均MIC值显著高于利福平敏感株的平均MIC值(t=9.955,P=0.000)。4株251单密码子突变(V251A[n=1],V251F[n=2],V251L[n=1])的利福平耐药株其MIC值范围在10~80 μg/mL之间;4株516单密码子突变株(D516G[n=1],D516V[n=3])的MIC值范围在40~80 μg/mL之间;12株526单密码子突变株的利福平MIC值范围在20~80 μg/mL之间,其平均MIC值为48.3 μg/mL;67株531单密码子突变株的利福平MIC值范围在20~80 μg/mL之间,其平均MIC值为49.6 μg/mL。526单密码子突变株的平均MIC值与531单密码子突变株的平均MIC值比较差异无统计意义(t=0.164,P=0.87)。90株单密码子突变株的利福平平均MIC值为48.4 μg/mL,19株多重密码子突变株的利福平平均MIC值为115.8 μg/mL,该二类菌株利福平平均MIC值比较t检验结果显示,多重突变株MIC值高于单密码子突变株的MIC值(t=2.659,P=0.016)。
3讨论
本次研究结果显示,所有利福平耐药株rpoB基因都发生了错义突变,而利福平敏感株未发生错义突变,该结果与以往研究结果一致,即rpoB基因编码的RNA多聚酶β亚基的氨基酸突变是利福平耐药的主要机制[2]。
序列分析显示,266株菌株的rpoB基因一共发生了36种错义突变形式,另外还发现8种无意义突变分别存在于4株利福平耐药株和4株利福平敏感株中,这类无意义突变将导致利用分子诊断技术检测利福平耐药的准确性降低。526和531仍然是常见突变密码子,其突变频率分别是17.4%和69.7%,与其他地区的研究结果类似[20-25]。109株利福平耐药株中,5.5%(n=6)的菌株rpoB基因突变皆位于RRDR之外的rpoB基因起始端,其利福平耐药程度与516、526、531单密码子突变株相近。由于本研究采用大肠杆菌编码系统进行密码子命名,如果采用结核分枝杆菌编码系统进行命名,251密码子在结核分枝杆菌编码系统中则是176密码子,该密码子是以往研究者们较常发现的一个位于rpoB基因起始端的密码子[19,26-28]。密码子253和247在结核分枝杆菌编码系统中分别是178和172密码子,皆位于rpoB基因起始端。本次研究发现位于rpoB基因起始端的密码子突变频率大于我们上次的研究结果[19],该结果提示,除RRDR之外,将rpoB基因起始端作为检测靶标诊断利福平敏感性将会提高现有分子诊断技术的敏感性。
t检验分析526和531单密码子突变株之间利福平平均MIC值结果显示差异无统计学意义(P=0.87),我们同时分析美国报道的516,526,531和533单密码子突变株之间的利福平平均MIC值,其结果也显示无显著性差异(F=2.587,P=0.058)[29],本次研究结果和美国的研究结果同时显示没有发现rpoB基因突变位置与利福平耐药水平有联系。美国和意大利分离到的526单密码子突变株和531单密码子突变株的利福平平均耐药水平分别显著高于本次研究分离到的526单密码子突变株和531单密码子突变株[16,29]。可见对于日本分离到利福平MIC值分别为1 μg/mL和0.5 μg/mL的533单密码子突变株而本次研究却分离到利福平MIC值为80 μg/mL的533单密码子突变株是不足为奇的[30]。并且日本研究者发现531突变株的利福平MIC值都≥64 μg/mL,相反,在利福平MIC值≤1 μg/mL的菌株中未发现516,526或531突变株存在[30]。但本次研究中发现65.7%(44/67)的531单密码子突变株其利福平MIC值都远低于日本研究者发现的64 μg/mL[30]。另外我们还发现251单密码子突变株的利福平MIC值范围为10~80 μg/mL。上述分析显示,不同地域之间的结核分枝杆菌即使rpoB基因突变位点相同,其利福平耐药水平也会不同,而同一地域中的菌株即使rpoB基因突变位点不同,其利福平耐药水平也可能会无显著性差异,综上所述,本次研究未发现rpoB基因突变位置与利福平耐药水平有联系,而不同地域的菌株可能由于其遗传背景的差异却可能导致其利福平耐药水平不同。
本次研究发现的多重突变株(两重突变至四重突变)皆由常见突变密码子516、526、531或533密码子联合1至3个其他密码子突变而来,其平均利福平MIC值为115.8 μg/mL。这些多重密码子突变株的平均利福平MIC值与单重密码子突变株的平均利福平MIC值t检验分析结果显示,多重密码子突变株的平均利福平MIC值显著高于单重密码子突变株的平均利福平MIC值(t=2.659,P=0.016),该结果恰好与美国研究者报道的多重密码子突变与高水平利福平耐药有关及其多重突变株利福平MIC值基本≥100 μg/mL的结果相符合[17]。但与国内谭守勇等报道的531密码子突变以及多重密码子联合突变与利福平高水平耐药有关[31],以及北京黄海荣等[32],上海庞茂银等[33]报道的531突变与利福平高水平耐药有关存在部分差异。本研究发现的531单密码子突变株利福平MIC值低于他们所报道的,可能与他们所测序列仅涵盖rpoB基因RRDR附近区域的核苷酸序列有关。其中谭守勇等所测序列仅为RRDR附近的184 bp[31],黄海荣所测序列为RRDR附近的588 bp[32],庞茂银所测序列为RRDR附近的213 bp[33],而本研究所测区域为rpoB基因起始密码子前-370 bp至+1 554 bp内的核苷酸序列(为1 924 bp),范围远大于他们所测序列,因此推测他们所发现的与利福平高水平耐药有关的531突变株可能不是单密码子突变株,也许在他们测定区域之外还有其他密码子突变存在。
总之,本研究结果提示结核分枝杆菌rpoB基因的突变位置可能只与利福平耐药表型有关而未发现其与利福平耐药水平有联系,但多重突变可能导致利福平耐药水平显著高于单重突变株,因此rpoB基因的突变位置不能用于预测利福平耐药水平,但多重突变却可能是利福平高水平耐药的标志。另外,rpoB基因起始端单密码子突变株的发现,提示除RRDR之外将rpoB基因起始端作为第二个靶标用以检测利福平敏感性有可能提高现有分子诊断技术的准确性。
参考文献:
[1]Cole ST, Telenti A. Drug resistance inMycobacteriumtuberculosis[J].Eur Respir J, 1995, 20(Suppl): 701s-713s.
[2]Telenti A, Imboden P, Marchesi F, et al. Detection of rifampicin-resistance mutations inMycobacteriumtuberculosis[J].Lancet, 1993, 341: 647-651. DOI: 10.1016/0140-6736(93)90417-F
[3]Fisher M. Diagnosis of MDR-TB: a developing world problem on a developed world budget[J].Expert Rev Mol Diagn, 2002, 2(2): 151-159. DOI: 10.1586/14737159.2.2.151
[4]Resch SC, Salomon JA, Murray M, et al. Cost-effectiveness of treating multidrug-resistant tuberculosis[J].PLoS Med, 2006, 3(7): e241. DOI: 10.1371/journal.pmed.0030241
[5]Pablos-Mendez A, Gowda DK, Frieden TR. Controlling multidrug-resistant tuberculosis and access to expensive drugs: a rational framework[J].Bull World Health Organ, 2002, 80(6): 489-500.
[6]Tupasi TE, Quelapio MI, Orillaza RB, et al. DOTS-plus for multidrug-resistant tuberculosis in the Philippines: global assistance urgently needed[J].Tuberculosis(Edinb), 2003, 83(1-3): 52-58. DOI: 10.1016/S1472-9792(02)00072-0
[7]O'Brien RJ, Nunn PP. The need for new drugs against tuberculosis. Obstacles, opportunities, and next steps[J].Am J Respir Crit Care Med, 2001, 163(5): 1055-1058.
[8]Poissy J, Aubry A, Fernandez C, et al. Should moxifloxacin be used for the treatment of extensively drug-resistant tuberculosis? An answer from a murine model[J].Antimicrob Agents Chemother, 2010, 54(11): 4765-4771. DOI: 10.1128/AAC.00968-10
[9]Katiyar SK, Bihari S, Prakash S, et al. A randomised controlled trial of high-dose isoniazid adjuvant therapy for multidrug-resistant tuberculosis[J].Int J Tuberc Lung Dis, 2008, 12(2): 139-145.
[10]Hwang HY, Chang CY, Chang LL, et al. Characterization of rifampicin resistantMycobacteriumtuberculosisin Taiwan[J].J Med Microbiol, 2003, 52(Pt3): 239-245. DOI: 10.1099/jmm.0.05045-0
[11]Taniguchi H, Aramaki H, Nikaido Y, et al. Rifampicin resistance and mutation of the rpoB gene inMycobacteriumtuberculosis[J].FEMS Microbiol Lett, 1996, 144(1): 103-108.
[12]Bodmer T, Zurcher G, Imboden P, et al. Mutation position and type of substitution in the β-subunit of the RNA plymerase influencein-vitroactivity of rifamycins in rifampicin-resistantMycobacterium.tuberculosis[J].J Antimicrob Chemother, 1995, 35(2): 345-348. DOI: 10.1093/jac/35.2.345
[13]Bartfai Z, Somoskovi A, Kodmon C, et al. Molecular characterization of rifampin-resistant isolates ofMycobacteriumtuberculosisfrom Hungary by DNA sequencing and the line probe assay[J].J Clin Microbiol, 2001, 39: 3736-3739. DOI: 10.1128/JCM.39.10.3736-3739.2001
[14]Huang H, Jin Q, Ma Y, et al. Characterization of rpoB mutations in rifampin-resistantMycobacteriumtuberculosisisolated in China[J].Tuberculosis, 2002, 82: 79-83. DOI: 10.1054/tube.2002.0326
[15]Mikhailovich V, Lapa S, Gryadunov D, et al. Identification of rifampin-resistantMycobacteriumtuberculosisstrains by hybridization, PCR, and ligase detection reaction on oligo nucleotide microchips[J].J Clin Microbiol, 2001, 39: 2531-2540. DOI: 10.1128/JCM.39.7.2531-2540.2001
[16]Pozzi G, Meloni M, Iona E, et al. rpoB mutations in multidrug resistant strains ofMycobacteriumtuberculosisisolated in Italy[J].J Clin Microbiol, 1999, 37(4): 1197-1199.
[17]Bahrmand AR, Titov LP, Tasbiti AH, et al. High-level rifampin resistance correlates with multiple mutations in the rpoB gene of pulmonary tuberculosis isolates from the Afghanistan Border of Iran[J].J Clin Microbiol, 2009, 47(9): 2744-2750. DOI: 10.1128/JCM.r00548-09
[18]Huitric E, Werngren J, Jureen P, et al. Resistance levels and rpoB gene mutations amonginvitro-selected rifampin-resistantMycobacteriumtuberculosismutants[J].Antimicrob Agents Chemother, 2006, 50(8): 2860-2862. DOI: 10.1128/AAC.00303-06
[19]Tan YJ, Hu ZQ, Zhao YL, et al. The beginning of the rpoB gene in addition to the RRDR might be needed for identifying RIF/Rfb cross resistance in multidrug-resistantMycobacteriumtuberculosisisolates from Southern China[J].J Clin Microbiol, 2012, 50(1): 81-85. DOI: 10.1128/JCM.05092-11
[20]Bakonyte D, Baranauskaite A, Cicenaite J, et al. mutations in the rpoB gene of rifampin-resistantMycobacteriumtuberculosisclinical isolates from Lithuania[J].Int J Tuberc Lung Dis, 2005, 9(8): 936-938.
[21]Kapur V, Li LL, Iordanescu S, et al. Characterization by automated DNA sequencing of mutations in the gene(rpoB) encoding the RNA polymerase β-subunit in rifampin-resistantMycobacteriumtuberculosisstrains from New York and Texas[J].J Clin Microbiol, 1994, 32(4): 1095-1098.
[22]Namaei MH, Sadeghian A, Naderinasab M, et al. Prevalence of primary drug resistantMycobacteriumtuberculosisin Mashhad Iran[J].Indian J Med Res, 2006, 124(1): 77-80.
[23]Mokrousov I, Otten T, Vyshnevskiy B, et al. Allele specific rpoB PCR assays for detection of rifampin-resistantMycobacteriumtuberculosisin sputum smears[J].Antimicrob Agents Chemother, 2003, 47: 2231-2235. DOI: 10.1128/AAC.47.7.2231-2235.2003
[24]Sajduda A, Brzostek A, Poplawska M, et al. Molecular characterization of rifampin- and isoniazid-resistantMycobacteriumtuberculosisstrains isolated in Poland[J].J Clin Microbiol, 2004, 42(6): 2425-2431. DOI: 10.1128/JCM.42.6.2425-2431.2004
[25]Telenti A, Honore N, Bernasconi C, et al. Genotyping assessment of isoniazid and rifampin resistance inMycobacteriumtuberculosis: a blind study at reference laboratory level[J].J Clin Microbiol, 1997, 35(3): 719-723.
[26]Heep M, Rieger U, Beck D, et al. Mutations in the beginning of the rpoB gene can induce resistance to rifamycins in bothHelicobacterpyloriandMycobacteriumtuberculosis[J].Antimicrob Agents Chemother, 2000, 44(4): 1075-1077. DOI: 10.1128/AAC.44.4.1075-1077.2000
[27]Heep M, Brandstatter B, Rieger U, et al. Frequency of rpoB mutations inside and outside the cluster I region in rifampin-resistant clinicalMycobacteriumtuberculosisisolates[J].J Clin Microbiol, 2001, 39(1): 107-110. DOI: 10.1128/JCM.39.1.107-110.2001
[28]Siu GK, Zhang Y, Lau TC, et al. Mutations outside the rifampicin resistance-determining region associated with rifampicin resistance inMycobacteriumtuberculosis[J].J Antimicrob Chemother, 2011, 66(4): 730-733. DOI: 10.1093/jac/dkq519
[29]Cummings MP, Segal MR. Few amino acid positions in rpoB are associated with most of the rifampin resistance inMycobacteriumtuberculosis[J].BMC Bioinformatics, 2004, 5: 137. DOI: 10.1186/1471-2105-5-137
[30]Ohno H, Koga H, Kohno S, et al. Relationship between rifampin MICs for and rpoB mutations ofMycobacteriumtuberculosisstrains isolated in Japan[J].Antimicrob Agents Chemother, 1996, 40(4): 1053-1056.
[31]Tan SY, Tan YJ, Li YQ, et al. Mutation of DNA fragment of rpoB gene in different degrees of rifampin-resistance inMycobacteriumtuberculosis[J].Chin J Pract Intern Med, 2004, 24(1): 27-29.(in Chinese)
谭守勇, 谭耀驹, 黎燕琼. 对利福平耐药的结核分枝杆菌rpoB基因突变与耐药程度关系的探讨[J].中国实用内科杂志, 2004, 24(1):27-29.
[32]Huang HR, Jin Q, Ma Y, et al. Characterization of rpoB mutation in rifampin2resistant clinicalMycobacteriumtuberculosisisolates from China[J].Chin J Tuberc Respir Dis, 2001, 24(4): 231-235.(in Chinese)
黄海荣, 金奇, 马玙, 等. 中国耐利福平结核分枝杆菌rpoB基因突变特点[J].中华结核和呼吸杂志, 2001, 24(4):231-235.
[33]Pang MY, Zhang WH, Chen S, et al. Relativity of rpoB gene mutations inMycobacteriumtuberculosismulti-drug resistance and degree of resistance to rifampin[J].Fudan Univ J Med Sci, 2004, 31(1): 228-230, 238.(in Chinese)
庞茂银, 张文宏, 陈澍, 等. 结核分枝杆菌rpoB基因突变与多重耐药及利福平耐药程度的相关性[J].复旦学报(医学版), 2004, 31(1):228-230,238.
DOI:10.3969/j.issn.1002-2694.2016.01.009
中图分类号:R378.91
文献标识码:A
文章编号:1002-2694(2016)01-0039-06
收稿日期:2015-05-04;修回日期:2015-08-17
Relationship between rpoB mutations and the levels of rifampicin resistance in M. tuberculosis
HU Zu-qiong1,LIU Yan-wen1,ZHOU Wen2,GAO Lu-lu3,CHEN Jun-yu1,XIE Wei-sheng1,ZHOU De-wang1,ZENG Shao-fang1,ZHONG Bing-tang1
(1.StateKeyLaboratoryofRespiratoryDiseases,DepartmentofClinicalLaboratory,GuangzhouChestHospital,Guangzhou510095,China;2.CentreforDiseaseControlandPreventionofYuexiuDistrict,Guangzhou510513,China;3.LogisticsDepartmentofGuangzhouAirForceMilitaryAreaCommand,Guangzhou510523,China)
Abstract:To learn about the correlation of mutation characterization of rpoB gene and the rifampicin(RIF) resistance levels in M. tuberculosis isolates, the 266 isolates were subjected to DNA sequencing of rpoB genes and to determining their rifampicin MICs, then the rifampicin MICs of isolates with different rpoB mutation profiles were analyzed by t-test. All of the 109 RIF-resistant(RIF-R) isolates had missense mutations in rpoB, but the 157 RIF-susceptible(RIF-S) isolates had not. Codons 531(69.7%) and 526(17.4%) were still the most frequent mutations. The 5.5%(6/109) RIF-R isolates had all mutations only at the beginning of the rpoB (at codon 247, 251 and 253). In addition, the MICs in the 531 single mutants and 526 single mutants showed no significant difference(P=0.87). But the MICs between single mutants and multiple mutants showed statistically significant difference(t=2.659, P=0.016). The 531 and 526 were the most frequent mutation codons in rpoB and the single codon mutation was the major characterization of rpoB in MTB. The significant difference of RIF MICs have not been found between the 531 single mutants and the 526 single mutants, but t-test showed that the RIF MICs of multiple mutants were significantly higher than that of single mutants. Therefore, DNA sequencing of rpoB are helpful for predicting rifampicin resistance phenotype and rifampicin resistance levels in MTB. In addition, mutations identified at the beginning of the rpoB may exhibit another hot-spot region except for RRDR conferring rifampicin resistance in M. tuberculosis.
Keywords:M. tuberculosis; rpoB mutation; rifampicin; resistance level; MICs
广东省医学科研基金项目(A2013530)资助
