RNA碱基的太赫兹光谱和拉曼光谱特征研究
2016-06-05赵东波孙海军刘云飞
王 芳、赵东波、蒋 玲、徐 莉、孙海军、刘云飞*
1. 南京林业大学信息科学技术学院、江苏 南京 210037 2. 南京大学化学工程学院、江苏 南京 210023 3. 南京林业大学现代分析测试中心、江苏 南京 210037 4. 三江学院电子信息工程学院、江苏 南京 210012
RNA碱基的太赫兹光谱和拉曼光谱特征研究
王 芳1、4、赵东波2、蒋 玲1、徐 莉3、孙海军3、刘云飞1*
1. 南京林业大学信息科学技术学院、江苏 南京 210037 2. 南京大学化学工程学院、江苏 南京 210023 3. 南京林业大学现代分析测试中心、江苏 南京 210037 4. 三江学院电子信息工程学院、江苏 南京 210012
应用傅里叶红外光谱仪和激光拉曼光谱仪测试了RNA碱基在太赫兹波段(1~10 THz)的红外和拉曼光谱、同时结合Guassian09软件和周期性边界条件下基于能量的分块方法(PBC—GEBF)、分析了RNA碱基晶体的红外和拉曼光谱特征、得到了所有特征峰位置及其对应的振动模式、且计算光谱与测试光谱一致吻合、表明碱基粉末样品为无定形晶体结构。通过对红外光谱的分析可知、在太赫兹波段、腺嘌呤和鸟嘌呤都有6个红外活性振动模式、胞嘧啶和尿嘧啶分别为6个和3个红外活性振动模式、与实验结果相比、除了鸟嘌呤6.35 THz处的弱吸收峰没能重现、4.83和5.39 THz处的吸收峰简并; 胞嘧啶4.3和4.79 THz处吸收峰简并; 尿嘧啶3.32和3.82 THz处的吸收峰简并外、其他吸收峰的位置和强度均被准确地模拟重现。通过对拉曼光谱的分析可知、理论和实验光谱基本一致、除了尿嘧啶3.52和4.48 THz处特征峰简并; 鸟嘌呤7.26和8.03 THz、3.57、4.02、4.49、4.89和5.98 THz处特征峰简并外、其他特征峰的位置和强度均被准确的模拟重现。通过对特征峰的分析和辨认、可知在1~10 THz、RNA碱基的振动模式均来源于晶格内分子的集体振动、分子间的氢键和弱相互作用力对振动模式的贡献很大、进一步分析可知、在1~5.5 THz、其振动模式来自所有原子参与的集体振动、在5.5~10 THz、振动模式来自于部分原子参与的集体振动。此项研究对揭示RNA碱基在构成生物大分子结构、生物大分子鉴定以及太赫兹波段光谱的形成机制等方面、具有重要的理论和实际参考价值。
RNA碱基; 太赫兹光谱; 拉曼光谱; PBC-GEBF
引 言
核糖核酸(ribonucleic acid、RNA)存在于大多数已知的植物病毒和部分动物病毒以及一些噬菌体中、由磷酸、五碳糖和碱基组成、是生物遗传讯息的中间载体、并参与蛋白质合成及基因表达调控。而作为RNA重要组成部分的碱基(腺嘌呤、鸟嘌呤、胞嘧啶、尿嘧啶)、通过氢键配对形成RNA独特的单链长分子结构。太赫兹(THz)光谱和拉曼光谱都是研究分子结构的有效手段。THz光谱测定的是红外光子照射到物质分子上、因分子振动、转动所产生的共振吸收光谱、适用于红外活性分子测量; 而拉曼光谱测定的分子共振散射光谱、适应于红外非活性分子测量。太赫兹光谱和拉曼光谱在研究上具有互补性、是生物大分子鉴定和结构研究的重要手段[1-2]。闫慧[3]等利用太赫兹时域光谱技术(terahertz time domain spectroscopy、THz-TDS)结合赝势平面波密度泛函方法、开展了DNA碱基分子中胞嘧啶和胸腺嘧啶的太赫兹光谱研究、获得了0.1~3.5 THz范围内所有的红外活性模式。Fisher等[4]利用太赫兹时域光谱技术和密度泛函理论、对DNA碱基的红外吸收谱进行了分析研究、获得了0.5~4.0 THz范围内的所有吸收峰位置、并对胸腺嘧啶的吸收峰进行了辨认。Nishizawa[5]等利用太赫兹波探测器、获得了0.4~5.8 THz范围内五种碱基的吸收谱。这些文献报道的实验数据、为RNA/DNA碱基分子结构及振动模式研究提供了依据、但选取不同的分子动力学模型、其计算出的理论值与实验结果有较大偏差。本工作应用周期性边界条件下基于能量的分块方法(generalized energy-based fragmentation approach under periodic boundary conditions,PBC-GEBF)[6-11]计算RNA碱基晶体的晶格参数、晶格能、几何结构、振动及转动光谱等、并与太赫兹光谱(1~10 THz)和拉曼光谱(3~10 THz)的测试结果进行比对、发现在太赫兹波段(1~10 THz)、RNA碱基的远红外和拉曼光谱的理论值和实验值一致吻合。此研究为揭示RNA碱基在生物大分子构成中的作用及生物大分子鉴定等方面提供非常重要的信息。
1 实验部分
1.1 仪器
拉曼光谱仪为美国Themor公司的DXR532型激光拉曼光谱仪、仪器配置532和780 nm双激光器系统、本研究选择的780 nm激光器; 样品点激光功率控制精度0.1 mW; 拉曼频移范围:100~3 300 cm-1(3~99 THz); 空间分辨率优于1 μm; 光谱重复性:≤±0.2 cm-1; 带有智能的OMNIC软件、可用于光谱分析与处理。
实验使用为德国Bruker 公司的VERTEX 80V型傅里叶红外光谱仪、频谱范围为:15 500~10 cm-1; 仪器配置3个分束器:6、25和50 μm、实验中选择6 μm分束器、有效频率范围为:680~30 cm-1(0.9~20.4 THz); 真空环境下的分辨率可达2 cm-1; 信噪比优于50 000∶1(峰-峰值)。
1.2 样品制备与方法
实验中使用的腺嘌呤A(adenine)、尿嘧啶U(uracil)、胞嘧啶C(cytosine)、鸟嘌呤G(guanine)均购于Sigma-Aldrich公司、样品均为白色或类白色粉末、使用前没有进一步提纯处理。由于样品在太赫兹波段的吸收很强、在进行红外光谱测试前、将纯样品粉末(纯度≥99%)与聚乙烯粉末以1∶10的质量比混合均匀后压成0.5~1 mm厚的薄片、直径为13 mm。同时制备纯聚乙烯样品薄片作为测试背景、纯聚乙烯样品的质量需与测试样品中聚乙烯含量(质量)相同、这样可以排除聚乙烯吸收的影响。拉曼光谱的测试相对简单、只需将纯样品粉末置于载玻片上、然后再盖上一块载玻片、将样品压平后置于拉曼显微镜下、调整焦距寻找最佳测试点即可完成拉曼光谱的测试。
2 分子结构与理论计算
利用Guassian09[12]软件、并应用密度泛函理论(density functional theory、DFT)对RNA碱基晶体进行结构优化和频率计算、使用B3LYP杂化泛函和6-311++G(d,p)基组、计算结果无虚频。在计算过程中引入基于能量分块和考虑周期性边界条件的PBC-GEBF方法、将分子晶体的能量和振动光谱的计算划分为一系列较小的分子团簇、“分而治之”、大大的节约了计算成本、计算光谱的拟合半宽高为4 cm-1。
RNA碱基中胞嘧啶晶体属于正交晶系、其他碱基晶体均属正斜晶系、其中腺嘌呤单晶胞中含有8个分子、其他碱基的单晶胞均由4个分子组成。RNA特有的尿嘧啶(uracil,U)的单分子和单晶胞结构如图1所示、其晶胞结构中含有丰富的N—H…O和C—H…O氢键。理论计算中使用的碱基晶胞几何结构均来自于剑桥晶体库(cambridge structural database、CSD)、对应的refcode分别为:腺嘌呤(KEMDOW)、尿嘧啶(URACIL)、胞嘧啶(CYTSIN)、鸟嘌呤(KOBFUD)。
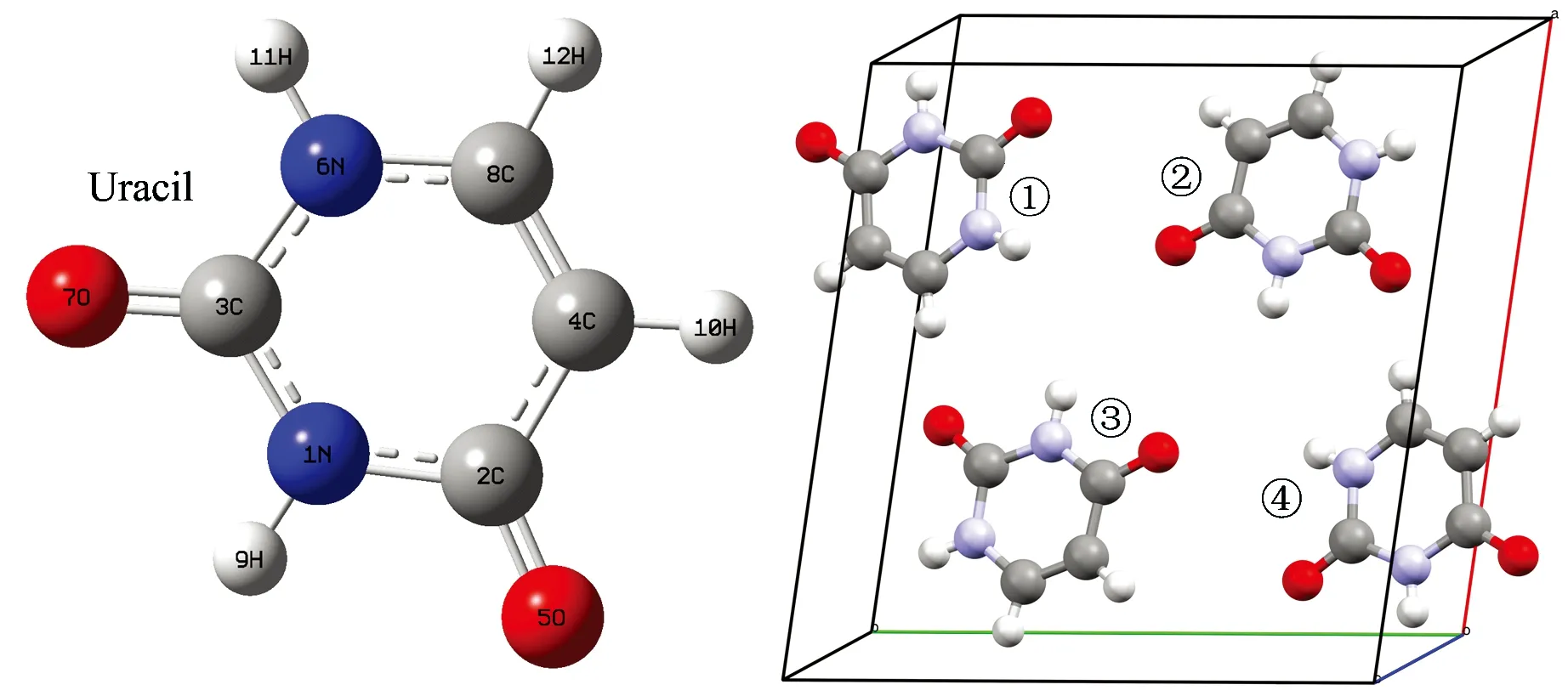
图1 尿嘧啶的单分子和单晶胞结构
表1为RNA碱基的晶格参数理论值和实验值的对比结果、采用PBC-GEBF及基于赝势平面波(pseudo-potential plane wave、PPW)的VASP方法[13]对晶格参数进行理论预测、从表1可以看出、理论值和实验值基本相符、且PBC-GEBF预测较VASP预测更接近实验值、说明PBC-GEBF方法的计算结果更为准确、误差在2%以内、为本研究的首选方法。原子编号参见图1。除了尿嘧啶的C3—N6—H11键角的理论值和实验值相差了0.6°、腺嘌呤的N1—C2—H11和N3—C2—H11键角的理论值和实验值相差了0.1°、其余的键长和键角的理论值和实验值几乎为零偏差、偏差产生的原因是因为在晶体环境中形成了氢键网络导致的结构差异、但是所有偏差都在合理的范围之内、进一步说明了计算结果的可靠性。
表1 RNA碱基晶格参数的理论值(基于PBC-GEBF及基于赝势平面波的VASP方法)和实验值的对比结果、实验数据已加粗
Table 1 Comparison of lattice parameters of RNA nucleobases which predicted by PBC-GEBF and VASP,respectively,with the experimental data as reference in bold
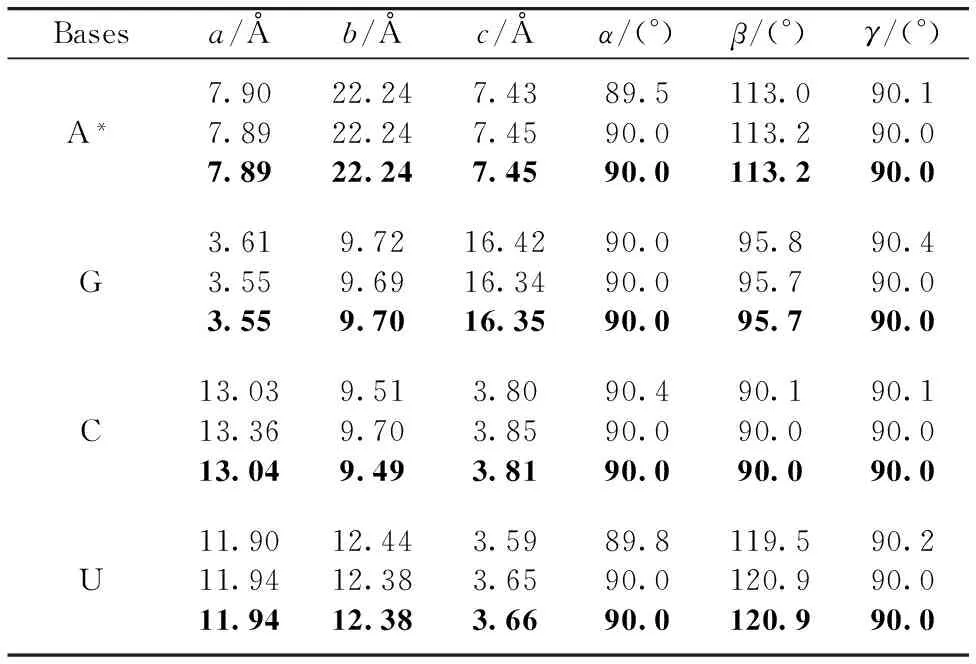
Basesa/Åb/Åc/Åα/(°)β/(°)γ/(°)A*7.907.897.8922.2422.2422.247.437.457.4589.590.090.0113.0113.2113.290.190.090.0G3.613.553.559.729.699.7016.4216.3416.3590.090.090.095.895.795.790.490.090.0C13.0313.3613.049.519.709.493.803.853.8190.490.090.090.190.090.090.190.090.0U11.9011.9411.9412.4412.3812.383.593.653.6689.890.090.0119.5120.9120.990.290.090.0
* Data in the first row corresponding to each nucleobase predicted by VASP and the second predicted by PBC-GEBF
3 结果与讨论
3.1 RNA碱基太赫兹光谱
RNA碱基太赫兹光谱的实验结果和理论计算结果见图2。
由图2可以看出、RNA碱基的实验光谱[图2(a)]和其晶体结构的计算光谱[图2(b)]有着相似的吸收剖面、表明RNA碱基粉末状样品属于无定形晶体结构。但实验光谱相对理论光谱有一定的频移、这是因为实验光谱是在室温下测得、而理论计算出的光谱是在低温(0 K)条件下模拟得到。另外、实验数据基于分子的固态结构、而理论模拟基于气相结构。在1~10 THz频段、应用PBC-GEBF方法计算RNA碱基晶体的红外共振吸收光谱、得到腺嘌呤和鸟嘌呤都有6个红外活性振动模式、胞嘧啶和尿嘧啶分别为6个和3个红外活性振动模式。与实验结果相比、除了鸟嘌呤6.35 THz处的吸收峰没能重现、4.83和5.39 THz处的吸收峰简并; 胞嘧啶4.3和4.79 THz处吸收峰简并; 尿嘧啶3.32和3.82 THz处的吸收峰简并、其他吸收峰的位置和强度均被准确地模拟重现。在这里需要说明的是:理论计算中、无论什么样的分子团簇(二聚体、三聚体、四聚体等等)均不能精确地描述分子晶体的整体性能、这也是造成理论结果和实验结果有一定偏差的原因。从图2还可以看出、RNA碱基在太赫兹波段的吸收峰均显示为较宽的吸收包络、这与分子内基团的振动吸收有很大的区别。分析认为、太赫兹波段的振动特性均来自于分子的集体振动、这一结果可以从理论计算得到的振动模式中得到验证。表2列出了尿嘧啶太赫兹光谱中所有吸收峰位置和对应的振动模式。
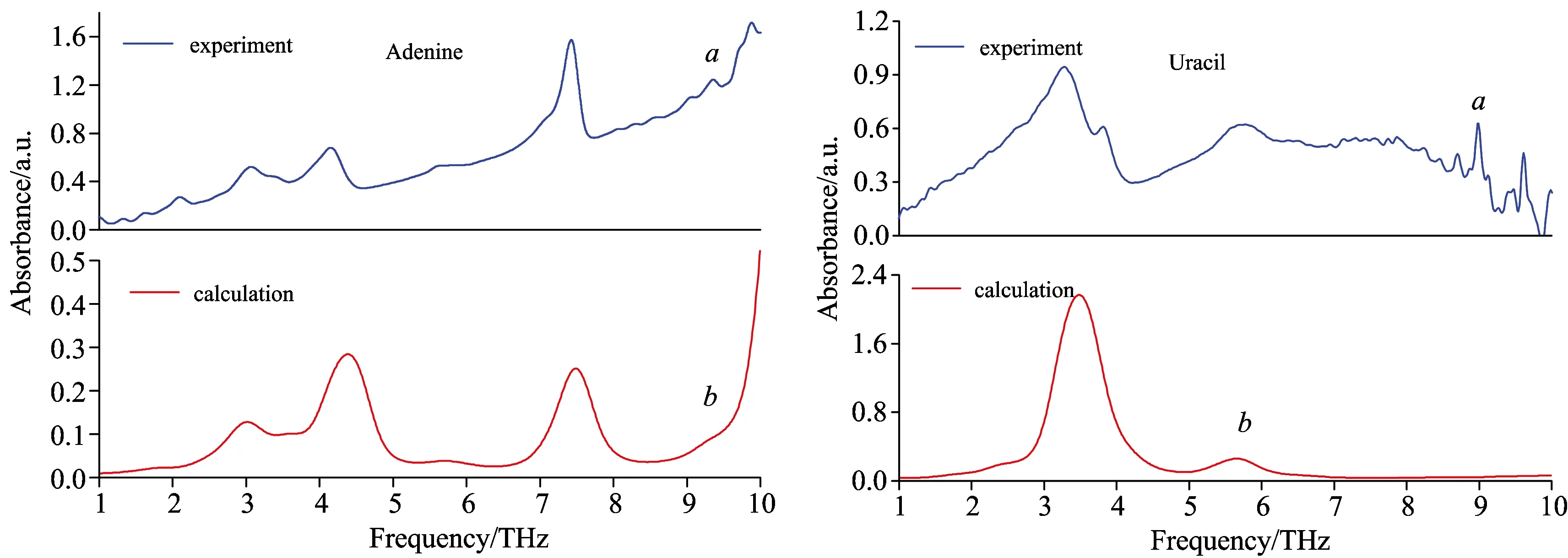
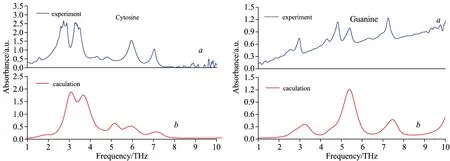
图2 RNA碱基晶体在1~10 THz波段的傅里叶红外光谱(a)与理论光谱(b)对比
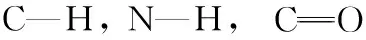
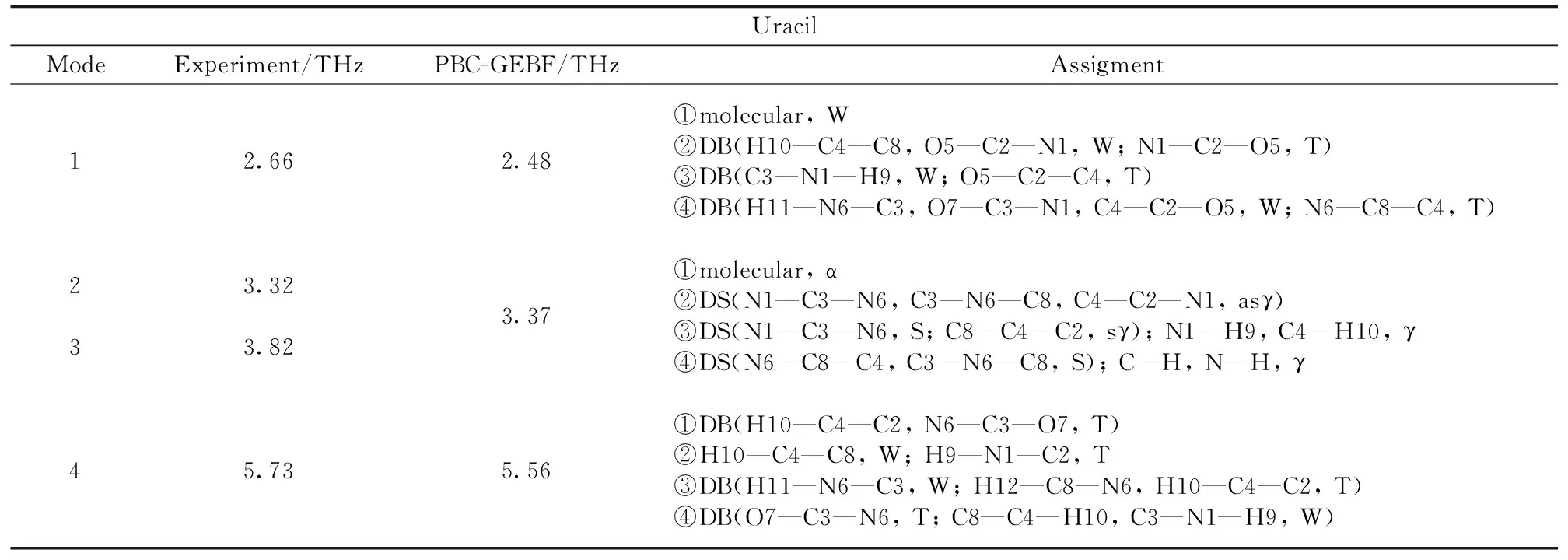
表2 尿嘧啶晶体在太赫兹波段所有红外活性振动模式
Abbreviations: γ:stretching; β:bending; α:rocking; as γ:asymmetrical stretching; sγ:symmetrical stretching; W:wagging; T:twisting; S:scissoring; DB:ring deformation (bending); DS:ring deformation (stretching); RB:ring breathing; BT:butterfly torsion; TS,translation; ①—⑧:Molecular number in the unit cell; The content in brackets is vibration modes of the ring; Ignore the distinction between single and double bonds
图3给出了尿嘧啶3.37 THz处的振动模型。RNA碱基所有分子结构、红外吸收峰对应的振动模式和模型以及动态视频见补充材料。
3.2 RNA碱基拉曼光谱
拉曼光谱作为红外光谱的有力补充、同样可以得到分子振动、转动信息、还可以显示红外光谱不能显示或者比较弱的吸收峰、常被用于分子结构的定性或定量分析。图4为RNA碱基在3~10 THz范围拉曼光谱理论和实验值、使用PBC-GEBF方法计算得到的拉曼光谱与实验结果吻合、其特征峰基本一致、除了尿嘧啶3.52和4.48 THz处特征峰简并; 鸟嘌呤7.26和8.03 THz、3.57、4.02、4.49、4.89和5.98 THz处特征峰简并外、其他特征峰的位置和强度均能准确的模拟重现、可以用解释太赫兹光谱的方法说明这一现象。作为红外光谱的一种补充手段、对RNA碱基晶体在太赫兹波段的拉曼光谱进行了研究。在4~10 THz、拉曼光谱呈现弱特征峰、肉眼难以看清、为此对该区域进行了放大处理、见图5。表3给出了尿嘧啶的拉曼特征谱理论和实验值、并对所有特征峰进行了指认、给出了对应的振动模式。
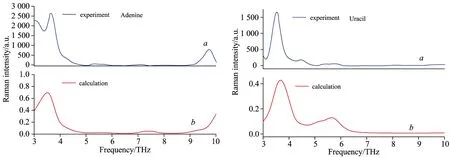
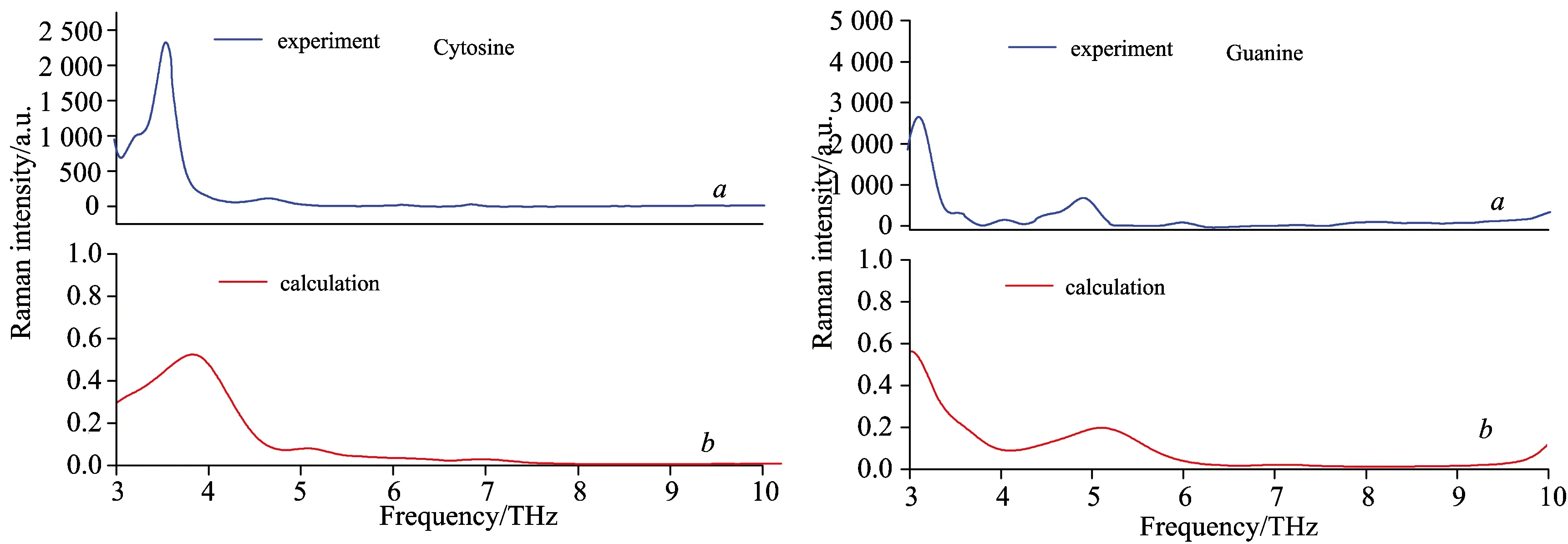
图4 RNA碱基晶体在3~10 THz波段拉曼光谱的实验(a)和理论(b)结果对比
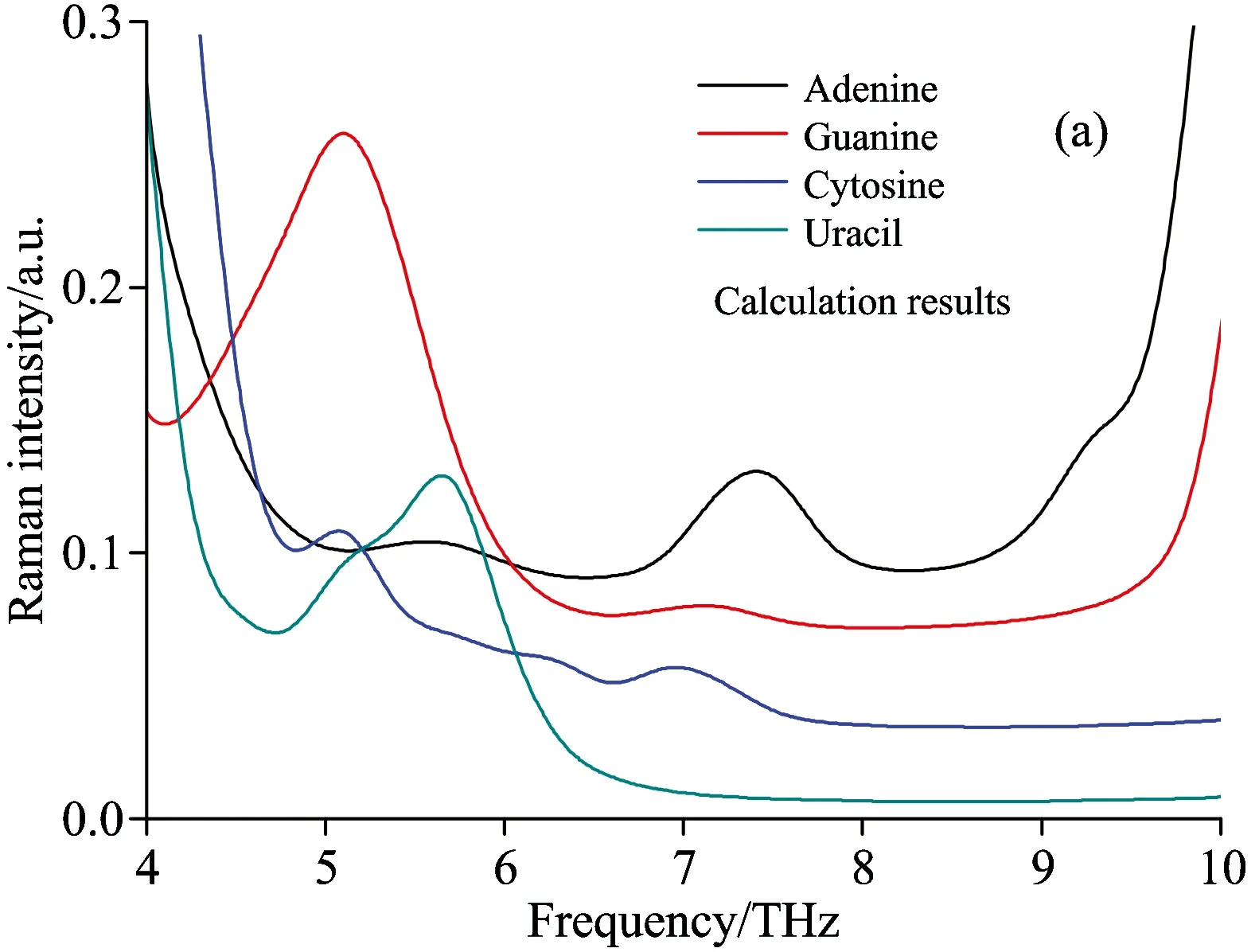
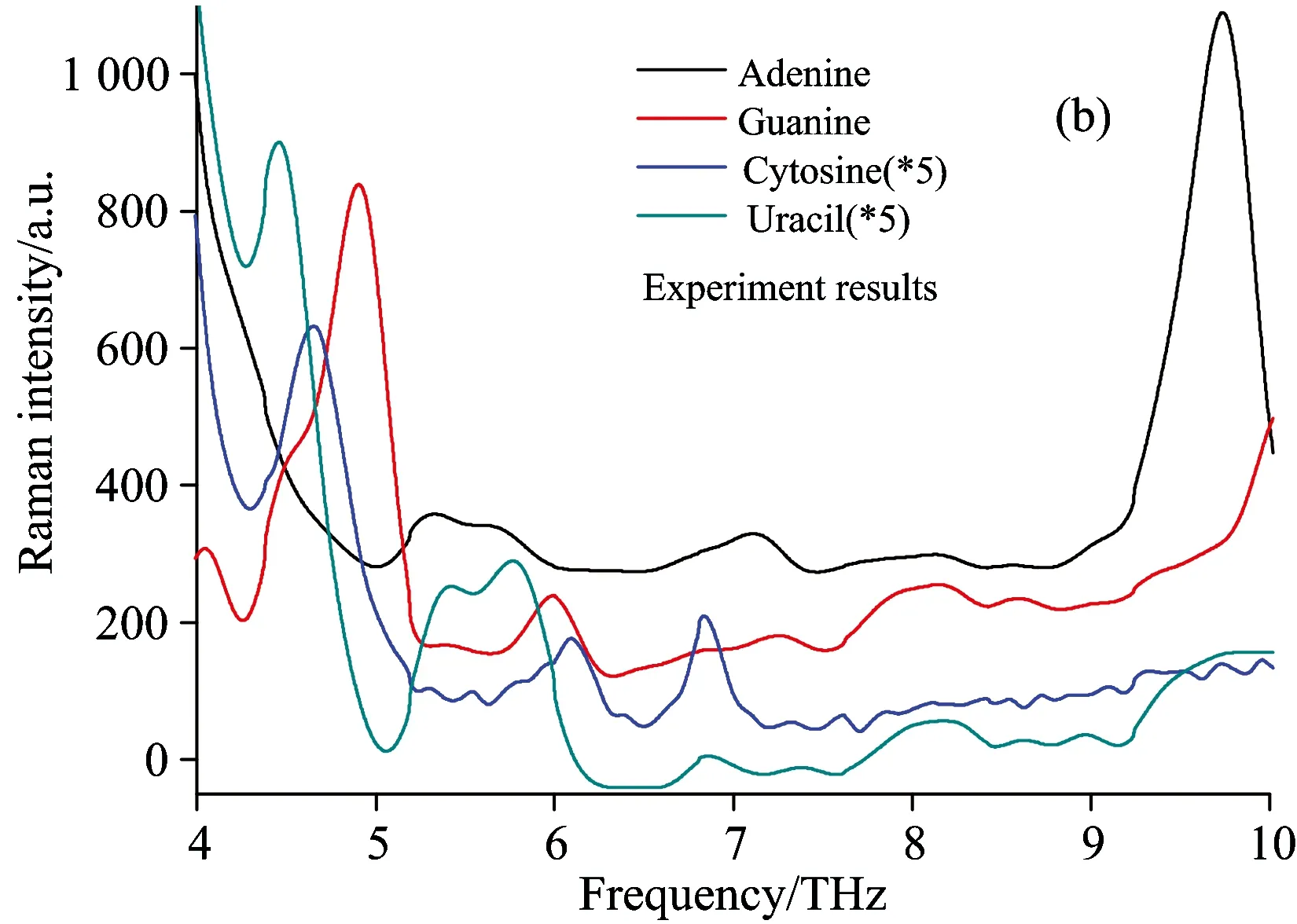
图5 RNA碱基晶体在4~10 THz波段拉曼光谱的理论(a)与实验(b)结果对比,实验结果中尿嘧啶和胞嘧啶的强度被放大了5倍
Fig.5 Theoretical (a) and experimental (b) spectra (Raman) of 4 RNA nucleobase crystals in the frequency range from 4~10 THz. The intensity of experimental results for cytosine and uracil (b) were magnified 5 times
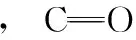
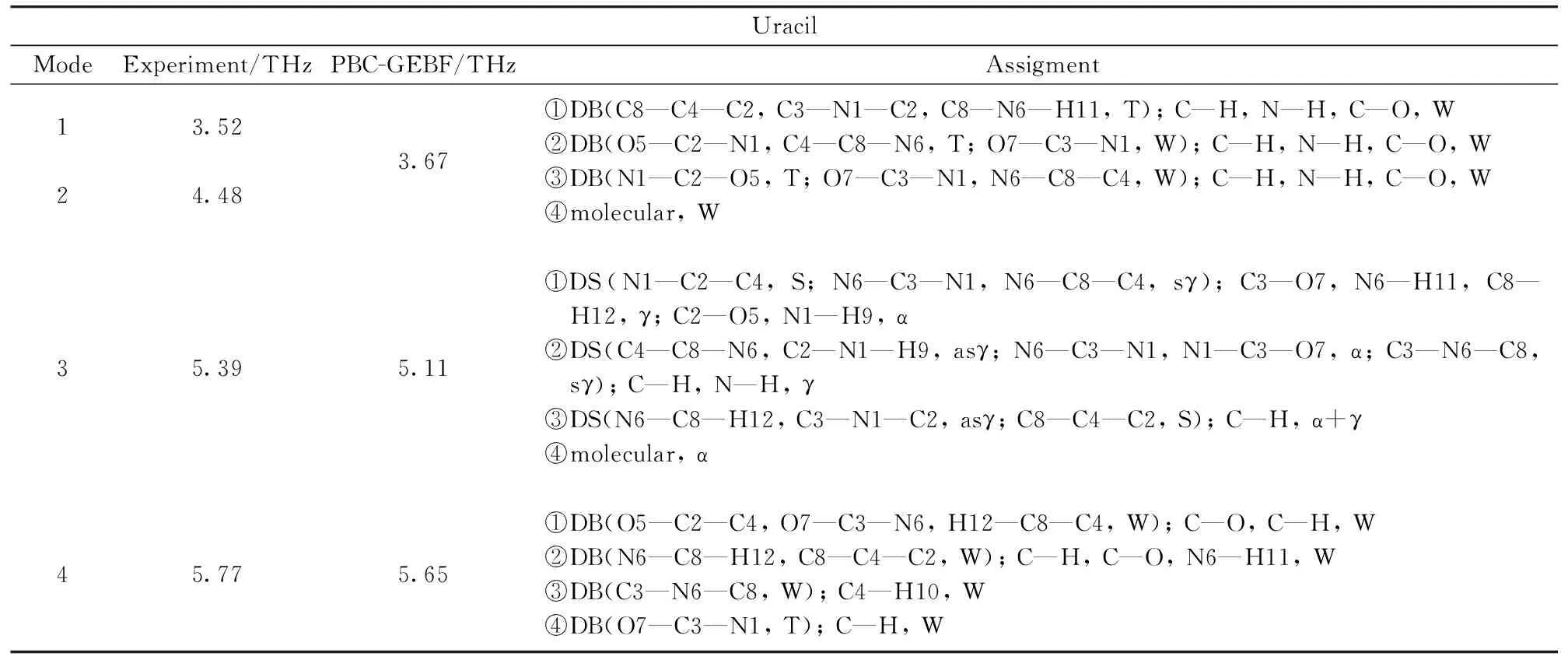
表3 尿嘧啶晶体在太赫兹波段所有拉曼活性振动模式
Abbreviations: γ:stretching; β:bending; α:rocking; as γ:asymmetrical stretching; sγ:symmetrical stretching; W:wagging; T:twisting; S:scissoring; DB:ring deformation (bending); DS:ring deformation (stretching); RB:ring breathing; BT:butterfly torsion; TS:translation; ①—⑧:Molecular number in the unit cell; The content in brackets is vibration modes of the ring; Ignore the distinction between single and double bonds
4 结 论
应用傅里叶红外光谱仪和激光拉曼光谱仪测试了4种RNA碱基在太赫兹波段的远红外和拉曼光谱、并利用PBC-GEBF方法结合Guassian09 软件对RNA碱基晶体结构的振动光谱进行了计算、得到了所有特征峰位置及其对应的振动模式、理论和实验结果相一致。拉曼光谱作为红外光谱的有力补充、在对RNA碱基振动光谱分析时、发挥了很大的优势、从RNA碱基太赫兹波段的振动光谱看出、在1~10 THz波段、所有振动模式均来源于分子的集体振动、分子间的氢键和弱相互作用对振动模式的贡献很大。从RNA碱基红外活性振动模式可以看出、1~5.5 THz的振动模式一般来源于所有原子参与的集体振动、5.5~10 THz的振动模式来自于部分原子参与的集体振动、这与拉曼光谱观测到的结果一致。这说明、随着振动频率的增加、振动模式由分子的集体振动向基团的振动过渡。
[1] Michalska,Katarzyna,et al. Journal of Molecular Structure,2016,1115: 136.
[2] Al-Zoubi N,Koundourellis J E,Malamataris S,et al. Journal of Pharmaceutical & Biomedical Analysis,2002,29(3): 459.
[3] YAN Hui,FAN Wen-hui,et al(闫 慧、范文慧、等). Spectroscopy and Spectral Analysis(光谱学与光谱分析),2013,33(10): 2612.
[4] Fischer B M,Walther M,UhdJepsen P. Physics in Medicine and Biology,2002,47(21): 3807.
[5] Nishizawa J、Sasaki T,Suto K,et al. Optics Communications,2005,244(1-6): 469.
[6] Li Shuhua,Li Wei,Fang Tao. Journal of the American Chemical Aociety,2005,127(19): 7215.
[7] Li Wei,Li Shuhua,Jiang Yuansheng. Journal of Physical Chemistry,2007,111(11): 2193.
[8] Li S,Li W,Ma J. Accounts of Chemical Research,2014,47(9): 2712.
[9] Fang T,Li W,Gu F,et al. Journal of Chemical Theory and Computation,2015,11(1): 1.
[10] Svensson M,Humbel S,Froese R D J,et al. Journal of Physical Chemistry,1996,100: 19357.
[11] Fang Tao,Jia Junteng,Li Shuhua. Journal of Physical Chemistry,2016,120(17): 2700.
[12] Frisch M J,Trucks G W,Schlegel H B,et al. Gaussian 09,Revision D.01,Gaussian,Inc.,Wallingford CT,2013.
[13] Kresse G,Furthmuller J. Comput. Mater,Sci. Physical Review B Condensed Matter,1996,6: 15.
[14] Mahapatra S,Nayak S K,Prathapa S J,et al. Crystal Growth and Design,2008,8: 1223.
[15] Stewart R F,Jensen L H. Acta Crystallographica,1967,23: 1102.
[16] Barker D L,Marsh R E. Acta Crystallographica,1964,17(12): 1581.
[17] Guille K,Clegg W. Acta Crystallographica,Section C: Crystal Structure Communications,2006,62: o515.
*Corresponding author
Research on THz and Raman Spectra of RNA Nucleobases
WANG Fang1,4、ZHAO Dong-bo2、JIANG Ling1、XU Li3、SUN Hai-jun3、LIU Yun-fei1*
1. College of Information Science and Technology,Nanjing Forestry University,Nanjing 210037,China 2. School of Chemistry and Chemical Engineering,Nanjing University,Nanjing 210023,China 3. Advanced Analysis and Testing Center,Nanjing Forestry University,Nanjing 210037,China 4. School of Electronic and Information Engineering,Sanjiang University,Nanjing 210012,China
The Infrared and Raman spectra of RNA nucleobases in terahertz (THz) band (1~10 THz) were detected with Fourier transform infrared spectroscopy (FTIR) and Raman spectroscopy. The position of all the characteristic peaks and corresponding vibration modes of RNA nucleobase crystals were obtained with Guassian09 software and energy-based fragmentation approach under periodic boundary conditions (PBC-GEBF) method. The computational results were verified to be in accordance with experimental data,which indicated that the powder of RNA nucleobases is amorphous crystal structure. The infrared spectra demonstrated that adenine,guanine and cytosine all have 6 infrared active vibrational modes,while uracil only has 3. Comparing to experimental results,the position and intensity of the absorption peaks were nicely corroborated by the predicted spectrum,except that one weak vibrational frequency at 6.35 THz is missing and two peaks (4.83 and 5.39 THz) merge in the predicted spectrum of guanine; two peaks in 4.3 and 4.79 THz merge into a single one in the calculated spectrum of cytosine; the peaks of thymine in 3.32 and 3.82 THz merged. The computational results of Raman spectra were also verified to be in line with the experimental data. The position and intensity of the characteristics peaks were exactly simulated except that two peaks of guanine in 3.52 and 4.48 THz merged; two peaks in 7.26 and 8.03 THz merge and five peaks (3.57,4.02,4.49,4.89,5.98 THz) merge in the calculated spectrum of guanine. Through the analysis and identification of the characteristic peaks,it is indicated that the vibration modes of DNA nucleobases in 1~10 THz were derived from collective vibration of molecules in the lattice. The intermolecular hydrogen bond and the weak interaction force contribute greatly to the vibration modes. In addition,as the frequency increases to over 5.5 THz,the vibration modes will change from the atoms collective vibration to some atoms vibration. This research has important theoretical and practical reference value to reveal the effect of RNA nucleobases in the areas of RNA molecular structure constitution,biological macromolecules identification and terahertz spectra formation mechanism and biological inheritance.
RNA nucleobases; THz spectra; Raman spectra; PBC-GEBF
Jun. 22,2016; accepted Sep. 28,2016)
2016-06-22、
2016-09-28
国家自然科学基金项目(31170668、31200541)、江苏省自然科学基金项目(2012417)、江苏省优势学科(PAPD)、江苏高校品牌专业建设工程项目(TAPP)资助
王 芳、1984年生、南京林业大学博士研究生 e-mail:582536573@qq.com *通讯联系人 e-mail:lyf@njfu.com.cn
O657.3
A
10.3964/j.issn.1000-0593(2016)12-3863-07
猜你喜欢
杂志排行
光谱学与光谱分析的其它文章
- Studies on the Interaction of Perfluorononanoic Acid with Human Serum Albumin by Multi-Spectroscopic,Molecular Docking and Isothermal Titration Calorimetry Techniques
- Study on Emission Spectrum of OH Radicals in a Combination System of Pulsed Discharge Plasma and Activated Carbon
- Simulating the Three-Dimensional Image of Cold Atomic Cloud
- K Shell Fluorescence Parameters by Impact of 5.96 keV Photons on Ti and Its Compounds
- Structure Effect on K Shell Fluorescence Parameters at Bis-4-Bromobenzyl-1,2,4-Triazol-3-Ones
- 动态应变场下相移光栅光谱特性及实验研究
