白鹭MHCⅡDABⅠ基因第二外显子的多态性与进化
2016-04-12罗斯特林清贤陈小麟
李 力,罗斯特,林清贤,2,陈小麟,2*
( 1.厦门大学生命科学学院,2.厦门大学环境与生态学院,滨海湿地生态系统教育部重点实验室,福建厦门361102)
白鹭MHCⅡDABⅠ基因第二外显子的多态性与进化
李力1,罗斯特1,林清贤1,2,陈小麟1,2*
( 1.厦门大学生命科学学院,2.厦门大学环境与生态学院,滨海湿地生态系统教育部重点实验室,福建厦门361102)
摘要:克隆测序白鹭( Egretta garzetta) 5个种群138份个体组织样本的主要组织相容性复合体Ⅱ类B基因( MHCⅡDAB Ⅰ)第二外显子( exon2)序列,分析探讨exon2的多态性、进化选择、系统关系和种群遗传结构.主要结果如下:白鹭MHC ⅡDABⅠexon2序列长度为270 bp,共计定义了139个等位基因;序列分析显示exon2有101个核苷酸变异位点( 37.4%)和31个氨基酸变异位点( 34.4%) ;基于贝叶斯法构建的系统树显示白鹭MHCⅡDABⅠexon2有5个高支持率的谱系;肽结合位点( PBR)、非肽结合位点( non-PBR)的非同义替换率( dN)和同义替换率( dS)比值计算显示,PBR的dN/dS为1.99 ( p<0.05),而non-PBR的dN/dS则略小于1( p>0.05),表明白鹭MHCⅡDABⅠexon2受到正选择作用;根据等位基因在群体中的分布频率作分子方差分析( AMOVA),得到群体间分化指数( F(ST))为0.194 1( p<0.000 1),提示白鹭MHCⅡDABⅠexon2存在显著的种群遗传结构分化.
关键词:白鹭;主要组织相容性复合体( MHC) ;遗传变异;进化;系统关系;种群结构
主要组织相容性复合体( major histocompatibility complex,MHC)是一类与免疫密切相关的基因家族,广泛存在于脊椎动物体内,负责编码细胞表面糖蛋白,在机体免疫和自身免疫耐受的形成过程中起着重要的作用[1-2].当抗原进入机体后,MHC分子可与之结合而被T细胞受体识别,进而激发机体产生特异性免疫反应;根据其化学结构、功能差异及编码蛋白种类的不同,MHC分为3大类,即MHCⅠ、MHCⅡ、MHCⅢ,目前在保护遗传学中的研究主要对象为MHCⅠ和MHCⅡ[3].MHCⅠ和MHCⅡ具有相似的结构,都是非共价结合的异源双链分子,包含α链和β链,具有4个胞外结构域,但结构域的组成有所不同[4-5].MHC基因具有丰富的多态性,其多态性最丰富的是MHCⅡ类基因,主要为β链基因( B基因),包括β1(DABⅠ)和β2( DABⅡ),两者均具有高度多态性,特别是其负责编码抗原结合区域的第二外显子( exon2)是目前公认的研究抗病标记和遗传育种的重要位点[6-7].由于不同外源病原会诱发MHC产生相应变化,因此MHC变异能够反映种群内、种群间受到自然选择压力时所产生的适应性,适用于研究进化生态学和保护遗传学方面的科学问题[8-9].有关鸟类MHC基因的研究,自Kaufman等报道了鸡( Gallus gallus)的MHC全基因序列[10]以来,目前已对多种鸟类的MHC基因进行了深入的研究,包括朱鹳( Nipponia nippon)[11]、白腰叉尾雨燕( Oceanodroma leucorhoa)[12]、日本鹌鹑( Coturnix japonica)[13]等.鸟类MHC的早期研究主要侧重于分析MHC分子结构和应用于物种进化等方面,近年来则倾向于MHC基因多态性与群体遗传学的研究.
白鹭( Egretta garzetta)是鹭科白鹭属的鸟类,为全球性分布的水鸟[14].近年来关于白鹭的研究,主要集中在其野外生活习性和系统分类地位[15-16],而有关白鹭群体遗传学的研究则少见报导.作为广布种,野外白鹭种群在不同生境下面对各不相同的环境条件,受到侵扰的病原也会有所差异,由此可能产生不同的免疫应答反应,进而引起MHC基因水平的差异.因此,本研究通过分析白鹭不同地理种群的MHC基因遗传多样性,探讨自然选择压力对白鹭MHC基因种群遗传结构的影响,以便更好地理解白鹭MHC基因遗传多样性的进化机制,完善鸟类适应性进化理论.
1材料与方法
1. 1材料
共取得白鹭个体血液和羽毛组织样品138份,分别采自福建宁德日屿( RY: 28)、福建厦门鸡屿( JY: 30)、河南信阳( XY: 30)、浙江舟山( ZS: 30)和贵州遵义南白( NB: 30) 5个地理种群,所有样品均通过无损伤性取样方法从野外自然繁殖种群个体中获取.
1. 2基因组DNA提取和PCR扩增
采用EasyPure基因组DNA提取试剂盒(北京全式金生物公司)提取基因组DNA.采用Li等[17]所设计的引物ARB2EN1( 5'-ACYKKCCYCCCTGCACAAACAGGG-3')和ARB2EC ( 5'-CCCCAGGGARATGTTCTGCCACGC-3'),扩增MHCⅡDABⅠexon2序列.PCR反应在C1000TMThermal Cycle PCR仪( Bio-Rad)完成,反应总体系为25 μL,其中包括10×PCR Buffer 2.5 μL、DNA模板0.3 μL( 30~50 ng)、0.2 mmol/L dNTP 0.5 μL、20 μmol/L引物各0.3 μL、1 U Taq DNA聚合酶( TaKaRa) 0.3 μL、ddH2O 20.8 μL,设不含DNA模板的空白对照.PCR反应条件为: 94℃预变性5 min; 94℃变性1 min,58℃退火30 s,72℃延伸40 s,共30个循环;最后72℃延伸10 min.
1. 3 SSCP电泳
通过单链构象多态性( single strand conformation polymorphism,SSCP)分析方法筛选不同的等位基因[18].操作过程如下:将已纯化的PCR产物10 μL混合等体积变性缓冲液(体积分数为95%的去离子甲酰胺、10 mmol/L NaOH、20 mmol/L EDTA、0.2 g/L溴酚蓝及0.2 g/L二甲苯菁),99℃加热10 min后迅速冰浴,以维持DNA单链状态.将变性后的DNA上样于体积分数为8%的聚丙烯酰胺凝胶中,在4℃、260 V条件下电泳19 h后银染、干燥.回收纯化所有存在区别的条带并作为二次PCR扩增的模板,以上一小节中的PCR反应体系和条件进行扩增.
1. 4克隆及测序
将上述二次扩增所得的PCR产物在2.0%(质量分数)的琼脂糖凝胶中进行电泳分离,用胶回收试剂盒( Omega)纯化目的片段.将纯化后的PCR产物连接到pMD18-T载体( TaKaRa)上,再将重组质粒转入大肠杆菌( Escherichia coli) DH5α菌株( TaKaRa),在含有氨苄青霉素的LB平板上进行涂板,37℃培养箱中培养16 h.通过蓝白斑筛选和菌落PCR法筛选阳性克隆.从每个样品挑选5~8个阳性克隆,采用载体通用测序引物M13,在上海美吉生物公司完成测序.
1. 5数据分析
使用软件Lasergene SeqMan Pro 7.1.0拼接校对测序结果,根据判别外显子-内含子的GT-AG法则,并参照黄嘴白鹭( Egretta eulophotes ) MHC序列( GenBank: KC282867.1),再用推导的氨基酸序列在GenBank数据库中进行BLAST检索来确定是否为MHC序列.使用MEGA 6.0软件对外显子核苷酸序列进行序列比对,分别计算外显子核苷酸和氨基酸的变异位点数、等位基因间的p遗传距离和平均遗传距离,评估外显子的变异程度[19].从GenBank数据库中下载3种鹭科鸟类的MHCⅡDABⅠexon2序列,包括黄嘴白鹭( E.eulophotes,HM991028.1)、岩鹭( E.sacra,HM991087.1 )、夜鹭( Nycticorax nycticorax,HM991040.1).基于这些序列,利用MrBayes 3.2.3软件构建贝叶斯树( Bayesian tree),计算相关参数,以后验概率( posterior probability value)评估贝叶斯树各分支的置信度[20].
根据等位基因在各个群体的分布频率,用Arlequin 3.1软件进行群体间分子方差分析( analysis of molecular variance,AMOVA)以检验群体间的遗传结构[21].利用PAML4.0软件包中的CODEML程序,根据选择参数ω(ω=dN/dS;其中dN表示非同义替换率,dS表示同义替换率)的大小推测目标序列是否经受选择压力(如果ω显著大于1,则表明存在正选择作用)[22];此外主要比较数据组在不同密码子进化模型M1与M2、M7与M8之间的差异,并根据贝叶斯经验贝叶斯路径( Bayes empirical Bayes,BEB)检测受到正选择作用的氨基酸位点[23].
2结果
2. 1 MHCⅡDABⅠexon2的序列测定与多态性分析
本研究共计对白鹭138份个体样本的630个阳性克隆进行了测序,所得序列在GenBank数据库中进行BLAST同源比对,检索后确定为MHCⅡDABⅠexon2序列片断.每条序列只有在2个或2个以上克隆序列完全一致时才被认定是可靠的序列,用于后续的分析[24].所得MHCⅡDABⅠexon2核苷酸序列长度为270 bp,共定义139个等位基因,命名为Egr-001~Egr-139(图1).139个等位基因经比对排列后,没有发现插入(缺失)单核苷酸或终止密码子.核苷酸序列变异位点的比例为3 7 .4 % ( 1 0 1 / 2 7 0),对应的氨基酸序列变异位点的比例为3 4 .4 % ( 3 1 / 9 0).等位基因间核苷酸位点的变异范围为1 2 ~2 8个,氨基酸位点的变异范围为3 ~1 4个.

图1 139个白鹭MHCⅡDABⅠ等位基因的exon2核苷酸序列变异位点Fig.1 Variable nucleotide sites among 139 MHCⅡDABⅠexon2 alleles in little egret
2. 2 MHCⅡDABⅠexon2的选择压力检测
MHCⅡ分子直接与抗原多肽结合的氨基酸残基称为肽结合位点( peptide binding residues,PBR).由于与抗原多样性相互作用,PBR序列变异较大,当dN/dS>1时该位点可认为受到了正选择作用.分别计算PBR、非肽结合位点( non-PBR)的dS和dN,结果如表1所示: PBR的dN/dS显著大于1( p<0.05),而non-PBR 的dN/dS则略小于1( p>0.05),这表明白鹭MHCⅡDABⅠexon2编码的PBR在进化过程中受到强烈的正选择作用.
分别利用PAML软件包CODEML程序中的模型M1与M2以及M7与M8检测exon2所受的选择作用.比较分析结果(表2)显示:模型M2相对于M1( p<0.001)、M8相对于M7( p<0.001)对本文数据有更好的拟合度;在模型M2当中共检测到10个氨基酸位点受到正选择作用,其后验概率均大于95%;而在M8检测到11个受正选择作用的氨基酸位点,其中有10个位点与M2的位点完全相同.
2. 3 MHCⅡDABⅠexon2等位基因的系统关系
基于白鹭139个等位基因序列与其他3种外群鹭科鸟类(黄嘴白鹭、岩鹭、夜鹭)的MHCⅡDABⅠexon2序列,通过贝叶斯方法所构建的系统发育树(图2)显示:白鹭的MHCⅡDABⅠexon2等位基因可分为5个谱系( RY、JY、ZS、NB、XY),均有较高的支持率,且各谱系内只包括与之相对应的地理种群序列;在这5个谱系中,XY与NB关系较亲近,并与RY聚为一支,而JY则与ZS关系更亲近.
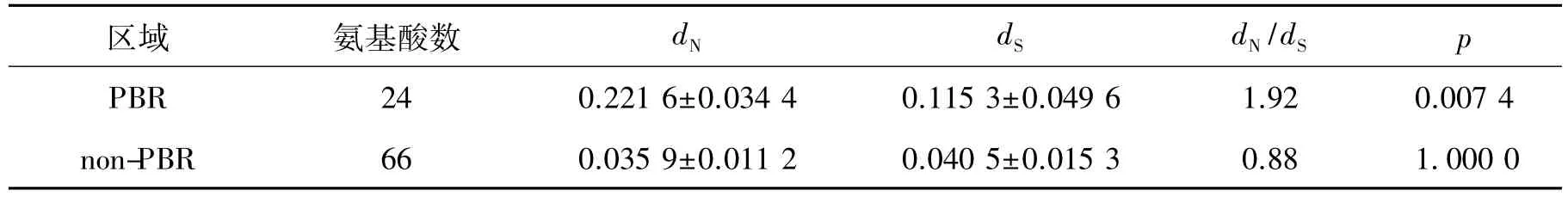
表1白鹭MHCⅡDABⅠexon2编码的肽结合位点( PBR)及非肽结合位点( non-PBR)的dN和dSTab.1 dNand dSof PBR and non-PBR encoded by MHCⅡDABⅠexon2 in little egret

表2不同密码子进化模型对白鹭MHCⅡDABⅠexon2拟合度检验的估算参数与似然值Tab.2 Parameter estimates and likelihood values of different codon evolution models for MHCⅡDABⅠexon2 in little egret
2. 4 MHCⅡDABⅠexon2的种群遗传结构
在白鹭MHCⅡDABⅠexon2等位基因分布中,ZS种群拥有49个,等位基因数量最多,然后依次是RY种群( 35)、NB种群( 20)、JY种群( 19),而XY种群最少( 16).用Arlequin v3.1软件分析群体间分化指数( FST),结果(表3)显示5个白鹭群体间的FST均较大,并且这5个群体两两之间FST值差异显著( p<0.05),表明这5个群体间的遗传分化显著,群体间缺乏基因交流.对5个群体的AMOVA结果(表4)显示群体内个体间的变异占总变异来源的80.59%,群体间的变异则占19.41%( p<0.000 1),进一步说明这5个群体间的遗传差异显著.
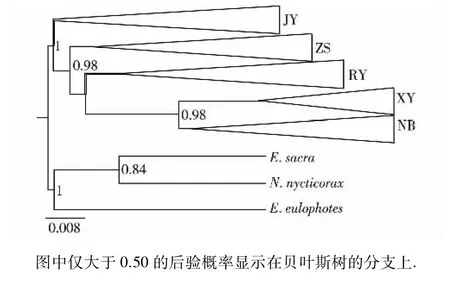
图2白鹭及3种鹭科鸟类MHCⅡDABⅠexon2序列的贝叶斯树Fig.2 Bayesian tree of MHCⅡDABⅠexon2 sequences among E.garzetta,E.eulophotes,E.sacra and N.nycticorax

表3白鹭5个不同种群群体之间的FST值Tab.3 FSTvalue between 5 different populations of E.garzetta
3讨论
PCR-SSCP能够检测DNA片段上不同位点的多态性及单个碱基的突变、缺失、插入或置换等变化[25].本文利用该技术研究白鹭5个地理种群的MHCⅡDABⅠexon2的遗传多态性.结果显示exon2拥有数量众多的等位基因序列,表现出较高的多态性.所有等位基因序列均未发现插入或缺失突变,翻译成氨基酸序列后,也未发现终止密码子,这间接表明所分离到的等位基因序列可能来源于表达的基因座,能够行使正常的功能表达.

表4白鹭5个种群MHCⅡDABⅠexon2基因遗传差异的分子方差分析Tab.4 AMOVA of MHCⅡDABⅠexon2 among 5 populations of E.garzetta
为了探究白鹭MHCⅡDABⅠ基因多态性的维持机制,本研究对exon2编码的抗原结合区PBR以及non-PBR的dN与dS值进行了统计分析.结果显示PBR的dN显著大于dS( p<0.01),提示白鹭MHCⅡDABⅠexon2曾经历过强烈的正选择作用.在CODEML程序选择压力检验模型的比较分析结果中,模型M2 和M8分别较M1和M7具有更好的拟合度( p<0.001),同样也支持上述的正选择作用.由此我们推测,正选择作用对维持白鹭MHCⅡDABⅠexon2多态性起着重要作用,该结论与其他鸟类MHC的研究报道一致.Alcaide等在研究黄爪隼( Falco naumanni)时发现其MHCⅡDABⅠexon2具有较高的遗传多态性,从21份个体样品中共分离获得26种等位基因,选择压力分析结果显示其受到了强烈的正选择作用[26].Bollmer等在比较分布于加拉帕戈斯群岛的特有种加岛鵟( Buteo galapagoensis)和其近缘广布种斯温氏鵟( Buteo swainsoni)在MHCⅡDABⅠexon2的遗传多样性差异时发现,广布种斯温氏鵟相对于特有种加岛鵟在MHCⅡDABⅠ上有更高的遗传多样性,他们在20个斯温氏鵟个体上共发现了20种等位基因,而在32个加岛鵟个体上则只有3种等位基因,同时也在斯温氏鵟的MHCⅡDABⅠexon2上检测到强烈的正选择作用[27].Dearborn等基于巢式PCR和高通量测序技术研究白腰叉尾海燕( Oceanodroma leucorhoa)的MHCⅡDABⅠexon2,共获得21种等位基因( 48个个体),研究结果揭示白腰叉尾海燕MHCⅡDABⅠexon2存在较高遗传多态性的重要原因是曾经长期受到病原体侵扰所引起的正选择作用[12].
基于贝叶斯树、FST和AMOVA结果,5个白鹭种群的MHCⅡDABⅠexon2等位基因存在显著的种群分化.有研究表明当动物在不同环境下的微生物和病原体侵扰时,由于受到选择压力的长期作用,会导致与机体免疫反应重要相关的MHC基因在不同种群的等位基因数量上出现较大差异[28-29].因此本研究推测,导致白鹭5个种群间MHC基因出现种群分化的原因可能是这5个地理种群在应对不同生境时受到了不同病原侵扰,诱发了不同的免疫反应.这种等位基因数量的差异反映了不同的环境选择压力的影响.
由于MHC基因的序列多样性、等位基因数量受不同生境的病原种类和数量所影响,因此MHC基因的多样性能够反映不同种群对各自环境的适应能力[7,30-31].今后的研究有必要针对不同种群生境中的寄生虫和微生物种类进行调查,以深入探讨白鹭种群MHC等位基因数量差异的成因.
参考文献:
[1]KLEIN J,SATTA Y,OHUIGIN C,et al.The molecular descent of the major histocompatibility complex[J].Annual Review of Immunology,1993,11: 269-295.
[2]NIKOLICH-ZUGICH J,FREMONT D H,MILEY M J,et al.The role of mhc polymorphism in anti-microbial resistance [J].Microbes and Infection,2004,6( 5) : 501-512.
[3]BOEHM T N,HESS I I.Evolution of the immune system in the lower vertebrates[J].Annual Review of Genomics and Human Genetics,2012,13: 127-149.
[4]BLUM A,MILLER H.The major histocompatibility complex and inflammation[J].Southern Medical Journal,2000,93 ( 2) : 169-172.
[5]KAUFMAN J,SALOMONSEN J,FLAJNIK M.Evolutionary conservation of MHC classⅠand classⅡmolecules: different yet the same[J].Seminars in Immunology,1994,6 ( 6) : 411-424.
[6]FURLONG R F,YANG Z.Diversifying and purifying selection in the peptide binding region of DRB in mammals [J].Journal of Molecular Evolution,2008,66( 4) : 384-394.
[7]YAO Y,DAI Q,LI J,et al.Genetic diversity and differentiation of the rhesus macaque ( Macaca mulatta) population in western Sichuan,China,based on the second exon of the major histocompatibility complex classⅡDQB ( MhcMamu-DQB1 ) alleles[J].BMC Evolutionary Biology,2014,14( 1) : 130.
[8]RADWAN J,BIEDRZYCKA A,BABIK W.Does reduced MHC diversity decrease viability of vertebrate populations? [J].Biological Conservation,2010,143( 3) : 537-544.
[9]VETEŠNÍKOVÁ ŠIMKOVÁ A,CIVÁˇNOVÁ K,GETTOVÁ L,et al.Genomic porosity between invasive Chondrostoma nasus and endangered endemic Parachondrostoma toxostoma ( Cyprinidae) : the evolution of MHCⅡB genes[J].PLoS One,2013,8 ( 6) : e65883.
[10]KAUFMAN J,MILNE S,GÖBEL T W,et al.The chicken B locus is a minimal essential major histocompatibility complex[J].Nature,1999,401( 6756) : 923-925.
[11]CHEN L,LAN H,SUN L,et al.Genomic organization of the crested ibis MHC provides new insight into ancestral avian MHC structure[J].Scientific Reports,2015,5: 7963.
[12]DEARBORN D C,GAGER A B,GILMOUR M E,et al.Non-neutral evolution and reciprocal monophyly of two expressed Mhc classⅡB genes in Leach's storm-petrel[J].Immunogenetics,2015,67( 2) : 111-123.
[13]SUZUKI S,HOSOMICHI K,YOKOYAMA K,et al.Primary analysis of DNA polymorphisms in the TRIM region ( MHC subregion) of the Japanese quail,Coturnix japonica[J].Animal Science Journal,2013,84( 1) : 90-96.
[14]HAFNER H,DUGAN P J,BOY V.Use of artificial and natural wetlands as feeding sites by little egrets ( Egretta garzetta L.) in the Camarge Southern France[J].Colonial Waterbirds,1986,9: 149-154.
[15]HASHMI M Z,MALIK R N,SHAHBAZ M.Heavy metals in eggshells of cattle egret ( Bubulcus ibis) and little egret ( Egretta garzetta) from the Punjab province,Pakistan[J].Ecotoxicology and Environmental Safety,2013,89: 158-165.
[16]林清贤,周晓平,方文珍,等.中国6种白色羽鹭科鸟类的系统归属研究[J].厦门大学学报(自然科学版),2010,49( 1) : 83-86.
[17]LI L,ZHOU X,CHEN X.Characterization and evolution of MHC classⅡB genes in Ardeid birds[J].Journal of Molecular Evolution,2011,72( 5/6) : 474-483.
[18]BINZ T,REUSCH T,WEDEKIND C,et al.SSCP analysis of Mhc classⅡB genes in the three spine stickleback[J].Journal of Fish Biology,2001,58( 3) : 887-890.
[19]TAMURA K,STECHER G,PETERSON D,et al.MEGA6: molecular evolutionary genetics analysis version 6.0[J].Molecular Biology and Evolution,2013,30 ( 12 ) : 2725-2729.
[20]RONQUIST F,HUELSENBECK J P.MrBayes 3: Bayesian phylogenetic inference under mixed models[J].Bioinformatics,2003,19( 12) : 1572-1574.
[21]EXCOFFIER L,LISCHER H E.Arlequin suite ver 3.5: a new series of programs to perform population genetics ana-lyses under Linux and Windows[J].Molecular Ecology Resources,2010,10( 3) : 564-567.
[22]YANG Z.PAML 4: phylogenetic analysis by maximum likelihood[J].Molecular Biology and Evolution,2007,24( 8) : 1586-1591.
[23]WONG W,YANG Z,GOLDMAN N,et al.Accuracy and power of statistical methods for detecting adaptive evolution in protein coding sequences and for identifying positively selected sites[J].Genetics,2004,168( 2) : 1041-1051.
[24]CUTRERA A P,LACEY E A.Major histocompatibility complex variation in Talas tucotucos: the influence of demography on selection[J].Journal of Mammalogy,2006,87( 4) : 706-716.
[25]ZHOU H,LI S,LIU X,et al.Haplotyping using a combination of polymerase chain reaction—single-strand conformational polymorphism analysis and haplotype-specific PCR amplification[J].Analytical Biochemistry,2014,466: 59-64.
[26]ALCAIDE M,EDWARDS S V,NEGRO J J.Characterization,polymorphism,and evolution of MHC classⅡB genes in birds of prey[J].Journal of Molecular Evolution,2007,65( 5) : 541-554.
[27]BOLLMER J L,HULL J M,ERNEST H B,et al.Reduced MHC and neutral variation in the Galapagos hawk,an island endemic[J].BMC Evolutionary Biology,2011,11 ( 1) : 143.
[28]EIZAGUIRRE C,LENZ T L,KALBE M,et al.Divergent selection on locally adapted major histocompatibility complex immune genes experimentally proven in the field[J].Ecology Letters,2012,15( 7) : 723-731.
[29]HUGHES A L,YEAGER M.Natural selection at major histocompatibility complex loci of vertebrates[J].Annual Review of Genetics,1998,32( 1) : 415-435.
[30]BORG A A,PEDERSEN S A,JENSEN H,et al.Variation in MHC genotypes in two populations of house sparrow ( Passer domesticus) with different population histories[J].Ecology and Evolution,2011,1( 2) : 145-159.
[31]DAVISON F,KASPERS B,SCHAT K A,et al.Avian Immunology[M].London,UK: Academic Press,2011: 159-182.
Polymorphism and Evolution of MHCⅡDABⅠGene Exon2 in Little Egret ( Egretta garzetta)
LI Li1,LUO Site1,LIN Qingxian1,2,CHEN Xiaolin1,2*
( 1.School of Life Sciences,Xiamen University,2.Key Laboratory of Ministry of Education for Coast and Wetland Ecosystems,College of the Environment&Ecology,Xiamen University,Xiamen 361102,China)
Abstract:The DNA fragments of major histocompatibility complex classⅡB gene ( MHCⅡDABI) exon2 alleles from 138 individual samples in 5 populations of little egret ( Egretta garzetta) were cloned and sequenced to investigate their polymorphism,evolution selection,phylogenetic and population genetic structures.The main results were as follows: sequence of MHCⅡDABⅠexon2 of little egret was 270 bp in length,and a total of 139 alleles were defined in the exon2: sequence analyses indicated that exon2 genes had 101 nucleotide acid variation sites ( 37.4%) and 31 amino acid variation sites ( 34.4%) ; the Bayesian phylogenetic tree showed that there were 5 distinct lineages with high bootstrap values in the exon2; when the proportion of synonymous substitution rate ( dS) to non-synonymous substitution rate ( dN) was calculated for peptide binding residues ( PBR) or non-PBR of MHCⅡDABⅠexon2,the dN/dSin PBR was 1.99 ( p<0.05),whereas the ratio in non-PBR was a bit lower than 1( p>0.05),implying that positive selection was acting on the MHCⅡDABⅠexon2 in the little egret; analyses of molecular variance ( AMOVA) based on allele distribution frequencies in different populations showed that F(ST)value was 0.194 1 ( p<0.000 1),suggesting that there was significant population structure differentiation of MHCⅡDABⅠexon2 in little egret.
Key words:Egretta garzetta; major histocompatibility complex ( MHC) ; genetic variation; evolution; phylogenetic relationship; population structure
*通信作者:xlchen@ xmu.edu.cn
基金项目:国家自然科学基金( 41476113,31272333) ;福建省自然科学基金( 2010Y2007)
收稿日期:2015-04-16录用日期: 2015-08-19
doi:10.6043/j.issn.0438-0479.2016.01.007
中图分类号:Q 958; Q 953
文献标志码:A
文章编号:0438-0479( 2016) 01-0030-07
引文格式:李力,罗斯特,林清贤,等.白鹭MHCⅡDABⅠ基因第二外显子的多态性与进化[J].厦门大学学报(自然科学版),2016,55( 1) : 30-36.
Citation: LI L,LUO S T,LIN Q X,et al.Polymorphism and evolution of MHCⅡDABⅠgene exon2 in little egret ( Egretta garzetta)[J].Journal of Xiamen University( Natural Science),2016,55( 1) : 30-36.( in Chinese)
